草原红牛FABP7 基因克隆、生物信息学及组织表达差异分析
吕 阳,曹 阳,高 一,刘理想,薛佳佳,张国梁
(吉林省农业科学院畜牧科学分院,吉林公主岭 136100)
草原红牛是我国第1 个肉乳兼用型优良品种,具有肉质性能好、口感细嫩、风味独特、遗传性能稳定等特点,是东北地区肉牛的主要品种[1],但与国外优质肉牛品种相比仍存在个体偏小、产肉性能偏低等缺点,因此加强选种选育工作、深入研究其脂肪沉积的分子调控机制,对了解草原红牛的肉用性能具有重要价值。脂肪酸结合蛋白(FABPs)是一种细胞内低分子量(14 ~16 ku)多肽,分布于脊椎动物、非脊椎动物细胞质中,主要有9种脂肪酸结合蛋白[2],并且在大多数组织中含量丰富,是胞内脂质结合蛋白超家族成员。FABPs 参与细胞内脂肪酸的运输和体内脂肪的合成与降解,包括长链脂肪酸和类视黄醇的摄取和细胞内运输,与长链脂肪酸紧密结合发挥重要作用。另外还在基因调控、细胞信号、细胞生长和分化中发挥作用[3]。FABPs 家族成员中,FABP7(Fatty Acid Binding Protein 7)是脑型脂肪酸结合蛋白(Brain Lipid Binding Protein),最初在脑组织发现分离,主要作用是调节脂肪酸的摄取和胞内运输,参与中枢神经系统神经胶质细胞的调节。FABP7 位于脂质代谢的经典通路PPAR 通路中[4],与脂质代谢密切相关,并在脂肪沉积中起作用[5]。近年来家畜脂肪沉积及脂代谢的分子机制研究广泛,FABP7 基因作为分子辅助标记参与其中,具有潜在研究价值。目前,关于FABP7基因的研究主要集中于鸡、猪的脂肪沉积分子机制,FABP7 基因对牛的脂代谢是否有影响鲜有报道。因此,本研究以草原红牛为研究对象,利用RT-PCR 技术克隆草原红牛FABP7 基因编码区序列,并使用多种生物软件和在线工具进行生物信息学分析,通过qPCR 技术检测FABP7 基因在各组织间mRNA 的表达水平,为进一步研究FABP7 基因在牛脂代谢及脂肪沉积等方面的调控作用提供参考。
1 材料与方法
1.1 实验材料 实验所用的草原红牛样品来自吉林省农业科学院畜牧分院,饲养于吉林省农业科学院畜牧分院草原红牛育种实验场,3 头健康的24 月龄草原红牛阉牛屠宰后,采集心、肝、肾、胃、肠、肌肉、皮下脂肪等组织,用锡箔纸包好并迅速放入液氮中冻存,后期一部分放入-80℃冰箱用于提取RNA。
1.2 主要试剂 Trizol 购自Invitrogen 公司;pMD19-T 载体购自TaKaRa 公司;Axygen DNA 凝胶回收试剂盒购自Axygen 公 司;Transcriptor First Strand cDNA Synthesis Kit、LightCycler 480 SYBR Green Ⅰ Master 购自罗氏(中国)有限公司。
1.3 实验方法
1.3.1 引物设计与合成 根据GenBank 数据库中牛FABP7(登录号:NM_001078162.2)的mRNA 序列,使用Primer Premier5.0 软件设计该基因的CDS 区序列特异性引物及荧光定量PCR 引物,选取GAPDH 基因(登录号:NM_001034034.2)为内参基因,序列见表1。引物由金唯智生物技术有限公司合成。
1.3.2 草原红牛FABP7 基因克隆 按照Trizol 试剂使用说明书对草原红牛的小肠组织进行总RNA 的提取,使用紫外分光光度计检测其浓度和纯度(OD 值在1.8~2.0),符合要求后进行反转录获得cDNA。PCR 扩增体系共20 μL:Mix 10 μL,10 pmol/L 上、下游引物各0.5 μL,100 ng/μL cDNA 1 μL,ddH2O 8 μL。PCR 反应程序:95℃预变性3 min;95℃变性30 s,49.9℃退火30 s,72℃延伸20 s,共35 个循环;72℃延伸5 min;4℃保存。PCR 产物用1.5%琼脂糖凝胶进行检测后使用PCR 清洁试剂盒回收目的条带。采用TA 克隆的方法,将回收产物与pMD19-T载体16℃孵育30 min 进行连接,随后转化至大肠杆菌DH5α 感受态细胞中,将细菌均匀涂布在预先准备好的氨苄青霉素平板上,倒置放于37℃培养箱中过夜培养,挑取白色单菌落,在含有氨苄青霉素的液体培养基中培养,摇床37℃ 200 r/min 振荡培养过夜,送往苏州金唯智生物公司测序。
1.3.3 FABP7 基因生物信息学分析 通过NCBI-BLAST 对草原红牛FABP7 基因编码区核苷酸序列与牛(登录号:NM_001078162.2)、羊(登录号:XM_004011152.3)、猪(登录号:NM_001025229.1)、人(登录号:NM_00131 9039.1)、鼠(登录号:NM_021272.3)、鸡(登录号:NM_205308.2)FABP7 基因核苷酸序列进行同源性比对,并利用Mega6.0 软件构建草原红牛、普通牛、羊、猪、鼠、鸡FABP7 基因系统进化树。使用DNAStar 中SeqMan 软件对测序结果与GenBank 数据库中公布的牛FABP7 基因序列进行比对,利用在线ProtParam 分析FABP7 基因编码蛋白质的基本理化性质;利用在线Net Phos3.1、NetOGlyc4.0、NetNGlyc1.2、SignalP 4.1 Server 分析预测FABP7 氨基酸序列中存在的潜在磷酸化位点、O-糖基化位点、N-糖基化位点及信号肽,使用Scratch Protein Predictor 对FABP7 蛋白进行二硫键预测;运用在线工具Prot Scale 进行氨基酸序列的亲疏水性分析;利用在线工具TMHMM2.0 和Tmpred 对蛋白质序列的跨膜区结构进行分析;TargetP 1.1 Server、PSORT II 预测蛋白质的亚细胞定位;PredictProtein 预测FABP7 蛋白的二级结构,利用SWISS-MODEL 分析预测FABP7 蛋白三级结构。
1.3.4 FABP7 基因各组织表达量测定 将提取的各组织总RNA 反转录后的cDNA 作为模板(共3 头),实时荧光定量PCR(qPCR)检测FABP7 基因在心、肝、肾、胃、肠、肌肉、脂肪中的相对表达量,qPCR 反应体系为20 μL:10 pmol/L 上、下游引物各0.5 μL,100 ng/μL cDNA 1 μL,SYBR 10 μL,H2O 8 μL;程序:95℃预变性5 min,95℃变性10 s,60℃退火15 s,72℃延伸20 s,40个循环;熔解曲线分析:95℃ 5 s,65℃ 1 min,每个待测样品设置3 个重复。
1.4 统计分析 采用2-ΔΔCt方法对实时荧光定量PCR 结果进行统计分析,采用Graph 软件对试验数据进行单因素方差分析,结果表示为平均值±标准误(或标准差),以P<0.05 作为差异显著性判断标准。

表1 引物信息
2 结 果
2.1 FABP7 基因CDS 区序列克隆鉴定 以反转录得到的小肠组织cDNA 为模板,进行PCR 扩增,产物经1.5%琼脂糖凝胶电泳检测,得到399 bp 左右的扩增片段,与预期大小一致(图1),同时用TA 克隆的方法,通过测序比对成功克隆草原红牛FABP7 基因编码区。
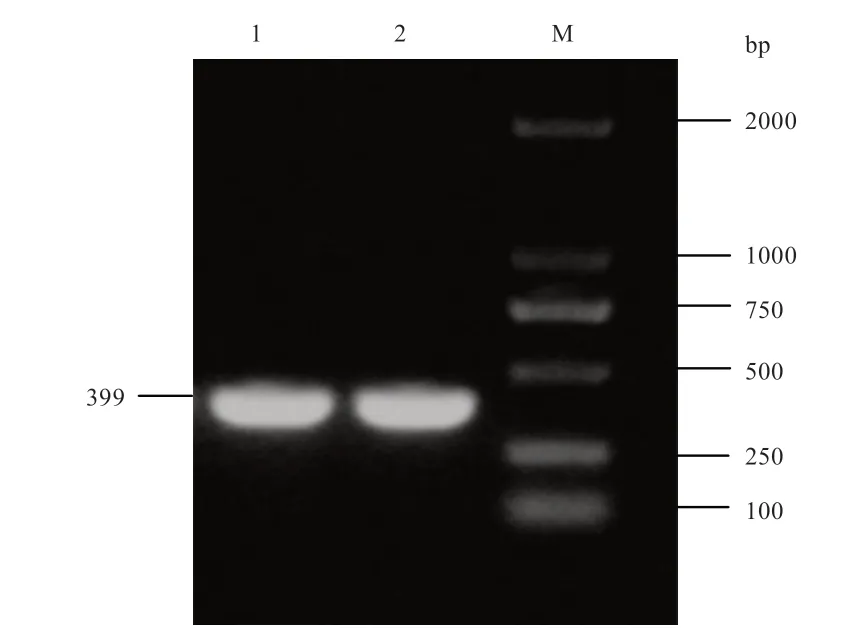
图1 草原红牛FABP7 基因编码区 PCR 扩增电泳图
2.2 FABP7 基因序列的生物信息学分析
2.2.1 FABP7 基因序列分析 如图2 所示,FABP7 基因与牛、羊、猪、人、鼠、鸡的同源性分别为99%、98%、94%、92%、85%、85%,比对发现同源性在85%~99%,说明该基因在生物进化过程中比较保守;并用Mega6.0 构建核苷酸系统进化树,差异度单位为0.02,98、93、49、72 代表每个分支的置信度,如果2个物种在同一竖线上,代表同源性越近,如果越长说明越远。因此,草原红牛与牛、绵羊的亲缘关系最近,与鸡的亲缘关系最远(图2),所构建的系统进化树可以直观表达不同物种之间的分子水平差异。
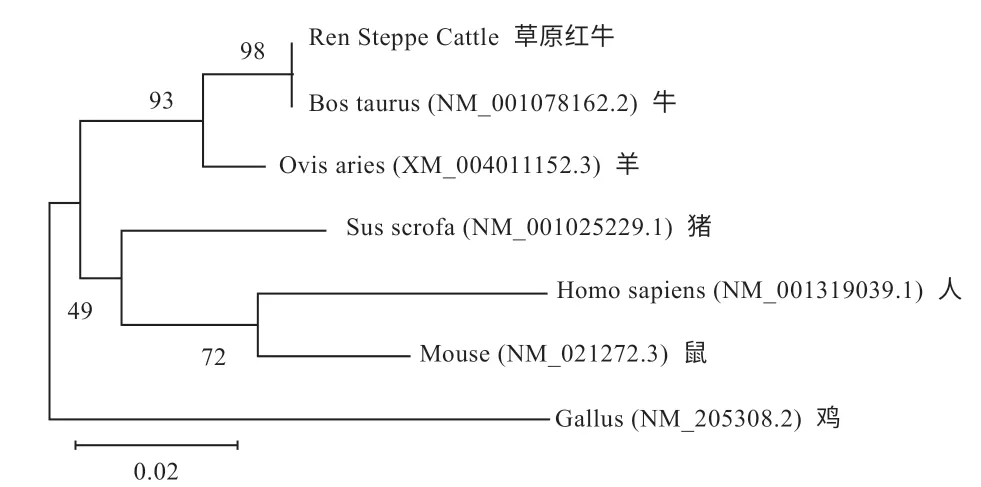
图2 FABP7 基因系统进化树
2.2.2 FABP7 蛋白质的理化性质 通过在线工具ProtParam分析草原红牛FABP7 蛋白的理化性质,结果显示,FABP7基因共编码132 个氨基酸,分子式C659H1037N179O206S6,总原子数2 087,分子量为14.96 ku,理论等电点为5.38。氨基酸组成结果显示,共包含20 种氨基酸,其中缬氨酸(V)所占的比例最高为11.4%(15 个氨基酸),脯氨酸(P)所占的比例最低为0.8%(1 个氨基酸),同时包含21 个负电荷氨基酸碱基(天冬氨酸D+谷氨酸E)和17 个正电荷氨基酸(精氨酸R+赖氨酸K),该蛋白整体偏向负电荷,消光系数14 105,不稳定系数为26.27,说明该蛋白为稳定蛋白,预计的半衰期为30 h,脂肪系数为73.71,总平均亲水性为-0.378,蛋白表现出亲水性,并使用Protscale 分析其蛋白亲疏水性发现,该蛋白为亲水性蛋白。
2.2.3 FABP7 蛋白潜在的磷酸化位点、糖基化位点、信号肽、跨膜区及二硫键分析 FABP7 氨基酸序列中共存在14 个潜在的磷酸化位点(图3),4 个丝氨酸磷酸化位点,9 个苏氨酸磷酸化位点,1 个酪氨酸磷酸化位点。分析表明不存在潜在位点O-糖基化,1 个N-糖基化潜在位点(图4)位于第35 氨基酸;SignalP 4.1 Server预测该氨基酸序列不存在信号肽;FABP7 氨基酸序列的跨膜区结构分析发现,FABP7 氨基酸序列不存在跨膜区结构。FABP7 蛋白在Cys6 和Cys81 位置存在1 个二硫键。
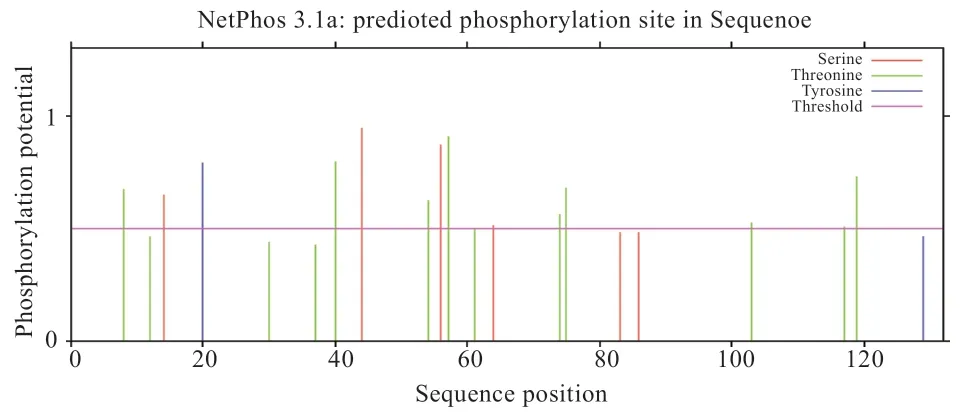
图3 FABP7 蛋白潜在的磷酸化位点分析
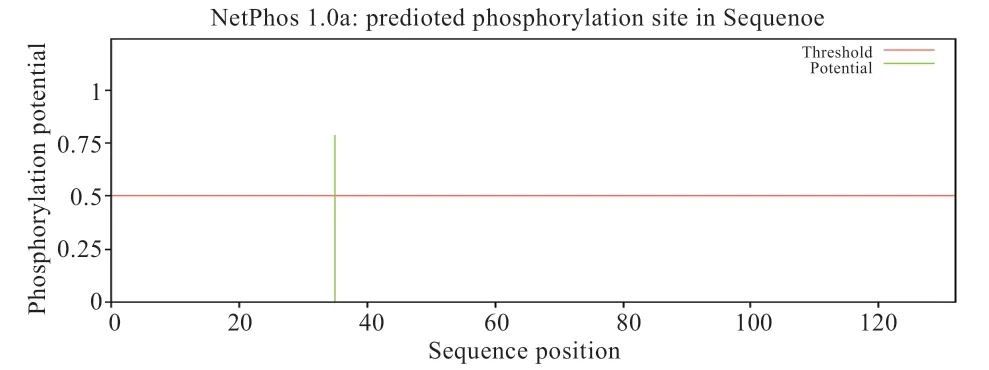
图4 FABP7 蛋白潜在的N-糖基化位点分析
2.2.4 亚细胞定位 使用TargetP 1.1 Server、PSORT II预测蛋白质的亚细胞定位,结果表明分泌信号通路位点(mTP)0.061、线粒体作用位点(SP)0.091,所占比例均很小,无信号肽,说明该蛋白不属于分泌型蛋白;FABP7 蛋白主要定位于细胞质(65.2%)上,细胞核(21.7%)和线粒体(13%)中也有分布(表2)。
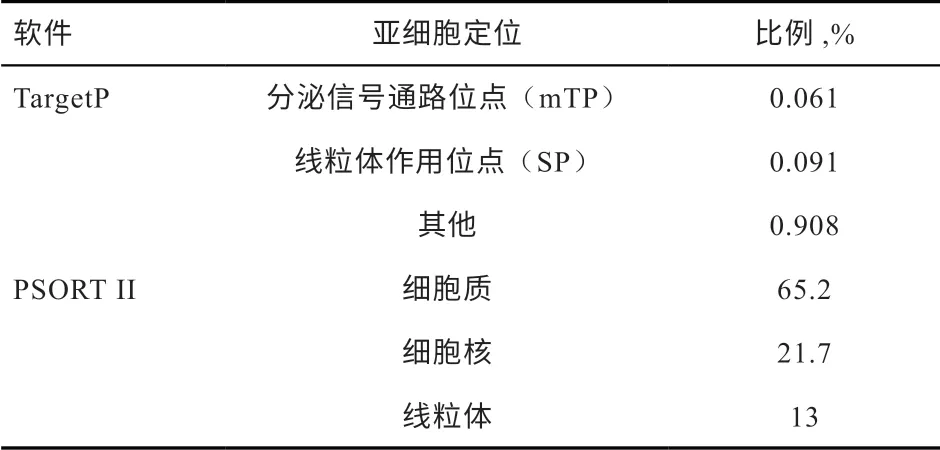
表2 FABP7 蛋白亚细胞定位
2.2.5 FABP7 蛋白高级结构预测 使用PSIPRED 预测FABP7 蛋白二级结构,结果显示,α-螺旋占12.12%,β-折叠占58.33%,无规则卷曲占29.55%,为混合型蛋白。利用SWISS-MODEL 分析预测FABP7 蛋白三级结构,可以发现存在2 个α- 螺旋结构和10 条β- 折叠(图6),结果与预测的二级结构基本一致。

图5 FABP7 蛋白的二级结构预测
2.3 FABP7 基因在草原红牛不同组织间的差异表达 如图7 所示, FABP7 基因在6 个组织中均有表达,在肠组织表达量最高,高于其他组织(P<0.05);在心脏、脂肪和胃中中度表达,在肝脏和肌肉中相对低表达。

图6 FABP7 蛋白的三级结构预测
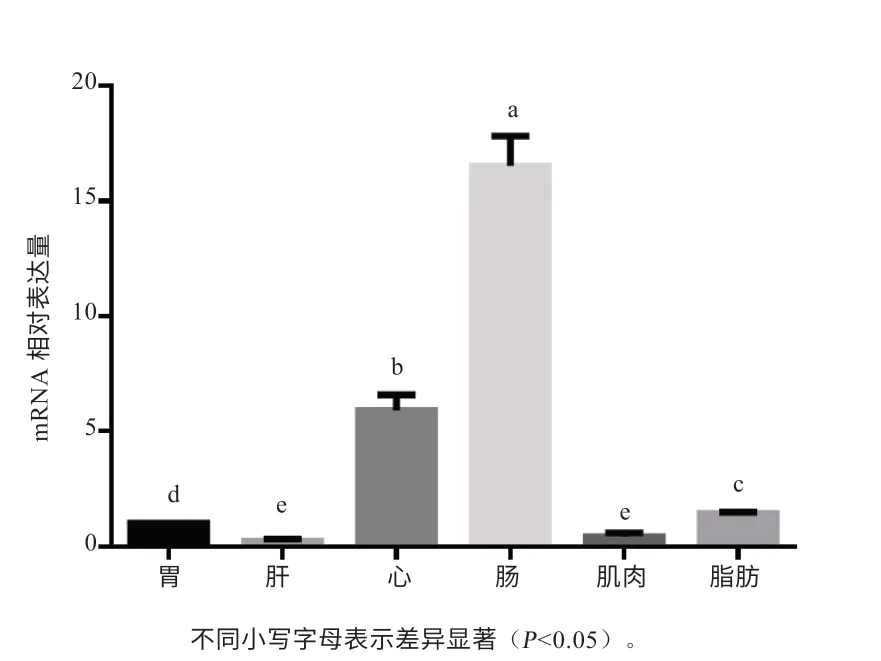
图7 FABP7 基因在草原红牛各组织中的mRNA 相对表达量
3 讨 论
目前,关于FABP7 基因的研究主要集中在神经胶质细胞方面,FABP7 在神经干细胞和发育中的脑星形胶质细胞中大量表达。多不饱和脂肪酸(PUFAs)是大脑的关键结构成分,对大脑的正常发育至关重要,PUFAs 参与细胞运输和其生理作用,与细胞内脂质结合蛋白基因家族编码的脂肪酸结合蛋白紧密相关,FABP7是ω-3 PUFA 的强结合剂,表明其通过调节脂质代谢或信号转导在胶质细胞的分化或增殖中发挥作用[6-8]。
此外,关于FABP7 基因在畜禽动物脂代谢作用的研究相对较少。在全基因组关联分析中发现FABP7 可能是影响北京油鸡公鸡体重以及胴体性状的候选基因[9],并且在鸡[10]、猪[5]的脂肪沉积分子机制研究发现,与脂质代谢相关的FABP7 基因上调,并参与PPAR 信号通路;同时,敲除FABP7 减少细胞对长链脂肪酸的摄取和Hepa1-6 细胞中脂质的积累[11-12]。这些研究说明FABP7 可能促进脂肪细胞分化,在脂质代谢中发挥作用。然而,关于FABP7 基因对牛脂代谢是否有影响的研究鲜有报道。
本研究通过对草原红牛FABP7 基因核苷酸序列与其他7 个物种进行同源性对比分析,发现草原红牛与普通牛、绵羊的亲缘关系最近,与鸡的亲缘关系最远,符合生物进化特征,并且具有广泛的同源性。信号肽预测发现,该氨基酸序列不存在信号肽,不属于分泌蛋白,说明该结构不具有把蛋白质引导到不同膜结构的亚细胞器内的功能。亚细胞定位分析结果表明,FABP7 蛋白主要分布于细胞质、细胞核和线粒体上,说明FABP7 蛋白可通过接受外来传入的信号,参与细胞内的调控代谢,从而维持细胞的脂质稳态等。磷酸化位点和糖基化位点预测发现,共有14 个潜在的磷酸化位点,而磷酸化主要发生于丝氨酸、苏氨酸和酪氨酸等氨基酸残基[13],蛋白磷酸化能够修饰蛋白,存在的磷酸化对于细胞信号转导发挥重要作用[14];N-糖基化潜在位点1 个,与磷酸化类似,蛋白糖基化同样具有修饰蛋白的作用,经过糖基化使之形成糖蛋白,使蛋白发挥作用,调节细胞功能[15],蛋白质的磷酸化和糖基化修饰对草原红牛FABP7 蛋白的生物学功能具有重要的调节能力,更进一步说明FABP7 基因在调节细胞功能上发挥着主要作用。根据分析预测FABP7 蛋白三级结构,发现存在2 个α- 螺旋结构和10条β- 折叠,这对于脂肪酸结合蛋白与脂肪酸分子结合的稳定性起到了一定的作用,有助于脂肪酸的摄取、转运等环节。
本研究通过qPCR 方法检测FABP7 基因在草原红牛6 个组织的相对表达情况,发现FABP7 在各个组织中均有表达,但是不同组织间的表达差异较大,肠组织表达量最高,显著高于其他组织,在心脏中的表达量仅次于肠,在脂肪和胃中中度表达,在肝脏和肌肉组织中低度表达。FABP7 在哺乳动物中的肝脏、乳腺、肾脏、皮下脂肪、肌肉中均有表达[16],在建鲤中FABP7主要在脑、肝脏和精巢中表达[17]。本实验中FABP7 在肠中表达量最高,而胃、肠为脂肪代谢的主要场所,参与脂肪的运输,与脂质代谢和脂肪沉积均密不可分,猜测不同组织间表达差异与FABP7 基因所发挥的作用有关,但目前对草原红牛FABP7 基因各个组织的相关研究较少,具体的功能和调控机制仍不清楚,有待进一步深入研究。本研究主要为FABP7 基因在草原红牛脂肪分化和脂质代谢后续研究提供基础资料,进一步研究FABP7 基因在草原红牛中脂质代谢及脂肪沉积等方面的调控作用提供参考。

