蛤蚧及其混伪品基于12S rRNA序列的Bar-HRM鉴定研究△
苏燕燕,丁丹丹,马婷玉,向丽
中国中医科学院 中药研究所 中药鉴定与安全性检测评估重点实验室,北京 100700
蛤蚧为我国名贵动物中药材,其基原为壁虎科Gekkonidae壁虎属动物蛤蚧(又名大壁虎)GekkogeckoLinnaeus,以除去内脏的干燥全体入药,具有止咳、平喘、保肝、降血糖、提高机体免疫力及激素样作用[1-4],为临床常用补虚药[5-6]。因市场需求增大、人们大肆捕捉及生存环境的改变,蛤蚧野生资源骤减,现已列为国家二级保护动物和广西省一级保护动物,市场上蛤蚧多为进口和养殖。由于蛤蚧、藏蛤蚧等形态相似,导致同名异物、同物异名的情况较为严重,市场常见壁虎、藏蛤蚧、石龙子、蜥蜴等混伪品[7-11]。近年来,越来越多的蛤蚧粉、蛤蚧胶囊等未经检验的药材在电商平台出售,严重威胁临床用药安全,亟需一种准确高效的鉴别方法规范药材市场、保障药材安全。
高分辨率熔解曲线(High Resolution Melting,HRM)技术是指采用饱和荧光染料,结合实时荧光定量PCR技术,通过核酸熔解曲线分析,进而实现基因检测、基因型分析的新型分子诊断技术[12-14]。因其具有灵敏度高、检测快速、闭管操作避免污染、可实现高通量检测、可选择性测序等优点,逐渐应用于基因突变扫描、基因型分析、单核苷酸多态性(SNP)分析等研究[15-18]。此外,结合DNA barcoding的Bar-HRM技术逐渐形成,并成功应用于沙棘、蜂蜜等中药、食品的鉴定研究中,具有重要应用价值[19-21]。
目前,蛤蚧相关的HRM鉴定研究未见报道,而动物通用条形码线粒体细胞色素c氧化酶亚基I基因(cytochrome c oxidase subunit I,COI)序列长度大于600 bp,相较之下,片段长度约400 bp且能实现蛤蚧与混伪品鉴别的12S核糖体核糖核酸(Ribosomal Ribonucleic Acid,rRNA)基因序列则更为适宜[22-25]。因此,本研究以蛤蚧与其6种常见混伪品为研究对象[11,26-27],基于12S rRNA序列对其进行HRM鉴别研究,并对市售蛤蚧粉进行检测,以验证该方法的可行性,完善了中药蛤蚧的分子快速鉴定体系。
1 材料
1.1 实验样品
本研究所用动物样品共48份,其中,蛤蚧10批次30份;蛤蚧常见混伪品无蹼壁虎Gekkoswinhonis、疣尾蜥虎Hemidactylusfrenatus、喜山岩蜥Laudakiahimalayana、石龙子Eumeceschinensis、东方蝾螈Cynopsorientalis和红瘰疣螈Tylototritonshanjing各3份。此外,购买市售蛤蚧粉10份。动物样品均经中国科学院昆明动物研究所蒋珂助理研究员鉴定,凭证标本保存于中国中医科学院中药研究所。本研究共得48条12S rRNA序列,均已提交至NCBI核酸数据库(表1)。

表1 蛤蚧及其混伪品样品信息
1.2 仪器及试剂
Scientz-48高通量组织研磨仪(宁波新芝生物科技股份有限公司);Qubit 3.0荧光定量仪[赛默飞世尔(美国)科技有限公司];Rotor-Gene Q MDx实时定量PCR仪[凯杰(德国)生物技术有限公司];血液/组织/细胞基因组DNA提取试剂盒[天根生化科技(北京)有限公司];HRM分析试剂盒[天根生化科技(北京)有限公司];T-A克隆试剂盒[宝生物工程(大连)有限公司]。
2 方法
2.1 DNA提取
采用试剂盒法提取样品总DNA[26]。所有样品DNA均利用Qubit 3.0测定浓度,并调整为30~50 ng·μL-1。采用微量紫外分光光度计测定样品DNA质量,OD260/280处于1.8~2.0的为优质;保存于-20 ℃冰箱备用。
2.2 蛤蚧12S rRNA序列扩增及数据分析
以10批次蛤蚧样品DNA为模板,进行12S rRNA序列扩增[24],采用凝胶电泳法检测扩增结果,送北京诺赛基因组研究中心有限公司双向测序。
通过软件CodonCode Aligner V3.0对10批次蛤蚧的12S rRNA序列峰图进行拼接剪切,在NCBI比对验证后采用MEGA 6.0进行序列分析。
2.3 蛤蚧及其混伪品HRM-PCR扩增与Bar-HRM数据分析
以10批次蛤蚧的主要单倍型和6种常见混伪品的DNA为检测对象。
HRM-PCR体系为25 μL,包含2×HRM PCR master mix 12.5 μL,正反向引物L1091/H1478(2.5 μmol·L-1)各1 μL,以1 μL DNA为模板,加RNase-Free Water补充至25 μL。
HRM-PCR反应条件为94 ℃预变性5 min;94 ℃变性1 min,52 ℃退火45 s,72 ℃延伸1 min,循环35次;PCR反应完成后进入扩增产物的HRM曲线分析程序:熔解温度从75 ℃逐渐升至95 ℃,每变化0.15 ℃收集一次荧光信号。蛤蚧及其混伪品的HRM-PCR产物均经测序验证。
2.4 蛤蚧及其混伪品Bar-HRM数据分析
实验中获得的高分辨率熔解曲线均通过Rotor-Gene Q MDx version 2.3.1.49(QIAGEN GmbH,Hilden,Germany)进行分析;蛤蚧与其6种常见混伪品的HRM-PCR产物均经测序验证,并采用MEGA 6.0进行序列分析。
2.5 灵敏度检测
任取1份蛤蚧DNA,将质量浓度调整为50 ng·μL-1,并稀释为25、10、5、1 ng·μL-1的DNA溶液,进行HRM-PCR实验,研究DNA模板浓度与鉴定结果的关系。
2.6 蛤蚧掺伪检测
以0、1%、5%、10%、25%、50%、75%、100%的比例将无蹼壁虎DNA与蛤蚧DNA混合,以掺伪DNA为模板进行HRM-PCR扩增,设蛤蚧为对照样品,研究DNA掺伪比例与HRM曲线的关系。
2.7 市场蛤蚧粉鉴定
在市场购买10份蛤蚧粉,取样约45 mg,以试剂盒法提取总DNA。利用HRM技术对其进行检测:每份设置3个物理重复,以蛤蚧对照药材DNA为阳性对照,并测序验证。针对出现套峰的样品进行T-A克隆检测。
3 结果
3.1 蛤蚧12S序列分析
10批次30条蛤蚧12S rRNA序列,长度均为396 bp,可分为2种单倍型(图1),其中,单倍型H1有21条,单倍型H2有9条;共存在5个变异位点,分别为64位点的C-T变异,211位点的T-A变异,228位点的C-T变异,325位点的G-A变异和358位点的A-G变异。表明序列较为保守。
3.2 蛤蚧及其混伪品HRM分析
因10批次30份蛤蚧样品中仅存在2种单倍型,因此,每种单倍型任选3份DNA与6种混伪品共同进行HRM实验。根据图2可知,蛤蚧与6种混伪品的12S rRNA-HRM曲线均具有不同的形状特点,在归一化熔解曲线(图2A)和差异化熔解曲线(图2B)中,蛤蚧的2种单倍型均可区分开,但相对于无蹼壁虎、疣尾蜥虎、喜山岩蜥、石龙子、东方蝾螈和红瘰疣螈6种混伪品,蛤蚧的2种HRM曲线聚在一起。无蹼壁虎、石龙子和红瘰疣螈的HRM曲线分别只有一种,表明这3个物种分别只有一种单倍型。而3份疣尾蜥虎的HRM曲线呈现出相近的2种,则

注:H1和H2表示不同单倍型。图1 蛤蚧不同单倍型12S rRNA序列
该样品的12S rRNA序列有2种单倍型;3份东方蝾螈和3份喜山岩蜥(藏蛤蚧)的HRM曲线均出现较小的差异,可能存在序列差异很小的不同单倍型,或者因实验操作中加样量不均一而导致曲线出现微小差异,需测序验证。总体而言,蛤蚧及其6种常见混伪品均可通过HRM曲线进行鉴别区分。

注:A.归一化熔解曲线;B.以蛤蚧为参考基因型的差异化熔解曲线。图2 基于12S rRNA序列蛤蚧与常见混伪品的HRM曲线
3.3 蛤蚧及其混伪品DNA barcoding分析
所有HRM-PCR产物均经测序验证。实验共得48条12S rRNA序列,其中蛤蚧30条,混伪品18条,所有12S rRNA序列均在NCBI核酸数据库进行BLAST比对验证,比对到的最相近物种为自身物种,比对值为99%~100%。序列特征见表2。蛤蚧和6种常见混伪品的序列长度均小于400 bp,其中,无蹼壁虎的序列长度最长,为399 bp;喜山岩蜥的最短,为362 bp;蛤蚧的序列长度为396 bp。7个物种的平均GC碱基含量(鸟嘌呤G和胞嘧啶C在DNA 4种碱基中所占的比率)在40%~50%,其中,疣尾蜥虎的平均GC碱基含量最高,为50.0%;东方蝾螈的最低,为41.4%;蛤蚧的平均GC碱基含量为43.5%。
在本研究的7个物种中,无蹼壁虎、石龙子、东方蝾螈和红瘰疣螈共4个物种的12S rRNA序列分别只有1种单倍型,种内无变异位点,序列较为保守;蛤蚧、疣尾蜥虎和喜山岩蜥分别有2种单倍型,其中,蛤蚧的12S rRNA序列含5个变异位点,喜山岩蜥有1个变异位点,疣尾蜥虎存在18个变异位点,种内差异较大;与HRM曲线分析结果相符。根据种内、种间遗传距离分析结果显示,7个物种的种内最大遗传距离均小于其种间最小遗传距离,表明蛤蚧及其6种混伪品可通过遗传距离分析进行鉴别。此外,在NJ树图中(图3),蛤蚧和6种常见混伪品分别聚为一支,而且,自展支持率都高达99%,表明,蛤蚧及6种常见混伪品可通过构建NJ树准确区分。
因此,经测序验证,基于12S rRNA序列的DNA barcoding分析,蛤蚧与其6种常见混伪品可准确鉴别,而且各物种间也能准确区分,与HRM曲线分析结果相符,表明基于12S rRNA序列的HRM技术可实现蛤蚧及其6种常见混伪品的准确区分。
3.4 灵敏度分析
为研究DNA浓度对鉴定效果的影响,将蛤蚧DNA浓度调整为50、25、10、5、1 ng·μL-1,以此系列浓度DNA为模板进行HRM-PCR扩增。图4显示不同浓度蛤蚧样品的HRM曲线聚在一起(图4A),Tm值为(81.11±0.03)℃(图4B),表明DNA浓度为1~50 ng·μL-1的蛤蚧样品均可通过HRM-PCR检出,且结果稳定。

表2 蛤蚧及常见混伪品12S rRNA序列特征
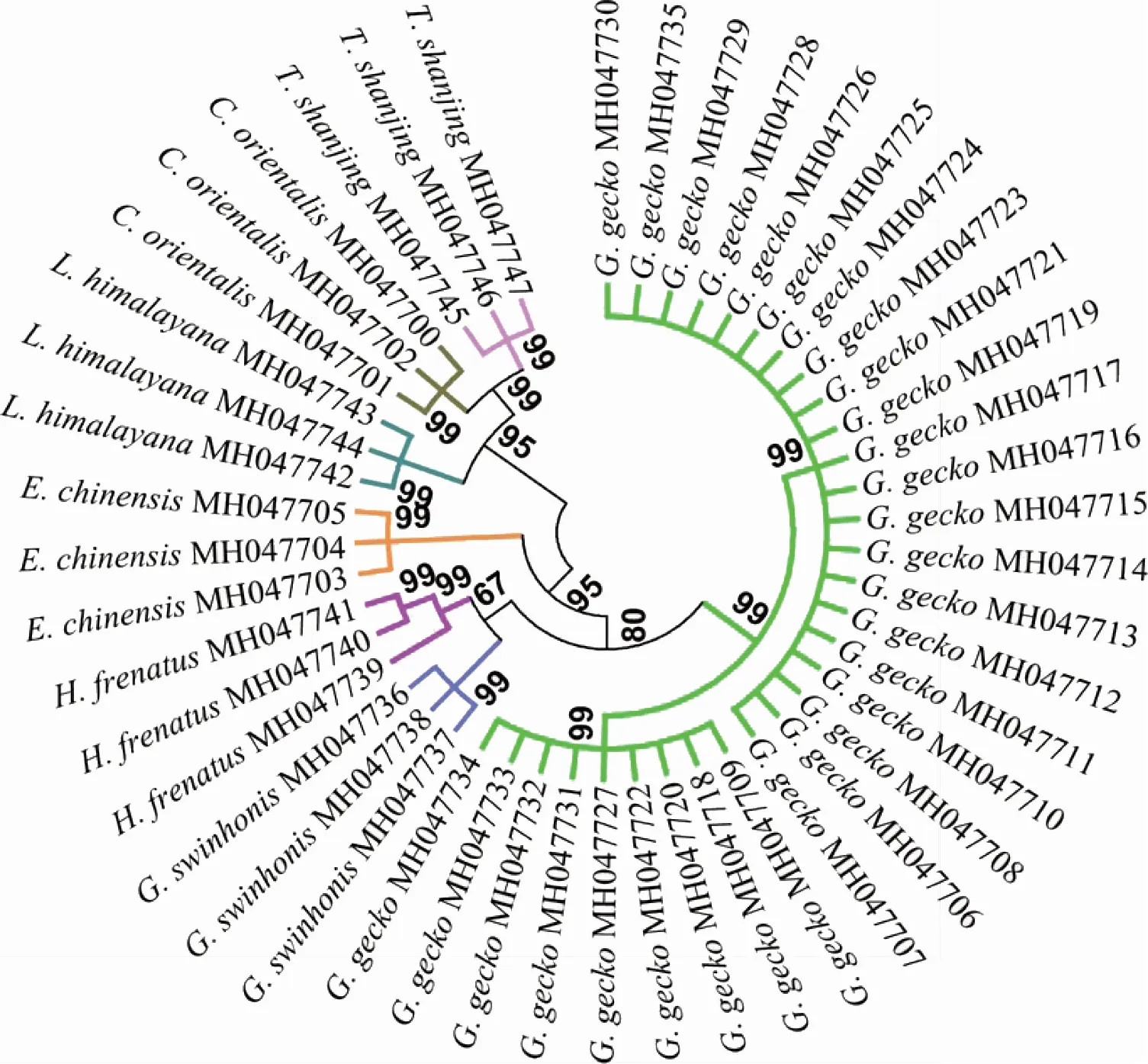
注:bootstrap 1000次重复,仅显示自展支持率≥50%。图3 基于12S rRNA序列蛤蚧及其混伪品的系统进化NJ树

注:A.归一化熔解曲线;B.DNA熔解曲线。图4 基于12S rRNA序列蛤蚧HRM曲线灵敏度检测
3.5 掺伪检测
设置0、1%、5%、10%、25%、50%、75%、100%的DNA掺伪比例(无蹼壁虎/蛤蚧),研究HRM技术对物种的检出能力。不同掺伪比例得到的曲线不同(图5),以蛤蚧的HRM曲线为基准,曲线相似性随着掺伪比例的增大而降低,当掺伪比例为1%时,该HRM曲线的相似性为99.07%(表3)。结果表明,基于12S r RNA序列的HRM曲线可实现蛤蚧的掺伪检测,最低检测限可达1%;而且,曲线相似性分析结果,对推算掺伪比例具有一定指导意义。

表3 不同掺伪比例HRM曲线基因型置信百分比 %
3.6 市场蛤蚧粉鉴定
在市场购买了10份蛤蚧粉,以基于12S rRNA序列的HRM技术其进行初步检测,以鉴定其真伪。每份样品进行3个物理重复,10份蛤蚧粉得30条HRM曲线(图6)。其中,SY8的3条曲线单独聚在一起,SY4、SY5、SY7的9条曲线聚为一类,其余6份样品的18条HRM曲线聚为另一类,没有与已知蛤蚧12S rRNA序列完全相同的样品。推测曲线聚为一起的蛤蚧粉来源接近。经测序验证,10份蛤蚧粉的测序峰图均出现不同程度的套峰,具体表现为部分位点出现套峰,底峰较高。对出现套峰的样品进行T-A克隆检测,10份市场蛤蚧粉均检测到多种蛤蚧单倍型,表明所购蛤蚧粉为混合样品。
此外,SY1、SY2、SY3、SY6、SY9、SY10曲线呈现异常,经Qubit 3.0测定DNA浓度低于1 ng·μL-1,表明DNA降解严重,其原因可能为:炮制过程中温度过高或时间过久;药材存放时间较久;药材肌肉组织含量少,如去尾后的蛤蚧。而且,经多次T-A克隆检测,比对到羽螨属Proctophyllodessp.物种和黑粪蚊。样品疑似不洁。结合价格分析,SY6价格最低,为0.39元/g,另外5份售价为0.67~0.74元/g;而SY4、SY5、SY7和SY8的售价分别为0.60、0.56、0.56、1.60元/g,检测结果无异常。推测SY1、SY2、SY3、SY9、SY10有以次充好的可能。

注:A.归一化熔解曲线;B.以蛤蚧为参考基因型的差异化熔解曲线;C.DNA熔解曲线。图5 基于12S rRNA序列蛤蚧掺伪检测HRM曲线

注:A.归一化熔解曲线;B.以蛤蚧为参考基因型的差异化曲线;C.DNA熔解曲线。图6 市售蛤蚧粉HRM曲线
4 讨论
蛤蚧作为我国名贵动物中药材,相关鉴定研究多有报道,常用鉴定方法为形态鉴定、显微鉴定、理化鉴定等传统鉴定方法[11,28-29]。形态鉴定和显微鉴定均需要长期从事中药鉴定工作的专家完成,专业难度大,不易掌握;目前未发现蛤蚧的专属特征性成分,《中华人民共和国药典》中也未收载相关成分的含量测定方法,鉴定难度大。
目前,以DNA barcoding和特异性PCR方法为代表的分子鉴别技术较为高效。张红印、GU等[26,30]均通过COI序列对蛤蚧及其10种易混物种和伪品进行了DNA barcoding鉴定研究,也有学者基于蛤蚧及其多种混伪品的COI、16S rRNA、Cytb、12S rRNA片段设计特异引物进行鉴别[25,27,31-32]。但是,DNA barcoding技术的精准鉴定主要依赖于PCR产物的测序,若大宗中药材均采用测序检测,一则检测周期延长,二则测序成本高。而且,存放较久的样品、炮制品以及深加工后的中成药,因DNA降解,很难经传统PCR扩增到较长的DNA片段,如700 bp左右的COI序列。特异性PCR检测方法对于掺伪品则无法准确鉴别。
本研究以蛤蚧与其6种常见混伪品为研究对象,采用HRM技术进行鉴定研究。HRM技术灵敏度高,理论上可实现单个碱基的差异分析,且检测快速[12,18,33]。结果显示,蛤蚧、疣尾蜥虎和喜山岩蜥分别生成了2种峰型的熔解曲线,推测存在不同的单倍型,经测序验证,与熔解曲线分析结果相符。因此,通过HRM鉴别,可实现同一个物种不同单倍型的区分,检测灵敏度和特异性较强。而且,经Barcoding验证分析,蛤蚧与6种混伪品均能通过BLAST分析、遗产距离分析、NJ树分析区分开,表明蛤蚧及其6种常见混伪品可通过基于12S rRNA序列的Bar-HRM方法进行高效鉴别。
基于12S rRNA序列,对蛤蚧和无蹼壁虎进行掺伪检测,根据曲线相似性分析,掺伪比例为1%时,曲线相似性为99%,表明掺伪最低检测限可达1%。而Sakaridis等[34]针对驴肉的掺伪检测研究,显示出了更高的灵敏度,最低掺伪比例仅为0.1%。虽然不同物种、不同片段的扩增偏好性不同,但曲线相似性分析结果,对于评估药材是否掺伪及掺伪比例仍具有一定参考价值。经对10份市场蛤蚧粉检测发现,其中6份DNA浓度较低,且在后续T-A克隆检测中发现螨属和黑粪蚊物种,疑似存放不当,造成污染。
本研究基于12S rRNA序列建立的蛤蚧的Bar-HRM鉴别方法,为蛤蚧的快速鉴别、蛤蚧药材的掺伪检测及其质量评价提供了一定的技术支持,但蛤蚧的12S rRNA序列长度约为400 bp,对于深加工后的相关产品则很难检测,还需进一步开发片段进行研究。

