拟南芥AtSWEET4基因启动子序列、编码蛋白结构分析及其在病原菌胁迫下表达特性检测
刘晓柱 李银凤

摘要:【目的】分析擬南芥糖运输载体蛋白基因(AtSWEET4)启动子序列及其编码蛋白的结构,并检测病原菌胁迫下AtSWEET4基因的表达特性,为研究AtSWEET4蛋白在拟南芥生长发育和生物胁迫中的作用机制提供理论依据。【方法】采用生物信息学方法分析AtSWEET4基因启动子序列及其编码蛋白的结构,并采用实时荧光定量PCR(qRT-PCR)和β-葡萄糖苷酸酶(GUS)组织染色法检测人工接种菜豆晕疫病菌(Pseudomonas syringae pv. phaseolicola,Psp)NPS3121和丁香假单胞菌番茄致病变种(Pseudomonas syringae pathovar tomato,Pst)DC3000后AtSWEET4基因的表达情况。【结果】AtSWEET4蛋白由251个氨基酸组成,分子量为27.8 kD,分子式为C1300H2038N304O346S11,理论等电点(pI)为8.71,脂肪氨基酸指数为118.76,不稳定指数为38.72,平均亲水性指数为0.629,为稳定的亲水蛋白,且含有7个跨膜结构域,无信号肽,为非分泌型蛋白。AtSWEET4蛋白与芥属的亚麻荠和荠菜SWEET4蛋白亲缘关系最近,其次是十字花科的甘蓝和萝卜SWEET4蛋白,与禾本科的二穗短柄草SWEET4蛋白亲缘关系最远。AtSWEET4基因启动子序列中包含核心元件TATA-box、增强子元件CAAT-box、调控植物生长发育相关元件、激素响应相关元件、非生物胁迫响应相关元件和生物胁迫响应相关元件。Psp NPS3121和Pst DC3000均可诱导AtSWEET4基因表达,但表达模式不同:接种Psp NPS3121后6和12 h的AtSWEET4基因表达明显上调,但至接种后24 h又迅速下降;接种Pst DC3000后AtSWEET4基因表达量逐渐增加。【结论】AtSWEET4基因的启动子序列特征及其编码蛋白结构与其参与植物免疫反应密切相关。
关键词: 拟南芥;运输载体蛋白(SWEET4);理化性质;结构特征;启动子序列;病原菌胁迫;表达分析
中图分类号: S432.2 文献标志码: A 文章编号:2095-1191(2019)09-1893-10
Abstract:【Objective】To provide theory basis for AtSWEET4 protein in regulating plant development and pathogensstress in Arabidopsis thaliana,the protein structural features and promoter sequence characteristics of A. thaliana sugars will eventually be exported transporters gene(AtSWEET4) were analyzed,expression pattern of AtSWEET4 were detected under pathogens stress conditions. 【Method】The sequence characteristics of AtSWEET4 gene promoter and structural features of its encoded protein were analyzed using bioinformatics methods. In addition,the expression of AtSWEET4 gene were also explored via real-time fluorescence quantitative PCR(qRT-PCR) after inoculation of Pseudomonas syringae pv. Phaseolicola(Psp) and Pseudomonas syringae pathovar tomato(Pst) DC3000 by beta glucuronidase tissue staining methods. 【Result】Sequence analysis demonstrated that AtSWEET4 protein was consistent of 251 amino acids, the molecular weight of AtSWEET4 was 27.8 kD, molecular formula was C1300H2038N304O346S11,and the isoelectric point(pI) was 8.71. Fatty amino acid index was 118.76, instability index 38.72,average hydrophilicity index was 0.629, which was a hydrophilic protein.It contained seven transmembrane domains, had no signal peptide and belonged to non secretion type protein. AtSWEET4 protein had the nearest relationship with SWEET4 protein of Camelina sativa and shepherd’s-purse in Brassica, followed by that of wild cabbage and carrot in Cruciferae, it had the farthest relationship with that of Brachypodium distachyon in Gramineae. AtSWEET4 promoter sequence contained core element TATA-box,enhancer element CAAT-box, plant growth and development related elements,hormone response related elements,abiotic stress response related elementsand biotic stress response related elements. PspNPS3121 and Pst DC3000 could induce expression of AtSWEET4 gene, but with different expression patterns. AtSWEET4 gene up-regulated 6 and 12 h after inoculation with PspNPS3121, but down-regulated after 24 h quickly. Expression of AtSWEET4 gene gradually increased after inoculation with Pst DC3000. 【Conclusion】The promoter sequence characteristics of AtSWEET4 and its encoded protein structural features are closed related to their defense function to plant immunity.
Key words: Arabidopsis thaliana; transport carrier protein(AtSWEET4); physicochemical property; structure features; promoter sequence; pathogens stress; expression analysis
0 引言
【研究意义】植物光合作用的主要产物,如蔗糖、葡萄糖和果糖等参与调控植物的多种生命活动,其跨生物膜系统运输时需要相应的蛋白质载体协助,如单糖转运蛋白、蔗糖转运蛋白和糖外排转运蛋白等(Slewinski,2011;Lahmidi et al.,2016)。其中,糖运输载体(Sugars will eventually be exported transporters,SWEETs)是近年来新发现的一类新型糖运输蛋白家族,参与调控植物生长发育和逆境胁迫响应(Li et al.,2018)。因此,分析拟南芥糖运输载体蛋白(AtSWEET4)基因及其启动子序列特征,检测病原菌胁迫下AtSWEET4基因表达特性,可为研究AtSWEET4基因在拟南芥生长发育和逆境胁迫中的调控机制提供理论依据。【前人研究进展】目前,主要利用模式植物拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)研究SWEETs基因家族功能(韩佳轩和姜晶,2015),但近年来在其他物种中也陆续被发现、克隆及表达(玄元虎等,2014;胡丽萍等,2017;Gao et al.,2018)。在植物体内SWEETs蛋白负责糖类物质的运输、再分配及贮藏等,从而调控植物多种生命活动。其中,水稻OsSWEET11是一种蔗糖低亲和型运输载体,参与蔗糖在水稻韧皮部装载的过程(Yuan and Wang,2013),且在OsSWEET5基因过表达植株中,叶片中可溶性糖和吲哚乙酸(IAA)的含量发生变化会导致幼苗期植株生长延迟和早衰(Zhou et al.,2014);葡萄VvSWEET4、VvSWEET7、VvSWEET10、VvSWEET11、VvSWEET15和VvSWEET17d等6个SWEETs基因家族成员在其浆果发育过程中表达量显著提高(Chong et al.,2014);芜菁BrSWEET9突变体中,BrSWEET9基因的表达被抑制,植株中花蜜分泌量显著减少(Lin et al.,2014);大豆中大多数SWEETs基因家族成员均在种子中表达,且随着种子灌浆期的发育其表达量逐渐提高,种子成熟后其表达量又逐渐下降(Patil et al.,2015);拟南芥atsweet8突变体植株四分体时期的小孢子因无法正常沉积孢粉素,而导致花粉外壁发育缺陷,植株雄配子育性降低(Guan et al.,2008)。此外,SWEETs参与植物的生物逆境胁迫反应,当细菌、真菌等植物病原菌入侵植物后,可导致植物SWEETs基因表达上调,使糖类物质从植物细胞进入病原菌细胞中,从而使其大量繁殖(Chandran,2015),如水稻感染白叶枯病菌后,白叶枯病菌分泌的特定转录激活(TAL)效应因子能结合在特定OsSWEETs基因的启动子区域,从而激活目标OsSWEETs基因的表达,促使更多糖类从植株细胞中进入病原菌中,促进病原菌增殖,导致植株感病(Li et al.,2013;Hu et al.,2014)。可见,SWEETs基因家族成员在宿主和病原菌互作过程中发挥重要作用。【本研究切入点】本课题前期研究发现,拟南芥AtSWEET4蛋白介导葡萄糖和果糖的运输,影响植物的生长发育过程(Liu et al.,2016),但目前关于AtSWEET4基因启动子序列特征及其表达对植物的抗性影响机制尚不清楚。【拟解决的关键问题】采用生物信息学方法分析AtSWEET4基因启动子序列及其编码蛋白的结构特征,并采用实时荧光定量PCR(qRT-PCR)和β-葡萄糖苷酸酶(GUS)组织染色法检测人工接种病原菌后AtSWEET4基因的表达情况,为研究AtSWEET4蛋白在拟南芥生长发育和生物胁迫中的作用机制提供理论依据。
1 材料与方法
1. 1 试验材料
供试拟南芥品种为Col-0生态型。拟南芥种子在冰箱内4 ℃春化2 d后播种于植物营养基质中(营养土∶蛭石=1∶1),22 ℃,12 h/12 h光照/黑暗恒温培养。菜豆晕疫病菌(Pseudomonas syringae pv. phaseolicola,Psp)NPS3121和丁香假单胞菌番茄致病变种(Pseudomonas syringae pathovar tomato,Pst) DC3000由湖南農业大学细胞生物学实验室提供。主要试剂:大肠杆菌JM109感受态细胞、植物基因组DNA提取试剂盒、植物总RNA提取试剂盒、M-Mulv逆转录酶试剂盒和荧光定量PCR试剂盒均购自生工生物工程(上海)股份有限公司。
1. 2 试验方法
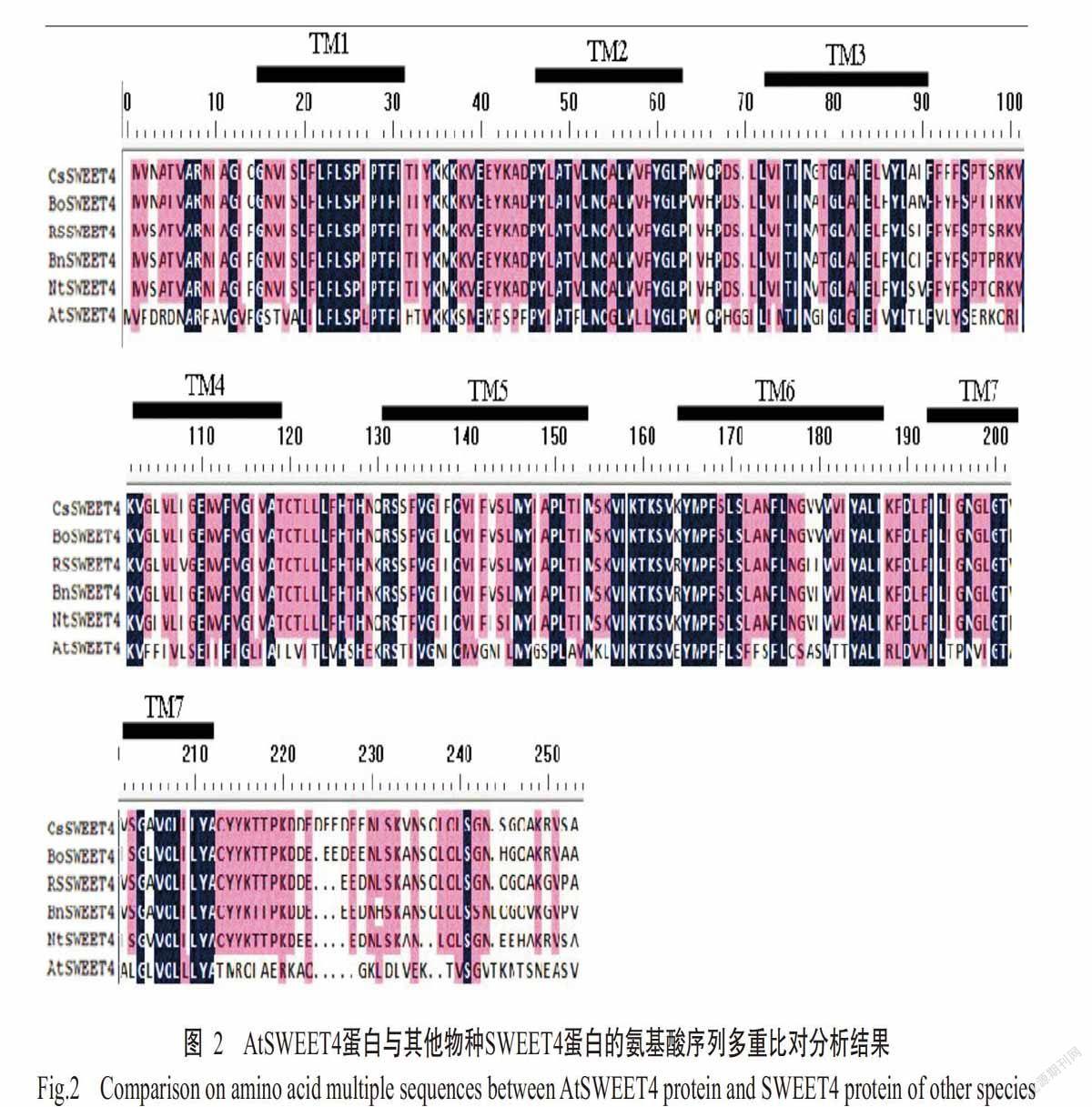
1. 2. 1 AtSWEET4基因启动子序列及其编码蛋白的生物信息学分析 从拟南芥信息资源库(The Arabidopsis Information Resource,TAIR)中下载获取拟南芥SWEET4基因(AT3G28007)编码区及其编码的氨基酸序列和起始密码子ATG上游2000 bp的启动子序列。采用瑞士生物信息学研究中心蛋白质专业分析系统(http://www.expasy.org/tools/pi_tool.html)、国际生物测量学与进化生物学实验室在线系统(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_dsc.html)和ProtScale(https://web.expasy.org/protscale/)等在线工具分析AtSWEET4蛋白的理化性质。采用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)、TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/)、PRABI-Lyon-Gerland(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_ automat.pl?page=/NPSA/npsa_dsc.html)和SWISS-MODEL(https://swissmodel.expasy.org/repository)等在线工具分析AtSWEET4蛋白的信号肽、跨膜结构域等结构特征;以MEAG 7.0构建AtSWEET4蛋白系统发育进化树;运用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)、NEW PLACE(http://www.dna.affrc.go.jp/PLACE/signalscan.html)和MethPrimer 2.0(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi)等在线工具分析AtSWEET4基因的启动子序列特征。
1. 2. 2 植物表达载体构建及拟南芥遗传转化
1. 2. 2. 1 AtSWEET4基因启动子克隆及植物表达载体pBI121-psweet4::GUS构建 采用植物基因组试剂盒提取拟南芥基因组DNA。利用提取的基因组DNA、拟南芥SWEET4基因启动子引物pSWEET4-F和pSWEET4-1391R(表1)进行SWEET4基因启动子序列PCR扩增。PCR反应体系(50.0 μL):10×PCR Buffer 5.0 μL,DNA模板2.0 μL,10 mmol/L dNTPs 2.0 μL,10 μmol/L上、下游引物(pSWEET4-F和pSWEET4-1391R)各1.0 μL,Taq DNA 聚合酶5 U,ddH2O补足至50.0 μL。扩增程序:95 ℃预变性5 min;94 ℃ 1 min,60 ℃ 1 min,72 ℃ 2 min,进行35个循环;72 ℃延伸10 min。PCR产物用1%琼脂糖凝胶电泳进行检测,并切胶回收,经Pst I和Nco I酶切后与载体pBI121连接,连接产物转化至JM109感受态细胞中,通过酶切验证后挑取阳性重组植物表达载体,送至生工生物工程(上海)股份有限公司进行测序。
1. 2. 2. 2 拟南芥遗传转化 利用冻融法将构建的重组植物表达载体pBI121-psweet4::GUS转化根癌农杆菌GV3101。通过花絮侵染法,利用携带重组载体pBI121-psweet4::GUS的根癌农杆菌介导转化Col-0生态型拟南芥,继续培养拟南芥植株,待其种子成熟后收取种子,通过卡纳霉素抗性筛选即可获得转基因植株。
1. 2. 3 转基因植株检测 用卡纳霉素筛选出抗性植株,并提取其基因组DNA,以GUS-R和GUS-R(表1)为引物进行PCR检测。PCR反应体系(25.0 μL):10×PCR Buffer 2.5 μL,DNA模板1.0 μL,10 mmol/L dNTPs 1.0 μL,10 μmol/L上、下游引物(GUS-F和GUS-R)各1.0 μL,Taq DNA聚合酶3 U,ddH2O补足至25.0 μL。扩增程序:95 ℃预变性5 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1.5 min,进行35个循环;72 ℃延伸10 min。设野生型Col-0叶片DNA为模板的PCR反应体系为对照(CK)。PCR产物用1%琼脂糖凝胶电泳进行检测。
1. 2. 4 病原菌接种 采用KBM液体培养基振荡培养Psp NPS3121和Pst DC3000,培养条件为28 ℃,200 r/min。待菌液培养至OD600 nm为1.0时,4000 r/min离心2 min收集菌体,10 mmol/L MgCl2溶液洗涤菌体,并用10 mmol/L MgCl2溶液懸浮菌体至OD600 nm为0.2(×108 CFU/mL),加入Silwet L-77,使其终体积为0.2%。选取约3周龄生长健壮的拟南芥植株,采集其完全伸展开的叶片用于真空渗透接种,将菌体注入叶片后,擦拭多余的菌液。接种后0、6、12和24 h分别取大小一致的叶片各3片,液氮迅速冷冻后-80 ℃超低温冰箱保存备用。
1. 2. 5 qRT-PCR检测AtSWEET4基因表达情况 参照植物总RNA提取试剂盒说明提取拟南芥叶片RNA,并利用M-Mulv逆转录酶试剂盒反转录合成cDNA,用于qRT-PCR检测AtSWEET4基因在3周龄拟南芥Col-0叶片接种非寄主菌Psp NPS3121后,具体操作参照SYBR Green Mix说明,所用引物序列(SWEET4-F和SWEET4-R)见表1。
1. 3 统计分析
采用Excel 2007进行数据整理和作图,使用Photoshop CS 6.0辅助制图。
2 结果与分析
2. 1 AtSWEET4蛋白氨基酸序列及其理化性质分析结果
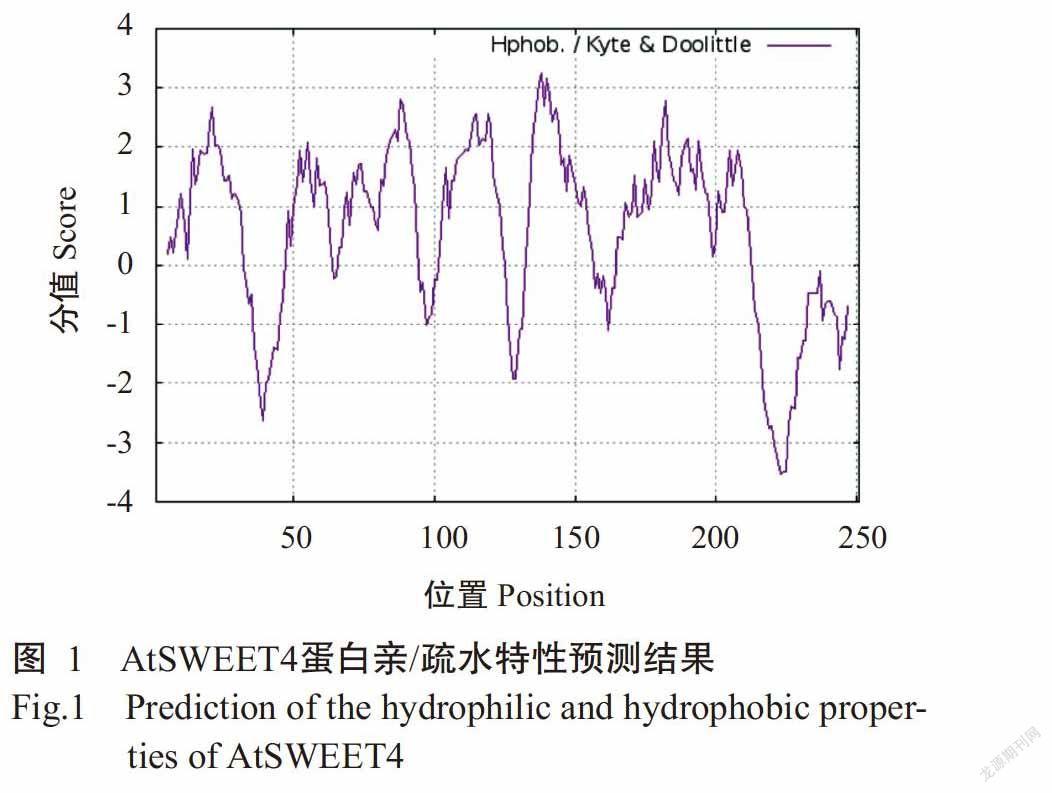
AtSWEET4基因序列分析结果发现,拟南芥AtSWEET4基因编码区全长756 bp,编码251个氨基酸。AtSWEET4蛋白分子量为27.8 kD,分子式为C1300H2038N304O346S11,理论等电点(pI)为8.71,脂肪氨基酸指数为118.76;不稳定指数为38.72,属于稳定蛋白。AtSWEET4蛋白中以亮氨酸、缬氨酸、异亮氨酸、苯丙氨酸、赖氨酸、丙氨酸、甘氨酸和苏氨酸含量较高,分别为12.4%、10.4%、8.8%、6.8%、6.4%、6.4%、6.4%和6.0%;其中,负电荷氨基酸残基(天冬氨酸和谷氨酸)数量为16个(6.4%),正电荷氨基酸残基(精氨酸和赖氨酸)数量为20个(8.0%),输水氨基酸(丙氨酸、异亮氨酸、亮氨酸、苯丙氨酸、色氨酸和缬氨酸)数量为115个(45.8%),极性氨基酸(半胱氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸和酪氨酸)数量为67个(26.7%)。
采用ProtScale预测AtSWEET4蛋白亲/疏水特性,结果如图1所示。该蛋白的最大亲水位点为第223位的天冬氨酸(D),分值为-3.544,最大疏水位点为第138位的半胱氨酸(C),分值为3.256,平均亲水性指数为0.629,即AtSWEET4蛋白属于亲水蛋白。
采用DNAMAN将AtSWEET4蛋白氨基酸序列与其他物种的SWEET4蛋白进行多重比对分析,结果发现AtSWEET4蛋白的氨基酸序列与亚麻芥(Camelina sativa)CsSWEET4、甘蓝(Brassica oleracea)BoSWEET4、萝卜(Raphanus sativus)RsSWEET4、苎麻(Brassica napus)BnSWEET4和烟草(Nicotiana tabacum)NtSWEET4的氨基酸序列相似性均超过70%(图2)。
2. 2 拟南芥AtSWEET4蛋白结构分析结果
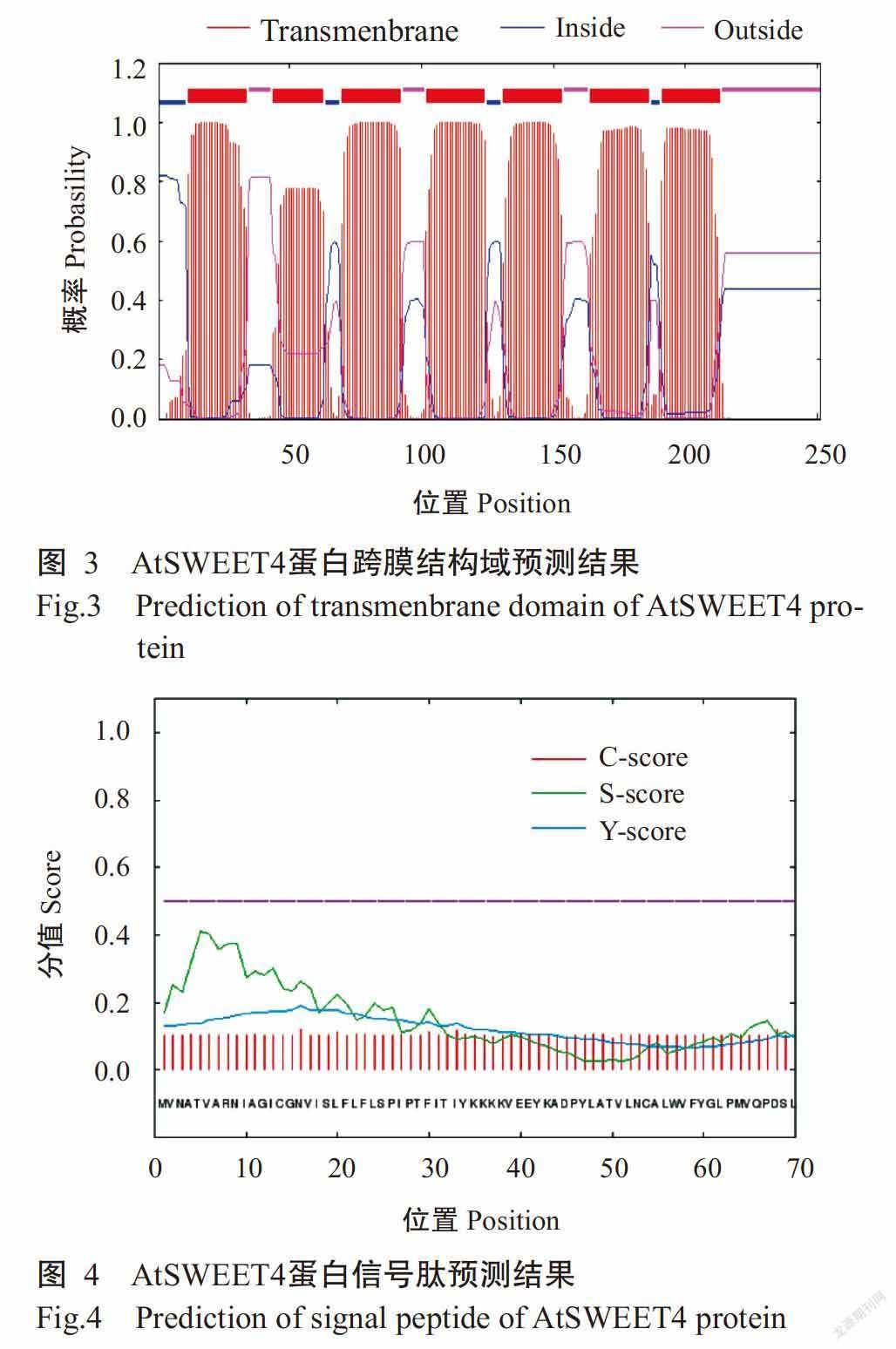
DNAMAN多重比对分析结果显示,AtSWEET4蛋白与CsSWEET4、BoSWEET4、RsSWEET4、BnSWEET4和NtSWEET4均包含7个典型的跨膜结构域(TM)序列(图2);采用TMHMM Server v. 2.0预测结果也显示AtSWEET4含有7个跨膜结构域(图3)。SignalP 4.1 Server预测结果显示,AtSWEET4蛋白无信号肽切割位点,表明其无信号肽,为非分泌型蛋白(图4)。AtSWEET4蛋白二级结构预测结果如图5所示,AtSWEET4蛋白二级结构中含α-螺旋数量最多,为122个(48.61%),无规则卷曲113个(45.02%),β-折叠16个(6.37%)。利用SWISS-MODEL预测AtSWEET4蛋白的三级结构,结果发现α-螺旋和无规则卷曲为该蛋白三级结构的主要成分(图6),与其二级结构预测结果吻合。
从GenBank下载不同物种SWEET4蛋白的氨基酸序列,并采用MEAG 7.0构建系统发育进化树,结果如图7所示。AtSWEET4蛋白与芥属的亚麻荠和荠菜(Capsella rubella)SWEET4蛋白亲缘关系最近,与十字花科的甘蓝和萝卜SWEET4蛋白亲缘关系较近,与禾本科的二穗短柄草(Brachypodium distachyon)SWEET4蛋白亲缘关系最远。此外,同科物种的SWEET4蛋白亲缘关系较近,如茄科的烟草(Nicotinana attenuate)和辣椒(Capsicum chinense)SWEET4蛋白处在同一分支,禾本科的水稻(Oryza sativa)、高粱(Sorghum bicolor)、糜子(Panicum miliaceum)、小麦(Triticum urartu)、玉米(Zea mays)和谷子(Setaria italica)SWEET4等处在同一分支,表明植物SWEET4蛋白在进化过程中相对较保守。
2. 3 AtSWEET4基因启动子序列分析结果
利用PlantCARE和NEW PLACE分析AtSWEET4基因启动子序列,结果如表2所示。该基因启动子序列中包含核心元件TATA-box、增强子元件CAAT-box、调控植物生长发育相关元件(MYB recognition site、MYB-binding site、T-box promoter motif、LFY consensus binding site motif、CCA1 binding site motif和I box promoter motif等)、组织特异性相关元件(GATA-box等)、激素响应相关元件(茉莉酸反应相关元件TGACG-motif、乙烯响应相关元件ERE、ABA信号途径ATHB5 binding site motif和生长素信号途径ARF binding site motif等)、非生物胁迫响应相关元件(厌氧诱导必要顺式作用元件ARE和低温反应相关元件LTR等)和生物胁迫响应相关元件(真菌诱导子响应元件W box)。
此外,采用Methprimer 2.0預测AtSWEET4基因启动CpG岛,结果发现在拟南芥AtSWEET4基因启动子中无CpG岛存在(图8),说明AtSWEET4基因未发生甲基化修饰和调控。
2. 4 重组植物表达载体pBI121-psweet4::GUS构建及纯合转基因植株获得
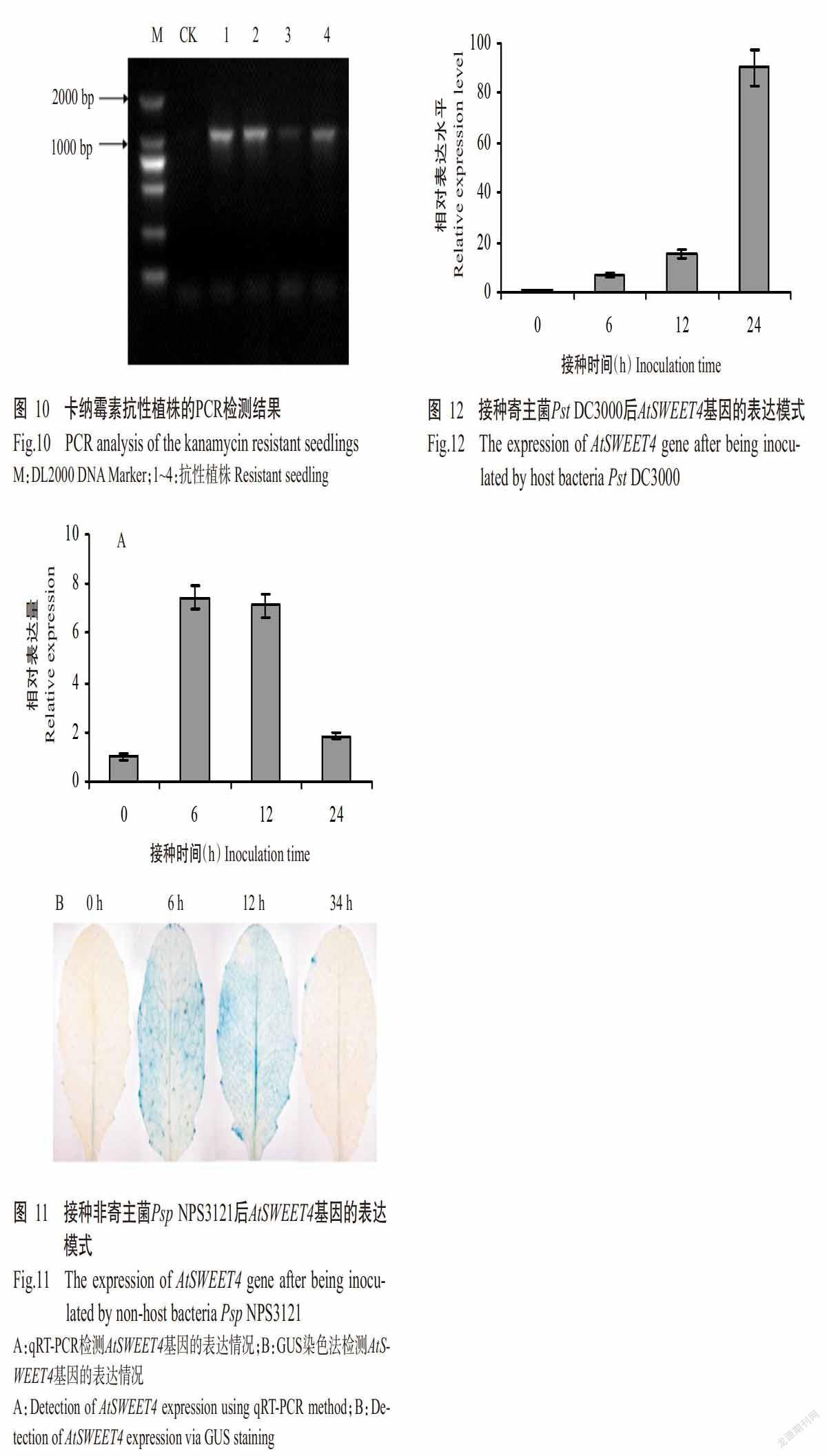
以拟南芥基因组DNA为模板,利用引物pSWEET4-F和pSWEET4-1391R进行PCR扩增,获得约2000 bp特异性条带,与预期结果相符(图9)。将其连接至表达载体pBI121上构建重组植物表达载体pBI121-psweet4::GUS,经根癌农杆菌介导转化拟南芥Col-0,通过卡纳霉素筛选,获得4株抗性植株,对其进行PCR检测,结果从4株抗性植株中均可扩增出约1500 bp特异性条带,而CK未检测出该条带(图10),表明筛选出的4株抗性植株均为转psweet4::GUS拟南芥植株。
2. 5 接种病原菌后AtSWEET4基因表达情况
采用qRT-PCR检测AtSWEET4基因在接种非寄主菌Psp NPS3121的拟南芥Col-0叶片中表达情况,结果如图11-A所示。接种Psp NPS3121可诱导AtSWEET4基因的表达,其中,接种后6和12 h的相对表达量约是接种前(0 h)表达量的7倍,接种后24 h的相对表达量又降至较低水平。为进一步证实该结果,转psweet4::GUS基因拟南芥植株接种Psp NPS3121,结果(图11-B)发现,接种6和12 h后植株叶片中GUS着色较深,说明AtSWEET4基因表达较强,而接种24 h后GUS着色较浅,暗示AtSWEET4基因的表达水平较弱。
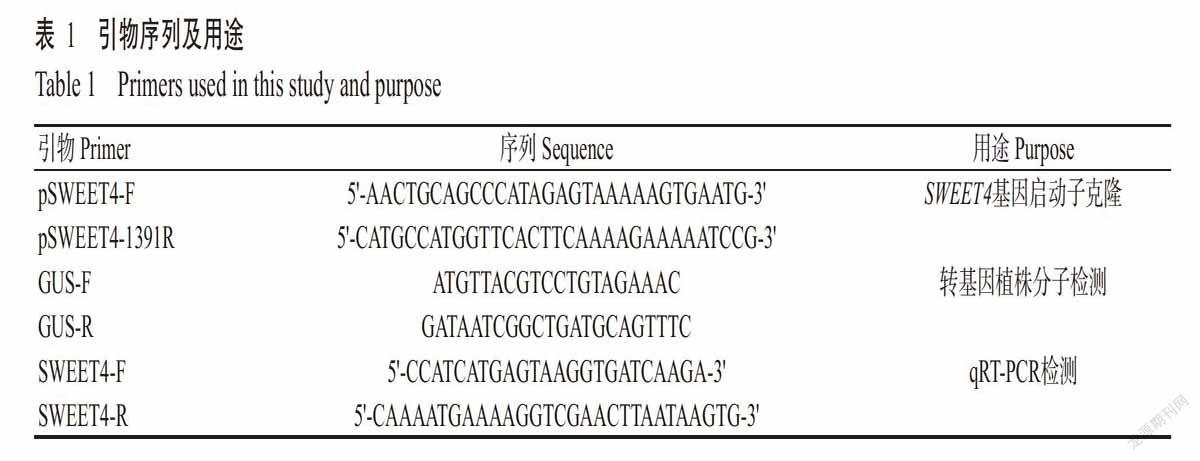
采用qRT-PCR检测AtSWEET4基因在3周龄拟南芥Col-0叶片接种寄主菌Pst DC3000后的表达情况,结果发现,接种Pst DC3000后,AtSWEET4基因的相对表达量不断升高(图12),说明Pst DC3000可诱导AtSWEET4基因的表达。
3 讨论
植物SWEETs蛋白属于MtN3/saliva家族,由7个跨膜结构域组成,其中前3个以TM1-TM3-TM2的形式排列,第4个跨膜结构域保守性较差,主要发挥连接作用,因此,形成3-1-3结构形式(Forrest et al.,2011;Xuan et al.,2013)。研究发现,水稻OsSWEET2b蛋白N端由TM4与THB1构成,C端由THB2构成(Tao et al.,2015);OsSWEET2蛋白中发挥葡萄糖运转功能的关键位点分别为TM2的半胱氨酸、TM3和TM7的天冬酰胺及TM6的苯丙氨酸(Tao et al.,2015)。虽然前人研究已证实AtSWEET4蛋白在拟南芥的生长和发育过程中发挥重要作用(Liu et al.,2016),但AtSWEET4蛋白理化性质和结构特征尚不清楚,为此,本研究采用生物信息学方法分析AtSWEET4蛋白的理化性质和结构特征,结果发现,AtSWEET4蛋白分子量为27.8 kD,理论等电点为8.71,分子式为C1300H2038N304O346S11,脂肪氨基酸指数为118.76,不稳定指数为38.72,属于稳定蛋白和亲水蛋白,无信号肽,含有7个跨膜结构域,其蛋白二级结构和三级结构中以α-螺旋最多(48.61%),其次是无规则卷曲(45.02%),表明AtSWEET4蛋白具有SWEET蛋白家族典型结构特征,但其分子结构还需借助电镜作进一步研究。
病原菌可诱导多种植物SWEETs基因成员的表达,如辣椒SWEET基因(UPA16)可被细菌性疮痂病菌(Xanthomonas campestris pv. vesicatoria)诱导表达(Kay et al.,2009);小麦SWEET基因可被条锈病菌(Puccinia striiformis)诱导表达(Slewinski,2011);木薯MeSWEET10a基因可被细菌性枯萎病(X. axonopodis pv. manihotis)的TAL20效应因子诱导表达(Cohn et al.,2014);甜橙CsSWEET1基因可被细菌性溃疡病(X. citris sp. citri)的PthA4和PthAw效应因子诱导表达(Hu et al.,2014);葡萄VvSWEET4基因参与调控灰霉病病原菌(Botrytis cinerea)的抗性(Chong et al.,2014)。本课题组前期研究发现,AtSWEET4基因表达上调,导致拟南芥对Psp NPS3121抗性降低(Liu et al.,2016)。本研究AtSWEET4基因启动子序列分析结果也显示,AtSWEET4基因启动子中存在病原菌诱导响应元件,因此,采用qRT-PCR和GUS组织染色法检测接种病原菌后AtSWEET4基因的表达模式,结果发现Psp NPS3121和Pst DC3000均可诱导AtSWEET4基因表达,但表达模式不同:接种Psp NPS3121后6和12 h的AtSWEET4基因表达明显上调,但至接种后24 h又迅速下降;接种Pst DC3000后,AtSWEET4基因表达量逐渐增加。二者表达模式的差异可能与病原菌的致病模式有关,Psp NPS3121属于拟南芥非寄主菌,侵染拟南芥后植株通过自身免疫系统抑制病原菌,导致AtSWEET4基因在接种后6和12 h呈上调表达,但接种24 h后AtSWEET4基因表达下调;Pst DC3000属于拟南芥的寄主菌,具有对抗植物免疫系统的效应因子,侵染拟南芥后可持续诱导AtSWEET4基因表达,为植株提供更多的糖分以抵抗病原菌侵染。
植物SWEETs家族蛋白不仅能为植物病原菌提供糖类物质,还可为有益微生物提供营养,如根瘤菌可诱导苜蓿MtSWEET11基因的表达,在形成共生根瘤形成时为其提供糖类物质(Kryvoruchko et al.,2016),说明SWEETs蛋白家族在植物与微生物的互作中发挥重要作用。但目前对该家族蛋白的研究尚处于起始阶段,其具体功能机理还需深入研究。
4 结论
AtSWEET4蛋白结构特征和启动子序列特征与其参与植物免疫反应密切相关。
参考文献:
韩佳轩,姜晶. 2015. 拟南芥、水稻和番茄SWEET/MtN3/saliva基因家族的分析[J]. 分子植物育种,13(3):581-588. [Han J X,Jiang J. 2015. Genome-wide analysis of SWEET gene family in Arabidopsis thaliana,Oryza sativa and Lycopersicum esculentum[J]. Molecular Plant Breeding,13(3):581-588.]
胡麗萍,张峰,徐惠,刘光敏,王亚钦,何洪巨. 2017. 植物SWEET基因家族结构、功能及调控研究进展[J]. 生物技术通报,33(4):27-37. [Hu L P,Zhang F,Xu H,Liu G M,Wang Y Q,He H J. 2017. Research advances in the structure,function and regulation of SWEET gene family in plants[J]. Biotechnology Bulletin,33(4):27-37.]
玄元虎,朱毅勇,胡一兵. 2014. SWEET蛋白家族研究进展[J]. 中国科学:生命科学,44(7):676-684. [Xuan Y H,Zhu Y Y,Hu Y B. 2014 Research advances of the SWEET proteins family[J]. Scientia Sinica Vitae,44(7):676-684.]
Chandran D. 2015. Co-option of developmentally regulated plant SWEET transporters for pathogen nutrition and abio-tic stress tolerance[J]. IUBMB Life,67(7):461-471.
Chong J,Piron M,Meyer S,Merdinoglu D,Bertsch C,Mestre P. 2014. The SWEET family of sugar transporters in grapevine:VvSWEET4 is involved in the interaction with Botrytis cinerea[J]. Journal of Experimental Botany,65(22):6589-6601.
Cohn M,Bart R S,Shybut M,Dahlbeck D,Gomez M,Morbitzer R,Hou B H,Frommer W B,Lahaye T,Staskawicz B J. 2014. Xanthomonas axonopodis virulence is promo-ted by a transcription activator-like effector mediated induction of a SWEET sugar transporter in cassava[J]. Molecular Plant Microbe Interactions,27(11):1186-1198.
Forrest L R,Kramer R,Ziegler C. 2011. The structural basis of secondary active transport mechanisms[J]. Biochimica Biophysica Acta,1807(2):167-188.
Gao Y,Wang Z Y,Kumar V,Xu X F,Yuan D P,Zhu X F,Li T Y,Jia B L,Xuan Y H. 2018. Genome-wide identification of the SWEET gene family in wheat[J]. Gene,642:284-292.
Guan Y F,Huang X Y,Zhu J,Gao J F,Zhang H X,Yang Z N. 2008. RUPTURED POLLEN GRAIN1,a member of the MtN3/saliva gene family,is crucial for exine pattern formation and cell integrity of microspores in Arabidopsis[J]. Plant Physiology,147(2):852-863.
Hu Y,Zhang J,Jia H,Sosso D,Li T,Frommer W B,Yang B,White F F,Wang N,Jones J B. 2014. Lateral organ bounda-ries 1 is a disease susceptibility gene for citrus bacterial canker disease[J]. Proceedings of the National Academy of Sciences of the United States of America,111(4):E521-E529.
Kay S,Hahn S,Marois E,Wieduwild R,Bonas U. 2009. Detailed analysis of the DNA recognition motifs of the Xanthomonas type III effectors AvrBs3 and AvrBs3Δrep16[J]. The Plant Journal,59(6):859-871.
Kryvoruchko I S,Sinharoy S,Torres-Jerez I,Sosso D,Pislariu C I,Guan D,Murray J,Benedito V A,Frommer W B,Udvardi M K. 2016. MtSWEET11,a nodule-specific sucrose transporter of Medicago truncatula[J]. Plant Physiology,171(1):554-565.
Lahmidi N A,Courty P E,Brulé D,Chatagnier O,Arnould C,Doidy J,Berta G,Lingua G,Wipf D ,Bonneau L. 2016. Sugar exchanges in arbuscular mycorrhiza:RiMST5 and RiMST6,two novel Rhizophagus irregularis monosaccharide transporters,are involved in both sugar uptake from the soil and from the plant partner[J]. Plant Physiology and Biochemistry,107:354-363.
Li T,Huang S,Zhou J,Yang B. 2013. Designer TAL effectors induce disease susceptibility and resistance to Xanthomonas oryzae pv. oryzae in rice[J]. Molecular Plant,6(3):781-789.
Li X,Si W,Qin Q Q,Wu H,Jiang H Y. 2018. Deciphering evolutionary dynamics of SWEET genes in diverse plant lineages[J]. Scientific Reports,8(1):13440.
Lin I W,Sosso D,Chen L Q,Gase K,Kim S G,Kessler D,Klinkenberg P M,Gorder M K,Hou B H,Qu X Q,Car-ter C J,Baldwin I T,Frommer W B. 2014. Nectar secretion requires sucrose phosphate synthases and the sugar transporter SWEET9[J]. Nature,508(7497):546-549.
Liu X,Zhang Y,Yang C,Tian Z,Li J. 2016. AtSWEET4,a hexose facilitator,mediates sugar transport to axial sinks and affects plant development[J]. Scientific Reports,6:24563.
Patil G,Valliyodan B,Deshmukh R K,Prince S,Nicander B,Zhao M Z,Sonah H,Song L,Lin L,Chaudhary J,Liu Y,JoshiT,Xu D,Nguyen H T. 2015. Soybean(Glycine max) SWEET gene family:Insights through comparative genomics,transcriptome profiling and whole genome resequence analysis[J]. BMC Genomics,16(1):520.
Slewinski T L. 2011. Diverse functional roles of monosaccharide transporters and their homologs in vascular plants:A physiological perspective[J]. Molecular Plant,4(4):641-662.
Tao Y,Cheung L S,Li S,Eom J S,Chen L Q,Xu Y,Perry K,Frommer W B,Feng L. 2015. Structure of a eukaryotic SWEET transporter in a homotrimeric complex[J]. Nature,527(7577):259-263.
Xuan Y H,Hu Y B,Chen L Q,Sosso D,Ducat D C,Hou B H,Frommer W B. 2013. Functional role of oligomerization for bacterial and plant SWEET sugar transporter family[J]. Proceedings of the National Academy of Scien-ces,110(39):E3685-E3694.
Yuan M,Wang S. 2013. Rice MtN3/Saliva/SWEET family genes and their homologs in cellular organisms[J]. Molecular Plant,6(3):665-674.
Zhou Y,Liu L,Huang W,Yuan M,Zhou F,Li X H,Lin Y J. 2014. Over expression of OsSWEET5 in rice causes growth retardation and precocious senescence[J]. PLoS One,9(4):e94210.
(責任编辑 陈 燕)

