18个双孢蘑菇核心种质的重测序初步分析
施肖垄 蔡志欣 郭仲杰 卢园萍 陈美元 廖剑华

摘要:【目的】通过对双孢蘑菇不同类型核心种质的基因组重测序分析,探讨双孢蘑菇不同类型菌株间基因组存在的差异及开发相关分子标记。【方法】对双孢蘑菇国内外杂交菌株、野生菌株、高产型或优质型传统菌株、棕色菌株、不育菌株等共18株核心种质进行基因组重测序,应用不同的生物信息学处理软件,对测序得到的原始reads序列与双孢蘑菇参考基因组H97序列进行比对,同时基于比对结果进行SNP、SV检测,通过检测结果对多态性标记分布进行统计并实现DNA水平差异基因挖掘和差异基因功能注释等。【结果】样品测序共获得21.63G数据量,Q30平均达到89.10%。样品的reads与参考基因组H97的比对效率平均为82.50%,基因组覆盖度为96.32%,平均深度分别在33X左右。基于测序数据与参考基因组的比对结果,共检测获得约813768个SNP,53840个InDel,平均每个个体获得924个SV变异。【结论】国内外菌株的亲缘关系表明As2796系列与ul系列是世界上并列的两大双孢蘑菇杂交品系。
关键词:食用菌;基因组;SNP;InDel;结构变异
中图分类号:S646.11文献标志码:A 文章编号:1008-0384(2019)10-1167-06
0引言
【研究意义】目前,多种食用菌已经完成了全基因组测序,包括双孢蘑菇(Agaricus bisporus)、草菇(Volvariellavolvacea)、香菇(Lentinusedodes)、黑木耳(AuricuLARIA Heimuer)等。在基因组测序完成并共享之后,利用新一代测序技术对基因组进行重测序变得简单易行且成本低廉。全基因组重测序是对已知基因组序列的物种进行不同个体的基因组测序,并在此基础上对个体或群体进行差异性分析。全基因组重测序的个体,通过序列比对,可以找到大量的单核苷酸多态性位点(Single NucleotidePolymorphisms,SNP)、插入缺失位点(Insertion/Dele.tion,InDel)、结构变异位点(structureVariation,SV)位点等。通过生物信息学手段,可以分析不同个体基因组间的结构差异,同时完成注释。【前人研究进展】通过全基因组测序及重测序,许多作物重要的农艺性状,如产量、抗逆性等基因被鉴定、克隆,这对改良主要农作物、提高产量、研究遗传变异、开发新的分子标记用以辅助育种等具有深远的意义。我国科学家对家蚕和水稻的重测序研究已经获得了重要的成果。福建农林大学通过草菇基因组的测序及重测序,获得基于SV位点的系列SCAR标记并用于构建遗传连锁图等。【本研究切入点】本研究从课题组所在单位保藏的400多个双孢蘑菇栽培与野生菌株中挑選了18个不同类型的代表性核心种质开展基因组重测序工作。【拟解决的关键问题】结合生物信息学分析,以期发现它们在基因组水平上存在的差异,开发SNP、SV等分子标记,并发掘相关基因,为进一步的双孢蘑菇遗传育种工作提供相关的理论依据与分子工具。
1材料与方法
1.1供试菌株
双孢蘑菇18个核心种质由福建省农业科学院食用菌研究所种质资源与遗传育种研究室保藏并提供,详见表1。
1.2试验方法
1.2.1菌株的培养将供试菌株从试管接入液体PDB培养基中,恒温24℃、转速220r·min振荡培养2~3周。
1.2.2基因组DNA提取与检测按OMEGA公司真菌基因组DNA提取试剂盒操作手册进行。提取的基因组DNA样品用0.8%琼脂糖凝胶电泳(电压5V·cm)检测其带型,用Nanodrop微量检测设备检测其浓度和杂质污染程度。
1.2.3基因组DNA重测序在北京百迈客(Biomarker)公司进行。提取检测合格的样品基因组DNA,用机械打断的方法(超声波)将DNA片段化,QIAquickPCR试剂盒纯化,末端修复、3’端加A、连接测序接头,再用琼脂糖凝胶电泳进行片段大小选择,最后进行PCR扩增,建好的文库用IlluminaHiSeqTM 2500进行测序。
1.2.4重测序数据分析应用不同的生物信息学处理软件,对测序得到的原始reads(双端序列)进行数据评估,然后将reads序列与双孢蘑菇参考基因组H97序列(https://genomejgi.doe.gov/Agabi_varbisH97_2/Agabi varbisH972.home.html)进行比对,并基于比对结果进行SNP、SV检测,通过检测结果对多态标记分布进行统计并实现DNA水平差异基因挖掘和差异基因功能注释等。使用samtools进行去重复(Mark Duplicates),GATK进行局部重比对(LocalRealignment),碱基质量值校正fBase Recalibration)等处理,再使用GATK进行SNP的检测,过滤,并得到最终的SNP位点集。使用GATK检测长度小于50bp的小片段InDel,使用snpEff软件对SNP和InDel变异位点进行注释。通过BreakDancer软件检测SV数据集,主要包含删除(Deletion,DEL)、插入(Insertion,INS)、倒位(Inversion,INV)、染色体内易位(Intra-chromosomal Translocation,ITX)、染色体间易位(Inter-chromosomal Translocation,CTX)。
2结果与分析
2.118个双孢蘑菇菌株重测序数据分析
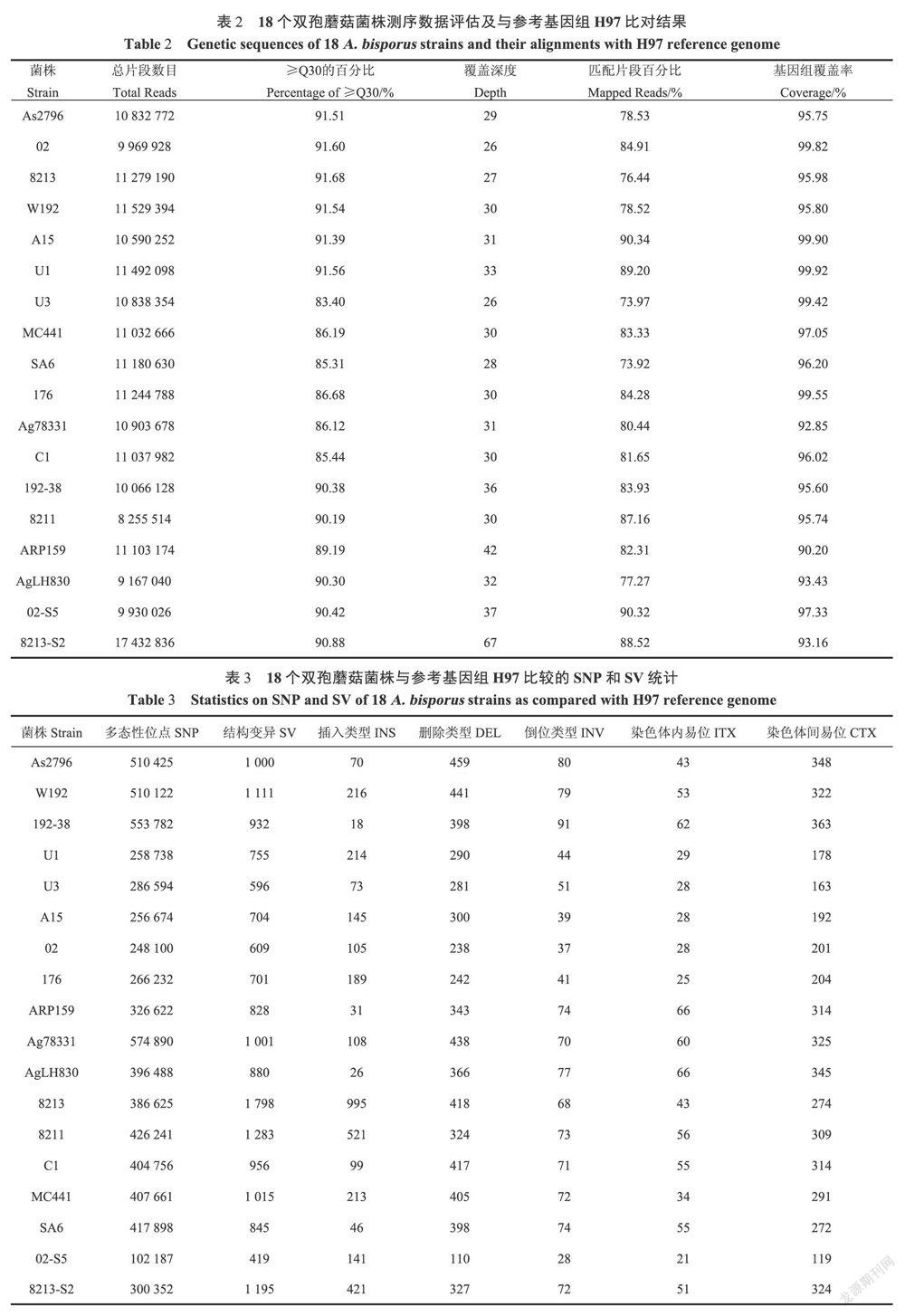
提取的基因组DNA质量好,Nanodrop检测主峰清晰,OD260/280及OD260/230数值显示杂质较少,符合重测序要求。对18个样品进行重测序分析,共获得21.63G数据量,Q30平均达到89.10%。通过bwa软件对二代基因组测序得到的短序列与参考基因组进行比对,定位测序reads在参考基因组上的位置,统计得到样品的测序深度、基因组覆盖度等信息。结果表明,样品的reads与参考基因组H97的比对效率平均为82.50%,基因组覆盖度为96.32%,平均覆盖深度约33X(表2)。染色体的覆盖深度分布图反映出样品的测序随机性较好。
2.2 18个双孢蘑菇菌株与参考基因组的SNP和SV分析
基于测序数据与参考基因组H97的比对结果,共检测获得约813768个SNP,53840个InDel,平均每个个体获得924个SV变异(表3)。从统计数据可以看出,国内杂交菌株如As2796、W192、国内野生菌株Ag78331、AgLH830等与H97比较的SNP、SV数量分别比国外杂交菌株如A15、u1、国外野生菌株ARPl59等大得多,表明国内野生菌株与H97的亲缘关系比国外野生菌株更远,而国内栽培菌株也与国外栽培菌株具有很大的差异。对这些变异进行了注释,发现存在于同义编码突变的SNP数量最多,其次是非同义编码突变和内含子区域。而InDel则主要存在于内含子区域,其次是基因的上游和下游区。
2.318个双孢蘑菇菌株DNA水平的差异基因挖掘
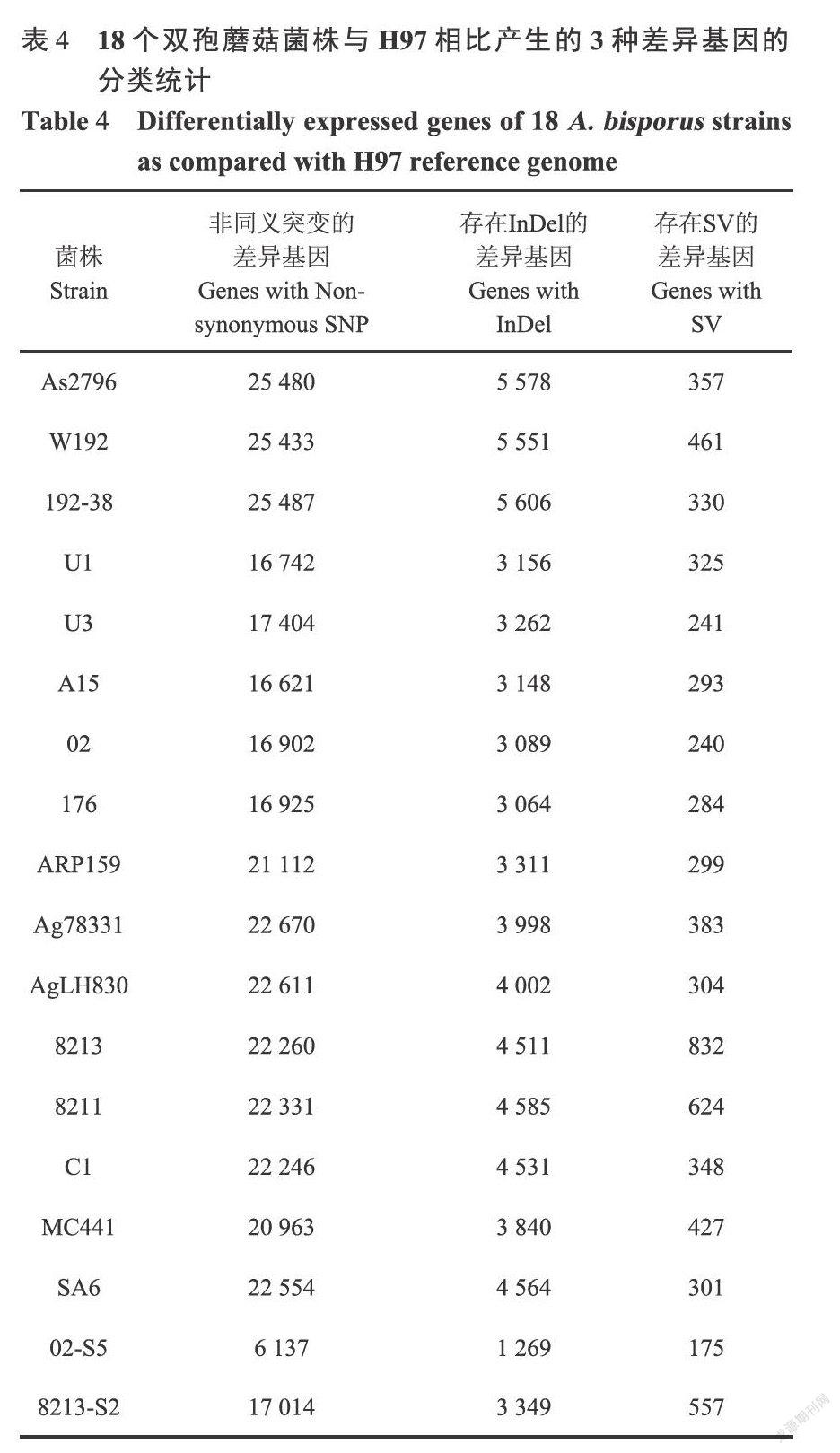
基因中碱基的差异就有可能造成其密码子的不同,此项分析用于寻找测序单株与参考基因组之间在基因层面的差异,包括SNP、InDel、SV等。较为典型的差异基因有如下3种:基因中存在非同义突变而产生蛋白差异、基因中存在小的InDel而导致基因功能改变或者丧失、基因中发生了结构变异而导致结构和功能的改变。在本研究的18个样品中,和参考基因组H97相比发生了以上3种变异的基因数,统计如表4所示。相比国外栽培菌株u1、A15系列,国内栽培菌株As2796、W192系列和参考基因组的差异基因数目较大,这与SNP、SV数量分析结果相似。从02分离的不育菌株02-$5与参考基因组有着明显较少的差异基因,这与它和H97同样是源自荷兰的高产类型菌株的单核体有关。使用BLAST将差异基因与NR、Swiss-Prot、GO、COG、KEGG等数据库比对,获得了这些基因的注释,可用于对差异基因功能作进一步的分析。
2.42个菌株之间比较的SNP和InDel数目
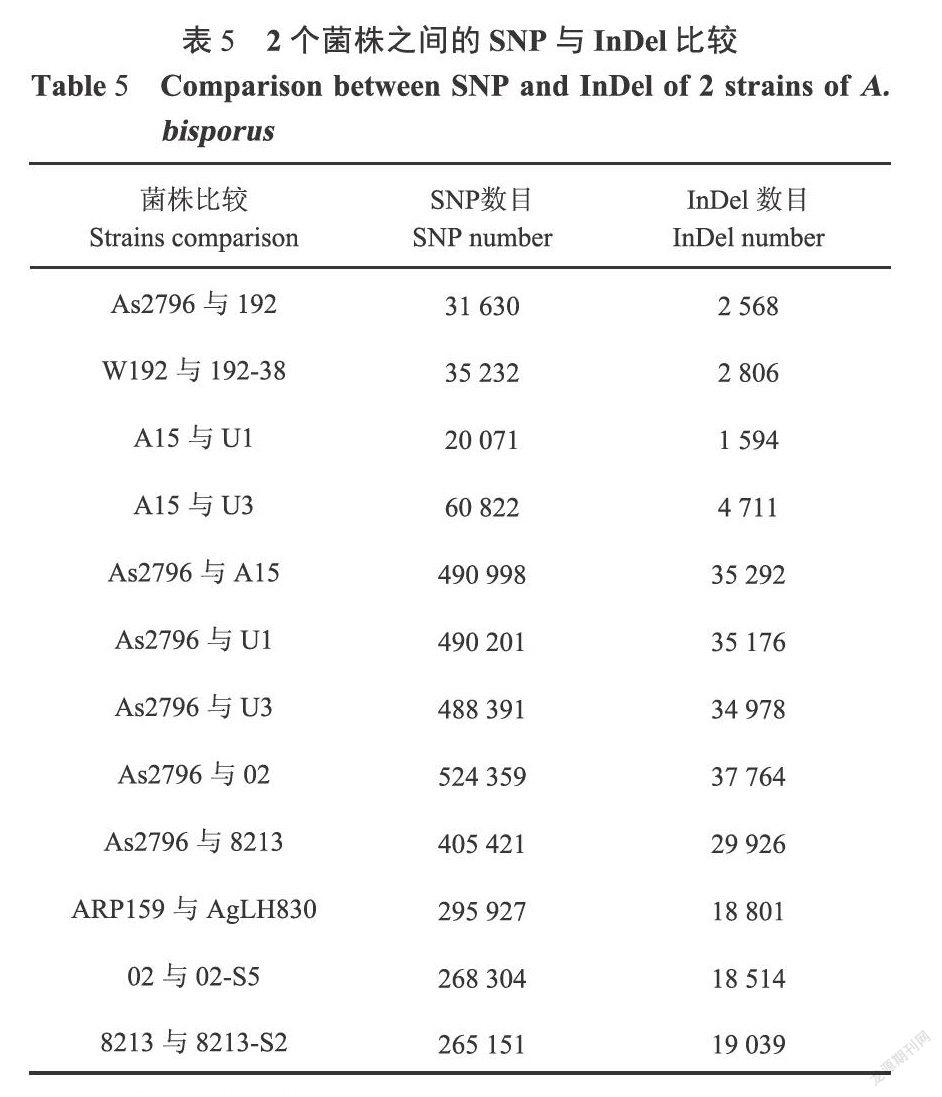
對部分菌株进行两两之间的SNP与InDel比较分析(表5),发现来源接近或农艺性状相近的菌株,其SNP和InDel数目就较少,比如As2796与其回交菌株W192、W192与其子代192-38、U1与其子代A15等。反过来说结果也是一致的,不同菌株间SNP和InDel数目较大的,其生物学特性和农艺性状差异就大,如国内杂交菌株As2796和国外杂交菌株U1、U3、A15,国内野生菌株AgLH830与国外野生菌株ARPl59,02,8213与其不育菌株等,其SNP和Indel的数目是相近菌株的数倍以上,其中SNP相差4~26倍,Indel相差4~24倍。
3讨论与结论
以As2796为代表的国内杂交菌株表现为较高产,质量优,耐粗放,适合农业与中国式工厂化栽培模式,适合鲜销与制罐,而以u1为代表的国外杂交菌株表现为高产,产量集中,质量一般,不耐粗放,适合欧美工厂化栽培模式,较适合鲜销。从SNP、SV及差异基因数目可以看出,国内外杂交菌株基因组差异很大。由于As2796的高产亲本02与u1的高产亲本Somycel9.2(其同核体H97即为参考基因组测序菌株)都是源自欧洲的高产传统菌株,SNP、SV及差异基因数目比较也说明它们之间的亲缘关系较为接近,所以可以推断国内外杂交菌株的差异主要源于另一个亲本的不同。As2796的优质亲本是老法国种8213,As2796很好地结合了02的高产与8213的优质特性,而u1的另一亲本为偏高产的米色菌株Somycel53,U1仍主要表现高产的特性,质量上不如As2796。因此,As2796系列与U1系列是世界上并列的两大双孢蘑菇杂交品系。
国内外野生菌株与H97比较的SNP、SV数量相差也较大,表明它们可能源自较远的分支,这对创新利用国内野生种质、育成具有自主知识产权的新品种意义重大。2个关系较近的菌株如As2796与W192、W192与192-38,它们之间的SNP、InDel标记可以开发成鉴别2个菌株的DNA标记,在菌株鉴别或菌种鉴定中比普通的DNA指纹标记更为适用。
本文对18个双孢蘑菇核心种质重测序数据进行了初步的统计和分析,获得了与参考基因组间大量的SNP、InDel和SV位点,提取了18个菌株的一致性序列,并进行了不同菌株之间的分组差异比较,为这些菌株的进一步分析奠定了基础。接下来将对部分感兴趣的差异和标记进行筛选和验证,以期为菌株鉴别、遗传分析、杂交育种等筛选一批有用的基因和分子标记。

