静宁鸡PPARa基因克隆与生物信息学分析
杨妍梅 李玉 覃圣 蒋刘芽 赵金潭 陆晓东 熊杰 陈伟基 扎西英派





摘要:为阐明静宁鸡PPARa基因的结构及功能,根据NCBI上登录的原鸡PPARc基因的CDS序列设计引物,以静宁鸡肾脏为材料采用PCR的方法,对目的基因进行克隆,并测序。根据测序的结果,采用各类分析软件,对所获得的DNA片段进行生物信息学分析。结果成功克隆了静宁鸡PPARa基因的全长CDS序列。生物信息学分析结果表明,该基因CDS全长1407 bp,所编码的蛋白质包含468个氨基酸。其分子量为52300,等电点为5.85。在二级和三级结构上,a-螺旋和无规则卷曲为蛋白的主要结构形式。PPARc蛋白是一个亲水性蛋白质,不存在跨膜区域,不含信号肽,没有N-糖基化位点,但存在16个0-糖基化位点,存在25个丝氨酸(Ser)磷酸化位点、12个苏氨酸(Thr)磷酸化位点和6个酪氨酸(Tyr)磷酸化位点。静宁鸡PPARa蛋白在第100~168个氨基酸之间有1个ZnF_C4结构域,在第264~449个氨基酸之间有1个HOLI结构域。同源性分析结果表明,静宁鸡PPARa基因与原鸡的亲缘关系最近。静宁鸡PPARa基因的成功克隆和功能预测为进一步研究PPARa基因在静宁鸡脂肪代谢中的作用提供了基础。
关键词:静宁鸡;过氧化物酶体增殖剂激活受体;生物信息学分析
中图分类号:Q785
文献标识码:A
文章编号:1000-4440(2019)02-0370-08
过氧化物酶体增殖剂激活受体(Peroxisome proliferators-activated receptors,PPARs)在1990年被英国科学家Isseman和Green首先发现"。它是一种新型的固醇类激素受体和配体依赖的核转录因子,属于核受体第一亚家族C群(NR1C)[2]。PPARs是一个由3种核受体组成的家族,目前已知有3种亚型:PPARa、PPARγ、PPARβ。这3种亚型由不同的基因编码,在组织中的表达和功能也不同[34。PPARs基因在心脏,脂肪组织,脑,肠,肌肉,脾脏,肺脏,肾上腺和大鼠脊髓中普遍存在。PPARs基因可被长链脂肪酸激活,广泛存在于体内脂肪代谢旺盛的组织中。
近年来,国内外学者对畜禽PPARs进行了深入研究,如Sundvold等[5]对猪10种组织PPARs的Northern blot检测发现,PPARa较高表达于肝脏和肾脏,中等程度表达于心肌、骨骼肌和小肠。Diot等[6]对鸡9种组织进行Northern blot检测,结果显示鸡PPARa mRNA在肝脏、心脏和肾脏中高表达,在尾脂腺也有较高的表达。孟和等[7]的研究结果表明,PPARa mRNA只在鸡心脏、肝脏、肾脏和胃这4种组织中表达,在肝脏杂交信号最强。田亚东等[8]的研究结果表明,PPARa基因影响鸡体脂肪代谢。孟和等[9]的研究结果表明,PPARa基因已在AA肉鸡中检测到单碱基突变位点,在3种中国地方品种中也检测到,如石岐杂鸡、北京油鸡和白耳鸡。该突变位点显著影响鸡群的腹脂质量和胴体性状,推测该基因是影响鸡体脂肪代谢的主效基因或与控制该性状的主效基因连锁,因此,它可作为鸡脂肪性状的标记辅助选择的分子标记[9],该研究结果与田亚东等[8]所得结果相似。
静宁鸡主产区分布在甘肃省静宁县和宁夏回族自治区固原市,甘肃省庄浪、通渭、华亭、秦安和会宁,宁夏回族自治区隆德、泾源和西吉等也有分布,静宁鸡以静宁县的鸡源多和品质好而得名。静宁鸡以低脂肪、高蛋白、肉质鲜嫩、鸡汁鲜美而闻名[10]。由于在实际生产中,静宁鸡生长速度较缓,在一定程度上影响了该品种的开发利用,以致静宁鸡日趋减少且表现出混杂退化的趋势。随着人们消费水平的提高,对低脂肪高蛋白质肉质的需求也逐渐提升。目前,PPARo的研究主要集中在啮齿动物和人体上,并且在家禽中也有PPARc的报道,但是没有关于静宁鸡PPARa的报道。因此,本研究以静宁鸡为研究对象,利用PCR获得PPARa基因序列,然后利用生物信息学手段对该基因的结构及功能进行分析和预测,以期为进一步研究静宁鸡脂质代谢中PPARa的调控作用提供基础。
1 材料与方法
1.1 試验动物
6月龄静宁鸡购于静宁绿洲生态农业科技发展有限公司。
1.2 试剂
Reverse Transcriptase M-MLV(RNase H-)等反转录相关试剂、TaKaRaExTaq等PCR反应相关试剂、pMD18-T Vector均购自宝生物工程(大连)有限公司,Trizol购自赛默飞世尔科技(中国)有限公司,氨苄青霉素购自北京索莱宝科技有限公司,Axyen胶回收试剂盒购自Axyen公司,BL2感受态细胞购于上海碧云天生物技术有限公司,质粒小量提取试剂盒购自生工生物工程(上海)股份有限公司。
1.3 方法
1.3.1 组织总RNA的提取和eDNA合成利用Trizol法提取静宁鸡肾脏的总RNA,以ddH2O溶解总RNA,-80°C保存备用;参考RTase M-MLV(RNaseH-)反转录操作步骤合成eDNA,-20°C保存备用。1.3.2引物的设计及PPARa基因的克隆根据NCBI公布的原鸡的PPARa基因的CDS序列(NM_001001464),用Primer 5.0软件进行引物设计。所设计的引物序列为:PPARa-F:5'-AGTGACCGCTC-TACTTGACCAA-3';PPARo-R:5'-CTCCGAAC-CGAGTGAACAGC-3 ',引物由宝生物工程(大连)有限公司合成。
以合成的eDNA第一链为模板,进行PCR扩增。反应体系为:TaKaRa Ex Taq (5 U/μl)0.5 μl,10xExTaq Buffer(mg2+ Plus)(20 mmol/L)2.5 μl,dNTPMixture(2.5 mmol/L)2.0 μl,引物PPARa-F(10μmol/L)0.5 μl,引物PPARa-R(10 μmol/L)0.5 μl,eDNA2.0μl,添加灭菌水至25.0μl。扩增反应程序为:95°C预变性5 min;95°C变性3 min,62.3°C退火45s,72°C延伸80s,35个循环,最后在72°C条件下进行10 min的延伸,4°C保存。扩增产物经过1.2%琼脂糖凝胶电泳检测后,切胶回收目的片段。与pMD18-T Vector载体16°C恒温连接0.5 h后,利用热激法转BL2感受态细胞,挑出阳性克隆进行培养,然后提取质粒进行PCR验证,并将质粒送至宝生物工程(大连)有限公司进行测序。测序结果用DNAStar、MegAlign和Editseq软件比对和拼接获得静宁鸡PPARa基因CDS序列。
1.3.3 PPARa基因的生物信息学分析采用在线软件Protparam(http://www.expasy.ch/tools/prot-param.html)预测PPAR蛋白的理化性质,采用Protscal(http://www.expasy.ch/tools/pr-otscale.ht-ml)预测PPARa蛋白的疏水性,采用SignalP4.1(http://www.cbs.dtu.dk/services/SignalP/)预测PPARa蛋白的信号肽,采用NetNGlye 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc/)预测PPARa蛋白的N-糖基化位点,采用NetOGlye 4.0Server(http://www.cbs.dtu.dk/services/NetOGlyc/)预测PPARa蛋白的0-糖基化位点,采用NetPhos-3.1(http://www.cbs.dtu.dk/services/NetPhos-3.1/)预测PPARo蛋白的磷酸化位点,采用TMpred(http://ch.embnet.org/software/TMPRED-form.html)预测PPARa蛋白的跨膜结构,采用PSORT II Prediction(http://psort.hge.jp/form.html)预测PPARo蛋白的亚细胞定位,采用APSSP2(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_gor4.html)预测PPAR&蛋白二级结构,采用SWISS-MODEL(http://swissmodel.Expasy.org/)预测PPARa蛋白三级结构,采用HMMER(https://www.ebi.ac.uk/Tools/hmmer/search/jackhm-mer)预测PPARa蛋白保守结构,采用NCBI中BLAST进行PPARa蛋白同源性分析,最后,通过CLUSTALX对PPARo氨基酸序列进行比对,然后采用MEGA 5.10软件基于NJ(Neighbor-joining)法构建PPARa蛋白的系统进化树,距离校正使用泊松模型,计算时将空位完全删除,自举分析通过1000次循环实现。
2 结果与分析
2.1 静宁鸡PPARa基因的克隆
采用TRIzol法对静宁鸡肾脏的总RNA进行提取,并进行琼脂糖凝胶电泳检测。结果表明,总RNA条带清晰,无拖尾,整体质量较高,符合基因克隆的要求。目的基因的PCR扩增产物进行琼脂糖凝胶电泳检测,结果显示,在1500 bp左右有明亮的条带,如图1所示。分析测序结果表明PPARa基因CDS区的24 bp处是起始密码子ATG,1430 bp处是终止密码子TAA。该序列BLAST的结果显示目的片段与NCBI上登录的原鸡PPARa基因的CDS序列(NM_001001464)99%相同,说明静宁鸡PPARa基因已被成功克隆出来。
2.2 静宁鸡PPARa蛋白的生物信息学分析
2.2.1 PPARx蛋白的氨基酸序列及理化性质分析通过在线软件Protparam分析PPARo蛋白的理化性质,结果表明,PPARo的CDS区包含1407个核苷酸,编码468个氨基酸,氨基酸组成见图2,分子量为52300,理论等电点为5.85,含强酸性氨基(Asp+Glu)61个,强碱性氨基酸(Arg+Lys)52个,PPARa蛋白的分子式为C2301H3661 N6250gS3z2,组成的原子总数为7317个。预测该蛋白质不稳定系数为44.20,表明该蛋白质为一个不稳定蛋白质。在哺乳动物细胞中的半衰期为30 h,在酵母中的半衰期大于20 h,在细菌中的半衰期大于10 h。
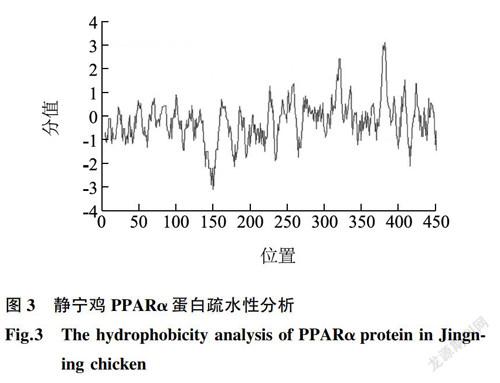
2.2.2 静宁鸡PPARa蛋白的疏水性和跨膜结构域分析利用在线软件Protscal预测静宁鸡PPARa蛋白氨基酸序列的亲水性/疏水性,结果如图3所示,第381位赖氨酸(Lys)疏水性最强(+3.156),第150位的丙氨酸(Ala)亲水性最强(-3.078),Z值为-0.225,表明PPARo蛋白为亲水性蛋白质。使用TMpred跨膜结构预测服务器对PPARa蛋白的氨基酸序列进行预测,结果发现,PPARo蛋白不存在跨膜区域,结果如图4所示。
2.2.3 静宁鸡PPARa蛋白的信号肽和糖基化位点分析采用在线软件SignalP 4.1 Server进行信号肽预测,结果显示,PPARa蛋白不包含信号肽。使用NetNGlyc1.0 Server和NetOGlyc 4.0 Server分别预测PPARa蛋白N-糖基化位点和0-糖基化位点,结果发现PPARo蛋白没有N-糖基化位点,但是在56、66、69、71、73、75、77、79、80、85、89、93、95、167、179、233处发现了O-糖基化位点。
2.2.4 静宁鸡PPARa蛋白磷酸化位点分析利用在线软件NetPhos-3.1 Server预测PPARa蛋白磷酸化位点,结果显示,有25个丝氨酸(Ser)磷酸化位点,分别在21、24、38、40、45、50、56、63、66、73、76、77、79、80、89、93、95、110、163、179、198、293、323、346和452位;有12个苏氨酸(Thr)磷酸化位点,分别在12、71、129、246、253、279、283、285、288、307、438和450位;有6个酪氨酸(Tyr)磷酸化位点,分别在9、194、214、314.464和468位,结果如图5所示。
2.2.5 静宁鸡PPARa蛋白的亚细胞定位预测利用PsortlIProtein Sorting Prediction软件对静宁鸡PPARa蛋白进行亚细胞定位预测,结果显示,静宁鸡PPARa蛋白主要定位于细胞质(47.8%),其他定位于細胞核(26.1%)、线粒体(17.4%)、内质网(4.3%)和高尔基体(4.3%)。由此预测静宁鸡PPARa蛋白可能为胞质蛋白。
2.2.6 静宁鸡PPARa蛋白保守结构域预测利用在线软件HMMER预测,结果发现静宁鸡PPARa蛋白在第100~168个氨基酸之间有1个核激素受体中的C4锌指结构(ZnF_C4结构域),在第264~449个氨基酸之间有1个核激素受体配体结合结构域(HOLI结构域)。
2.2.7 静宁鸡PPARa蛋白二级结构预测使用在线软件NPSA对静宁鸡PPARa蛋白二级结构进行分析,结果如图6所示,176个氨基酸参与ax-螺旋,占比为37.61%;83个氨基酸参与β_折叠,占比为17.74%;209个氨基酸参与无规则卷曲,占比为44.66%。由此说明,在静宁鸡PPARa蛋白二级结构中,a-螺旋和无规则卷曲是主要的结构形式。
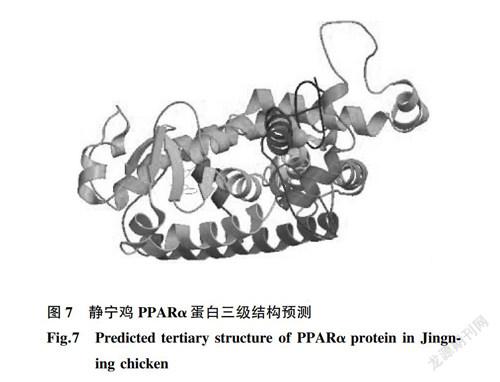
2.2.8 静宁鸡PPARa蛋白的三级结构预测利用在线软件SWISS-MODEL的自动建模功能,对静宁鸡PPARx蛋白三级结构进行预测,三维模型如图7所示。用于建立该模型的氨基酸残基范围为101~419位,該模型以3e00.1.B(Peroxisome proliferator-activated receptor gamma)蛋白为模板,序列同源性为64.39%。应用NCBI网站中Conserved domainsearch工具分析,证明PPARa蛋白是过氧化物酶体增殖剂激活受体。
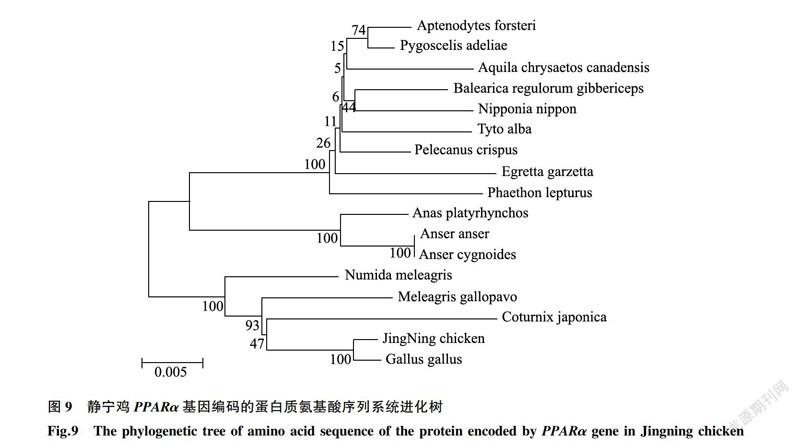
2.2.9 静宁鸡PPARa多序列比对分析及系统进化树对静宁鸡PPARa核苷酸序列和其他从NCBI下载的PPARa 核苷酸序列进行多序列比对。经BLAST在线分析,与静宁鸡PPARa核苷酸序列同源性高达99%的为原鸡(Gallus gallus,NM001001464.1),同源性高达98%的分别为火鸡(Meleagris gallo-pavo,XM_010716016.2)和吐绶鸡(Numida melea-gris,XM_0213932-56.1),同源性高达97%的为鹌鹑(Coturnix japonica,XM_015870635.1),同源性高达96%的分别为绿头鸭(Anasplatyrhynchos,XM_021273687.1)、阿德利企鹅(Pygoscelis adeliae,XM009323646.1)、卷羽鹈鹕(Pelecanus crispus,XM.009484690.1)、帝企鹅(Aptenodytes forsteri,XM_009283535.2)、仓鹗(Tyto alba,XM_009965270.1)、鸿雁(Anser cygnoides,KJ010765.1)、灰雁(Anseranser,AF481797.1)、白尾鹨(Phaethon lepturus,XM_010290610.1)、白鹭(Egretta gaettar,XM_009648790.1)、灰冠鹤(Balearica regulorum gibbericeps,XM_010300944.1)、朱鹦(Nipponia Nippon,XM_009465045.1)和加拿大金雕(Aquila chrysaetos Cana-densis,XM_011583046.1)。静宁鸡与原鸡相比,PPARa基因编码的氨基酸在98(亮氨酸/谷氨酸)和186(脯氨酸/亮氨酸)处发生突变,结果如图8所示。利用MEGA6.10软件构建NJ进化树,如图9所示。分析结果表明静宁鸡与原鸡,火鸡,吐绶鸡和鹌鹑的PPARa基因亲缘关系较近,其中与原鸡的亲缘关系最近。
3 讨论
PPARx是脂肪酸氧化酶基因的主要转录调控子,在调节脂质代谢平衡中起主要作用”。在本研究中,基于NCBI公布的原鸡的PPARa基因的CDS序列,通过Primer 5.0软件设计特异性引物,从静宁鸡肾脏中成功克隆了该基因的全长CDS序列。该CDS序列全长1407 bp,所编码的蛋白质包含468个氨基酸。其分子量为52300,理论等电点为5.85,分子式为C2301H3661N625O698S32,组成的原子总数为7317个。马云等研究发现鸭PPARa基因的cDNA全长1430 bp[13],最长开放阅读框为1407 bp,共编码468个氨基酸[12]孟和等研究发现鹅PPARa基因的cDNA序列长度为1407 bp[13]林森等从藏鸡肝脏组织中成功克隆得到PPARa基因,该基因编码区大小为1404 bp,编码468个氨基酸[14]。因此本研究结果与查阅的文献结果一致,说明已经成功克隆出静宁鸡PPARa基因。
磷酸化是蛋白质最常见和最重要的一种蛋白翻译后的修饰[15],并在许多生物反应中通过磷酸基团的变化,即添加或去除,发挥着“开关”作用[16]。糖基化具有调节蛋白质功能的作用,例如改善凝胶性和水合性等[17]。本研究中,静宁鸡PPARa蛋白为亲水性蛋白质,且不存在跨膜区域,不包含信号肽,没有N-糖基化位点,但存在16个0-糖基化位点,存在25个丝氨酸(Ser)磷酸化位点、12个苏氨酸(Thr)磷酸化位点和6个酪氨酸(Tyr)磷酸化位点,静宁鸡PPARa蛋白主要位于细胞质中。据报道,PPARa通过锌指结构域与视黄酸受体形成异源二聚体,然后与配体结合区(HOLI)连接。配体激活后的异源二聚体与靶基因启动子上的PPARa反应元件结合以激活靶基因活化,调节转录表达,然后调节脂类的氧化[18-19]。本研究中,利用在线分析软件预测结果显示静宁鸡PPARx蛋白在第100~168个氨基酸之间有1个ZnF_C4结构域,在第264~449个氨基酸之间有1个HOLI结构域,与查阅的文献结果[4]一致。
在静宁鸡PPARo蛋白的二级和三级结构中,a-螺旋和无规则卷曲是其主要的结构形式,β-折叠占比相对较少。多系列比对分析及系统进化树结果表明,静宁鸡PPARa基因与原鸡,火鸡,吐绶鸡和鹌鹑的PPARa基因亲缘关系较近,其中与原鸡的亲缘关系最近。
家禽PPARo基因调节脂肪代谢,影响家禽脂肪的沉积,从而影响腹脂率和胴体品质[2],因此,静宁鸡PPARo基因的成功克隆和功能预测为进一步研究PPARo基因在静宁鸡脂代谢中的作用和揭示PPARo基因功能提供了基础,为低脂高质量鸡育种提供基础研究资料。
参考文献:
[1]ISSEMANN L,GREEN S.Activation of a member of the steroid hormone receptor superfamily by perxisome proliferators[J].Nature,1990,347(6294):645-650.
[2]黄春红,肖调义,刘巧林,等.畜产动物过氧化物酶体增殖物激活受体基因的表达模式及功能研究进展[J].动物营养学报,2013,25(5):923-931.
[3]BRAISSANT O,FOUFELLE F,SCOTTO C,et al.Differential expression of peroxisome proliferator activated receptors(PPARs):Tissue distribution ofPPAR-a,β,and-y in the adult rat[J].Endocrinol,1996,137:345-349.
[4]ELBRECHT A,CHEN Y,CULLINAN C A,et al.Molecular cloning,expression and characterization of human peroxisome proliferator activated receptors γl and γ2[J].Biochemical and Biophysical Research Communicatons,1996,224:431-437.
[5]SUNDVOLD H,GRINDFLEK E,LIEN S.Tissue distribution of porcine peroxisome proliferator-activated receptor 0:detection of an alternatively spliced mRNA[J].Gene,2001,273:105-113.
[6]DIOT C,DUAIRE M.Characterization of a cDNA sequence encoding the peroxisome proliferator-activated receptor a in the chicken[J].Poultry Science,1999,78:119-1202.
[7]孟和,李輝,王宇祥.鸡PPARs基因组织表达特性的研究[J].遗传学报,2004,31(7):682-687.
[8]田亚东,亢娟娟,孙桂荣,等.PPARa基因对安卡x固始鸡资源群胴体品质的遗传效应分析[J].华北农学报,2010,25(6):14-18.
[9]孟和,王桂华,王启贵,等.鸡PPARa基因单核苷酸多态与脂肪性状相关的研究[J].遗传学报,2002,29(2):119-123.
[10]李宏强.静原鸡的地方品种形成及其种质资源保护措施[J].中国家禽,2009,31(3):52.
[11]张玥,姜宁,苏丽,等.PPARa与运动改善脂质代谢的关系[J].中国康复医学杂志,2008,23(6):495-504.
[12]马云,王云云,张晓婷,等.鸭PPARa基因结构及功能的生物信息学分析[J].浙江大学学报(农业与生命科学版),2011,37(4):371-379.
[13]孟和,李辉,王宇祥.鹅PPARa基因全长eDNA的克隆和序列分析[J].遗传,2004,26(4):469-472.
[14]林森,徐亚欧,林亚秋,等.藏鸡PPARax基因克隆与生物信息学分析[J].西南民族大学学报(自然科学版),2015,41(6):661-666.
[15]KIMJH,LEEJ,OH B,et al.Prediction of phosphorylation sites using SVMs[J].Bioinformatics,2004,20(17):3179-3184.
[16]EMANUELSSON O,NIELSEN H,BRUNAK S,et al.Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J].Journal of Molecular Biology,2000,300(4):1005-l016.
[17]MARCHAL I,GOLFIER G,DUGAS 0,et al.Bioinformatics in glycobiology[J].Biochimie,2003,85(1/2):75-81.
[18]NEVE B P,FRUCHART J C,STAELS B.Role of the peroxisome proliferator-activated receptors(PPAR)in atherosclerosis[J].Biochemical Pharmacology,2000,60(8):1245-1250.
[19]LALLOYER F,STAELS B.Fibrates,glitazones and peroxisome proliferator-activated receptors[J].Arteriosclerosis Thrombosis andVascular Biology,2010,30(5):894-899.

