OrchidBase 3.0:一个兰花基因功能和基因组进化研究的数据库
蔡文杰, 付志雄, 萧郁芸, 吴宛霖, 章迪杨, 兰思仁, 刘仲健, 陈虹桦,3
(1.台湾成功大学热带植物研究所,台湾 台南 701;2.台湾成功大学兰花研究与发展中心,台湾 台南 701;3.台湾成功大学生命科学学院,台湾 台南 701;4.福建农林大学兰科植物保护与利用国家林业与草原局重点实验室,福建 福州 350002;5.福建农林大学园林学院, 福建 福州 350002)
兰科是被子植物最大的科,兰科植物具有重大的园艺和药用价值,其种类、花形态、营养生长和生理特征的研究,特别是在花的形态与传粉者之间的特殊互作关系[1]、自然选择与遗传漂变的相互作用[2]、兰花—根菌之间的“奴役关系”[3]、景天酸代谢以及兰花的附生性等方面的研究[4]对植物进化模式研究方面提供重要的科学依据.
有几种兰花已作为模式物种并对其进行了研究,其中包括了蝴蝶兰(Phalaenopsis).由于蝴蝶兰及其杂交种对兰花育种及获取园艺性状的突变体具有重要的科学价值,通常被用于兰花发育调控的研究[5-6].其中,小兰屿蝴蝶兰(P.equestris)和台湾蝴蝶兰(P.aphroditesubsp.formosana)常常被用作育种亲本.小兰屿蝴蝶兰为二倍体,有38条染色体(2n=2x),其单倍体基因组的大小为1.16 Gb,在蝴蝶兰属种类中其基因组相对较小[7-8].此前,本研究团队建立了初代OrchidBase,用于储存和管理3种蝴蝶兰品种(台湾蝴蝶兰、小兰屿蝴蝶兰、原生蝴蝶兰) 的11个cDNA文库的转录组序列,它包括种子、原球茎、营养组织、叶片、冷处理和病原体接种的系列植株以及花序和花苞等组织的转录组数据[9].OrchidBase的第2个版本包括来自5个亚科的10种兰花,包括拟兰亚科的深圳拟兰(Apostasiashenzhenica)和麻栗坡三蕊兰(Neuwiediamalipoensis)、香荚兰亚科的深圳香荚兰(Vanillashenzhenica)和山珊瑚(Galeolafaberi)、杓兰亚科的兜兰(Paphiopedilumsp.)和杓兰(Cypripediumsp.)、兰亚科的厚瓣玉凤兰(Habenariadelavayi)和长距舌喙兰(Hemipiliaforrestii)、树兰亚科的墨兰(Cymbidiumsinense)和小兰屿蝴蝶兰的花器官转录组数据.
第3代OrchidBase的建立是基于对小兰屿蝴蝶兰全基因组数据的深入研究.小兰屿蝴蝶兰的全基因组大小约为1.16 Gb,有29 431个预测的蛋白质编码基因[10].该物种也是第一个全基因组测序的景天酸代谢植物.为了整合新增的小兰屿蝴蝶兰基因组数据,本研究团队建立了第3代可视化和具有实时对话功能的OrchidBase数据库,并提供易于使用的用户界面.用户可通过同源词或关键词搜索(如通过常见的Web浏览器)获取序列数据和注释信息,为研究兰花基因功能和基因组进化提供高质量的兰花基因组数据.
1 OrchidBase 3.0的架构和使用方法
OrchidBase 3.0是基于OrchidBase 2.0原有的架构,由Web端口、SQL Server数据库管理系统和Windows应用程序组成.其中,Web端口依托于静态的HTML页面和最新的.NET 102(Microsoft .NET framework 4.5)软件技术.不同于OrchidBase 2.0的是,OrchidBase 3.0由模型—视图—控制器(MVC)构成,利用ASP.NET开发的MVC 4框架[11]和Visual C编码#编程语言,可以动态执行检索任务,并使用交互式可视化网页显示小兰屿蝴蝶兰的基因组数据和基因功能注释结果.
主操作系统使用Microsoft Windows Server 2008 R2上的IIS 8.0版本,致使Genome Browser基因组浏览器可在Ubuntu 12.04上的Apache Web Server中实现可视化.此外,在XML和简单对象访问协议(SOAP)Web服务的基础上,该系统还提供了Web服务端口,而SQL Server系统被用于存储和管理收集的序列信息和注释数据.具体实施过程为:Windows应用程序执行序列分析,应用Perl和C#程序自动解析数据并构成数据库,利用开源工具和技术改进数据库覆盖范围、用户界面和系统性能.交互式数据可视化网页可通过D3和ASP.NET MVC框架体现.此外,JavaScript强大的工具包D3.js被应用于创建跨平台矢量图形,也为Web的可视化和以交互式的方式提供数据,并使其具有最高的兼容性数据.JBrowse是基于AJAX界面的浏览器,被用于定向导航蝴蝶兰基因组数据.该浏览器对服务器的性能要求较低,避免了不连续的过渡,从而增加用户在使用过程中的流畅度,实现平滑的动画滚动、缩放、导航和磁道选择[12-13].研究人员可以利用这些强大的桌面应用程序作为数据创作工具并进行资源共享.作为一个开放的、可通过Web访问的门户网站,OrchidBase 3.0大大简化了生物数据分析与可视化的工作流程,并将小兰屿蝴蝶兰基因组和转录组学等相关信息的可用数据集成一体.
1.1 扩展的数据库内容
小兰屿蝴蝶兰全基因组的组装支架(scaffold)序列(WGS_SCAFLDAPLD01S000001-APLD01S089583)是从NCBI数据库(http://www.ncbi.nlm.nih.gov/nuccore/APLD00000000.1/)中下载得到的.该数据集包含1.086 Gb的组装支架,约占小兰屿蝴蝶兰总基因组大小的93%.其中, 6 359个最长的组装支架包含了大约90%的总组装基因组(~980 Mb)[10].denovo基因预测、同源性预测和RNA序列辅助预测被用于注释组装基因组中的29 431个预测蛋白质编码基因和翻译氨基酸序列[10].这29 431个预测基因中的每个基因都分配了特定的基因ID(PEQU_ID)作为预测基因的标识符,这些基因可被用来研究进化历程中兰花生物过程的特异性功能.本研究团队还将所有RNA测序的读数映像到注释基因中,并统计叶片、根系、花茎和整花4个组织及萼片、花瓣、唇瓣和蕊柱4种花器官中每个基因的每千碱基读数每百万映射读数(reads per kilobase per million mapped reads information, RPKM),最后将这些信息都整合到OrchidBase 3.0中以提供小兰屿蝴蝶兰每个基因的完整表达谱.
1.2 小兰屿蝴蝶兰基因组信息的搜索
OrchidBase 3.0中包含的小兰屿蝴蝶兰基因组的组装支架和预测基因的相关信息可以通过Web界面搜索并通过“蝴蝶兰基因组”图标进行访问.此界面允许用户访问Genome Browser基因组浏览器、基因注释、基因定位、基因结构、基因表达、代谢途径和BLAST工具等,方便用户查询、获取各类基因组和预测基因数据(图1).
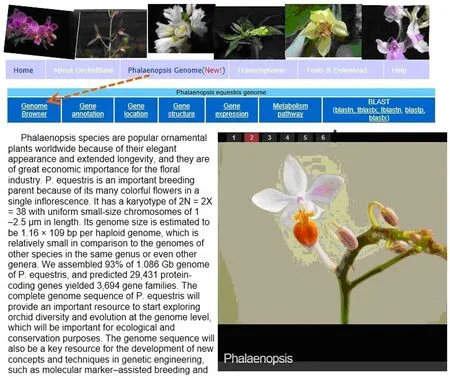
图1 OrchdBase 3.0的蝴蝶兰基因组页面Fig.1 A general overview of the Phalaenopsis genome page of OrchdBase 3.0
1.3 Genome Browse基因组浏览器
Genome Browser界面用于展示小兰屿蝴蝶兰基因组数据的信息(图2).该浏览器是新一代的基因组浏览器和JBrowse浏览器的结合体[13].JBrowse工具提供了所有的组装支架,使用户可以访问任何感兴趣的支架区域.标题部分通常包含支架标识符,下拉列表中通常包含支架编号.如果基因位置已知,可以将其作为“Scaffold ID:start..stop”输入搜索框.Genome Browser基因组浏览器还显示了预测基因的内含子/外显子结构并将Unigenes信息与基因组序列整合.当用户单击一个基因的时候会同时打开一个含有附加信息的弹出窗口,如用于下载外显子FASTA序列的链接或基因注释信息.
1.4 基因注释
“基因注释”页面列出了基因ID、相应支架的位置、同源的最佳匹配、E-value、Kyoto Encyclopedia of Genes and Genomes(KEGG)途径、Gene Ontology(GO)、Pfam和Interpro分类.用户可以在Web界面通过向服务器输入和提交关键词或Gene ID来查询目标信息,在关联数据库中能查询到基因序列和注释信息,并以适当的格式反馈给用户(图3).
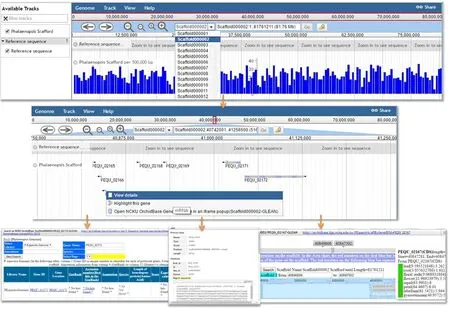
图2 JBrowse对蝴蝶兰基因组注释的可视化图Fig.2 Visualization of Phalaenopsis genome annotation by JBrowser
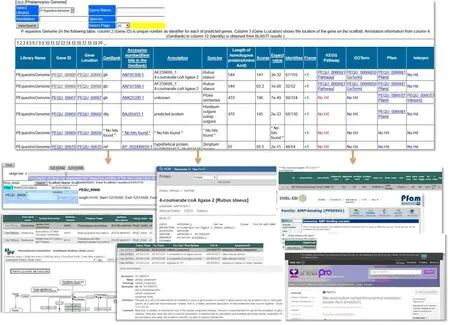
图3 蝴蝶兰注释基因的交互式网页Fig.3 Interactive web pages for Phalaenopsis annotated genes
1.5 基因定位、基因结构、基因表达和代谢途径
用户可以输入基因ID以检索一个支架上特定基因的位置(图4A),并分别通过“基因位置”和“基因结构”页面得到外显子位置的详细信息(图4B).用户也可以通过弹出的对话框得到注释信息页面及检索核苷酸和氨基酸序列的链接.另外,具有RPKM的注释基因列表可以在“基因表达”页面中看到(图5).用户可以输入基因ID或选择不同的页面以找到具有RPKM的8种组织和花器官的基因.“代谢途径”页面提供了相应KEGG途径的预测基因的信息.单击通路名称时,界面内容会被替换成KEGG途径中参与的预测基因ID,而图像显示的是数据库中被标识为红色的酶(图6).彩色途径图像是交互式的,允许用户访问KEGG数据库,搜索更多信息.
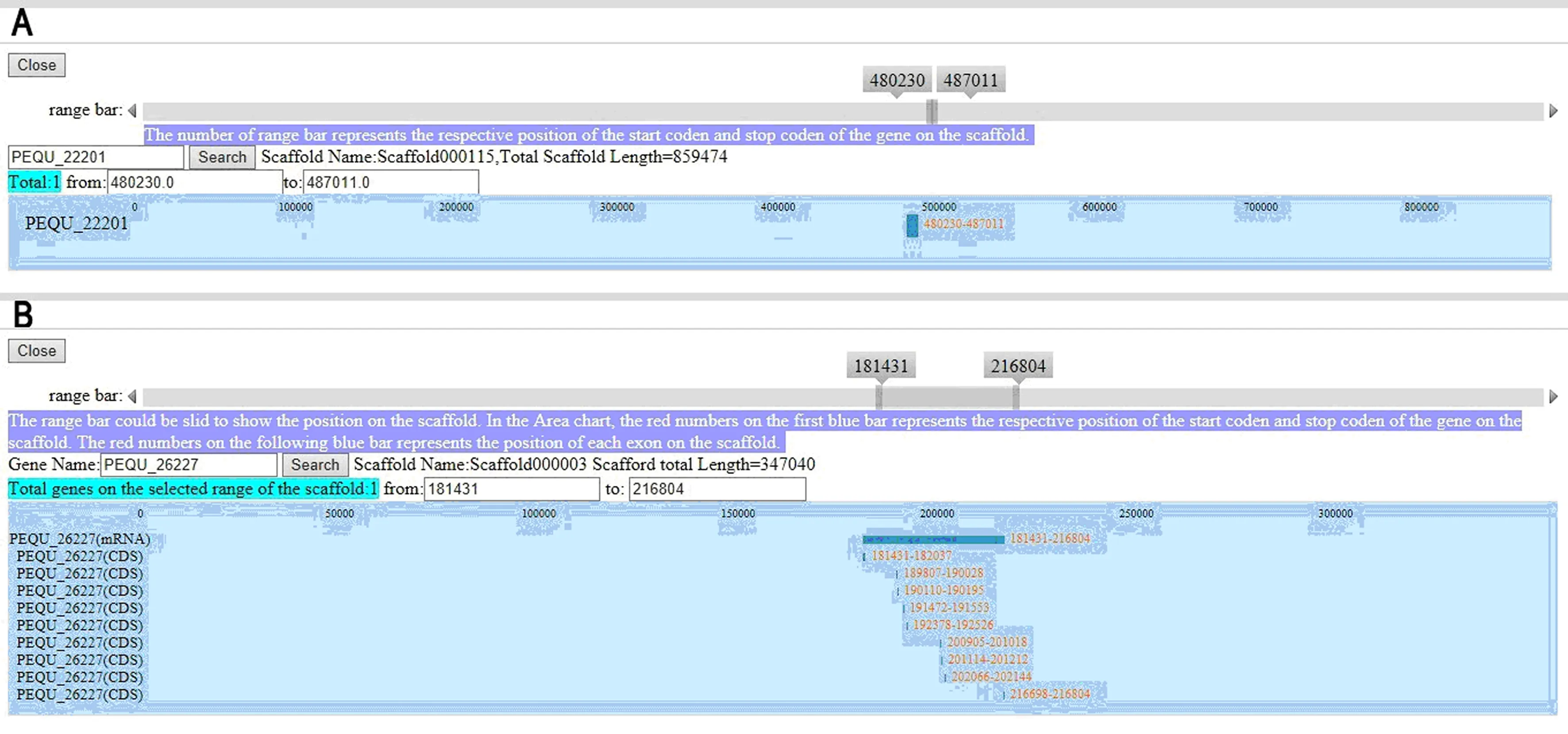
A:位置界面;B:结构界面.图4 “基因位置”和“基因结构”页面Fig.4 Interface for gene location and gene structure
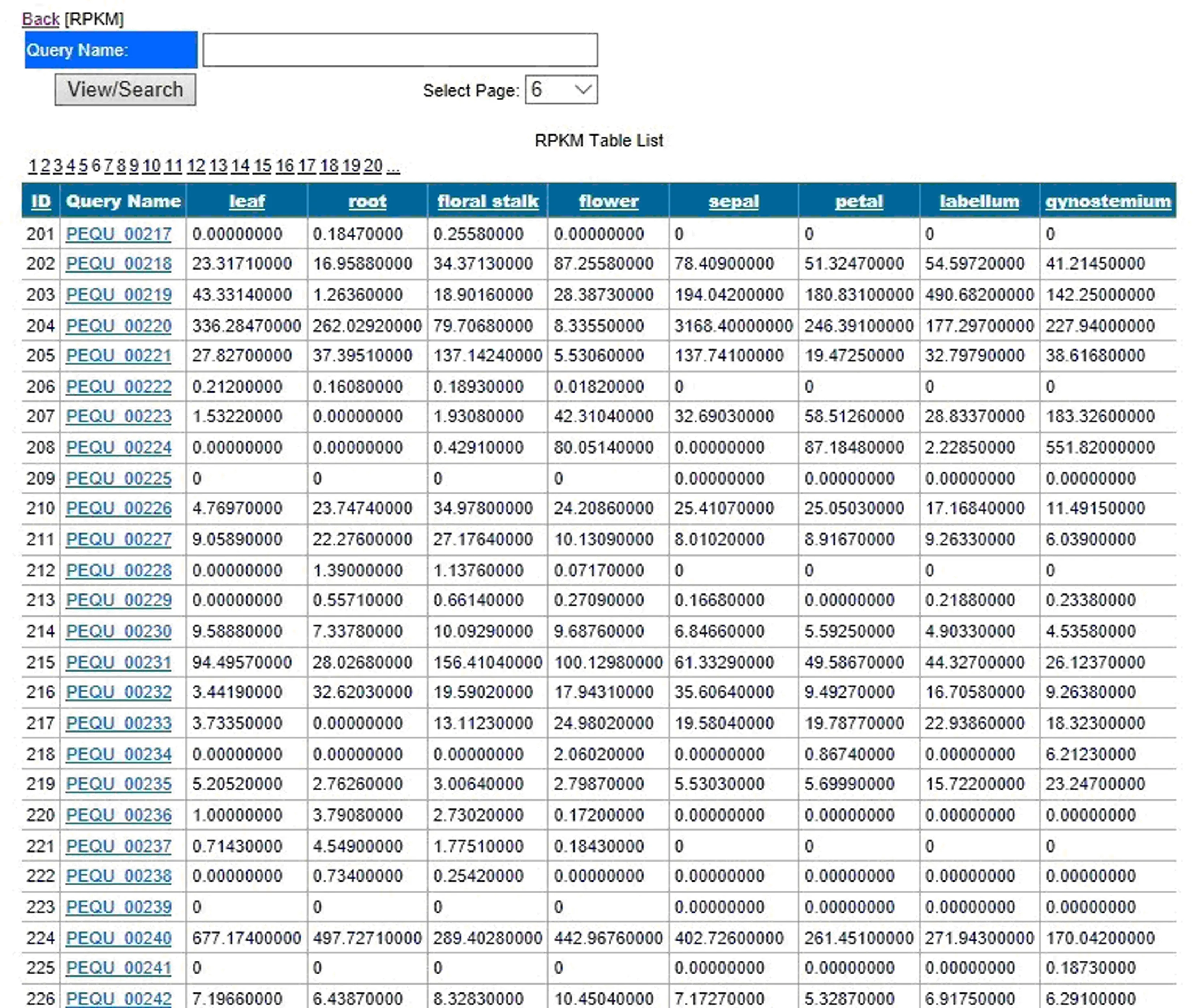
图5 “基因表达”页面Fig.5 Interface for gene expression
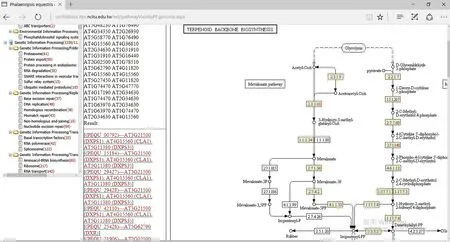
图6 “代谢途径”页面Fig.6 Interface for metabolism pathway
1.6 BLAST搜索
为了帮助用户进行序列比对,OrchidBase 3.0将BLAST嵌入在数据库中,通过Web表格为用户提供图像界面(图7).由此,组装的支架序列、预测的基因和蛋白质序列均可用于BLAST搜索[14].用户也可以使用各种BLAST搜索形式(BLASTn、tBLASTx、tBLASTn、BLASTp、BLASTx)对每种类型的序列进行相似性搜索,而序列可以通过在网页中输入数据来提交.用户可以在执行搜索之前设置适当的参数或只选择默认参数,BLAST搜索的结果会提供一个可以查询结果界面的链接,查询到的序列连同它们在组装支架上的位置将会根据预期值进行排序.
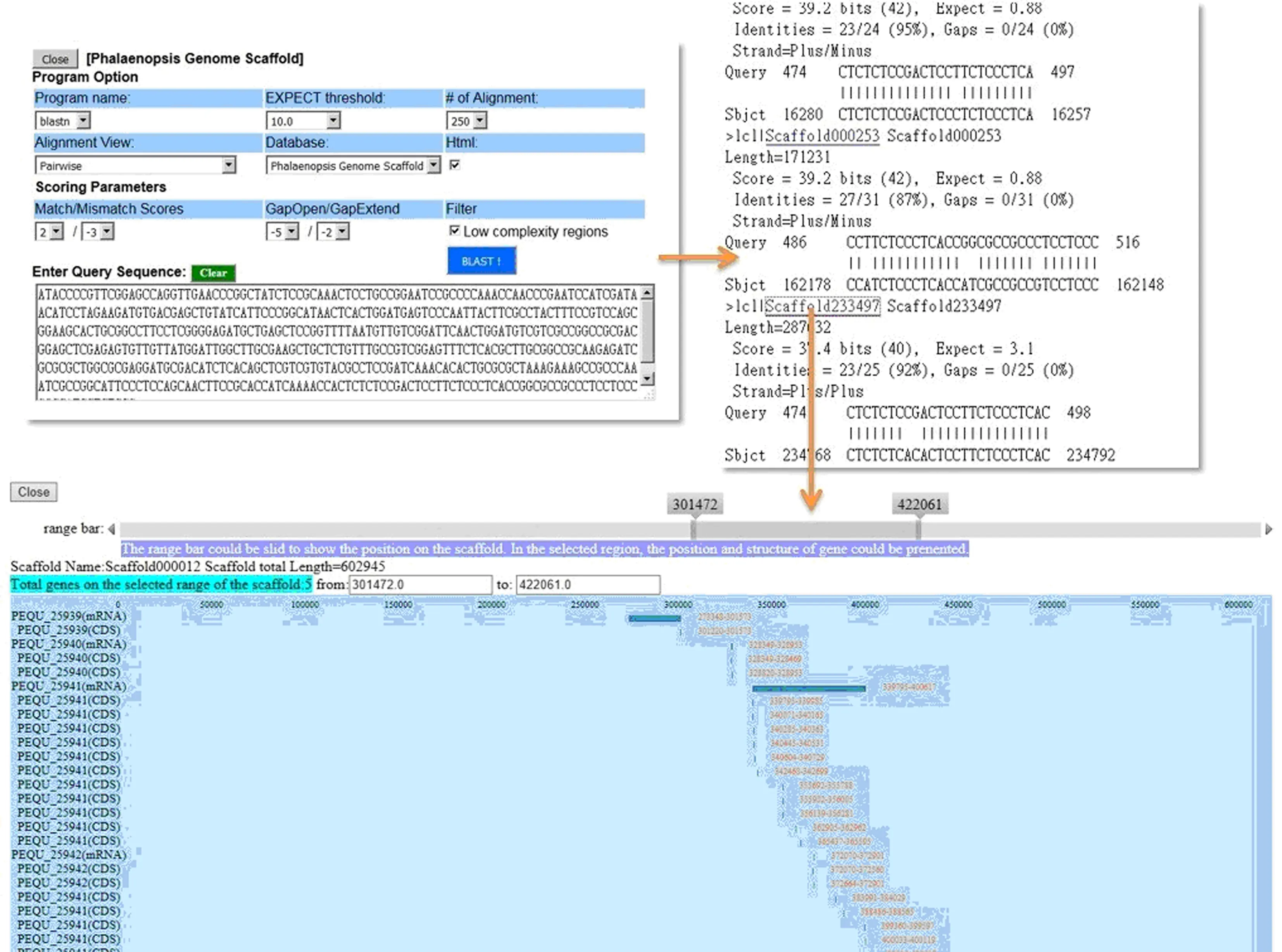
图7 BLAST搜索功能的页面Fig.7 Interface for BLAST-based search function
2 总结和今后的发展方向
蝴蝶兰全基因组序列的增加和整合,以及OrchidBase中详细的注释信息和易于使用的网络界面,可以帮助用户更有效地找到目标序列,如与花发育相关的基因[15]、花色素沉着模式的相关基因[16]、参与兰花胚珠发育的TCP转录因子基因的全基因组鉴定[17]等.此外,蝴蝶兰基因组序列已经为植物基因组进化和比较基因组研究提供了有价值的信息[18-19].OrchidBase使用基因组数据,有助于进一步了解兰花的基础信息.随着遗传和物理图谱构建技术的应用,将会构建蝴蝶兰基因组遗传和物理图谱,以更新OrchidBase,并引入新分析工具,以建立更加全面的兰花基因功能和基因组进化的数据库.

