基于末端脱氧核糖核酸转移酶的纳米界面上DNA的生长策略及用于恩诺沙星的适配体传感器构建研究
杜玉梅,周洋洋,卞晓军,2,3,颜 娟,2,3*
(1.上海海洋大学 食品学院,上海 201306;2.上海水产品加工及贮藏工程技术研究中心,上海 201306;3.农业部水产品贮藏保鲜质量安全风险评估实验室(上海),上海 201306)
纳米生物技术如今在多种领域发挥着重要作用[1-3]。1996年Mirkin等[4]首次提出通过金硫共价键在纳米金颗粒表面组装巯基DNA。纳米金(Au nanoparticles,AuNPs)具有功能性的表面、良好的生物相容性、较低的毒性等特殊性质,可以和寡核苷酸链、蛋白质等组装[5]。与生物材料相结合后的纳米金化学性质更加稳定,较大的表面积形成的自组装平台可提供更多的结合位点。基于纳米金的多重优越性,以金属纳米颗粒为载体构建的各种功能性纳米生物探针或者传感器已被广泛应用于生物成像以及分子检测[6-7]。
适配体[8](Aptamer)是一段通过指数富集的配体系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)筛选而来的寡核苷酸序列,能够高特异性和高选择性的识别细胞、核酸、蛋白质和小分子等靶分子[9-10]。相比于抗体,适配体易于合成和修饰、热稳定性好、成本较低、无毒性。因此,基于适配体的生物传感器已被广泛应用于各种分析检测[11-12]。单纯的依赖适配体捕获靶标前后构象变化[13]产生的信号,在检测方面的应用十分有限。为了进一步提升适配体生物传感器应用的广泛性,常常需要结合信号放大策略。尤其是基于核酸扩增的信号放大策略[14],包括聚合酶链式反应[15](Polymerase chain reaction,PCR)以及滚环扩增技术[16-17](Rolling circle amplification,RCA)和环介导等温核酸扩增(Loop-mediated isothermal amplification,LAMP)等。然而PCR技术不仅需要严格的温度控制,也需要扩增模板。等温核酸扩增中[18-19],RCA不仅需要环状模板,还需要特殊的引物设计;LAMP[20]虽不需要模板,但其需要多条引物以及设计复杂等弊端对该技术的广泛使用造成了一定的限制。末端脱氧核糖核酸转移酶(Terminal deoxynucleotidyl transferase,TdTase)[21-24]是一种在寡核苷酸链的3′-OH 端重复添加单核苷酸的酶,该操作无需模板和温度控制,常温下就能实现DNA链的末端延伸。
恩诺沙星(Enrofloxacin,ENR)别称乙基环丙沙星,是一种人工合成的氟喹诺酮类抗菌药物[25],现已被广泛应用于各种动物感染性疾病的预防和治疗,尤其近年来,在水生动物疾病防治中的应用发展迅猛。然而ENR的大量使用,造成了其在动物源食品中的过多残留,这种残留通过食物链的传递对人体造成毒副作用[26]。本研究使用ENR的核酸适配体组装在纳米金表面作为识别纳米生物探针,结合磁珠(Microbeads,MBs)分离技术构建针对ENR的适配体传感器(Aptasensor),通过TdTase介导的常温下纳米界面DNA的生长,实现了ENR识别信号的放大,从而建立了一种简单易操作的用于食品中ENR残留的检测分析方法。
1 实验部分
1.1 试剂与仪器
末端脱氧核糖核酸转移酶(20 U/μL,上海碧云天生物技术有限公司);磷酸盐缓冲液(20×PBS,pH 7.4~7.6)、琼脂糖IV(TM)、电泳缓冲液(50×TAE,pH 8.4)、4S Red Plus 核酸染色剂(10 000×水溶液)、脱氧核苷酸(dNTP,10 mmol/L)、三磷酸脱氧腺苷(dATP,100 mmol/L)、酪蛋白(Casein)、恩诺沙星(ENR)、诺氟沙星(Norfloxacin)、氧氟沙星(Ofloxacin)及环丙沙星盐酸盐(Ciprofloxacin hydrochloride)均购于生工生物工程(上海)股份有限公司;生物素标记的混合碱基(含有Biotin-dUTP,1 mmol/L,赛默科技有限公司);3,3’,5,5'-四甲基联苯胺(TMB,美国Neogen公司);亲和素标记的辣根过氧化物酶(avidin-HRP,eBioscience);8600-10氨基磁珠(美国Polysciences,Inc.);牛血清白蛋白(BSA,美国Amresco公司);氯金酸(美国Strem Chemical 公司);实验用水均为Milli-Q水(电阻率18.2 MΩ·cm,Millipore纯化系统,美国密理博公司);涉及的DNA序列均由生工生物工程(上海)股份有限公司合成并纯化,纯度为HPLC级,其中DNA1(Aptamer1)为 5'SH-AAAAAAAAAA-CCCATCAGGGGGCTAGGCTAACACGGTTCGGC-OH-3;DNA2(Aptamer2)为5’-TCTCTGAGCCCGGGTTATTTCAGGGGGA -NH2-3’。
恒温振荡金属浴(HCM100-Pro,大龙兴创实验仪器(北京)有限公司);Synergy2 SLFPTAD 多功能酶标仪(美国伯腾仪器有限公司);UV-2450紫外可见光光度计(日本岛津公司);凝胶成像分析系统(美国伯乐BIO-RAD Gel Doc XR);840-030-602-R原子力显微镜(布鲁克(北京)科技有限公司);miniG手掌型离心机;Lab dancer 振荡器;移液器(Thermomixer comfort Eppendorf,德国);EOS 50D数字照相机(日本佳能)。
1.2 实验方法
1.2.1 纳米金的制备参考经典的Frens方法[27],在圆底烧瓶中倒入49 mL水,加入1 mL 1%氯金酸,加热回流直至沸腾。待溶液沸腾后加入3.5 mL 1%的现配的柠檬酸三钠,反应20 min后停止加热。制备完成后将胶体金置于4 ℃备用。
1.2.2 纳米生物探针Au-DNA1的制备在1 mL 15 nm胶体金(2.3 nmol/L)中加入20 μL 100 μmol/L 的SH-DNA1充分混合,室温下轻微振荡过夜孵育[28]。次日加入0.1 mol/L的PB溶液室温下轻轻振荡30 min使得终浓度为0.01mol/L,分次加入2 mol/L氯化钠使得终浓度为0.15 mol/L进行老化,室温下过夜。之后用0.01 mol/L PB溶液进行离心洗涤(4 ℃,12 000 r/min,20 min)3次,沉淀重悬在200 μL 1×PBS(含137 mmol/L氯化钠、2.7 mmol/L氯化钾、10 mmol/L磷酸盐缓冲液、2 mmol/L磷酸钾)中4 ℃保存待用。
1.2.3 纳米生物界面DNA的生长建立20 μL的反应体系,分别含有10 μL Au-DNA1,4 μL反应缓冲液,1 μL末端转移酶,2 μL 1 mmol/L dNTPs(Biotin-dUTP∶dATP=1∶3)[25],以及3 μL水。将该混合物充分混匀,37 ℃水浴中孵育1 h后用1×PBS缓冲液洗涤3次,最终重悬在20 μL 1×PBS溶液中备用。
1.2.4 琼脂糖凝胶电泳的表征配制1%琼脂糖凝胶,取扩增前后的样品8 μL加入预染色(含0.01%4S Red Plus 核酸染色)的凝胶中,室温下在1×TAE中以90 V恒定电压运行30 min,并在凝胶成像系统中拍照记录。
1.2.5 原子力显微镜的表征取5 μL稀释的样品滴加至新鲜剥离的云母片,静置5 min,缓慢滴加水数滴,用干净的洗耳球或者氮气吹干待测。大气环境下在智能成像模式中使用SNL系列的扫描探针对样品进行扫描。
1.2.6 MBs的活化磁珠活化过程参照产品说明推荐方法进行:取2 mL 50 mg/mL氨基磁珠溶液加入16 mL PWB缓冲液(Pyridine wash buffer,PWB)洗涤后涡旋振荡充分混合。将离心管放在磁力架上静置1 min,待上清液澄清后弃掉,重复洗涤3次。之后用PWB稀释戊二醛,向磁珠中加入8 mL 5%戊二醛并涡旋混合,室温下振荡3 h,期间不断地涡旋振荡,以防磁珠下沉。磁分离下多次洗涤直至上清液澄清,之后PWB重复洗涤4次,最终磁珠重悬在10 mL的1×PBS中备用。
1.2.7 MBs-DNA2的制备取40 μL醛基化磁珠,磁响应后弃上清,加入8 μL 100 μmol/L的NH2-DNA2和80 μL 1×PBS充分混匀,37 ℃反应过夜孵育。次日,磁分离后加入100 μL 1×PBS洗涤1次。之后100 μL磁珠中加入100 μL 2%BSA室温封闭1 h,磁分离后沉淀重悬在200 μL 1×PBS溶液中备用。
1.2.8 MBs-ENR-AuNPs三明治夹心适配体传感器的构建建立50 μL的反应体系,其分别为10 μL MBs-DNA2,30 μL ENR(空白组是缓冲液)以及10 μL Au-DNA1,充分混匀后在振荡金属浴37 ℃反应1 h。反应结束后,磁分离下收集上清液对比颜色,沉淀物中加入50 μL 1×PBS溶液洗涤1次后备用。
1.2.9 比色测定基于TdTase的MBs-ENR-AuNPs三明治夹心结构表面的DNA生长后,加入50 μL 2%BSA和50 μL 0.25%Casein溶液联合封闭30 min。之后磁分离去上清液,加入稀释1 000倍的avidin-HRP溶液,室温孵育30 min。反应结束后加入100 μL 1×PBS(0.2%Tween-20)充分洗涤6次,弃上清液后加入50 μL TMB(含H2O2)避光反应5~10 min。磁响应后,收集上清显色溶液于96孔板中使用酶标仪(波长600 nm)测定吸收值。
2 结果与讨论
2.1 实验原理
纳米界面DNA生长过程如图1所示:以15 nm的纳米金作为载体,5'端修饰巯基的DNA1链通过金硫共价键组装在纳米金的表面,剩余活性位点用BSA进行封闭。加入Biotin-dUTP和dAPT的混合溶液[25],在TdTase的催化作用下,DNA1链的3′-OH末端进行延伸生长,得到一段嵌入生物素位点的ssDNA产物。

图1 基于TdTase的纳米界面上的DNA生长示意图
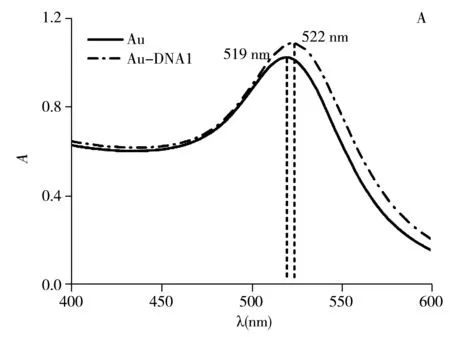
2.2 AuNPs-DNA1与MBs-DNA2的紫外可见光谱
为考察纳米金和磁珠表面组装DNA后表面结构发生的变化,实验对AuNPs以及AuNPs-DNA1进行紫外可见吸收光谱的扫描。结果显示,裸金在519 nm处有明显吸收峰,当纳米金和SH-DNA1通过金硫键共价结合后,其最大紫外吸收峰发生红移,在约522 nm处出现明显的吸收峰(图2A),表明纳米金和DNA序列成功组装。同时,收集磁珠和DNA2组装后的上清液(即溶液中未与磁珠组装的游离NH2-DNA2),测试其在波长260 nm处的吸收峰值为0.53;对比加入的总DNA2量(吸收值为0.70),紫外吸收值降低,即DNA2浓度降低(如图2B)。DNA2吸收值的变化说明醛基化的磁珠和NH2-DNA2孵育后,一部分的DNA2成功组装在磁珠表面,从而使得体系中游离NH2-DNA2的数量减少。
2.3 纳米界面DNA生长产物的表征
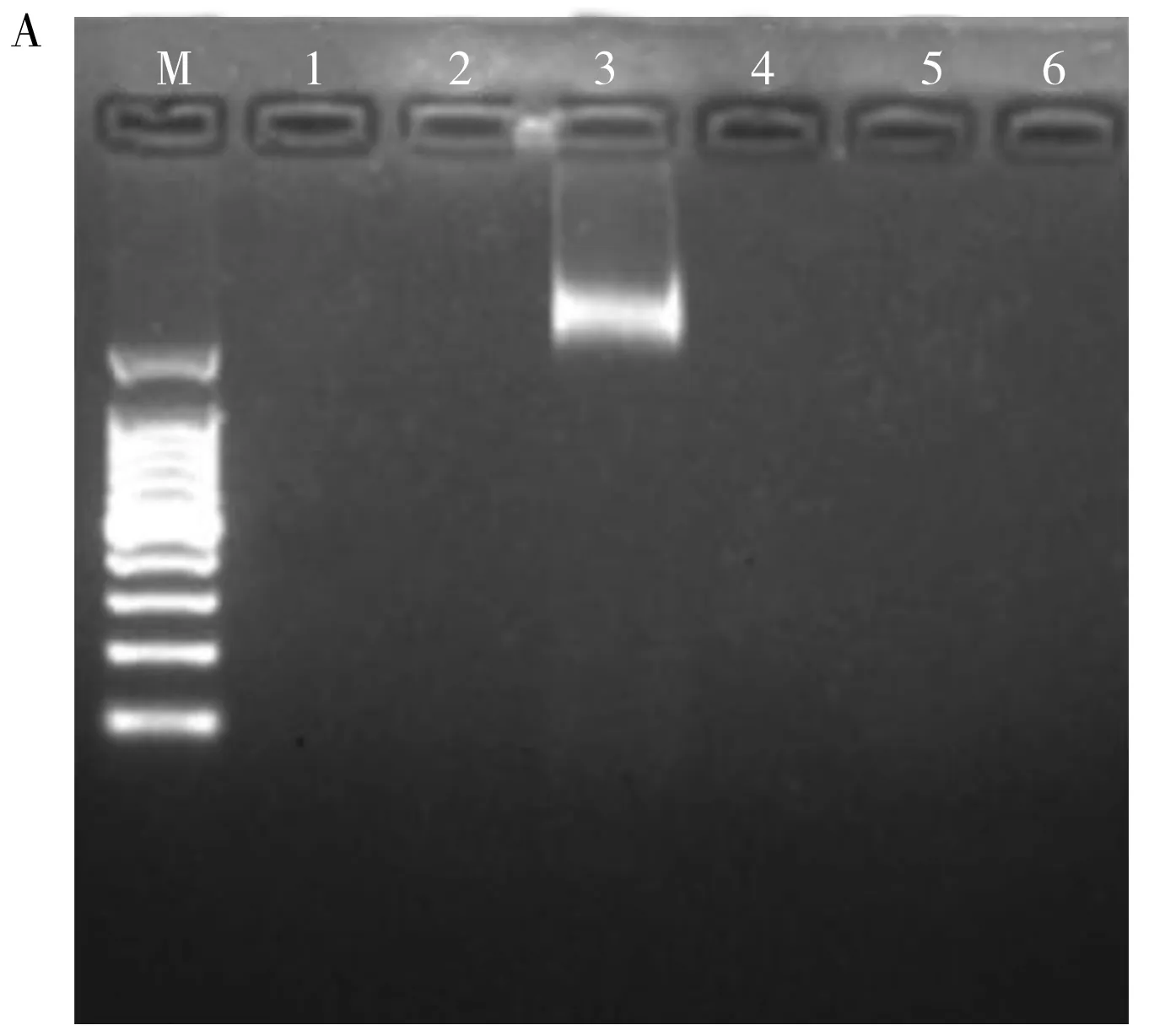
采用琼脂糖凝胶电泳成像和AFM成像技术表征了DNA在纳米界面的生长情况。图3A和B分别为琼脂糖凝胶在紫外光和白光下的成像结果。其中M为DNA marker,泳道1的上样样品为AuNPs-DNA1;泳道2的样品为AuNPs-DNA1中加入dATP,以及TdTase进行生长后的产物;泳道3的样品为AuNPs-DNA1中加入dNTPs,以及TdTase进行生长后的产物;泳道4为MBs-DNA2;泳道5的样品为MBs-DNA2中加入dATP,以及TdTase进行生长后的产物;泳道6的样品为MBs-DNA2中加入dNTPs,以及TdTase进行生长后的产物。对比图3A、B两图中的1~3三条泳道成像发现,2、3样品位置比样品1更接近进样孔,由于扩增产物分子量变大后发生滞后,说明在TdTase的作用下纳米金表面DNA成功延伸生长。然而在白光下虽能发现泳道2中纳米金的条带(图3B),在紫外成像中的相应位置却无DNA条带(图3A,泳道2),这是因为该样品使用dATP进行DNA生长得到的产物为碱基A组成的长单链DNA(poly A),该链由同种碱基组成,不易形成DNA二级结构,从而DNA染色剂(GelRed)不能有效嵌入,致使不能得到明显的DNA条带。
实验继续观察了泳道4~6的成像结果。结果发现,无论生长时添加dATP,还是dNTPs,泳道中均未发现明显的DNA条带。这是因为MBs-DNA2探针表面无供TdTase作用的3′-OH端的位点,无法进行DNA的延伸生长。此结果不但表明了本研究的可行性,同时也避免了检测体系中由于MBs表面可能的DNA延伸生长所带来的背景信号。进一步采用AFM成像技术提供了更为直观的表征结果。图4A为 AuNPs-DNA1的AFM成像结果,可观察到纳米金颗粒的粒径较为均一、分散性良好;然而其表面组装的单链DNA1由于链长较短,无法形成明显成像。经过TdTase介导的DNA生长过程之后,纳米金颗粒周围延伸出来多条ssDNA(图4B)。经过测量,在单链本身的卷曲状态下,产物ssDNA的长度已达850 nm,显示TdTase在室温下即已具备高效扩增性能。
2.4 基于TdTase的适配体传感器用于恩诺沙星的检测
2.4.1 检测原理将TdTase-aptasensor应用于恩诺沙星标准样的检测,其检测过程如图5所示。MBs-ENR-AuNPs三明治复合结构表面经过TdTase介导的DNA生长过程之后,avidin-HRP可与扩增产物ssDNA链中嵌入的生物素位点高特异性识别。最后,加入底物TMB,HRP催化H2O2介导的TMB发生氧化反应,产生肉眼可见的从无色到蓝色的颜色变化。
2.4.2 MBs-ENR-AuNPs三明治复合结构的验证将恩诺沙星溶液(1 mg/mL)稀释成0.25、0.1、0.05、0.025、0.01 mg/mL系列质量浓度溶液,并将制备的AuNPs-DNA1与MBs-DNA2两种探针混合溶液依次加入上述系列溶液中,混匀并振荡孵育后,利用磁分离技术收集上清液。结果显示,随着ENR浓度的递减,上清液的颜色逐步加深。这表明当ENR浓度减少时,无足够的ENR用于形成MBs-ENR-AuNPs三明治复合结构,在磁分离后,上清液中滞留更多的AuNPs-DNA1探针,溶液呈现的红色加深。此结果也表明所使用的DNA1与DNA2两条适配体片段对ENR分子具有良好的识别性能。

图5 基于TdTase的适配体传感器用于恩诺沙星检测的示意图Fig.5 Schematic diagram of TdTase-aptasensor for ENR detection
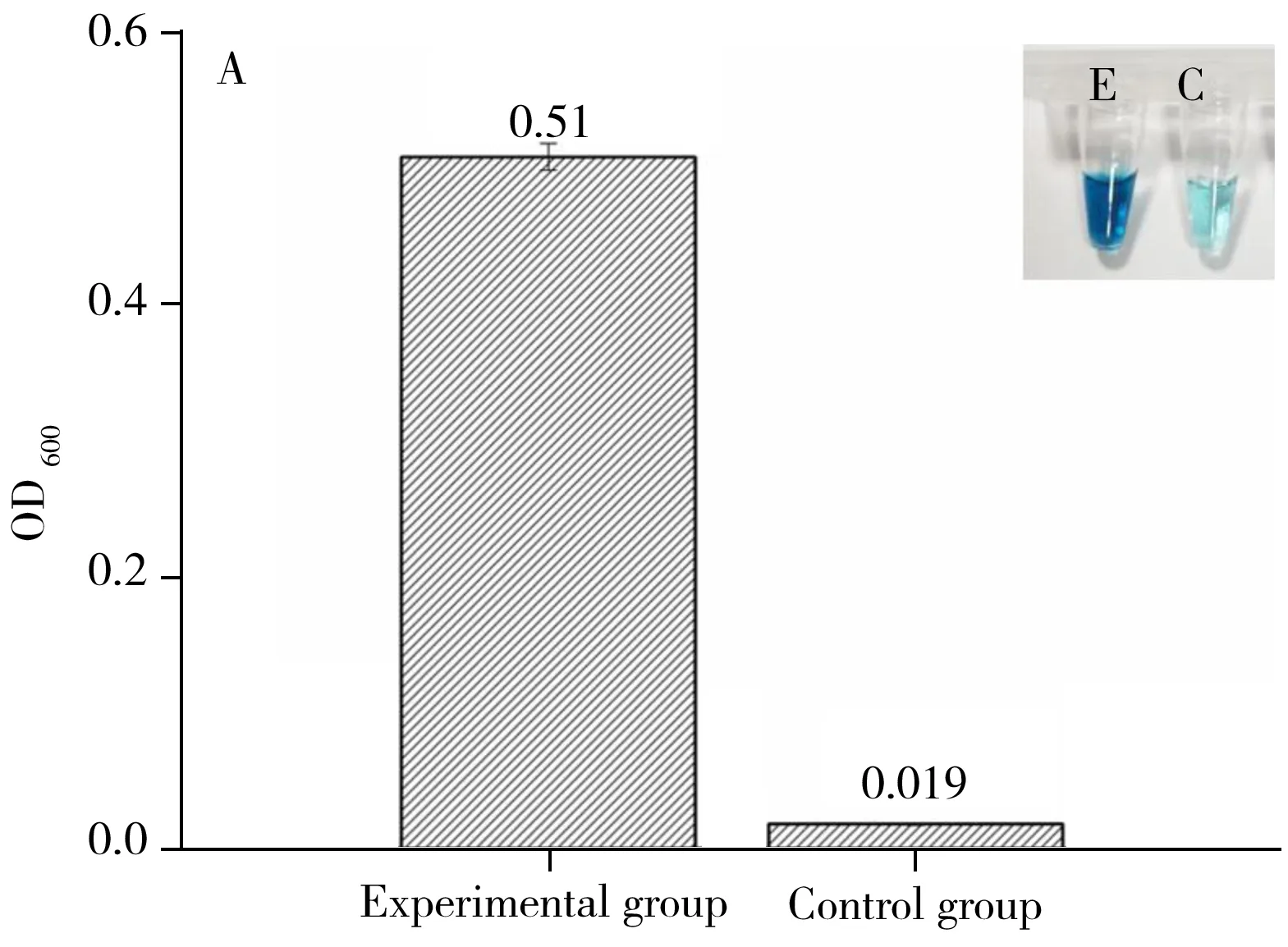
2.4.3 纳米界面的DNA生长策略用于ENR适配体传感器的可行性验证将同等体积的0.1 mg/mL ENR(实验组)与1×PBS(背景组)分别加入基于TdTase的适配体传感检测体系中,按照实验方法中所述的靶标识别、界面生长、比色测定等一系列步骤后,肉眼观察并用酶标仪测定两组吸收值结果。结果如图6A所示,实验组催化TMB后上清颜色明显深于背景组(插入图);且测定的实验组的吸收值为0.51,背景组吸收值为0.019。数据表明基于TdTase的纳米界面DNA生长策略能很好地适用于ENR的适配体传感器的体系构建,通过DNA的生长、生物素位点的嵌入、酶催化信号的测定,可实现对体系中ENR的检测;并且该体系背景信号较低,实验组信号明显优于背景组。
DNA生长后产物ssDNA的链长决定了产物链中嵌入的生物素位点的数目,从而影响到体系的检测性能。因此,该实验中通过采用琼脂糖凝胶电泳,考察了不同生长时间所得产物ssDNA的分子量,以优化实验中DNA的生长时间。结果如图6B所示,泳道1上样样品为AuNPs-DNA1,泳道2~4样品分别是生长时间为30 min、1 h、12 h的扩增产物。通过比较可以看出,与泳道1的AuNPs-DNA1相比,泳道2~4的分子量明显增大,说明DNA成功延伸生长;进一步对比2~4三条泳道中产物的位置,发现泳道3,4的产物条带比产物2更为弥散,说明生成了更多较大分子量的产物,然而产物3和4无明显差别。这可能是由于实验使用了较高活性的TdTase(20 U/μL),其在较短时间内即可实现DNA的高效延伸。因此选定1 h作为DNA的最优生长时间,并用于后续ENR的检测。
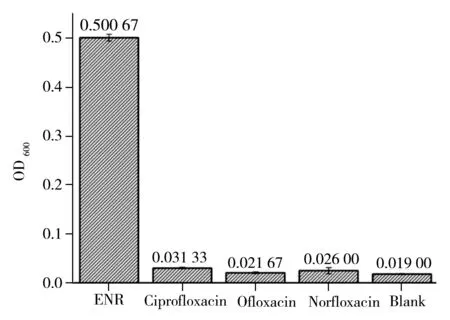
图7 适配体传感器对0.1 mg/mL ENR的特异性检测Fig.7 Specificity of the aptamer sensors for 0.1 mg/mL ENR the other antibiotic concentrations:0.5 mg/mL
2.4.4 基于TdTase的适配体传感器对ENR的响应性能分析在上述优化实验条件下,本研究测试了基于TdTase的适配体传感器对不同浓度ENR的响应能力以及特异性。在适配体传感器中,加入不同浓度ENR溶液。发现随着ENR浓度的增大,比色测定所得的吸收值越高。这是因为溶液中ENR分子越多,所形成的MBs-ENR-AuNPs三明治夹心复合结构数量越多,纳米界面TdTase介导的DNA生长得到的ssDNA和生物素位点就越多,进而和生物素结合的avidin-HRP就越多,而更多的HRP催化TMB变色后得到的信号值越高,且波长600 nm处的吸收值与10~1.0×105ng/mL范围内的ENR质量浓度呈良好的响应关系;当ENR溶液质量浓度低至1.0 ng/mL时,发现其检测信号与空白组信号相比,仍有较好的区分。表明该适配体传感器不但对不同浓度的ENR具有很好的响应能力,同时也具备检测较低浓度靶分子的良好能力。
另外,实验也测试了该传感器的检测特异性,即在适配体传感器中加入0.1 mg/mL ENR和加入0.5 mg/mL喹诺酮类其他抗生素(环丙沙星、氧氟沙星、诺氟沙星)及缓冲液进行对比。如图7所示,溶液中的ENR信号值显著高于其它对照组(环丙沙星、氧氟沙星和诺氟沙星),表明本研究所构建的传感器对ENR具备较好的检测特异性。
3 结 论
本文构建了一种基于TdTase催化的纳米界面上DNA生长策略的适配体比色传感器。该方法在操作上无需贵重仪器,无需复杂的样品前处理和严格的变温操作。在初步应用于恩诺沙星标准样的检测过程中,发现基于Aptamer的MBs-ENR-AuNPs三明治夹心复合结构使得该传感器具备良好检测特异性;此外,由于DNA在纳米界面的高效生长,体系中引入更多的HRP分子,使得该传感器对不同浓度的ENR具备良好的响应性能。这为课题组后续将其应用于实际样品中药物残留的检测奠定了良好基础。本研究发展的基于TdTase的适配体传感器在食品安全检测、临床诊断、环境监测等方面均具备良好的应用前景。

