基于SSR分子标记的桉树父本鉴定技术研究
王建忠,兰俊,陆珍先,杨珺,吕华丽,潘志新,费晓云
基于SSR分子标记的桉树父本鉴定技术研究
王建忠1,兰俊1*,陆珍先1,杨珺2,吕华丽1,潘志新1,费晓云1
(1.广西国有东门林场,广西 扶绥 532108;2.北京林业大学生物科学与技术学院,北京 100083)
利用筛选出的11对SSR引物进行SSR分子标记分析,通过比对子代与母本相同引物对应标记位点的等位基因配置,鉴别自由授粉过程中可能由母本自交产生的个体,并借助Cervus软件应用最大似然值法和遗传排除法,从154个基因型候选父本中鉴定出D2、D3、D4、D7、D11、D19和D20等7个半同胞子代的遗传父本分别为同一试验林内定植的尾巨桉DH18-04、DH18-20、DH32-29和DH241-2。研究对于今后进一步深入探究桉树自交不亲和与近交衰退机制,准确进行子代父本鉴定和杂交亲本重建,以及品种选育等工作具有重要指导意义。
桉树;父本鉴定;SSR
20世纪80年代,中澳技术合作东门桉树示范林项目引进了大量桉树基因资源,建立了亚洲最大的桉树基因库,并对遗传材料进行了卓有成效的改良,特别是在桉树人工杂交育种方面最为显著。通过制订杂交育种策略,培育出了一大批优良桉树杂交种,育种技术处于世界先进水平[1]。杂交育种过程中,如果缺乏相应繁育亲本间的杂交系谱信息或者遗传数据,就会增加近亲交配的机率,导致后代遗传相似度增大,致使遗传基础狭窄。因此,在进行桉树人工杂交育种之前,首先应该明确亲本遗传信息,为亲本选择提供理论基础。
通常情况下,亲本分析主要采用观察记录和遗传标记分析两种方法开展[2]。由于育种工作中,往往可以通过观察记录种子的来源从而直接获得母本的信息,因此亲本分析以父本鉴定为主。对于木本植物而言,通过对每个子代及其候选父本进行遗传标记分析,采用遗传排除法或最大似然值法,可鉴定出候选父本。前者是基于孟德尔遗传定律,即子代的等位基因一定来自杂交亲本,根据多位点遗传数据直接排除与后代具有不同位点的亲本,从而逐次在候选父本中鉴定得到遗传父本[3]。后者则最早由MEAGHER[4-5]提出,通过估计亲本的似然性模型来预测最可能亲本来源,可用于分析已知母本,或亲本均不明确的子代对应的遗传亲本,是父本鉴定中最常用的方法。
SSR分子标记为共显性标记且遵从孟德尔遗传定律,利用Cervus软件中已知性别群体鉴定模型[6-7],并结合遗传排除法比对子代基因型与母本的对应标记点的等位基因配置[8],可以对自交或杂交子代进行判定[9],并在大量候选父本中鉴定出杂交子代的遗传父本[10-11]。CHAIX等[12]曾利用SSR分子标记对巨桉()种子园进行亲本分析。PATTERSON等[13]则进一步通过鉴定1 954个蓝桉()子代的遗传父本,对种子园花粉授粉方式进行研究。GRATTAPAGLIA[14]采用SSR分子标记,对桉树种子园中经自由授粉产生的优良子代进行父本鉴定,通过计算亲本特殊配合力来评估种子园中亲本植株的优越性,进而通过剔除对子代优良性状贡献率低的亲本实现对种子园的改建,从而建立一种向前选择的策略来提高选育优良品种的效率[10]。
本研究通过筛选得到的高多态性SSR引物,结合SSR分子标记分析和遗传排除法,对前期诱导获得的尾巨桉()半同胞材料的亲本来源进行鉴定,从中鉴别出自交和杂交个体,并进一步对杂交个体的父本来源进行鉴定,从而建立一套桉树子代父本鉴定及杂交亲本重建的技术方法,为进一步开展桉树遗传改良工作奠定技术基础。
1 材料与方法
1.1 材料来源
研究材料来源于2015年收集的尾巨桉优树经自然授粉结实得到的种子,经播种育苗得到的半同胞子代。随机选择其中20个基因型作为半同胞子代父本鉴定的实验材料。
上述半同胞子代来源于共同的母本M(尾巨桉无性系DH18-19),其定植于广西国有东门林场基因库试验林。该试验林建于1999年5月,共收集桉树无性系(家系)200余个,包含东门杂交无性系/家系(DH系列编号)、尾叶桉()家系(U系列编号)、佛罗里达杂交桉家系(F系列编号)、巴西杂交桉家系(E系列编号)等,各无性系(家系)单行5株,株行距为2 m × 3 m(图1)。因来源于自然授粉结实,所以这些子代基因型的父本未知,候选父本可能来源于母本M定植的试验林内部,也可能来源于试验林周边行道树或农户种植的杂交桉无性系商品林。

图1 广西国有东门林场基因库采样示意图
注:M为母本(尾巨桉无性系DH18-19),空位为因无性系植株死亡导致的缺失
1.2 实验材料采集、DNA提取与SSR分析
上述半同胞子代,以及包含有它们共同母本M在内的基因库试验林内的所有无性系(家系)均是用于父本鉴定研究材料的采集对象。该试验林保护行所用品种尾巨桉DH32-29,试验林周边商品林造林品种尾巨桉DH33-9、DH32-26、DH32-28等,以及周边道路行所用品种雷林1号(No.1)等候选父本均是材料样本的采集对象。除子代和母本M以外,共采集到154个基因型的候选父本。采集时,选择幼嫩新叶作为样本材料,每个基因型的叶片样本经编号后置入液氮内速冻后放入足量的干冰中保存、运输,并尽快转入-80℃超低温冰箱进行长期保存。
叶片样本在液氮内低温研磨,使用植物基因组DNA提取试剂盒(天根生化科技(北京)有限公司,中国)进行总DNA提取,并经琼脂糖凝胶电泳检验提取质量。共使用3种引物,具体包括5’端包含有M13序列(5’TGTAAAACGACGGCCAGT3’)的上游引物,普通下游引物,以及标有FAM(6-carboxyf-luorescein)、HEX(hexachloro-6-carboxyfluorescein)、ROX(6-carboxy-x-rhodamine)和TAMRA(tetrachloro-6-carboxyrhodamine)荧光标记的M13引物(Schuelke,2000)。PCR总反应体系为20 μL(2 μL DNA模板,10 μL PCR Master Mixes(北京擎科新业生物技术有限公司,中国),7.2 μL ddH2O,0.08 μL正向引物,0.32 μL反向引物,0.4 μL荧光引物),程序设计为:94℃持续4 min;94℃持续30 s,退火温度设置为56℃持续30 s,72℃持续30 s,共计25次循环;94℃持续30 s,退火温度设置为53℃持续30 s,72℃持续30 s,共计25次循环;72℃持续10 min;4℃避光保存。使用ABI-3730XL基因分析仪(Appli-ed Biosystems公司,美国)对PCR产物进行测定,测定结果使用GeneMarker(version 2.2.0)软件(Soft-Genetics,USA)进行读取和分析[15]。
1.3 多态性引物筛选
以已有文献报道的240对桉树SSR引物作为筛选对象[16]。针对上述引物,以采集到的所有桉树基因型DNA为模板依次分别进行TP-M13-SSR PCR,筛选在不同基因型之间有良好多态性的SSR引物,并通过POPGENE (version 1.32)软件(University of Alberta,Canada)计算所有多态性引物的等位基因位点数(NA)、有效等位基因数(NE)、观察杂合度(HO)、期望杂合度(HE)等遗传参数[17]。
1.4 自交个体的排除
桉树雌雄同花,可能存在自交。基于孟德尔遗传定律理论,通过比对子代与母本的等位基因配置,若在子代的等位基因配置中存在至少一个在母本的相应标记位点上未被发现的等位基因,则表明这一子代特有等位基因可能来源于其他亲本,即可初步判定这一子代基因型并非来源于自交。
1.5 桉树半同胞子代父本鉴定
本研究采用SSR多态性引物对已知母本的尾巨桉半同胞家系子代进行父本鉴定。使用Cervus(version 3.0.7)软件(Field Genetics,UK),利用已知性别的群体鉴定模型进行分析[6-7],对半同胞家系中杂交子代的父本进行分析,通过似然比值(LOD)来筛选最似父本。其中,LOD值表示与随机候选父本相比,疑似父本传递给子代基因的可能性的大小。LOD值越大,表明鉴定结果的准确性越高。当LOD值大于0时,表明所鉴定出的亲本为遗传亲本的可能性大于假设亲本。若鉴定过程中出现相同LOD值的候选父本时,则进一步采用△-统计对候选父本LOD值之间的差距进行分析,通过在85% ~ 95%的置信区间内运行10 000次模拟来确定△-临界值[6]。同时,使用Cervus软件计算亲本排除率EP1和EP2,其中EP1表示在双亲未知情况下,在候选父本群体中随机取样,被排除为疑似父本的概率,EP2表示在母本已知情况下,在候选父本群体中随机取样,随机父本被排除为疑似亲本的概率[3,18-19]。
2 结果与分析
2.1 尾巨桉半同胞子代中自交个体的排除
经过对已有文献报道的240对SSR引物的初步筛选[16],其中具有良好多态性的96对SSR引物被用来鉴定尾巨桉半同胞子代是否源于自交。结果显示(图2),在7个半同胞子代D2、D3、D4、D7、D11、D19和D20中存在至少一个在母本M中的相应标记位点上未发现的等位基因,表明这些子代特有等位基因来源于母本以外的其他亲本,即这些子代基因型并非来源于母本M的自交。

图2 尾巨桉半同胞子代与母本SSR等位基因配置示意图(部分示意)
2.2 尾巨桉半同胞子代父本鉴定引物的筛选
以半同胞子代、试验林及周边采集到的共163个基因型DNA样本为模板,筛选得到表1中的11对具有较高多态性的引物:EUCeSSR0037、EUCeSSR0166、EUCeSSR0176、EUCeSSR0224、EUCeSSR0465、EUCeSSR0539、EUCeSSR0592、EUCeSSR0606、EUCeSSR0703、EUCeSSR1001、EUCeSSR1063。上述引物被用于7个半同胞子代的父本鉴定,并通过POPGENE软件对每个多态性位点的等位基因数(NA)、有效等位基因数(NE)、观察杂合度(HO)、预期杂合度(HE)和多态性信息含量(PIC)进行估算,结果显示:在163个基因型中每个位点检测到3至16个等位基因;观察和预期杂合度(HO和HE)分别为0.119至0.748(平均值为0.527)和0.311至0.814(平均值为0.618);多态性信息含量为0.281至0.784(平均值为0.572)。通过Cervus软件计算双亲未知情况下在候选父本群体中随机取样的排除率EP1,和已知母本情况下在候选父本群体中随机取样的排除率EP2,结果显示11对引物对父本的排除率EP2大于99.8%,表明可以有效排除候选父本中的非遗传父本。

表1 广西国有东门林场基因库内无性系群体SSR位点遗传变异参数
注:HO为观察杂合度,HE为预期杂合度;PIC为多态性信息含量,EP1为在候选父本群体中随机取样,被排除为疑似父本的概率;EP2为当母本已知时,在候选父本群体中随机取样,被排除为疑似父本的概率。
2.3 尾巨桉半同胞子代父本鉴定
筛选得到的11对SSR引物被用于7个半同胞子代的父本鉴定,经过Cervus软件计算,结果显示(表2):在母本已知的情况下进行父本鉴定,7个基因型均未出现与母本对应SSR位点上相异的等位基因;7个半同胞子代的LOD值均为正值,介于1.34至5.09之间;7个基因型均未与最似父本出现位点错配情况,因而鉴定得到唯一父本。
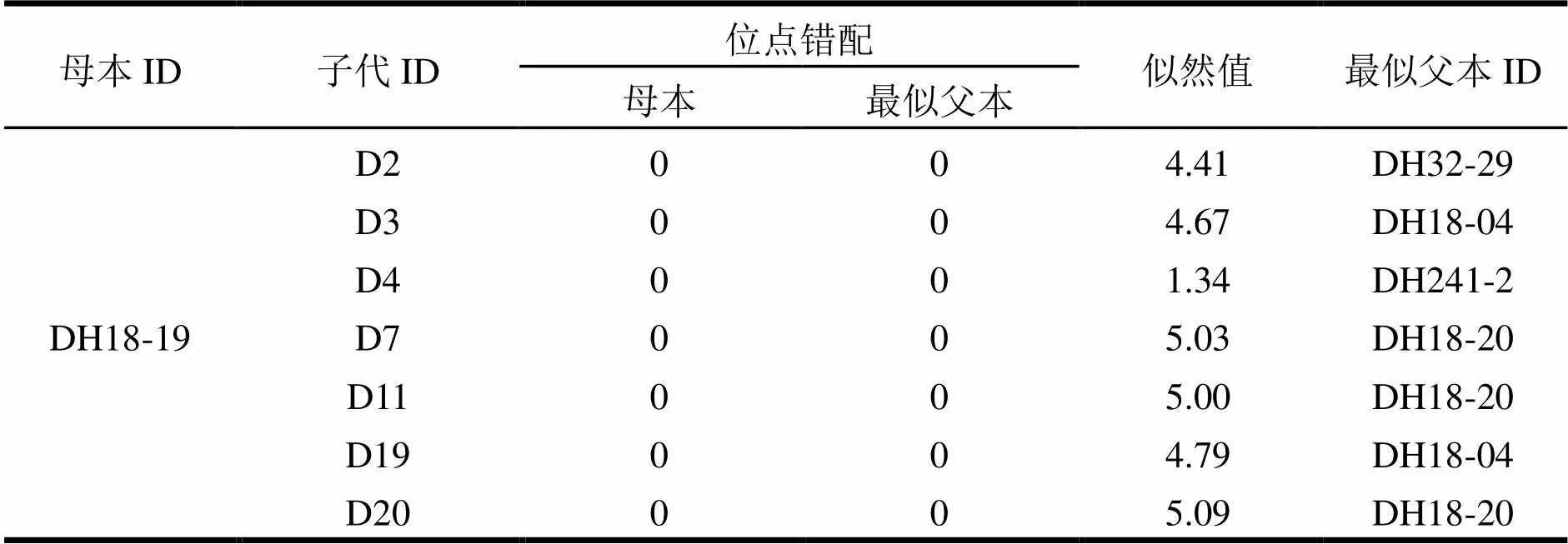
表2 Cervus软件父本鉴定分析表
3 讨论
由生殖生物学特性所决定,桉树具有较高的可能性发生自交[20-22]。尽管不同种桉树均有不同步的花期[22]、特异性的花丝或柱头长度[23]、胚珠或合子早期败育[24]等自交不亲和机制来减少自交的发生,但桉树在自然授粉时仍有较高比率的自交后代产生[25-26]。近年来,由于不需要繁琐的去雄工作,桉树SVP(Single Visit Pollination)[27]、OSP(One Stop Pollination)[28]等“一步授粉”技术被广泛应用于杂交育种工作,该类方法虽然在蒴盖开裂前提前授粉,但仍有接近5%自交个体的产生[13,29]。由于它们当中的一些个体在8个月后才表现出胸径生长的劣势[30],因此在育种工作中,自交个体对后续的苗期评价和选择工作产生着负面影响。SSR分子标记为共显性标记且遵从孟德尔遗传定律。基于子代的等位基因一定来自其双亲的定律,通过比对子代基因型与母本的对应标记位点的等位基因配置,可以对自交或异交子代进行判定,准确率可达97% ~ 99%[9-10]。本研究应用此方法,在20个半同胞基因型中,排除了自交个体,鉴定得到7个半同胞杂交子代。
在桉树中,已被确认并定名的种、亚种或变种超过900个[31],并且亚属内各种之间可以自由杂交[32]。在开展桉树遗传改良工作时,通过明确现有优良个体的亲本来源并实现杂交亲本重建,可有效提升杂交育种工作效率。SSR分子标记可以有效地分析子代个体某个位点的基因组成,进而快速的检测其亲本来源,并在桉树、杨树()等上得到应用[10-11,33]。本研究应用此方法,并结合采用位点逐一排除的遗传排除法[8],鉴定出同一母本M的7个半同胞子代分别来源于4个遗传父本。其中构成半同胞子代D7、D11和D20的雄配子来源于定植于母本M附近的同一遗传父本DH18-20,这是因为桉树作为典型虫媒传粉植物且蜜源丰富,在实验中观察到该母本在散粉季节吸引了大量蚂蚁吸食花蜜,并往返于同一或相邻植株的不同花枝和蚁穴之间,这可能是在鉴定异交子代时发现了较多自交个体的原因;其余4个基因型的遗传父本则与母本相距超过60 m,这是由于借助蜜蜂等飞行昆虫,桉树花粉传播距离最远可达数十公里[34-36]。
本研究利用筛选出的11对SSR引物进行SSR分子标记分析,通过比对子代与母本相同引物对应标记位点的等位基因配置,可用来鉴别自由授粉过程中可能由母本自交产生的个体;借助Cervus软件应用最大似然值法可以对尾巨桉半同胞子代的遗传父本进行鉴定,此基础上结合遗传排除法,鉴定出7个半同胞子代的遗传父本分别为同一试验林内定植的尾巨桉DH18-04、DH18-20、DH32-29和DH241-2。上述研究对于未来进一步深入探究桉树自交不亲和和近交衰退机制,准确进行子代父本鉴定和杂交亲本重建,以及开展品种选育等工作具有重要指导意义。
[1] RICHARD P,李海佳,李宏伟,等.广西桉树人工杂交育种研究[J].广西林业科学,2006,35(4):238-242.
[2] 陈小勇.自然植物种群的亲本分析及其在生态学研究中的应用[J].生态学杂志,1999,18(2):30-35.
[3] POLLEGIONI P, WOESTE K, MUGNOZZA G S, et al. Retrospective identification of hybridogenic walnut plants by SSR fingerprinting and parentage analysis[J]. Molecular Breeding, 2009, 24(4): 321-335.
[4] MEAGHER T R. Analysis of paternity within a natural population of. 1. Identification of most-likely male parents[J].The American Naturalist, 1986, 128(2):199-215.
[5] MEAGHER T R, THOMPSON E. The relationship between single parent and parent pair genetic likelihoods in genealogy reconstruction[J].Theoretical Population Biology,1986, 29(1):87-106.
[6] MARSHALL T C, SLATE J, KRUUK L, et al. Statistical confidence for likelihood-based paternity inference in natural populations[J]. Molecular Ecology, 1998, 7(5): 639-655.
[7] JONES A G, SMALL C M, PACZOLT K A, et al. A practical guide to methods of parentage analysis[J]. Molecular Ecology Resources, 2010, 10(1): 6-30.
[8] ROBLEDO-A J J, GIL L. Patterns of pollen dispersal in a small population ofL. revealed by total- exclusion paternity analysis[J]. Heredity, 2005, 94(1): 13-22.
[9] REIZ C, BRETO M P, ASINS M J. A quick methodology to identify sexual seedlings in citrus breeding programs using SSR marker[J]. Euphytica, 2000, 112(1): 89-94.
[10] GRATTAPAGLIA D, RIBEIRO V J, REZENDE G D S P. Retrospective selection of elite parent trees using paternity testing with microsatellite markers: an alternative short term breeding tactic for[J]. Theoretical Applied Genetics, 2004, 109(1): 192-199.
[11] HAN Z, GAO P, GENG X, et al. Identification of the male parent of superior half-sibindividuals based on SSR markers[J].Molecular Breeding,2017, 37(12):155-166.
[12] CHAIX G, GERBER S, RAZAFIMAHARO V, et al. Gene flow estimation with microsatellites in a Malagasy seed orchard of[J]. Theoretical Applied Genetics, 2003, 107(4): 705-712.
[13] PATTERSOM B, GORE P, POTTS B M, et al. Advances in pollination techniques for large-scale seed production in[J]. Australian Journal of Botany, 2004, 52(6): 781-788.
[14] GRATTAPAGLIA D. Molecular breeding ofMolecular biology of woody plants[M].Netherlands: Springer, 2000.
[15] HULCE D, LI X, SNYDER-L T.GeneMarker® genotyping software: Tools to increase the statistical power of DNA fragment analysis[J]. Journal of Biomolecular Techniques:JBT, 2011, 22(Suppl): 35-36.
[16] ZHOU C, HE X, LI F, et al. Development of 240 novel EST-SSRs inL’Herit[J]. Molecular Breeding, 2014, 33(1): 221-225.
[17] YEH F, YANG R, BOYLE T. POPGENE Version 1.32 Microsoft Windows-based freeware for populations genetic analysis[M]. Edmonton: University of Alberta, 1999.
[18] ISAGI Y, KANAZASHI T, SUZUKI W, et al. Highly variable pollination patterns in Magnolia obovata revealed by microsatellite paternity analysis[J]. International Journal of Plant Sciences, 2004, 165(6): 1047-1053.
[19] VAN E A L, WEABER R L, DRAKE D J, et al. DNA-based paternity analysis and genetic evaluation in a large, commercial cattle ranch setting[J]. Journal of Animal Science, 2007, 85(12): 3159-3169.
[20] GRIFFIN A R, HAND F C.Post-anthesis development of flowers ofF. Muell and the timing of artificial pollination[J].Australian Forest Research,1979,9: 9-15.
[21] ELDRIDGE K G, DAVIDSON J, HARDWOOD C, et al. Eucalypt domestication and breeding[M].Oxford: Clarendon Press, 1993.
[22] POTTS B M, GORE P L. Reproductive biology and controlled pollination of–a review[M]. Tasmania: School of Plant Science UTAS, 1995.
[23] LARCOMBE M J, COSTA J, TILYARD P, et al. On the persistence of reproductive barriers in Eucalyptus: The bridging of mechanical barriers to zygote formation by F1 hybrids is counteracted by intrinsic post-zygotic incompatibilities[J]. Annals of Botany, 2016, 118(3): 431-444.
[24] POUND L M, WALLWORK M A B, POTTS B M, et al.. Early ovule development following self- and cross-pollinations inLabill. ssp. globules[J]. Annals of Botany, 2002, 89(5): 613-620.
[25] HARDNER C M, POTTS B M. Postdispersal selection following mixed mating in[J]. Evolution, 1997, 51(1): 103-111.
[26] COSTA E S J, HARDNER C, TILYARD P, et al. The effects of age and environment on the expression of inbreeding depression in[J]. Heredity, 2011, 107(1): 50-60.
[27] WILLIAMS D R, POTTS B M, BLACK P G. Testing single visit pollination procedures forand[J]. Australian Forestry, 1999, 62(4): 346-352.
[28] HARBARD J L, GRIFFIN A R, ESPEJO J. Mass controlled pollination of Eucalyptus globulus: a practical reality[J].Canadian Journal Forestry Research,1999, 29(10): 1457-1463.
[29] HORSLEY T N, JOHNSON S D, MYBURG A A. Comparison of different control-pollination techniques for small-flowered eucalypts[J]. New Forests, 2010, 39(1): 75-88.
[30] HARDNER C M, Potts B M. Inbreeding depression and changes in variation after selfing inssp. globules[J]. Silvae Genetica, 1995, 44: 46-54.
[31] DOUGHTY R W. The Eucalyptus. A natural and commercial history of the gum tree[M]. Baltimore: John Hopkins Univ Press, 2000.
[32] WILCOX M D. A catalogue of the eucalypts[M]. Auckland: Groome Poyry, 1997.
[33] DONG C B, SUO Y J, KANG X Y. Assessment of the genetic composition of triploid hybridusing SSR markers with low recombination frequencies[J]. Canadian Journal Forestry Research, 2014, 44(7): 692-699.
[34] BARBOUR R C, POTTS B M, VAILLANCOUNT R E. Gene flow between introduced and native Eucalyptus species: crossability of native Tasmanian species with exotic E. nitens[J]. Australian Journal of Botany, 2005, 53(5) :465-477.
[35] BARBOUR R C, POTTS B M, Vaillancourt RE. Gene flow between introduced and nativespecies: Early-age selection limits invasive capacity of exotic×F1 hybrids[J]. Forest Ecology and Management, 2006, 228: 206–214.
[36] BARBOUR R C, POTTS B M, VAILLANCOUNT R E. Gene flow between introduced and native Eucalyptus species: Morphological analysis of tri-species and backcross hybrids involving[J]. Silvae Genetica, 2007, 56(3/4) :127-133.
Study on Using SSR Markers for Paternal Identification in
WANG Jianzhong1, LAN Jun1, LU Zhenxian1, YANG Jun2, LV Huali1, PAN Zhixin1, FEI Xiaoyun1
(1.,,,; 2.,)
A technique for male parent identification using SSR markers inhalf-sib progenies was developed in the study reported here. Eleven selected SSR loci with high polymorphism were used to identify the selfed individuals in an open pollinated population, based on the comparison of marker loci with the same primers between progenies and female parents. According to maximum likelihood scores obtained from analyses by Cervus software,clones DH18-04, DH18-20, DH32-29 and DH241-2 were identified as the male parents of the outcrossed progenies D2, D3, D4, D7, D11 and D20, respectively. This research has important significance for guiding further examination of self-incompatibility, paternal identification and hybrid parental reconstruction in these eucalypt taxa.
; paternal identification; SSR
Q943
A
广西创新驱动发展基金项目(桂科AA17204087-3)
王建忠(1985― ),男,硕士,工程师,主要从事桉树遗传改良及无性系开发研究,E-mail: 379760245@qq.com
兰俊(1981― ),男,硕士,高级工程师,主要从事桉树遗传改良及无性系开发研究,E-mail: lanjun107@aliyun.com

