基于MiSeq高通测序技术的米酒真菌多样性分析
周书楠,王玉荣,周亚澳,廖 华,张振东,郭 壮,3,*
(1.湖北文理学院食品科学技术学院,鄂西北传统发酵食品研究所,湖北襄阳 441053;2.恩施市农业局,湖北恩施 445000;3.恩施市公共检验检测中心,湖北恩施 445000)
米酒是以糯米、大米或籼米等谷物为主料,添加酒曲发酵制成的一类风味独特的低酒精度饮料,因其具有清香醇厚、营养丰富和工艺简单等特点,湖北省一直保留着制作和饮用米酒的习惯[1-2]。传统米酒的制作多在开放环境下进行,除酒曲中的根霉和酵母菌会进入成品中外,原料及环境中的多种微生物亦会掺入其中,由此可见,米酒的发酵过程实际上是多种微生物之间以及微生物与原料中的淀粉、蛋白质、脂肪和矿物质等化学成分之间相互作用的过程。传统工艺制作的米酒易受微生物、原料和环境等因素的影响,其成品品质很难保持一致,有时甚至易受致病菌污染[3]。因此,对米酒中微生物种类及多样性进行全面解析显得尤为重要。多年来,国内学者对米酒中酵母菌[4-5]、乳酸菌[6]和霉菌[7-8]进行了多项卓有成效的研究,其研究结果均表明米酒中微生物以霉菌和酵母菌等真菌为主。然而以上研究均采用的是基于纯培养的传统微生物学手段,该方法存在一定局限性,不能全面真实的反映米酒中微生物多样性。
以MiSeq为代表的第二代高通量测序技术具有操作简单、通量高和结果可信度高的特点,一次可对多个样本中的微生物群落信息进行平行分析[9]。相较于富集培养、分离纯化和生理生化鉴定等传统微生物手段,MiSeq高通量测序技术可检测出样品中低丰度、难培养的微生物类群,能够更加真实准确的反映样品中微生物群落结构,并且该技术已在窖泥[10-11]、泡菜[12]、酒曲[13]和白酒[14]等中得到广泛应用,这些研究的开展为研究米酒中微生物多样性提供了很好的思路。
本研究以采集自恩施土家苗族自治州的农家自酿米酒为研究对象,在提取样品宏基因组的基础上,采用MiSeq高通量测序技术对其真菌多样性进行解析,同时采用多元统计学手段对样品间差异性进行分析,以期为米酒中微生物资源发掘提供一定理论依据。
1 材料与方法
1.1 材料与仪器
米酒 于2018年2月采集自恩施市10个农户家中,且发酵时间均在10 d以上,依照采集次序,依次将样品编号为MJ1~MJ10;QIAGEN DNeasymericon Food Kit基因组DNA提取试剂盒 德国QIAGEN公司;5×TransStartTM、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mix、FastPfu Buffer和FastPfu Fly DNA聚合酶(5 U/μL) 北京全式金生物技术有限公司;DNA 1000试剂盒 美国Agilent公司;正向引物SSU0817F(5′-TTAGCATGGAAT AATRRAATAGGA-3′)、反向引物SSU1196R(5′-TCTGGACCTGGTGAGTTTCC-3′) 武汉天一辉远生物科技有限公司。
5810R台式高速冷冻离心机 德国Eppendorf公司;2100芯片生物分析仪 美国Agilent公司;ND-2000C微量紫外分光光度计 美国NanoDrop公司;Vetiri梯度基因扩增仪 美国AB公司;DYY-12电泳仪 北京六一仪器厂;PowerPacTMBasic稳压仪和UVPCDS8000凝胶成像分析系统 美国BIO-RAD公司;MiSeq高通量测序平台 美国Illumina公司;R920机架式服务器 美国DELL公司。
1.2 实验方法
1.2.1 宏基因组DNA提取与检测 取50 g米酒样品置于无菌离心杯中,400×g离心10 min以去除米粒等固体杂质,取上清液于4000 r/min下,离心10 min以收集菌体。参照DNA提取试剂盒中的方法,对收集的菌体进行宏基因组DNA提取,使用1.0%的琼脂糖凝胶电泳,对提取的宏基因组DNA的质量进行检测,同时使用微量紫外分光光度计检测其OD260/280是否在1.8~2.0之间,记录各样品DNA浓度,将符合要求的样品DNA置于-20 ℃的冰箱中备用。
1.2.2 PCR扩增及测序 参照王丹丹等[15]的方法,对样品真菌18S rRNA的V4-V5区进行PCR扩增,体系为20 μL:5×PCR buffer 4 μL,2.5 mmol/L dNTPs Mix 2 μL,5 μmol/L正反向引物(正向引物上加7个核苷酸标签)各0.8 μL,5 U/μL FastPfu Fly DNA聚合酶 0.4 μL,DNA模板10 ng,剩余体积用无菌超纯水补齐;扩增条件参照陈庆金等[16]的方法略作改动:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,30 个循环;72 ℃完全延伸10 min。扩增结束后,用无菌超纯水将1.0%琼脂糖凝胶电泳检测合格的扩增产物浓度稀释至100 nmol/L,并用干冰将其寄至上海美吉生物医药科技有限公司进行MiSeq高通量测序。
1.2.3 序列质量控制及分析 MiSeq测序完成后,根据序列间的重叠关系将返回的正反向成对单链序列拼接成一条完整DNA序列,要求拼接时满足以下任一条件的序列予以剔除:重叠区的碱基数小于10 bp;核苷酸标签碱基发生错配;引物碱基错配数大于2 bp或最大错配比率大于0.2[17]。剩下的序列再按核苷酸标签序列,将其划分至对应的样品中并校正序列方向,最后切除正反引物及核苷酸标签,若切除的引物和核苷酸后序列长度小于50 bp,则该序列亦舍弃。
序列质控合格后采用QIIME(v1.7.0)分析平台对序列进行分析[18],用UCLUST软件[19]首先以100%的相似度得到单一序列,然后以97%的相似度建立操作分类单元,紧接着从每个OTU中挑选一条代表性序列与SILVA[20-21]数据库进行比对,确定各序列和各OTU的微生物学分类地位,进而对米酒真菌的α多样性指数和β多样性指数进行计算。
1.3 数据处理
使用发现物种数和香农指数对真菌丰度和多样性进行分析,采用基于分类操作单元加权UniFrac距离的聚类分析和基于欧氏距离的差异分析,对样品真菌群落结构差异进行研究,采用曼-惠特尼检验(Mann-Whitney test)对不同聚类间的组间差异显著性进行分析。使用软件Matlab R2016b和OriginPro 2017C绘图。
2 结果与分析
2.1 基于门和属水平的真菌多样性分析
采用MiSeq高通量测序技术,从10个米酒样品中共检测出85672条有效序列,本研究首先对测序深度进行分析,结果如图1所示。
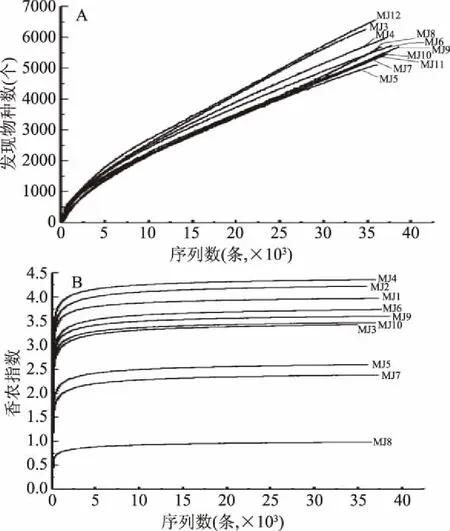
图1 样品真菌序列稀释曲线(A)和香农指数曲线(B)
由图1A可知,稀释曲线随着序列数的增加呈现逐渐上升的趋势,而由图1B可知,香农指数曲线在序列数为10000条左右时就已全部进入稳定期,说明随着测序序列量的增加样品中可能会有新的物种被发现,但其真菌多样性不会再有明显变化,表明本研究测得的85672条序列满足后续生物信息学分析要求。由图1B可知,所有样品中香农指数最大的为MJ4,最小的为MJ8,经α多样性分析发现,在测序深度为34810条序列时二者的香农指数分别为4.36和0.99,说明样品MJ4中真菌丰富度最高而样品MJ8中最低。本研究检测出的合格序列在97%的相似度下可被划分至10627个OTU中,从每个OTU中挑选一条代表性序列进行比对,然后统计其界、门、纲、目、科和属的种类和数量,其中真菌门水平的分析结果如图2所示。

图2 米酒中优势真菌门相对含量分析
由图2可知,所有米酒样品中主要检测出隶属于子囊菌门(Ascomycota)、毛霉亚门(Mucoromycotina)、担子菌门(Basidiomycota)、壶菌门(Chytridiomycota)和芽枝霉门(Blastocladiomycota)的真菌,其中Ascomycota和Mucoromycotina的累积平均相对含量高达99.20%,而Basidiomycota、Chytridiomycota和Blastocladiomycota仅在一个或少数几个样品中存在。由图2亦可知,MJ1和MJ4中Ascomycota的相对含量明显低于其他样品,而MJ5、MJ7和MJ8中Basidiomycota含量亦明显低于其他样品,说明各样品中真菌门的种类和含量存在差异。进一步在属水平上进行研究时发现10个样品共检测出17个真菌属,其中平均相对含量大于0.1%的有4个,在本研究中将这4个真菌属定义为优势真菌属,则其在各样品中的分布情况如图3所示。
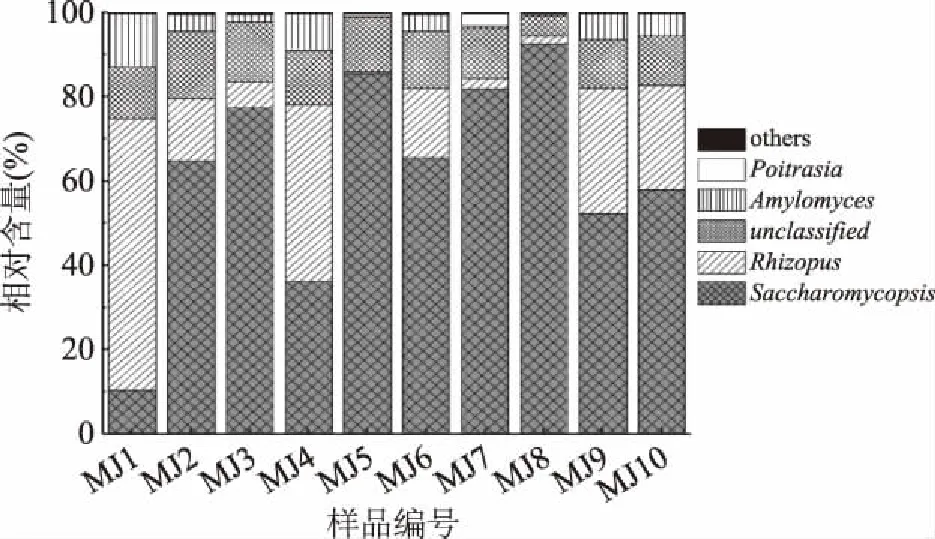
图3 米酒中优势真菌属相对含量分析
由图3可知,米酒样品中的优势真菌属主要为复膜孢酵母属(Saccharomycopsis)、根霉属(Rhizopus)、淀粉菌(Amylomyces)和Poitrasia,其平均相对含量分别为62.44%、20.28%、4.42%和0.47%,所有合格序列仅有12.17%不能鉴定到属水平。值得一提的是,Saccharomycopsis在MJ1中的相对含量仅有10.39%,而在MJ8中的相对含量高达92.54%;Rhizopus在MJ1中的相对含量为64.32%,而在MJ5、MJ7和MJ8中的相对含量分别为0.43%、2.43%和2.02%;样品MJ1和MJ10中均不含有Poitrasia。由此可见,虽然米酒样品中含有大量真菌菌群,但这些优势真菌在各样品中并非均匀分布。焦晶凯[22]采用DGGE技术对不同发酵时期传统酿造米酒中微生物多样性变化进行分析,发现米酒中含有的真菌主要为酵母属(Saccharomyces)、毕赤酵母属(Pichia)和克鲁维酵母属(Kluyveromyces),与本研究结果存在一定差异性;但王丹丹等[15]采用MiSeq高通量测序技术对孝感凤窝酒曲真菌微生物多样进行研究时发现淀粉霉(Amylomyces)、小克银汉霉属(Cunninghamella)、毛霉属(Mucor)、复膜孢酵母属(Saccharomycopsis)、曲霉属(Aspergillus)、念珠菌(Candida)、拟威尔酵母(Cyberlindnera)和接合酵母(Zygosaccharomyces)是其内主要真菌,与本研究结果较为相似,出现这种现象的原因可能是原料来源和实验方法等因素造成的。
2.2 基于OTU水平的真菌多样性分析
本研究从10个米酒样品中得到10627个OTU,各样品中OTU数量及种类并非完全相同,如图4所示为各OTU在所有样品中出现次数统计结果。
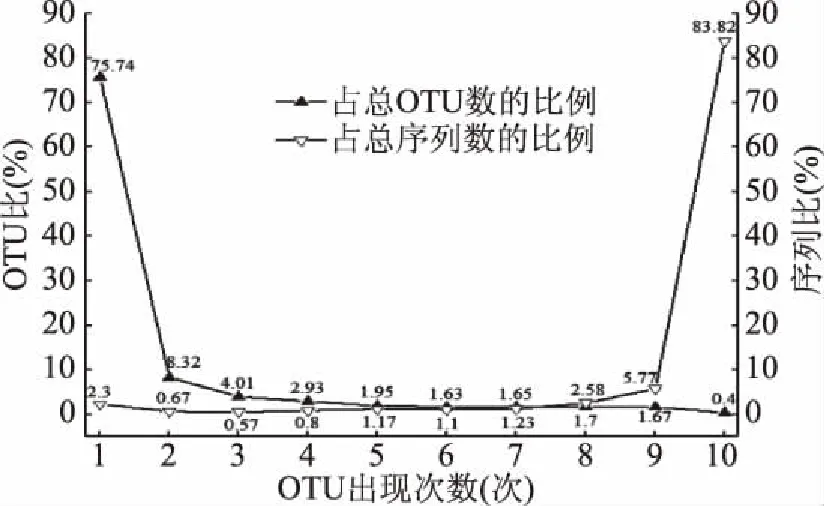
图4 OTU出现次数统计分析
由图4可知,随着OTU出现频次从1到10逐渐升高,其所含OTU数占总OTU数的比例整体上呈现逐渐下降的趋势,而所含序列数占总序列数的比例趋势恰好相反,尤以出现频次为1和10的OTU最为明显。某个样品中特有的OTU即图4所示,仅出现1次的OTU占总OTU数的比例为75.74%,但其所含序列数仅为2.30%;出现10次的OTU即所有样品共有OTU占总OTU数的比例仅为0.40%,而其所含序列数占到总序列数的83.82%;其他频次的OTU即部分样品中共有OTU的OTU比和序列比值总体上较为相近。说明样品中特有真菌种类多但在各样品中的含量少,而多数为共有真菌菌群。值得一提的是,10627个OTU中序列平均相对含量大于0.1%的OTU有32个,其中相对含量大于0.5%有7个,这7个OTU在各米酒样品中的相对含量如图5所示。
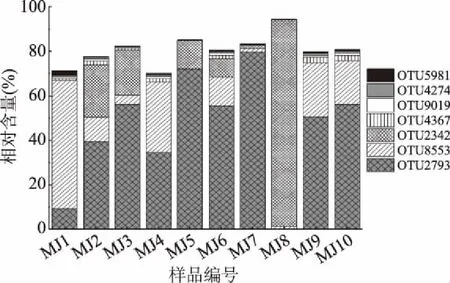
图5 平均相对含量大于0.5% OTU统计分析
由图5可知,平均相对含量大于0.5%的OTU及其平均相对含量分别为OTU2793(45.40%)、OTU8553(16.41%)、OTU2342(15.90%)、OTU4367(1.18%)、OTU9019(0.63%)、OTU4274(0.59%)和OTU5981(0.53%),其中OTU2793、OTU2342和OTU4274隶属于Saccharomycopsis,OTU8553、OTU4367和5981隶属于Rhizopus,OTU9019隶属于Amylomyces,且这些OTU在各样品中均存在。由图5亦可明显看出,MJ8中OTU2342的相对含量已达92.47%,明显高于其他样品,而其OTU2793的相对含量仅为0.02%。由此可见,米酒样品中的核心优势真菌属为Saccharomycopsis、Rhizopus和Amylomyces,且各样品相对含量存在差异。王艳萍等[8]采用分离纯化的方法,对荆州自然发酵米酒中微生物进行分析,发现米酒发酵过程中的主要优势菌为霉菌;焦晶凯[22]的研究亦与本研究结果均有一定的相似性。但目前鲜有针对米酒中Amylomyces进行分离纯化的研究,这些结果为后续微生物资源发掘提供了一定研究靶点。
2.3 样品差异分析
通过计算样品间的距离,UniFrac显著性检验可利用各样品序列类型,来比较样品在特定的进化谱系中是否有显著的微生物群落差异[23]。本研究在对不同米酒样品中真菌进行分析鉴定的基础上,进一步采用基于分类操作单元加权UniFrac距离显著性检验对样品间差异性进行了分析,结果如图6所示。

图6 样品UPGMA聚类分析
由图6可知,采集的10个米酒样品大致可以分为两类,聚类Ⅰ包括MJ1、MJ2、MJ4、MJ6、MJ9和MJ10,聚类Ⅱ包括MJ3、MJ5、MJ7和MJ8。在加权UniFrac中,分支的长度是根据对该分支上两个群落的相对丰度差异进行加权计算得到的,由图6亦可知,聚类Ⅰ中各分支长度整体上要长于聚类Ⅱ各分支,为探究其差异显著性,本研究进一步采用欧氏距离对样品真菌群落结构组间差异进行分析,结果如图7所示。
由图7可知,采用欧氏距离计算不同聚类真菌微生物群落结构差异时,聚类Ⅰ的组内距离为0.082±0.028,聚类Ⅱ的组内距离为0.047±0.014,即聚类I中的欧氏距离大于聚类Ⅱ,说明聚类I中样品相似度低于聚类Ⅱ。经Mann-Whitney检验发现两组数据差异显著(p=0.038<0.05),说明聚类Ⅰ样品真菌群落结构组内差异要显著高于聚类Ⅱ(p<0.05)。本研究进一步在属水平上对造成上述现象的因素进行了探究,结果如表1所示。

表1 不同聚类间优势真菌属差异分析Table 1 Difference analysis of dominant fungi among different clusters

图7 真菌群落结构组间差异分析
由表1可知,聚类Ⅰ和聚类Ⅱ中Saccharomycopsis、Rhizopus、Amylomyces和Poitrasia的相对含量差异均极显著(p<0.01)。整体上看,聚类Ⅰ中Rhizopus和Amylomyces的相对含量要高于聚类Ⅱ,而聚类Ⅱ中Saccharomycopsis和Poitrasia要高于聚类Ⅰ。由此可见,Saccharomycopsis、Rhizopus、Amylomyces和Poitrasia是造成米酒样品两个聚类微生物群落结构差异显著的关键真菌类群。
3 结论
MiSeq高通量测序结果表明,恩施地区米酒中的真菌微生物主要隶属于5个门和17个属,其中核心优势真菌属为Saccharomycopsis、Rhizopus和Amylomyces,且有12.17%的序列未能鉴定到属水平,说明该地区米酒中含有大量的真菌资源且仍有多种真菌未被发掘。在对样品中真菌进行研究时发现,采集自恩施的10个米酒样品的真菌组成及含量均不相同,所有样品大致可以分为两个聚类,采用多元统计学手段分析得知造成这种差异的关键真菌类群主要为Saccharomycopsis、Rhizopus、Amylomyces和Poitrasia,而这些因素亦可能是造成不同传统农家自酿米酒产品品质不稳定的主因,这为后续农家自酿米酒微生物资源的发掘以及后期米酒制作工艺改进提供了一定的参考。

