草鱼自噬相关基因Beclin1的克隆及其在MC-LR胁迫下的表达特征
何 丽 阮记明 刘 毅 付建平 刘 林 隗黎丽
(1. 江西农业大学动物科学技术学院, 南昌 330045; 2. 江西师范大学生命学院, 南昌 330022)
微囊藻毒素(Microcystins, 简称MCs)是一类具有生物活性的单环七肽化合物, 是铜绿微囊藻等蓝藻产生的次生代谢产物[1]。到目前为止, 在已知结构的100多种MCs变体中, 微囊藻毒素-LR (Microcystin-LR, MC-LR, 其中L代表亮氨酸)是毒性最强、危害最严重的一种肝毒素[2]。此外, MC-LR还能够在水生动物的肾脏、心脏、性腺和肌肉等组织器官中积累, 通过食物链的传递最终威胁人类健康[3,4], 已有的研究报道表明其主要的致毒机理是抑制丝∕苏氨酸蛋白磷酸酶l和2A(PP1和PP2A)的活性以及诱导氧化应激[5,6], 从而引起细胞骨架重组、DNA受损及氧化和抗氧化系统平衡失调等, 最终导致细胞凋亡和细胞坏死[7]。尽管已取得了一系列的研究进展, 但仍难以全面诠释MC-LR毒性机制。现已有零星报道表明自噬与MC-LR的肝毒性密切相关, 如Menezes 等[8]发现人(Homo sapiens)的肝脏细胞暴露在低浓度的MC-LR中时, 可见自噬小体的产生; MC-LR也可引起小鼠(Mus musculus)肝细胞产生自噬[9]。然而, MC-LR诱导鱼类自噬的研究相对较少, 其诱导自噬发生的机制也尚不明确。
已有的研究表明自噬是细胞体内多余的蛋白质和亚细胞成分在溶酶体内消化降解的过程, 被认为是一种选择性的非caspase依赖的程序性细胞死亡[10]。大量研究表明, 自噬可由饥饿、内质网应激、低氧、病原体感染和细胞器受损等多种因素诱导[11], 该过程受30多种自噬相关基因的调控。其中Beclin1, 是酵母自噬相关基因Atg6的同源基因,是在1998年由Liang等在致死性Sinbis病毒性脑炎的大鼠(Rattus norvegicus)中发现的[12], 1999年Aita等[13]首次成功克隆了大鼠Beclin1基因。然而,Beclin1在鱼类中的研究却相对较少, 目前只有在斑马鱼(Danio rerio)[14]、牙鲆(Paralichthys olivaceus)[15]、荷包红鲤(Cyprinus carpio)[16]和稀有鲫(Gobiocypris rarus)中[17]有相关报道。
草鱼(Ctenopharygodon idella)作为一种重要的淡水经济鱼类, 是我国养殖规模最大、产量最高的淡水养殖鱼类, 在池塘集约化养殖模式下, 草鱼极易遭受MCs的威胁。在我们先前的研究中, 经转录组测序研究发现, 草鱼受MC-LR胁迫后, 肝脏差异表达基因中包括Beclin1等自噬相关基因[18]。此外,到现在为止还未见Beclin1在草鱼中的克隆及其对MC-LR响应的报道, 因此, 本文在此研究基础上, 通过RACE-PCR技术获得草鱼Beclin1(命名为CiBeclin1)基因的全长cDNA序列, 并对其cDNA和推导的氨基酸进行了生物信息学分析, 同时分析在MCLR胁迫下草鱼肝脏Beclin1基因的表达变化情况,为进一步研究MC-LR的毒性机制奠定基础。
1 材料与方法
1.1 材料与试剂
试验草鱼购于江西省南昌神龙渔业公司, 平均体重为(22.13±2.17) g。试验所用MC-LR为 Taiwan Algal Science Inc 公司产品, 间氨基苯甲酸乙酯甲烷磺酸盐(MS-222)为Sigma公司产品, RNA提取试剂盒TRIzol reagent为Invitrogen公司产品, 用于合成不同组织表达的逆转录试剂盒RevertAidTMFirst Strand cDNA Synthesis Kit 以及荧光定量PCR 的SYBR Green Real-time PCR Master Mix为Promega公司产品, 进行RACE扩增的第一链cDNA用Super SMART®PCR cDNA Synthesis Kit (Clontech)试剂盒合成、DL2000 DNA Marker、pMD18-T 载体和T4 DNA Ligase购自TaKaRa公司。
1.2 试验鱼的处理及样品的分离
试验草鱼买回后先在室内暂养2周, 暂养期间每日按照鱼体重的2.0%进行投喂, 并保持水温在(20 ± 0.2)℃。试验前48h停止投喂并将草鱼进行随机分组, 即对照组和2个剂量处理组, 每组设置3个重复。在试验前, 根据产品说明书将MC-LR (纯度≥95%)粉末用甲醇溶解成1 μg/μL的母液保存备用, 在正式染毒之前用0.8%生理盐水稀释成为25 µg MC-LR/kg体重(25 µg MC-LR/kg body weight, 25 µg MC-LR/kg BW)和100 µg MC-LR/kg BW, 每尾鱼注射量为0.1 mL, 对照组草鱼注射等量的0.8%的生理盐水。在染毒24h、48h、72h和96h后, 分别从实验组和对照组中各取6尾鱼。在解剖前, 先用100 μg/mL的MS-222将鱼麻醉, 然后迅速分离新鲜的肝脏, 置于液氮中保存。
1.3 核酸的提取、单链cDNA及SMART cDNA合成
取出保存在液氮中的样品, 采用Invitrogen公司的Trizol试剂盒提取总RNA, 具体提取方法参考试剂盒的说明书进行。用于RACE 扩增的cDNA按照Super SMART®PCR cDNA Synthesis Kit 操作手册的方法合成, 用于检测不同组织表达以及MC-LR诱导表达的第一链cDNA用RevertAidTMFirst Strand cDNA Synthesis Kit 试剂盒合成。
1.4 CiBeclin1 cDNA全长的获得
根据转录组测序数据获得Beclin1一段序列[18],先设计简并引物验证该EST序列(表1), 引物由上海生工生物技术有限公司合成。PCR扩增条件为95℃预变性3min, 94℃ 30s, 58℃ 30s, 72℃ 90s, 共35个循环, 72℃延伸10min。PCR产物经1.0%琼脂糖凝胶电泳、切胶和回收纯化后克隆到pMD18-T载体(TaKaRa, Tokyo, Japan) , 然后再转化到感受态TOP10, 进行蓝白斑筛选, 菌液PCR检测阳性克隆,并送上海生工生物技术有限公司测序, 获得中间片段。
CiBeclin1 cDNA 3′端片段克隆根据已获得中间片段序列扩增共设计2条特异性引物RC3-1和RC3-2, 并且RC3-1在RC3-2的上游(表1)。扩增时,用RC3-1和3′RACE 接头引物做第一次PCR扩增, 扩增条件为94℃预变性3min; 94℃ 30s, 58℃ 30s,72℃ 90s, 共20个循环; 72℃延伸10min。产物稀释后再用RC3-2和3′RACE 扩增试剂盒中的接头引物做巢式PCR, 随后筛选阳性克隆进行测序。
CiBeclin1 cDNA 5′端片段克隆根据已获得中间片段序列而设计的特异性引物(RC5-1、RC5-2和RC5-3)(表1)。按照SMARTTMcDNA Amplification Kit (Clontech) 说明书推荐的反应体系及反应条件进行5′端扩增, 最后将获得的5′扩增片段回收, 纯化后连入pMD18-T载体, 转化TOP10扩培, 挑选阳性克隆测序获得基因5′端序列。
1.5 CiBeclin1序列分析与系统发育树的构建
利用ExPASy网站(http://www.expasy.org)的Translate和ProtParam程序分别进行氨基酸序列的推导和蛋白理化性质的分析, 采用SignalP4.0(http://www.cbs.dtu.dk/services/SignalP/) 预测蛋白的信号肽; 跨膜区预测用TMpred (https://embnet.vital-it.ch/software /TMPRED_form.html)软件, 使用InterProScan (http://www.ebi.ac.uk/interpro/search/sequence-search)在线分析CiBeclin1蛋白可能存在的结构域。氨基酸序列比对用ClustalW1.81软件分析, 系统发育树则采用Mega7.0软件中的N-J法进行构建。
1.6 荧光定量PCR
取3尾草鱼分离其肝脏、头肾、脾脏、肝脏、肠道、心脏、胸腺、鰓、脑和肌肉组织各50—100 mg以及血液100 µL, 分别提取总RNA, 随后取1 µg RNA逆转录成cDNA作为检测CiBeclin1在不同组织中的表达。另外, 取出液氮中保存的经不同剂量MC-LR处理的对照组和实验组草鱼肝脏组织提取总RNA, 反转录合成cDNA, 用于分析MCLR对草鱼肝脏CiBeclin1表达的影响。使用Quantitative real-time PCR (qRT-PCR)方法检测CiBeclin1的表达量, 并以草鱼β-actin作为内参基因进行校正,CiBeclin1和β-actin引物见表1。qRT-PCR反应在伯乐公司CFX96 TouchTMReal-Time PCR Detection System上完成。20 µL PCR反应体系包括5.0 μL 50倍稀释的cDNA模板, 10 μL SYBR Green Realtime PCR Master Mix, 上下游引物各0.5 μL, 4.0 μL ddH2O。反应条件: 94 ℃变性5min, 94℃ 10s, 58℃15s, 72℃ 20s 进行45个循环后, 72℃延伸5min。采用2-ΔΔCt法计算CiBeclin1基因的相对表达量。
1.7 数据分析
实验数据均以平均值±标准差表示,CiBeclin1在不同组织中的相对表达采用One-way ANOVA分析(SPSS 16.0), MC-LR对CiBeclin1表达的影响采用多因素方差分析进行统计学检验(SPSS 16.0), 统计学显著性水平设定P<0.05表示差异显著。
2 结果
2.1 CiBeclin1基因全长cDNA序列特征分析
根据草鱼转录组测序得到一段长约1000 bpBeclin1的序列, 随后设计引物对该段序列进行验证并在NCBI 上进行BLAST 分析, 进一步确定为CiBeclin1 cDNA的中间序列, 随后通过RACE法扩增CiBeclin1基因的5′和3′末端序列。经序列拼接获得全长序列。CiBeclin1基因全长1590 bp, 包括5′非编码区和3′非编码区, 分别为54 bp和192 bp, 开放阅读框(Open reading frame, ORF)为1344 bp, 其在GenBank 上的登录号为MG797682。对其cDNA序列进行分析, 发现该序列含有ATTAAA Ploy(A)加尾信号。对其编码氨基酸序列预测分析, 发现CiBeclin1含有447个氨基酸, 分子量为51.2 kD, 理论等电点为4.88, 无信号肽。结构域分析表明,CiBeclin1蛋白有一个Bcl2同源结构域3(BH3, 111—120 aa)和一个进化保守结构域(ECD, 238—447 aa),并有一段不完整的富含亮氨酸的出核序列(LX3LX2LXX, 177—186 aa), 此外, 在http://smart.embl-heidelberg.de/上还预测到CiBeclin1蛋白有一个卷曲螺旋结构域(CCD, 171-211 aa)。
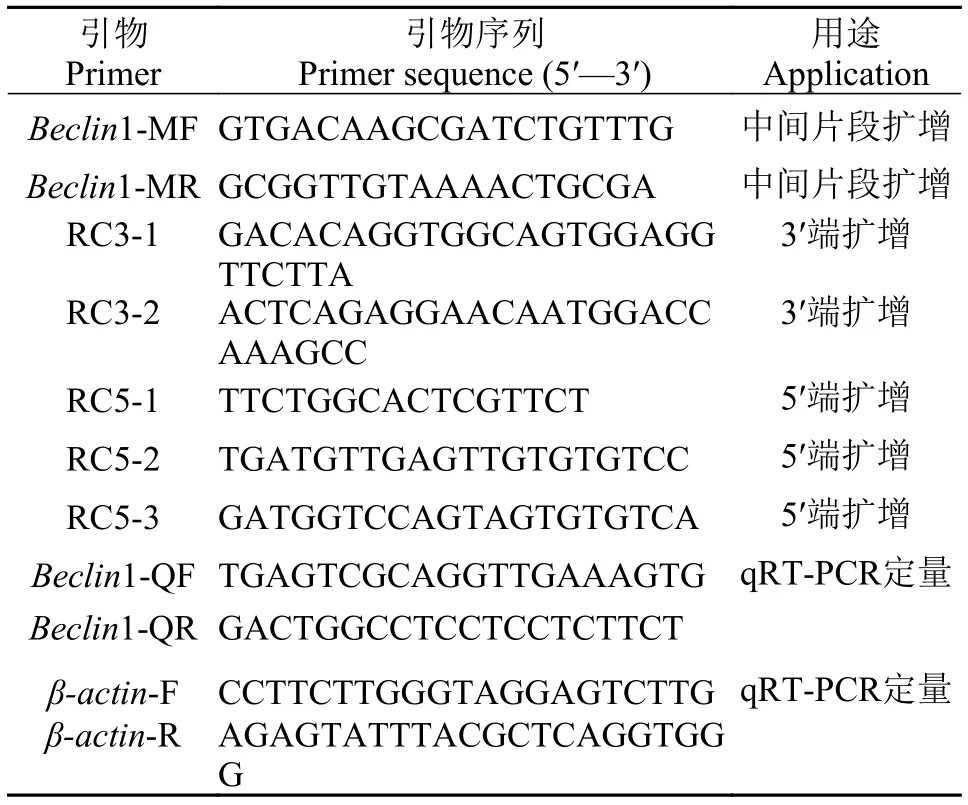
表1 CiBeclin1基因扩增和表达的引物Tab. 1 Primers used to amplify and express CiBeclin1 gene
2.2 CiBeclin1氨基酸同源性与系统进化分析
应用MEGA7.0软件Neighbor-joining(NJ)法构建系统进化树(图1),CiBeclin1与稀有鲫的Beclin1进化关系最近, 首先聚为一小支, 之后与斑马鱼和鲤鱼聚类形成一支, 再与其他鱼类聚为一大支。哺乳类、两栖类、爬行类和鸟类等其他动物聚为另一大支。
2.3 CiBeclin 1 基因组织分布特征分析
通过qRT-PCR方法研究了CiBeclin1在肝、肾、头肾、脾、肠、心脏、鳃、肌肉、皮肤以及血液10种组织中的表达, 结果表明,CiBeclin1在所检测的组织中均可表达, 呈组成型表达(图2)。在肝脏中的表达量最为丰富, 其次为肌肉, 在头肾中表达量相对较低。以相对表达量最低的头肾作为参照进行One-way ANOVA分析, 肝脏、肌肉、肠道以及心脏中CiBeclin1的表达量显著高于头肾表达量(P<0.05)。
2.4 微囊藻毒素-LR对草鱼肝脏CiBeclin1 mRNA水平的影响
草鱼暴露于不同剂量MC-LR(25和100 μg MCLR/kg BW)不同时间后(图3), 不同MC-LR诱导剂量对CiBeclin1 mRNA表达水平的影响差异显著(F=170.811,P=0.000), MC-LR诱导不同时间(24h、48h、72h和96h)对CiBeclin1 mRNA水平的影响也有显著差异(F=4.733,P=0.004); 作用浓度和作用时间之间有交互作用(F=3.967,P=0.002)。注射MCLR 24h, 48h, 72h和96h后, 2个剂量诱导组与对照组相比, 表达均显著下调(P<0.05); 但诱导96h 后, 100 μg MC-LR/kg BW剂量组中CiBeclin1表达量相对于诱导24h、48h和72h的表达量有上升的趋势, 但仍显著低于对照组(P<0.05)。
3 讨论
3.1 CiBeclin1基因cDNA全长的克隆及生物信息学分析
目前, 细胞自噬已成为生命科学研究领域的热点问题。然而, 自噬相关研究在鱼类中较少有报道。Beclin1作为细胞自噬的重要的调节因子, 在细胞自噬过程中发挥着重要作用[19]。本研究成功克隆了草鱼CiBeclin1基因的全长cDNA序列, 其包含一个1344 bp的开放阅读框, 编码447个氨基酸, 含有一个BH3结构域和一个ECD结构域, 这两个结构域在人类和哺乳类的Beclin1序列中也有[20], 对荷包红鲤和牙鲆Beclin1结构域的研究同样发现有这2个结构域[15—17]。与其他脊椎动物不同, 鱼类Beclin1基因的BH3结构域的117位氨基酸由丝氨酸取代了甘氨酸[16]。在酵母和哺乳动物细胞中, 抗凋亡蛋白家族成员如Bcl-2以及Bcl-XL等能与Beclin1 BH3结构域处结合而抑制自噬的形成[21], 此外,Beclin1的ECD结构域与Vps34结合诱导其他自噬相关蛋白定位于该蛋白复合体参与自噬的形成[22]。推测草鱼中的Beclin1可能发挥着同样调控自噬水平的功能。除此之外,CiBeclin1蛋白还有一个预测的卷曲螺旋结构域(CCD), 位于171—211 aa, 这与稀有鲫Beclin1的报道一致[17], 然而草鱼和稀有鲫CCD 结构域均由 41 个氨基酸残基构成, 而人类的由 97 个氨基酸组成[23]。CiBeclin1经分析发现其也具有一个41个氨基酸组成的CCD结构域。另外,CiBeclin1基因没有出核信号序列(PKKKRKV), 然而, 在177—186 aa位置有一段不完整的富含亮氨酸的出核序列(LX3LX2LXX, 其中X代表任何氨基酸),这与牙鲆和稀有鲫Beclin1的结构也相似[15], 另外, 该结构在人类和哺乳类的Beclin1序列中也有[20]。经过氨基酸同源性比对分析,CiBeclin1氨基酸序列与已报道的物种的氨基酸具有较高的相似性和一致性, 其中与稀有鲫氨基酸序列的相似性最高为98%。由以上分析可知,CiBeclin1蛋白序列和结构与其他鱼类及哺乳动物都比较相似。
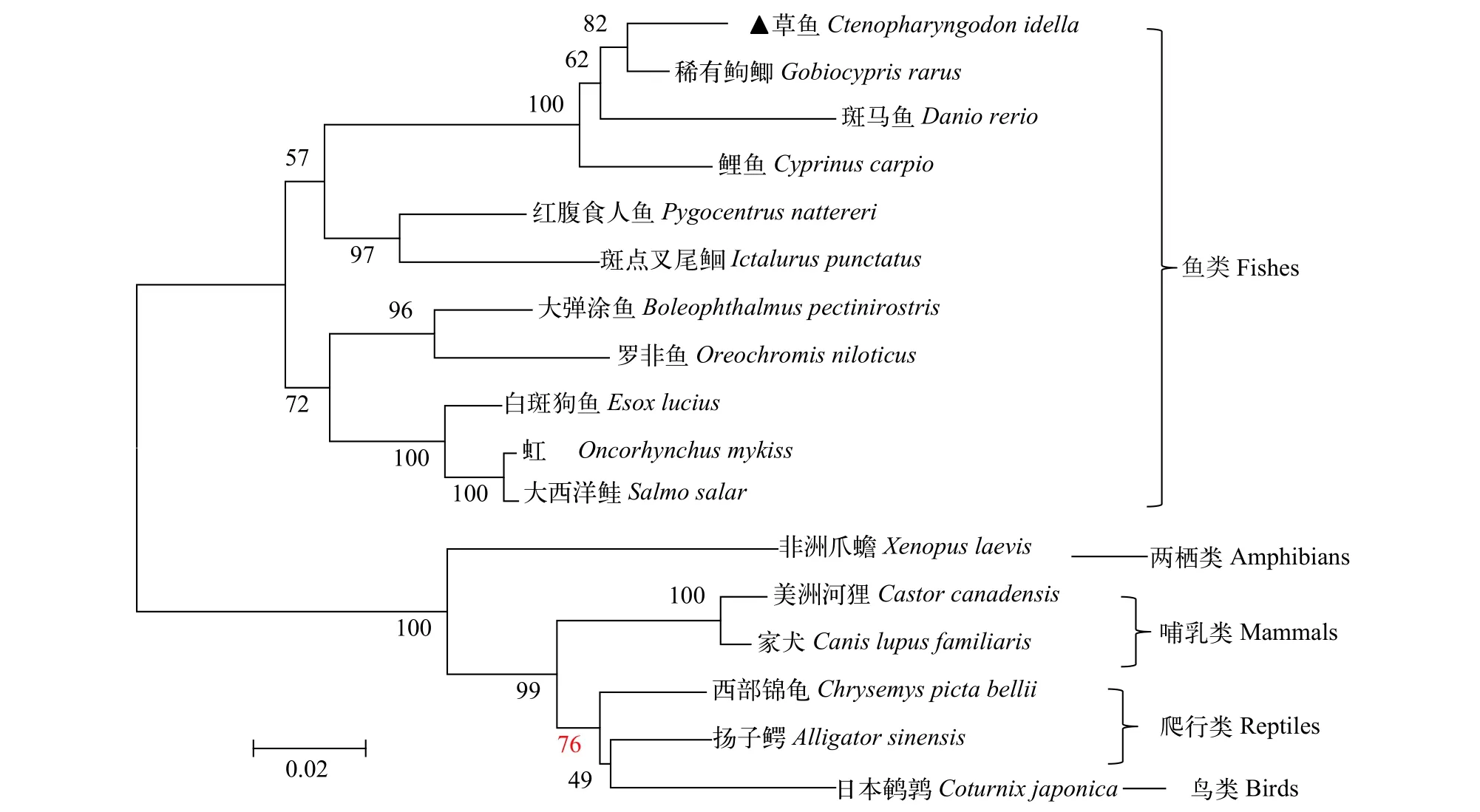
图1 草鱼及其他脊椎动物Beclin1基因的系统进化树Fig. 1 Phylogenetic tree of Beclin1 of grass carp and other species

图2 CiBeclin1的组织特异性表达分析Fig. 2 Tissue-specific expression of CiBeclin1 mRNA in grass carp

图3 MC-LR对草鱼肝脏CiBeclin1表达的影响Fig. 3 The effects of MC-LR on the expression of CiBeclin1 in liver of grass carp
3.2 CiBeclin1基因在不同组织中的表达分析
已有的研究报道表明,Beclin1可在健康牙鲆脑、鳍条、心脏、肝脏、肠道、胃和肾脏组织中广泛表达[15], 对稀有鲫Beclin1的表达分析也发现其可在不同组织中表达[17]。本研究同样发现CiBe-clin1也在所有检测的组织中表达, 包括肝、肾、头肾、脾、肠、心脏、鳃、肌肉、皮肤以及血液。Beclin1在各组织中的广泛表达提示了它在鱼类中具有非常重要的作用。
3.3 MC-LR对草鱼肝脏CiBeclin1 mRNA水平的影响
目前的研究报道表明细胞自噬除了在维持细胞内环境的稳态以及调节细胞的生长、分化和发育中起着重要作用外[24], 还在抵抗外界各种刺激,如饥饿、紫外线辐射、缺氧、病原体感染以及环境污染等方面起着自我保护作用[25—27]。其中有一些研究通过Beclin1的表达变化探讨了自噬在机体抵御环境污染物中的影响, 如Gao 等[16]检测了镉暴露后荷包红鲤肾脏组织中Beclin1基因的表达情况,结果发现在镉暴露后, 肾脏中Beclin1 mRNA和蛋白水平都有剂量和时间依赖性的升高, 因此作者认为Beclin1在应对镉的生物毒性上具有一定的作用。尽管MC-LR引起机体自噬的报道不多, 但有文献报道从微囊藻中提取的MC-LR可诱导肾Vero-E6细胞内质网空泡形成, 自噬小体大量聚集[28]。在原代培养的支持细胞中, 电镜结果显示MC-LR能够引起自噬小体增多, 说明MC-LR能促进自噬的发生[29]。也有研究报道Cho细胞经2.5、5和10 μmol/L MCLR处理后,Beclin1 表达升高, 且与对照组相比, 差异都有统计学意义(P<0.05), 并通过一系列实验证实了MC-LR诱导的自噬作为一种保护机制可抑制凋亡的发生[30]。然而, 吴珍等[9]采用qRT-PCR 分别检测自噬相关基因Beclin1 和LC3α的转录水平, 结果发现, MC-LR 在上调LC3α的同时, 抑制了Beclin1 的转录水平, 这与本实验结果类似。本研究通过qRT-PCR检测到不同剂量MC-LR诱导草鱼后,CiBeclin1的表达均被显著抑制。这些结果表明Beclin1在受到MC-LR胁迫后, 其表达会产生变化, 但在今后我们还需要更多的实验进一步验证Beclin1在抵御MC-LR胁迫中的具体作用。

