蛋白N-糖基化分析方法研究进展
张小倩, 张腾腾, 闫 攀, 杨毕超, 安凤霞
亳州学院中药学院, 安徽 亳州 236800
糖基化(glycosylation)是真核细胞最常见的蛋白转译后修饰方式之一,对蛋白质生物学功能的发挥起着至关重要的作用,如蛋白质折叠、细胞识别、癌细胞转移、薄膜保护作用、血型决定系统、物质转运等[1~3]。不仅如此,以重组蛋白质为基础研制的药品(例如单克隆抗体)成为当下社会快速发展的制药方向,许多新型药物都是糖蛋白,包括一些癌症标志物和疫苗等。自从1986年市场引入了第一个治疗型的单克隆抗体之后,治疗型蛋白药物的市场飞速增长。2017年,仅Humira一种单抗药物全球销售额就已高达184亿美元[4]。据不完全估计,人类蛋白中有50%以上都发生了糖基化修饰,生物制药中最亮眼的单克隆抗体100%都是糖蛋白药物,因此针对蛋白糖基化的研究愈发重要[5]。
生物体中最常见的糖蛋白是N-糖苷键型糖蛋白,即寡聚糖与肽链上的天冬酰胺(asparagine,Asn)侧链的酰胺氮连接而修饰的蛋白[6]。N-糖链在整个糖蛋白中只占很小的一部分,但是对整个糖蛋白的性质影响巨大。关于N-糖基化的研究通常可以从三个方面考量:①研究整体糖蛋白的糖链;②糖蛋白经过酶切后,糖肽水平的研究;③经过化学手段或酶切释放寡糖链,对游离N-糖的研究,其中对酶切游离后N-糖链的研究也是最简单、高效的方法[7,8]。目前,N-糖链主要是用肽N-糖苷酶F(PNGase F)酶解[9],得到游离的N-糖链,再结合毛细管电泳(capillary electrophoresis, CE)、高效液相色谱(high performance liquid chromatography, HPLC)和质谱(mass spectrum, MS)等技术对其进行分析。但由于游离N-糖链为多羟基醛类化合物,本身没有发色基团,紫外检测器几乎无法检测,且质谱响应也较差,常规电喷雾离子源(electrospray ionization, ESI)正负离子模式下灵敏度都不高,这也造成常规高效的检测方法如CE、HPLC及液质联用(LC-MS)等对其分析困难。针对这种情况,科学家多是通过柱前衍生法使糖链带上紫外或荧光基团,增加其检测的灵敏度[10],常规N-糖基化分析流程见图1。
糖基化作为蛋白翻译后修饰的重要步骤,其异常表达与疾病的发生发展、生理状态的变化之间具有很高的关联性,例如在炎症反应、恶性疾病、衰老等[11]不同的病理、生理条件下,糖链的结构和表达量都会发生变化。不仅如此,大量研究表明,单抗药物的N-糖基化修饰对其稳定性、清除率、免疫原性、抗体依赖细胞毒性(antibody-dependentcellular cytotoxicity,ADCC)及补体依赖细胞毒性(complement-dependent cytotoxicity,CDC)等都有一定的影响[12]。例如,缺少核心岩藻糖的糖型能够增强对FcγRⅢa受体的亲和力,从而提高ADCC[13];而高水平的唾液酸化的糖型则会减少对FcγRⅢa受体的亲和力,从而降低ADCC[14]。因此,对糖蛋白中N-糖基化修饰的研究具有重要意义,基于上述理念,本文对近年来科学家分离分析游离N-糖链的方法做了归纳与总结,这其中CE、HPLC以及MS联用技术在N-糖链的分离分析上有着巨大优势,对糖蛋白上N-糖的定性定量分析、结构表征具有重要意义。
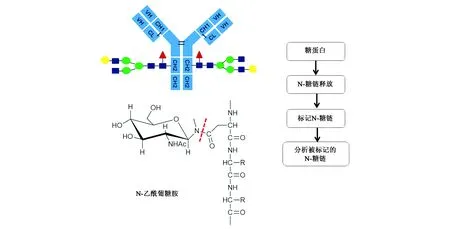
图1 N-糖链分析流程图Fig.1 Analysis flow chart of N-glycans.
1 N-糖链的释放
所谓N-糖链的释放就是将N-糖链从糖蛋白上切下来,其主要是通过酶解或化学方法将N-糖链和糖基化的多肽分开,然后再对释放的N-糖链单独分析,从而获得待检测样本中的糖链表达和变化情况,这样有目的性的对N-糖链进行分离分析可以大大提高分析效果[15,16]。
酶切释放主要依赖于PNGase A/F/H+和糖苷内切酶(endo-β-N-acetylglucosaminidase,Endo)F/H。PNGase可特异性识别并水解连接N-糖链的Asn残基侧链的氨基与羰基间的酰胺键[17]。其中,PNGase F是一种具有普适性的肽N-糖苷酶,已被广泛应用于释放糖蛋白的N-糖链。例如,Kozak等[18]采用PNGase F酶切人免疫球蛋白G(Ig G)中的N-糖链,然后用2-氨基苯甲酰胺(2-AB)衍生,19个N-糖链被稳定释放并检测。为了进一步分析β-伴大豆球蛋白(β-conglycinin)糖肽的肽链序列和糖链结构,李玲梅等[19]结合使用PNGase F与Endo H两种酶切技术鉴定糖基化位点。PNGase F可以特异性酶切Asn与糖链之间的糖肽键,而Endo H可以特异性酶切糖链五糖核心结构中2个N-乙酰葡萄糖胺(N-GlcNAc)残基之间的糖苷键,这2种方法酶解同一种糖肽所得肽段在分子量上相差202 Da,这一特点可用于精确鉴定糖基化位点。酶解后的N-糖链与肽段经MS分析,发现PNGase F 酶解所得肽链共有16个质谱峰,Endo H 酶解所得肽链共有14个质谱峰,对Endo H 与PNGase F 酶解后所得肽链进行比较发现,2个样品均有9条肽链,每条肽链都含有1个糖基化位点,而且每种Endo H 水解产物的分子量均比相应的PNGase F 水解产物大202 Da。最后通过数据库检索对分析结果进行验证,发现β-conglycinin上含有5个糖基化位点,分别为α亚基199位和455位的Asn,α′亚基215位和489位的Asn以及β亚基326位的Asn。
针对研究目的不同,也有学者采用其他不同的酶来切割N-糖链,比如,徐莎等[20]为了简化被切下来的N-糖链的衍生过程,使用非特异性酶链酶蛋白酶E(pronase E)对牛胰核糖核酸酶B(ribo B)和鸡白蛋白(chicken albumin)进行酶切,得到只带一个天冬氨酸的闭环N-糖链,这样由于引入了天然的-NH2活性基团,再以9-氯甲酸芴甲酯(Fmoc-Cl)对其衍生,可以省略N-糖链进一步分析前必须先衍生成糖胺的步骤,进而提高了整个实验过程的稳定性。不管采用何种酶切与衍生手段,最终都是为了提供一个高灵敏度的检测,便于后面的分析,但真正关键的地方在于N-糖链的分离与分析。N-糖链是一类极性很强的化合物,在反相色谱(reversed phase chromatography, RPLC)中基本没有保留,实验多采用亲水色谱(hydrophilic interaction chromatography, HILIC)来对其分析,再结合荧光检测、MS等对其检测[21]。
酶解释放N-糖链具有高效、快速、专一的特点,但成本高昂,近年来化学法释放N-糖链因其较低的成本与广谱性,也得到了一定的发展。N-糖链的化学释放主要依赖于肼、次氯酸钠和氨水等化学试剂。肼解反应能在95℃反应4 h的条件下释放N-糖链,但它需要严格的无水肼条件,并且反应伴有严重的脱乙酰基副反应[22]。次氯酸钠是通过氧化来释放N-糖链,此方法虽然能大规模释放N-糖链,但会使N-糖链还原末端的N-GlcNAc丢失[23]。针对上述情况,袁江北等[24]开发了一种简单、经济、快速的非特异性释放N-糖链的方法,实验采用NaOH体系对N-糖链进行完整的释放,糖蛋白样品置于50℃、0.5 mol/L NaOH溶液中反应16 h。MS分析结果表明该方法具有通用性好、释放效率高、无副反应的优点,N-糖链的释放效率能达到PNGase F酶的90%以上,并且通过此方法得到的N-糖链都是还原性寡糖,可以标记荧光试剂,目前已成功的用此方法对Ribo B、鸡白蛋白、人胎球蛋白和人血浆的N-糖链进行了解析。
2 N-糖链的分离分析方法
2.1 电泳法
CE以毛细管为分离通道、以高压直流电场驱动带电荷样品移动实现分离,具有分离效率高、样品消耗少等优点,在复杂糖链结构表征中具有很大的应用潜力[25]。硫酸化、唾液酸化的糖链自身带有负电荷,可用电泳法分离纯化,中性糖链通过与8-氨基芘-1,3,6-三磺酸(APTS)等作用形成带电荷且具有荧光基团的复合物,然后电泳分离[26]。王文波等[27]采用CE对原研抗CD20人鼠嵌合单抗和4个生物类似药的N-糖谱进行分析,比较它们之间的差异性。实验首先使用PNGase F对单抗Fc上的N-糖链进行酶切释放,然后用APTS标记;毛细管电泳使用Beckman N-CHO涂层毛细管,以分离胶为电泳缓冲液,在30 kV电压下分离,最后使用激光诱导荧光检测器(LIF)检测,最终原研抗CD20单抗中6种N-糖链被分离检测到。整个分离过程在20 min就可以完成,且经过APTS衍生的N-糖在LIF检测器下灵敏度可以达到amol级别。
电泳技术不仅可以对糖蛋白糖链进行一定的分离纯化,还可以给出其相关的结构信息。荧光辅助糖电泳(fluorescence-assisted carbohydrate electrophoresis, FACE)、CE等是糖链分离纯化、结构解析的常用工具,糖链在荧光标记后进行的聚丙烯酰胺凝胶电泳(PAGE)电泳即FACE,通过外切糖苷酶阵列法(reagent array analyses method,RAAM)和标准糖链的比对可以给出目标糖蛋白糖链的结构信息。CE可快速分离分析微量糖链,且不同缓冲体系中标准糖链的CE行为构建的糖谱,可以用于相关未知糖链的结构鉴定。Karger等[28]报道了基于CE-LIF检测的糖链检测方法,实现了7 min内Ig G中12种糖链异构体的高效分离,并将该方法成功应用于单抗类药物糖型表征,为抗体类药物的质量控制提供了简单、快速、灵敏的分析方法。李凤等[29]使用自制的聚乙烯醇中性涂层柱,乙酸铵缓冲体系,建立了寡聚葡萄糖的分析方法,通过拟合多项式分布曲线发现葡聚糖的聚合度值(glucose units, Gus)与各组分电泳迁移时间相关性良好,相关系数(R2)为0.99。然后采用此方法对5 μL人血清中N-糖链进行分析,初步分离得到了17个N-糖链组分,并建立指纹图谱,为探索血清蛋白的糖基化变化提供了分析方法。
PAGE胶上酶解法可同时分析糖蛋白糖链结构和糖基化位点,适用于不易纯化或量少的样品,是糖组学糖蛋白糖链结构研究的有力工具。Rudd等[30]利用PAGE将待分析糖蛋白分离后,切割感兴趣的条带并用PNGase F酶解释放糖链,再对糖链进行标记、分析。
2.2 液相色谱法
HPLC在分析化学、有机化学以及生物化学等领域应用很广泛[31]。N-糖链作为一类极性化合物,易溶于水、甲醇等极性溶剂,特别适合HPLC的分析。但是由于N-糖类化合物极性较强,在经典的RPLC上几乎无法保留,大都会被一起洗脱出来,并不适用于分离,即使是经过一些弱极性集团衍生过的N-糖也没有较好的保留。基于这种情况,科学家发明了HILIC用于这种强极性化合物的分析,它与正相色谱(normal phase liquid chromatography, NPLC)的保留机理相似,但却可以使用RPLC的流动相,使整个分离条件更温和,且有利于后续样品检测的顺利进行。
Henry等[32]采用超高效液相色谱(ultra performance liquid chromatography, UPLC)分离曲妥珠单抗上的N-糖链,并采用FLR与MS表征被分离的N-糖。实验通过PNGase F酶将单抗中的N-连寡糖释放出来,游离的寡糖先用2-AB进行标记,然后在HILIC模式下进行分离并使用FLR和串联四极杆飞行时间质谱(quadrupole-time of flight mass spectrometry,QTof)进行检测。对比3个批号曲妥珠单抗的N-糖的HPLC-FLR分析结果,被标记的N-糖均得到较好的分离,17个糖型在35 min内完成分离,各峰之间都实现了基线分离,结合FLR可以很好地用于各糖型的定量,且整个分析结果表现了出了很好的重现性。王文波等[33]利用UPLC串联单四极杆检测器(QDa)对抗CD20人鼠嵌合单抗的N-糖谱进行定性和定量分析。CD20单抗的N-糖链经PNGase F酶切释放后用RapiFluor MS试剂进行标记,通过QDa对N-糖进行糖型鉴别、FLR进行定量检测。UPLC条件采用酰胺色谱柱、乙腈-乙酸铵流动相体系,在荧光检测器下检测到15种N-糖类型,在35 min内实现基线分离,并对其进行定量,再结合QDa质谱检测器定性,很好的实现了抗CD20人鼠嵌合单抗中N-糖的表征情况。
HPLC因其高效稳定的分离能力在N-糖的分析中非常重要,经过HPLC的分离后,结合高灵敏度FLR检测的数据可以实现对N-糖的定量分析,同时MS又能帮助确认N-糖的结构信息。铁偲等[34]为了满足IgG药物N-糖分析的实际需要,建立了稳定、灵敏的IgG药物N-糖的分析方法,采用全新的肼基衍生化试剂对游离N-糖进行衍生化,并对衍生化后的N-糖进行LC-MS分析。实验同样采用酰胺色谱柱,乙腈-碳酸氢铵流动相体系。14个类型的N-糖被检测到,其中麦芽五糖在30 ng/mL的低浓度下仍然实现了检测。整个分析方法高效快速,而且具有极高的灵敏度。
2.3 质谱
ESI和基质辅助激光解析(matrix-assisted laser desorption ionization, MALDI)是目前用于糖链电离的两种主要方法,并且在糖链离子化方面具有很强的互补性。此外,通过与多种高性能质量分析器(三重四极杆、时间飞行、离子阱、傅里叶变换离子回旋共振等)串联后,可以实现串联质谱(MS/MS)或多级质谱(MSn)分析,从而获得更多的糖结构信息。
2.3.1电喷雾电离质谱 电喷雾电离质谱(electrospray ionization mass spectrometry, ESI-MS)使溶液中的被分析化合物在电喷雾针里带电、雾化后形成带电荷的分子离子峰,离子再经过质量分析器分析获得分子量相关信息。ESI-MS根据样品的性质可以产生多电荷离子,使得其质量检测范围成倍增加,并且能够获得比单电荷母离子更多的结构信息,对于大分子化合物的分子具有独特优势。但是对于ESI-MS而言,样品的制备是十分复杂的,主要是因为其容易受到样品添加剂和缓冲盐的影响[35]。因此,其常常需要与HPLC、CE等技术进行联用,结合HPLC与CE的分离能力对组分更为复杂的样品进行分离分析。例如Sethi等[36]通过使用PNGase F酶对3种不同表型的结肠癌细胞中的糖蛋白进行酶解,得到的游离N-糖采用LC-ESI-MS/MS在负离子模式下进行分析,结果发现3种表型的结肠癌细胞中的甘露糖均有较高的表达(70%~90%),同时揭示了二天线α-2,3-唾液酸N-糖结构对于肿瘤细胞分型的影响。
N-糖链结构复杂,不同单糖之间具有多种连接方式,而一级质谱数据只能提供N-糖的质量数,对其链接方式较难确定,串联质谱丰富的数据可以判断、确定糖链的线性结构。如潘丽英等[37]使用ESI-MS/MS分析人肝癌细胞HepG2和正常肝细胞L02中N-糖的差异,被PNGase F酶酶切下的游离N-糖链经过串联四极杆质谱分析后,通过分析母离子与碎片离子,26种N-糖链被检测并确认,通过比较发现,HepG2的大多数高甘露糖型糖链、唾液酸化糖链和岩藻糖基化糖链的数量较L02都明显升高。
对于糖链结构的完全解析,则需要多级质谱的数据来确定单糖的组成、序列、糖链的连接方式、糖链拓扑结构等信息。在多级质谱模式下,可以选择特定的母离子,使用气体进行碰撞,依次产生子离子碎片。通过分析单糖与单糖之间的裂解信息,可以确定糖链序列,分析单糖的环内裂解信息,可以确定糖链的分支情况。在确定碎片结构后,将所有的碎片信息重组,可以获得完整的糖链结构信息。Reinhold团队[38~40]就应用多级质谱对糖链进行测序,并提供了三种分析的策略和算法,对糖链的连接位点、单体、空间构型等信息进行了推测与判断。
2.3.2基质辅助激光解析电离质谱 MALDI-MS是一种直接气化并离子化非挥发性样品的质谱离子化方式,整个电离过程可在极短的时间内完成,且不产生热分解。而且这种温和的电离方式只产生单电荷分子离子,产生的数据简单,在糖组学分析中具有重要意义[41]。对N-糖链结构分析时,酸性末端如唾液酸常在高能量的碰撞中丢失,MALDI因其温和的电离方式可以得到准确的分子离子峰。MALDI离子源常用Tof进行检测,这种组合成的质谱被称为基质辅助激光解析飞行时间质谱(MALDI-TOF-MS),主要用于完全甲基化的糖链分析[42]。MALDI-TOF-MS具有灵敏度高、分子质量检测精确度高、检测速度快、成本低等特点。基于这些特征,Canis等[43]先用凝胶色谱和亲和色谱对血清中的血管性血友病因子(vWF)进行纯化,并对酶解游离出的N-糖进行了正负离子两种模式的MALDI-TOF-MS分析,分析了300个人血管性血友病因子携带的N-糖谱,同时也证明了H抗原不受N-糖基化位点的影响。此外,MALDI-TOF-MS也是用于大规模体外诊断和生物标志物研究的有力工具,Holst等[44]应用MALDI-TOF-MS对25种不同的结肠直肠癌细胞系的N-糖谱进行了大规模研究,发现了α-2,3和α-2,6连接的唾液酸糖肽在不同细胞膜蛋白的改变,并证明了高甘露糖型N-糖不仅存在于细胞内前体上,也存在于细胞膜上。
3 展望
蛋白质的糖基化作为调控蛋白质功能的重要因素之一,糖基化位点的改变或者糖链结构的改变都会引起蛋白质功能的改变。无论是基于病变机理对糖基化的研究,还是糖蛋白药物质量稳定性中关于糖基化的研究都至关重要。HPLC、CE与MS都是N-糖分析的有力手段,HPLC与CE用于不同糖型的分离,高效稳定,且使用性非常广,基本的N-糖都可以通过二者来分离,同时由于二者分析机理的差异性,又可以互补使用。FLR与MS作为N-糖分析时常用的检测方式,具有非常高的灵敏度,尤其是经过衍生后的N-糖被检出的灵敏度更是大大增加。N-糖基化作为糖蛋白中最主要的糖基化类型,对其的定量定性分析既重要又困难。糖复合物中糖类分子的异常表达,重组糖蛋白生产时,生物合成共价寡糖出错都是非常致命的问题。而且N-糖链在整个糖蛋白中占比非常少,准确、可重复的将其富集出来有较大的难度,在N-糖链的分离与解析时,因空间立体结构差异较大,同样也存在非常大的困难。应该着重发展更稳定可靠的N-糖释放、富集方法,再结合现代高效分析手段,以及有针对性的N-糖解析措施,为N-糖基化高通量、高准确性的分析提供依据。

