基于近红外光谱的大鲵黏液粉掺伪的定性定量研究
辛 茜, 陈德经,2*, 夏冬辉, 杨 慧, 金文刚,2, 贾少杰, 贺屹潮
(1.陕西理工大学 生物科学与工程学院, 陕西 汉中 723000; 2.陕西理工大学 陕西省资源生物重点实验室, 陕西 汉中 723000; 3.陕西理工大学 化学与环境科学学院, 陕西 汉中 723000)
中国大鲵(AndriasdavidianusBlanchard)属大型两栖纲、有尾目、隐鳃鲵科,属国家二级保护动物,2017年起大鲵被列入人工繁育国家重点保护水生野生动物名录,人工繁殖的子二代大鲵允许加工和利用[1]。大鲵体表多皮肤腺,受到微生物感染、外界压力或电刺激时,会分泌一种具有特殊性气味的白色黏稠状黏液,该黏液含有黏糖蛋白、低聚糖肽、无机盐以及“蛙皮素”等生物活性成分,具有抗菌[2-3]、抗肿瘤[4]等药理作用。徐伟良[5]采用MTT法检测黏液糖蛋白对人肺腺癌细胞A549的抑制增殖作用,发现大鲵黏液具有良好的杀伤肺癌细胞的作用。黏液在冻成干粉后,其粘附性极强,有较好的生物相容性,并且可降解,也可以作为止血材料[6]、粘合剂[7]及软组织填充材料等[8]。所以,作为新的“抗生素”药物和生物粘合剂的大鲵黏液,是目前市场上最具前景的天然医用生物粘合材料[9]。在大鲵黏液粉中掺入低成本的土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉,掺入物与大鲵黏液粉很难区别,但在生物活性以及应用价值上相差甚大。因此,如何快速、简捷、有效地鉴别大鲵黏液粉,成为大鲵黏液合理开发应用的关键。
本文以大鲵黏液粉为鉴别对象,选择土豆淀粉、大鲵肉粉、大鲵蛋白粉、大鲵皮粉作为掺伪物质,分别运用偏最小二乘线性判别法(PLS-DA)和偏最小二乘回归分析法(PLSR)建立掺伪大鲵黏液粉的定性判别模型和定量分析模型[10],达到快速鉴别掺伪大鲵黏液粉及预判掺伪含量的目的。
1 材料与方法
1.1 材 料
1.1.1 试验材料
大鲵黏液、土豆淀粉、大鲵肉粉、大鲵皮粉、大鲵蛋白粉均由陕西省资源生物重点实验室自制。
1.1.2 仪器与设备
VERTEX 70型傅里叶红外光谱仪,配有OPUS 7.5光谱采集软件和漫反射积分球附件(德国Bruker);FA3204B型电子天平(上海精科实业有限公司);LGJ-10B型冷冻干燥机(北京四环科学仪器厂有限公司);BCD-649WE型双开门冰箱(青岛Haier);DFY-200小型粉碎机(温岭市林大机械有限公司);100目不锈钢标准筛(临沂市科航实验设备有限公司)。
1.2 方 法
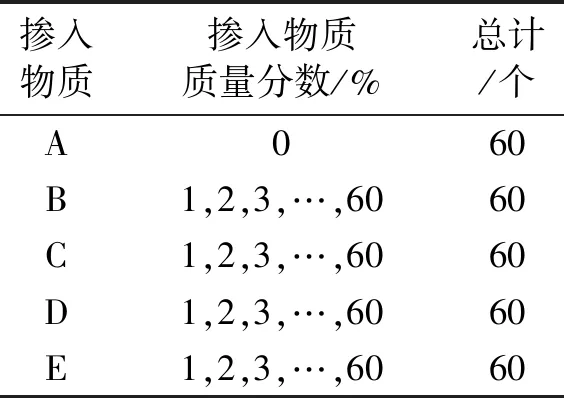
表1 掺伪样品的配比分布
1.2.1 样品制备
刺激大鲵收集黏液,用真空冻干机干燥,冻干的黏液研磨成粉末状过100目筛;土豆淀粉、大鲵蛋白粉,研磨过100目筛。以大鲵黏液粉(A)为基础,掺入物土豆淀粉(B)、大鲵肉粉(C)、大鲵蛋白粉(D)、大鲵皮粉(E),共配制4类掺伪样品。60个纯黏液粉样品和240个掺伪黏液粉样品,共计300个样品。样品质量均为0.3 g,称量误差不超过0.001 g。置于2 mL的离心管中,旋涡混合器上混匀,样品的配比方法见表1。
1.2.2 光谱采集
试验前需先打开机器预热30 min左右,然后将样品放入石英样品杯中混合均匀,以仪器内置背景为参比,采用积分球漫反射方式采集300个样品的近红外光谱,所有样品均重复采集3次,取其平均值,获得样品的平均近红外光谱。光谱扫描范围12 500~4000 cm-1,分辨率为8 cm-1。
1.2.3 定性分析
采用MATLAB R2014a分析软件。将采集的300个光谱进行2分类和5分类的分类建模分析(2分类是将样品粗分为纯样品和掺伪样品;5分类将样品细分为纯大鲵黏液粉、掺入物土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉)。采用KS(kennard-Stone)方法选择240个样本作为校正集,剩余的60个样本作为预测集。采用多元散射校正(MSC)、标准正态变量变换(SNV)、一阶导数(1SGD)、二阶导数(2SGD)、1SGD+MSC和1SGD+SNV对原始光谱经预处理后,通过比较校正集及预测集定性判别模型的准确率,来确定定性模型最佳预处理方法。
1.2.4 定量分析
采用偏最小二乘回归分析法(PLSR)分别建立掺入不同比例土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉的掺伪大鲵黏液粉的定量分析模型。各类掺伪样品的总数均为60个。建模时,每类分别采用KS(kennard-Stone)方法选取42个样本作为校正集,建立定量校正模型,其余18个样本作为预测集,检验预测模型的性能。通过交互验证均方根误差(Root mean square error of cross-validation,RMSECV)确定建模所需的主因子数,对所建模型的质量采用校正/预测相关系数(Correlation coefficient of calibration/prediction,Rc2/Rp2)、校正均方根误差(Root mean square error of calibration,RMSEC)和预测均方根误差(Root mean square error of prediction,RMSEP)来评价。校正/预测相关系数越大,模型线性相关性越好;校正均方根误差与预测均方根误差越小,模型拟合、预测效果越好[11]。定量数据处理软件与定性处理软件相同。
2 结果与分析
2.1 纯大鲵黏液粉和掺伪物的平均近红外光谱
图1为60个纯大鲵黏液粉样品在12 500~4000 cm-1光谱区间内的平均近红外光谱图。由于不同的大鲵黏液粉样品内部化学成分和组织结构不同,所以光谱的波峰强度会有所差异。在5774、6670、8416 cm-1等处有明显的特征吸收峰。5774 cm-1是C—H键倍频的倍频吸收和CH3基团吸收[12],6670 cm-1是N—H键的倍频吸收,8416 cm-1是C—H键伸缩振动的二倍倍频。
图2是纯土豆淀粉、大鲵肉粉、大鲵皮粉、大鲵蛋白粉和大鲵黏液粉的平均近红外光谱图。12 500~4000 cm-1区间信息量丰富,能反映出5种样品的差异性。在7500~5500、5000~4000 cm-1范围内,土豆淀粉的光谱图与其他4种样品的光谱图差异显著。可能是因为土豆淀粉的主要成分是多糖,其他4种样品主要以蛋白质为主。大鲵肉粉、大鲵蛋白粉、大鲵皮粉和大鲵黏液粉的光谱尽管趋势相同,但在相同的波长下吸收度是不同的。因此,可以进行定性判别。
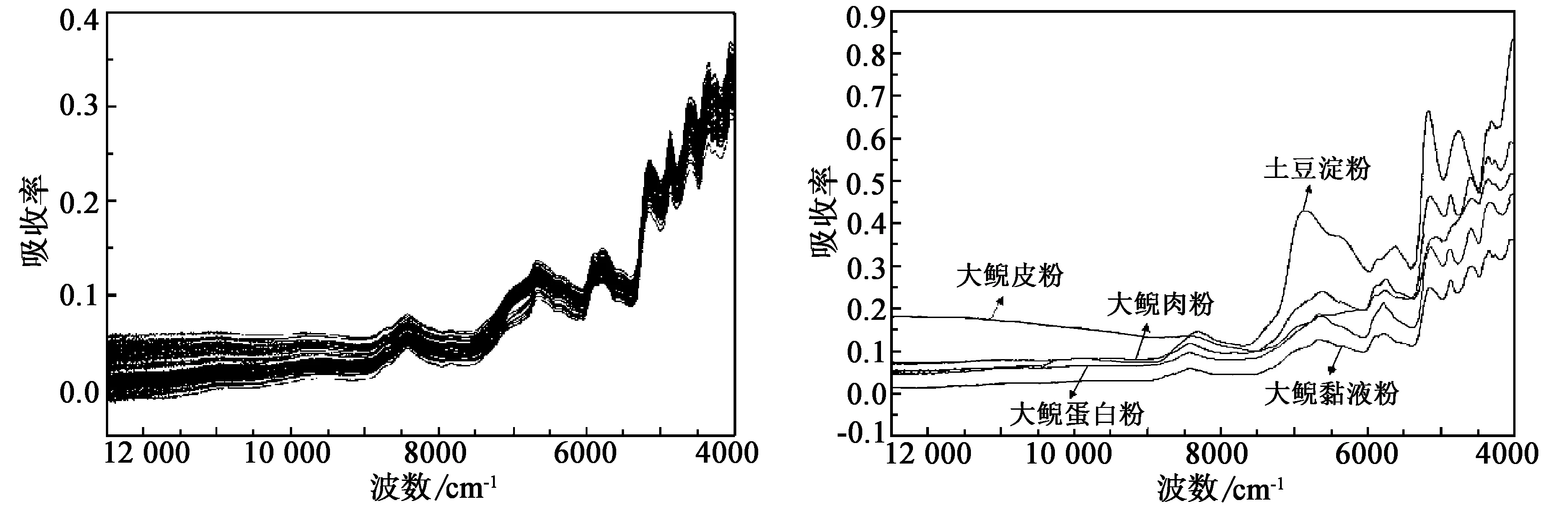
图1 大鲵黏液粉的平均近红外光谱 图2 5种纯样品的平均近红外光谱
2.2 掺伪大鲵黏液粉的平均近红外光谱
图3是掺入不同比例的4种物质后形成的近红外光谱图。由图可见,样品与样品间近红外光谱的谱峰重叠非常严重,并且含有大量的噪声信号,无法直接获得光谱中有效的信息。Mabood F等[13]采用近红外光谱结合化学计量法很好地解决了掺假骆驼奶的鉴别研究,因此,建立光谱与化学参考值之间相关联的化学计量学模型来处理数据是必不可少的。
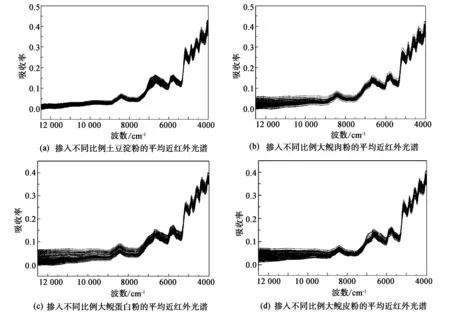
图3 4类掺伪大鲵黏液粉的平均近红外光谱
2.3 定性分析结果
近红外光谱在采集过程中会产生基线漂移、散射、噪音等问题,严重影响建模的精确度,采用适当的光谱预处理方法会有效提高模型的精确度[14]。经不同的预处理方法处理后产生的效果不同,有的可消除基线漂移和背景影响,有的可消除噪音,有的可消除由于颗粒大小不均产生的散射影响。并且不同的方法可叠加使用,叠加的效果也是有好有坏[15]。本文采用1SGD、2SGD、MSC、SNV、1SGD+MSC和1SGD+SNV共6种光谱预处理方法对原始光谱进行优化,结果见表2。
表2 PLS-DA法建立2分类掺伪大鲵黏液粉模型的判别结果
表2结果表明,采用6种预处理方法建立模型,其预测效果不同,且预处理模型的准确率均高于原始光谱的准确率。其中经2SGD方法预处理后校正集和预测集的准确率最低,分别为99.17%和98.33%;经1SGD+SNV方法光谱预处理后,模型校正集和预测集的准确率均为100%,该模型可以准确区分纯样品和掺伪样品。
表3是5分类掺伪大鲵黏液粉模型判别情况。结果表明,1SGD+SNV光谱预处理的效果最优,校正集和测试集的准确率高达100%。其他预处理方法均有误判数,但准确率也在原始光谱之上。该模型可以区分纯大鲵黏液粉和掺入土豆淀粉、大鲵肉粉、大鲵蛋白粉、大鲵皮粉的掺伪样品。
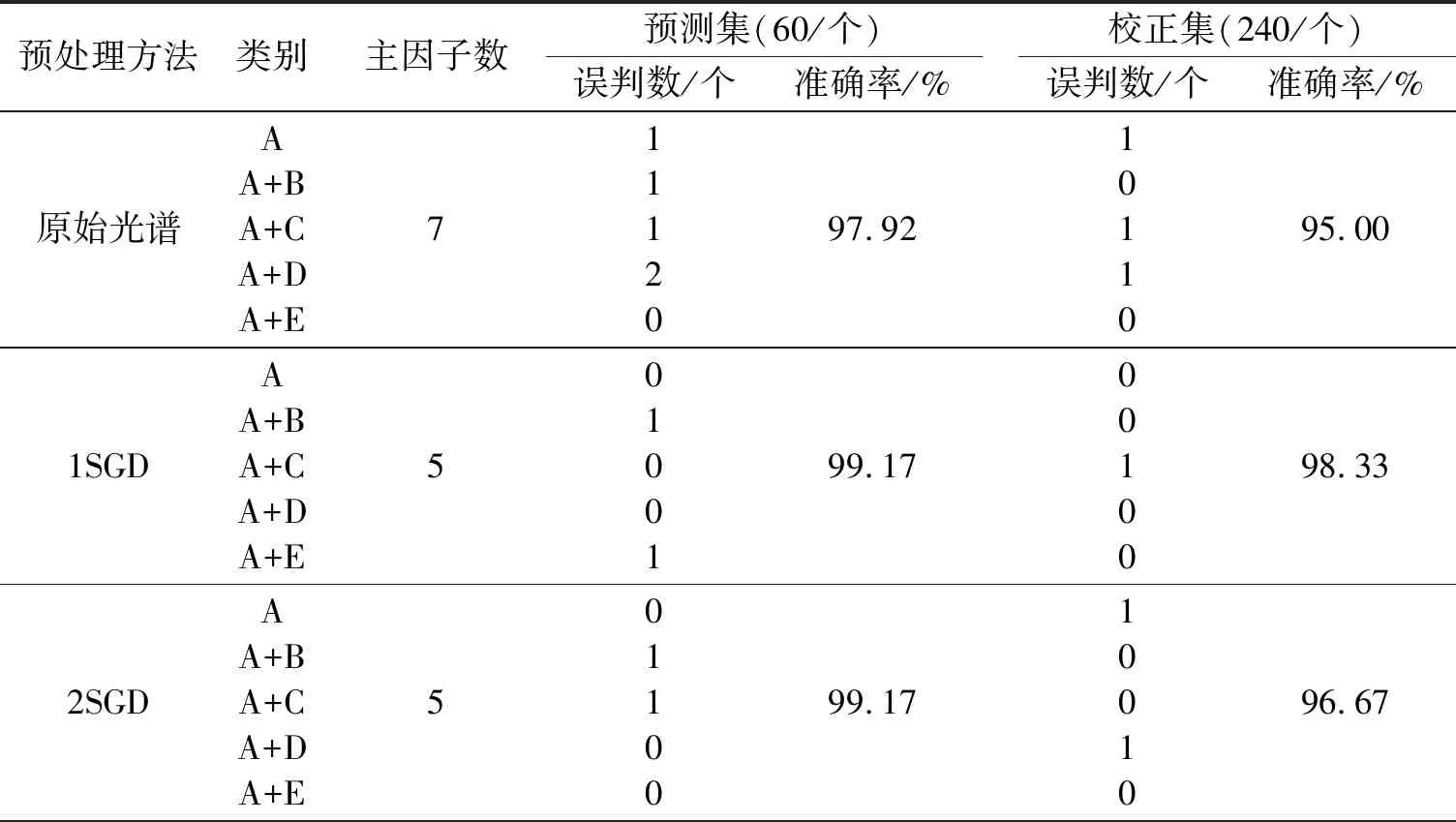
表3 PLS-DA法建立5分类掺伪大鲵黏液粉模型的判别结果
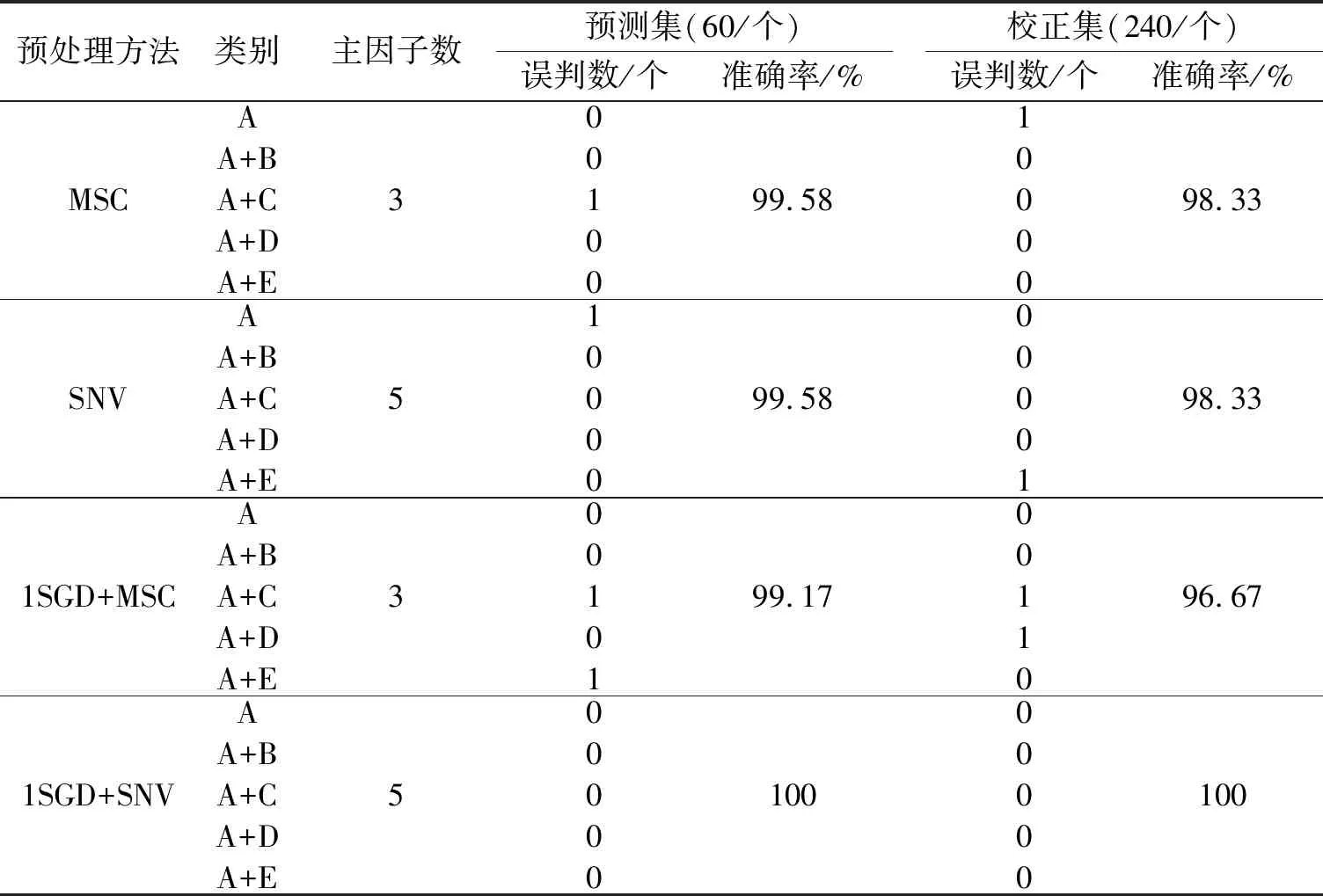
续表
因此,确定一阶导数+标准正太变量变换为大鲵黏液粉掺伪定性判别模型的较佳预处理方法。这是因为一阶导数可以消除基线漂移和背景干扰,SNV能够消除固体颗粒大小、表面散射和光程变化对光谱的影响[16]。索少增等[17]比较了消除常数偏移量、减去一条直线、矢量归一化、最小最大归一化等10种预处理方法对PLS建模效果的影响,得到SNV预处理后综合参数最好。徐文杰等[18]为了建立最优的淡水鱼鱼种鉴别模型,选择了20余种不同光谱预处理对3种建模方法进行优化。结果表明,其最佳的光谱预处理方法为数据标准化。所以,光谱预处理方法的选择是因检测对象的不同而存在差异的。
2.4 定量分析结果
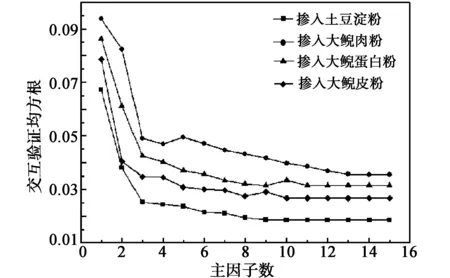
图4 主因子数与RMSECV的相关图
主因子数目的大小影响建模效果的好坏。主因子数目过多,建模时会将无用的噪音当作有用信息来用,此时模型过拟合;主因子数目过少的话,会出现欠拟合,是因为光谱中的有用信息未能完全反馈[19]。本文采取交互验证的方法确定合适的主因子数目。交互验证均方根(RMSECV)的值可以反映模型预测的精准性,RMSECV值越小,说明模型的预测能力越强;RMSECV值最小时,此时的主因子数目最合适[20]。用优化后的主因子数建立掺入土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉的掺伪样品定量校正模型,用预测集对模型效果进行验证。
图4是校正集建模时前15个主因子数与所对应的RMSECV值的相关关系图。从图中可知,当RMSECV值最小时,掺入土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉的掺伪大鲵黏液粉模型合适的主因子数分别为10、13、11和10。确定了合适的主因子数后对4个掺假样品建立定量分析模型。
大鲵黏液分别掺入土豆淀粉、大鲵肉粉、大鲵蛋白粉和大鲵皮粉后定量模型测出的实际值与预测值的相互关系如图5。由图可知,这4类掺伪大鲵黏液粉定量校正模型的线性方程:y=1.003 4x+0.294 9,y=0.987x+1.153 5,y=1.023 5x-1.071 5,y=0.984 7x+1.385 6;相关系数(Rc2)分别为0.992 0、0.969 1、0.976 8和0.982 2,校正均方根误差(RMSEC)分别为0.015 9、0.031 1、0.028 0和0.024 6。用预测集对校正模型进行检验,得到预测线性方程:y=1.011 7x-0.795 9,y=0.996 3x+0.895 5,y=0.977 7x+1.870 8,y=0.987 6x+1.122 7;相关系数(Rp2)分别为0.990 6、0.961 7、0.973 8和0.979 9,预测均方根误差(RMSEP)分别为0.019 5、0.035 3、0.030 2和0.022 7。
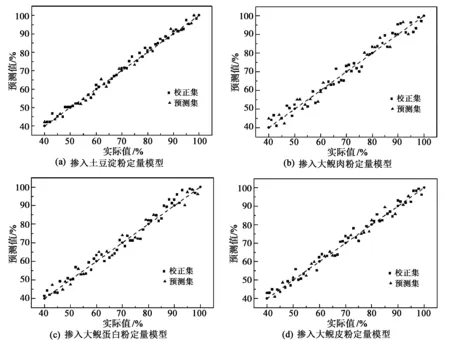
图5 4类掺伪大鲵黏液粉的实际值与预测值关系图
4类掺伪大鲵黏液粉模型的Rc2和Rp2都达到了0.9以上,RMSEC和RMSEP趋近于0,说明所建的定量模型均呈线性相关,且具有较高的预测精确度和稳定性。周晓璇等[21]也通过近红外光谱结合PLSR法建立的掺低档米定量模型和掺矿物油米定量模型均能很好实现对两种掺伪大米的定量分析。因此,本研究采用近红外光谱技术结合偏最小二乘线性回归法可以量化掺伪大鲵黏液粉的水平。
3 结 论
运用近红外光谱结合PLS-DA建立了掺伪大鲵黏液粉的定性分析方法。PLS-DA判别模型中,比较了大鲵黏液粉及其掺伪物的原始光谱以及经1SGD、2SGD、MSC、SNV、1SGD+MSC和1SGD+SNV等光谱预处理后所建模型判别效果。确定1SGD+SNV为较佳光谱预处理方法,所建2分类和5分类模型校正集与预测集的准确率均为100%。
运用近红外光谱结合PLSR建立了掺伪大鲵黏液粉的定量分析方法。大鲵黏液粉分别掺土豆淀粉、大鲵肉粉、大鲵蛋白粉、大鲵皮粉模型预测精度高、相关性好,能实现对掺伪大鲵黏液粉的定量分析。

