医学外显子组测序检测遗传病拷贝数变异的初步探索
张 彦,孙樱桐,许艺明,丁红珂,张 巍,尹爱华
(1.广东省妇幼保健院医学遗传中心,广东广州 511442;2.广州嘉检医学检测有限公司,广东广州 510300)
遗传病种类繁多,临床表现大多不特异,常规手段诊断困难,常常需要对整个基因组或基因组特定区域进行检测才能起到辅助诊断的作用。染色体微阵列技术和高通量测序技术作为基因组检测技术的代表,对于遗传病的临床诊断具有重要价值[1-2],但由于费用和检测周期等问题,临床病例急需一种相对单一的解决方案。对于孟德尔遗传病,除了种类繁杂的单核苷酸变异(single nucleotide variation,SNV)相关的遗传病外,拷贝数变异(copy number variation,CNV)也与多种遗传病相关[3]。高通量测序技术虽然针对不同种类的疾病有不同的检测方案,但大多是针对SNV的检测,对于CNV的检测,仍然有较多的争议,大多数的检测方案是依据全外显子组(whole exome)或特定基因包(panel)进行算法优化[4-5]。虽然不同的算法在特定的病种或群体中显示出一定的有效性,但并未得到广泛认可。医学外显子组(medical exome)测序,也叫临床外显子组(clinical exome)测序,是指针对目前已知的与遗传病相关的所有基因的编码区及其侧翼区进行捕获后测序[6]。不同的检测机构依据的数据库版本有差异、采用的捕获策略不同,因此方案设计上会有部分区别,但对于大多数遗传病的检测,尤其是SNV的检测,设计上的差别基本上可以忽略。然而对于CNV的检测,目前可获得的资料相对较少。本单位从2015年开始应用医学外显子组测序进行遗传病基因诊断,逐渐建立了较为完善的CNV检测方法,已经在较多病例中得到不断验证。本文仅以两个家系为例对医学外显子组测序在CNV相关的遗传病基因检测中的可行性进行分析。
1 材料与方法
1.1 研究对象
家系1(图1A)正常个体1-II-3于2017年8月来我中心咨询,自诉有智力低下家族史:2位哥哥(1-II-1和1-II-2)均为患者。患者1-II-1(36岁)和1-II-2(34岁)表现为头型偏长(似长方形),智力轻度低下,语言能力差,运动能力正常。患者1-II-2伴有单侧眼睑下垂,可识汉字,读书到小学5年级,期间成绩较差。
家系2(图1B)患者2-II-1,男,9岁,2016年8月来我中心咨询。患儿出生时手指、脚趾偏长,指关节屈曲。1岁时大脑CT提示脑发育异常(未见影像资料),智力评估低下,发育落后,并伴有房间隔缺损,三尖瓣反流(未见影像资料)。父母带患儿来我中心咨询时身高126 cm,体质量19 kg,身型较瘦,呈特殊面容,眉毛粗短,左眼斜视,舌无法正常外伸,不会讲话,外耳廓呈半圆形,耳位偏低(图2)。患者弟弟2-II-2正常,男,7岁。
1.2 方法
1.2.1 样本采集 经患者及家属知情同意,采集家系1、2患者和父母及部分正常个体外周血2 mL(EDTA-Na抗凝)。取得家系1患者基因结果后,抽取个体1-II-3妻子腹中胎儿1-III-1脐血1 mL(孕26+周时)。使用德国Qiagen公司生产的Qiamp DNA Blood Mini Kit提取试剂盒进行基因组DNA提取(操作步骤参照试剂盒说明书)。
1.2.2 脆性X综合征基因检测 FMR1基因CGG三碱基重复数检测,采用德国Roche公司生产的Expand Long Template PCR System试剂盒进行PCR扩增,反应条件:98 ℃ 10 min,循环数1;97 ℃ 35 s,64 ℃ 35 s,68 ℃ 4 min,循环数 10;97 ℃ 35 s,64 ℃ 35 s,68 ℃ 4 min(每个循环增加20 s),循环数25;68℃10 min。后经毛细管电泳(ABI 3 500-XL测序仪)进行片段长度判断,最后计算CGG三碱基重复数。
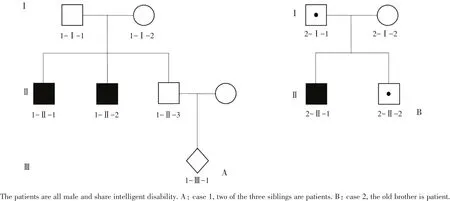
图1 两个家庭的家系图Fig.1 Pedigree for the two families
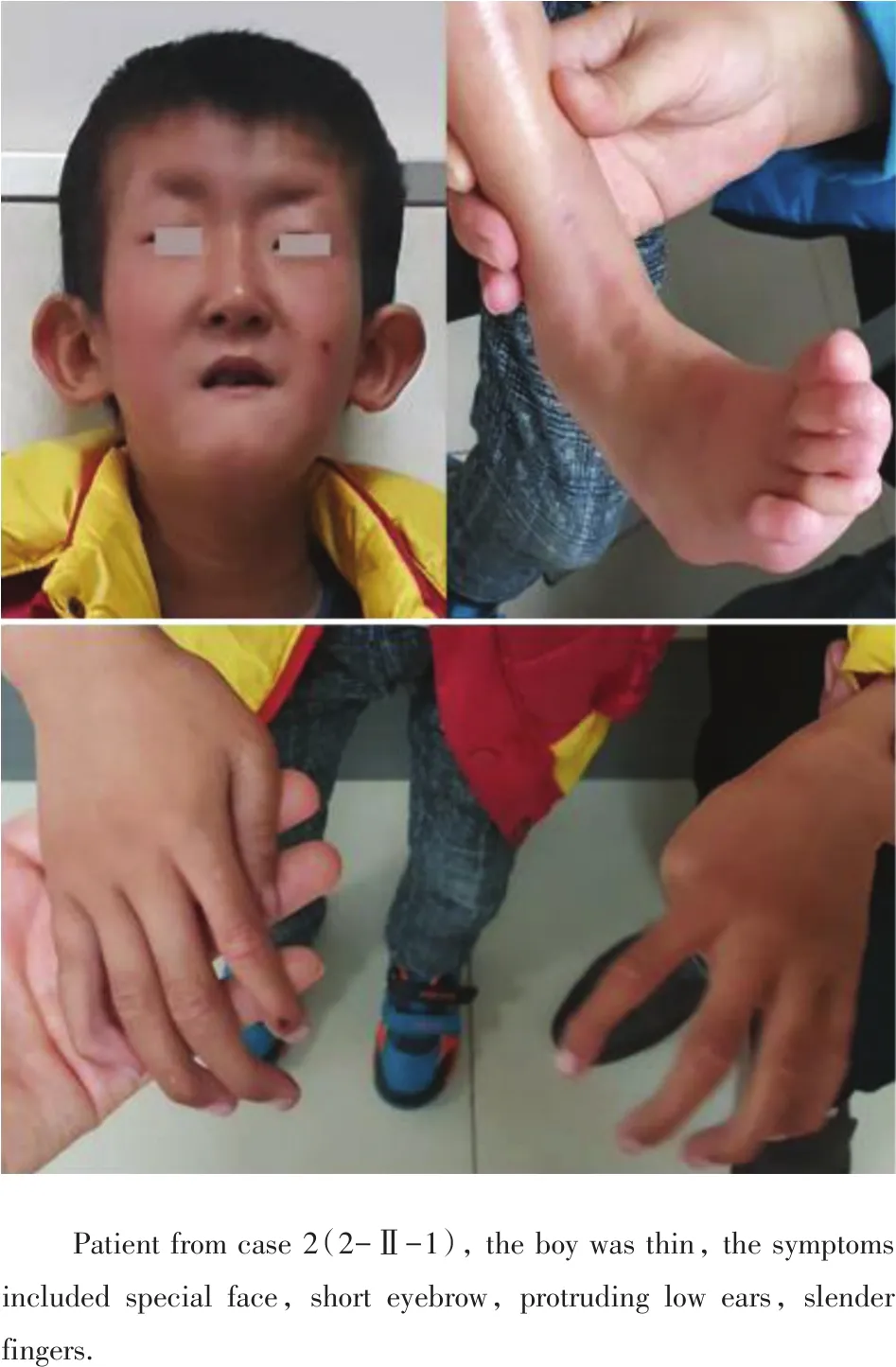
图2 病例2-Ⅱ-1部分表型Fig.2 Photos of patient 2-Ⅱ-1
1.2.3 染色体核型分析 采用G显带技术对外周血进行常规核型分析(500~550条带)。
1.2.4 染色体微阵列分析(chromosome microarray analysis,CMA) 使用美国Affymetrix公司生产的CytoScan 750 k芯片和扩增、杂交试剂盒进行CMA检测。参照Infinium HD Assay标准操作流程进行操作,检测结果使用Chromosome Analysis Suite(Ch AS;version 2.1)软件进行分析。结果判读参照DGV、ISCA、OMIM、DECIPHER等数据库。
1.2.5 医学外显子组测序 取质检合格的1 μg基因组DNA于Q800R超声破碎仪中打断,电泳质检打断后DNA片段大小为500 bp以下,峰值在350 bp左右。参照KAPA library Preparation kit Illumina platforms条件及方法进行文库构建:在DNA扩增酶的作用下进行末端修复,3’端加“A”,并在两端加上特定序列的接头,不同样本连接不同序列的barcode,经PCR对带有接头的文库扩增。Qubit测定文库浓度,每例样本取300 ng进行混合。往混合后样本中加入基于NimbleGen SeqCap技术的定制探针(Roche NimbleGen,Madison,Wis),杂交 24 h后,采用 Streptavidin Dynabeads进行目标区域捕获,并将捕获后产物进行纯化,PCR富集目标区域基因。琼脂糖凝胶电泳测定文库大小,Qubit3.0及荧光定量PCR测定文库浓度。将质检合格的文库稀释至上机测序浓度,使用Illumina Nextseq 500测序仪进行测序分析。
1.2.6 捕获区域CNV分析 使用NextGene对测序原始数据进行转换,生成质控文件,对低质量的测序数据(样本编码序列平均覆盖度小于3×的数据)进行剔除。然后,通过均一化计算,分析出单(多)个外显子的拷贝数变化,方法依据Feng等[7]发表文献,简要如下:收集每个样本的单个外显子所有DNA单个碱基信号,得出单个外显子的平均覆盖深度,然后对比参考样本中特定外显子的覆盖深度,进而得到拷贝数变异数据。理论上,正常水平标注为1,即正常;缺失一个拷贝标注为0.5,即杂合缺失;缺失2个拷贝标注为0,即纯合/半合缺失。实际检测到的杂合缺失平均值为0.55±0.07。参考序列版本号:GRCh37/hg19,根据美国医学遗传学学会(American college of medical genetics,ACMG)指南[8],对 SNVs和 CNVs进行评估和致病性分类。
2 结果
2.1 家系1
2.1.1 脆性X染色体综合征基因检测 首先对患者1-II-2进行脆性X基因检测,结果为阴性(结果未显示)。
2.1.2 医学外显子组测序 考虑家系正常个体1-II-3妻子当时已妊娠20周,为节省检测时间,征得家属同意后,行核心家系临床医学外显子组(约4 000个已知致病基因)的综合检测和分析(1-I-1、1-I-2、1-II-2)。共检测50 585 个编码区,8 423 367 bp。平均覆盖深度344×±218,大于10×覆盖区间占99.1%,大于20×覆盖区间占98.8%。结果发现患者1-II-2 7q36.3区域有约3.14 Mb的杂合缺失片段(chr7:155 595 594-158 738 470;图3A),16p13.3区域有大约11.4 kb的杂合缺失片段(chr16:215997-227410;图3B),同时19p13.3区域有大约330.8 kb的重复片段(拷贝数为3)(chr19:589946-920748;图3C)。其中7q36.3区域3.14 Mb片段的杂合缺失和19p13.3区域330.8 kb片段的重复未在父母中检测到,16p13.3区域11.4 kb片段的杂合缺失遗传自母亲。
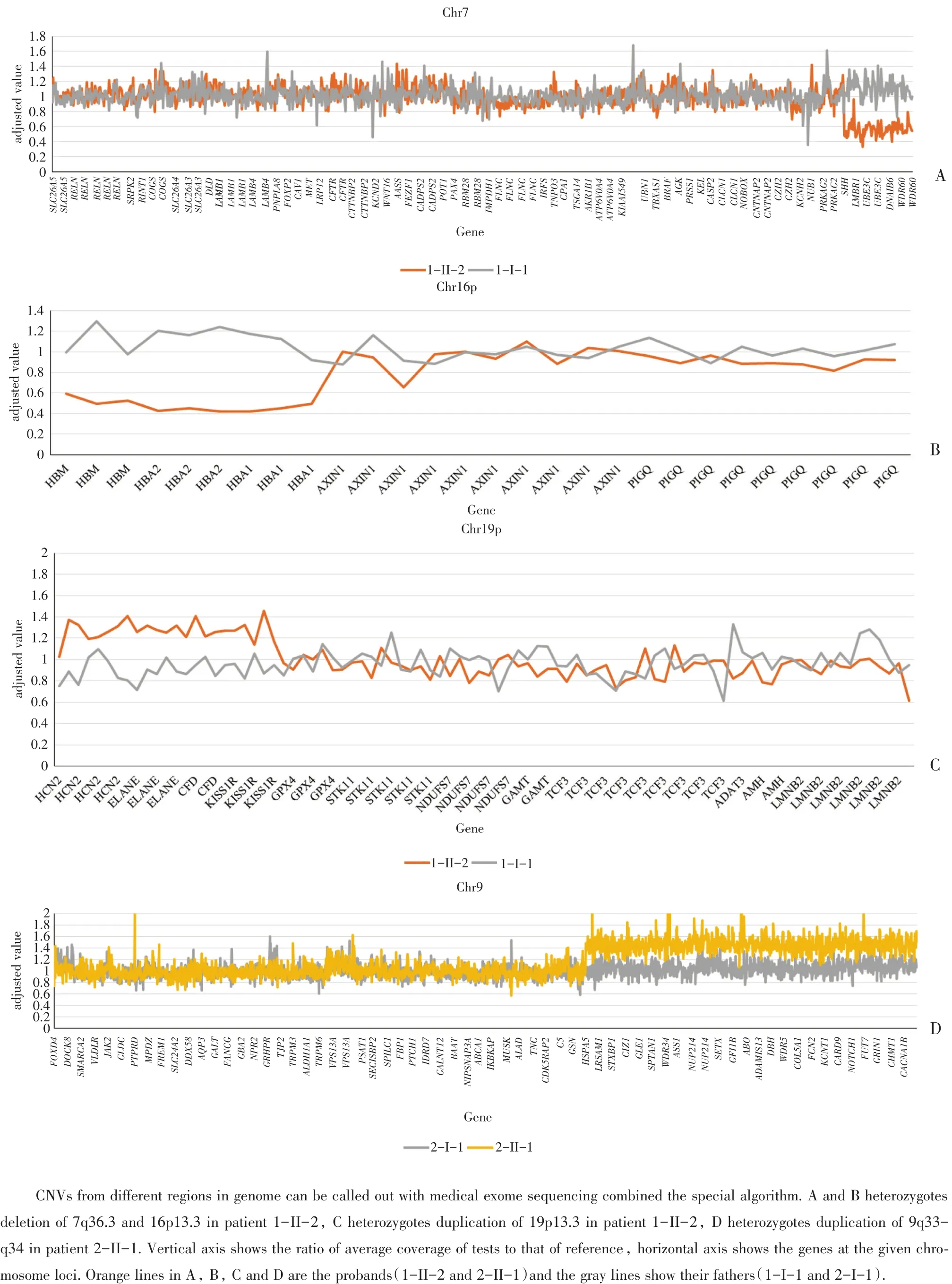
图3 利用NGS覆盖深度进行综合CNV分析Fig.3 CNV analysis data after medical exome sequencing

图4 家系2患者胞弟(2-II-2)外周血核型图Fig.4 Karyotype of 2-II-2
2.1.3 染色体微阵列分析 为明确家系患者病因,根据高通量测序结果,对患者1-II-1、正常个体1-II-3及其妻子腹中胎儿1-III-1同时进行了CMA检测。结果发现患者1-II-1存在7q36.2-q36.3区域约4.5 Mb的杂合缺失片段(chr7:154655991-159119707)合并19p13.3区域约841 kb的重复片段(chr19:260911-1102063);正常个体1-II-3及其胎儿未检测到明确异常。
2.2 家系2
2.2.1 医学外显子组测序 核心家系(2-I-1、2-I-2、2-II-1)进行临床医学外显子组(约4000个已知致病基因)综合检测和分析。共检测50585个编码区,8423367 bp。平均覆盖深度220×±127,大于10×覆盖区间占99.0%,大于20×覆盖区98.7%。经过CNVs分析(方法同2.1.2),发现患者2-II-1 9q33-q34区域有大约13.02 Mb的重复片段(拷贝数为 3)(chr9:127998871-141016451;图3D),父母未检测到明确异常。
2.2.2 染色体核型分析 为进一步明确家系遗传情况,对患者父母及弟弟进行外周血染色体核型分析,发现患者父亲2-I-1和弟弟2-II-2存在平衡易位:46,XY,t(3;9;q29;q33;图4,父亲核型未显示),患者母亲2-I-2核型未见异常。
3 讨论
CNV的检测大多通过染色体微阵列或高通量测序实现。本文的病例所涉及的染色体区域均依据GRCh37/hg19数据库。
家系1家族多发智障,两个患者均为男性,脆性X染色体基因(FMR1基因CGG串联重复)筛查阴性,患者1-II-2医学外显子组测序,发现三段CNV。其中16p13.3的11.4 kb缺失包含HBA2基因全长及HBM和HBA1基因的大部分,与α地中海贫血相关,同时该片段在患者母亲中也被检测到,该区域的缺失不是导致家系1患者智力低下的原因。由于与家系主要症状无关,未进行家系其他个体CNV验证。7q36.3的3.14 Mb缺失和19p13.3的330.8 kb重复则出现于两个病人中,患者1-II-2通过测序发现并在患者1-II-1通过CMA得到验证,1-II-3及其胎儿1-III-1均为阴性,两人均无症状(胎儿已出生,随访时4个月大,暂未发现明显异常)。7q36.3缺失区域含有5个OMIM基因,部分CNV缺失病例报道与本家系中患 者 症 状 类 似(nssv1604681,nssv706407,nssv1608164),且SHH和MNX1基因突变多表现为 显 性 遗 传(OMIM ID:600725,142994)[9-10];19p13.3区域(chr19:589946-920748)含有 3个OMIM基因,报道较少。根据临床表现及生物信息学分析,可以将7q36.3的缺失判定为疑似致病CNV,而19p13.3的重复则为意义不明。结合家系信息可推断,该家系中父母之一可能存在7号和19号染色体的平衡易位,需要进行后续荧光原位杂交(fluorescence in situ hybridization,FISH)验证。
家系2通过医学外显子组测序发现2-II-1带有9q33-q34重复,目前与9q33-q34区域重复相关的病例报道,描述的主要临床症状有智力障碍、并指、室间隔缺损、蜘蛛样指/趾等(DECIPHER ID:256749、269998)以及血液系统疾病或多系统畸形[11-12]。该家系患儿9q33-q34区域13.02Mb的重复片段,包含了LMX1B、LRSAM1、SPTAN1、COL5A1、NOTCH1、CARD9、EHMT1等OMIM致病基因。根据数据库现有资料、患儿临床表现及家系遗传学分析,提示9q33-q34区域13.02 Mb的重复片段很可能是导致该家系患者表型的原因,判定该重复片段为疑似致病CNV。结合家系信息推断,患者异常CNV来自父亲发生易位的3号染色体,而弟弟同时遗传了来自父亲的2条易位染色体,为平衡易位携带者,无临床症状。本家系患者未检测到3q29区域缺失,可能由于该区域不涉及OMIM致病基因,高通量测序未有提示,需进一步实验验证。
本文仅通过两个不相关的案例,在不同疾病、不同个体中通过相同的医学外显子组测序技术分别发现了位于不同染色体区段的CNVs,得到了其他技术手段,如核型分析和染色体微阵列等的验证,并初步分析了与疾病的相关性。初步得到医学外显子组测序技术用于临床罕见遗传病辅助诊断的证据,由于染色体微缺失微重复等拷贝数变异可以分布于基因组中任何位置,因此,该技术的临床应用效果评价仍然需要更进一步的大量数据和病例验证。
(致谢:感谢郭莉对本文图4的提供;感谢两个家系成员的信任和配合)

