辽河口芦苇湿地细菌和古菌群落周期日变化特征
李明月,米铁柱*,甄 毓,王勋功
辽河口芦苇湿地细菌和古菌群落周期日变化特征
李明月1,2,3,米铁柱1,2,3*,甄 毓1,2,3,王勋功2,3
(1.青岛海洋科学技术国家实验室海洋生态与环境科学功能实验室,山东 青岛 266071;2.海洋环境与生态教育部重点实验室,山东 青岛 266100;3.中国海洋大学环境科学与工程学院,山东 青岛 266100)
采用实时荧光定量PCR和Illumina高通量测序技术对秋季辽河口芦苇湿地水体中一昼夜细菌和古菌群落特征进行了研究.测序共获得52802条细菌和106091条古菌高质量序列,以97%的相似性聚类并抽平后共划分了530和979个OTU.结果表明,湿地水体中浮游细菌和古菌群落丰度均在早上达到最高值,夜晚达到最低值.细菌和古菌群落多样性指数均存在周期日变化,不同时间细菌和古菌群落结构和组成具有显著差异.此外,古菌群落组成特征表明辽河口芦苇湿地具有较高的潜在甲烷生成能力.本研究为进一步认识河口湿地细菌和古菌群落特征和功能奠定了基础.
湿地;16S rRNA;细菌;古菌;群落结构
湿地是位于陆地和水体的过渡地带,包括多种类型[1],其中河口湿地位于河流与海洋的交汇处,长期受到冲刷和断流等影响,处于不断的动态变化之中.河口湿地兼具淡水与海水生态系统的特点,各种生物地球化学过程交织,为大量生物提供了充足的栖息地[2].
微生物是河口湿地的重要组成部分,对河口湿地生态系统具有重要作用.研究表明,微生物群落及其多样性影响着湿地生态系统的特性和功能,是实现湿地生态功能、表征湿地环境变化的关键因素[3].细菌和古菌群落多样性、组成和结构与各种环境因子(例如温度、盐度、pH值、氮、硫、总有机碳、Fe、碳和氮的矿化、水体有机污染和富营养化等)[4-8]的交互作用,是其影响河口湿地生态系统的重要途径之一.对于河口湿地细菌和古菌群落的研究大多集中于湿地土壤和沉积物中[4-8],而对湿地积水中微生物的研究则相对较少.湿地积水中的微生物群落特征及其与环境因子关系的研究,有助于全面理解河口湿地生态系统的作用机制,可为河口湿地环境的原位修复和污染物微生物处理提供研究基础,这对河口湿地生态系统的可持续利用与综合管理,实现生态、经济、社会的协调发展具有重要意义.
辽河口湿地位于我国东北部,辽河入海口处,属于半湿润大陆季风性气候,其芦苇覆盖密度较大.同时,该区域具有丰富的天然气和石油资源[9].这里生长着许多物种,包括珍稀濒危物种,其生态系统的稳定具有重要意义.
目前,对辽河口湿地微生物群落的相关研究多集中于湿地土壤[10-12]或芦苇根际[13],且多采用传统分子生态学方法[10-11,13],如PCR-DGGE技术[10-11].本研究拟采用实时荧光定量PCR和Illumina Miseq高通量测序技术,结合环境因子的测定,分析辽河口芦苇湿地水体中细菌和古菌群落丰度、多样性、组成和结构的周期日变化规律.本研究将为辽河口湿地区域内生态系统保护与可持续发展提供基础资料和理论依据.
1 材料与方法
1.1 研究位点及样品采集
研究区域位于辽宁省盘锦市双台子河口湿地羊圈子苇场(图1).于2015年9月13日8:00、12:00和23:00采集300mL水样(S08、S12和S23)并依次用1μm和0.2μm聚碳酸酯滤膜(47mm, Whatman, UK)过滤,所有样品储存于-80℃,以备后续DNA提取实验.现场采集水样并测定温度(T)、pH值、盐度(Sal)和溶解氧(DO).用于测定溶解有机碳(DOC)、总氮(TN)、氨氮(NH4-N)、亚硝酸盐氮(NO2-N)、硝酸盐氮(NO3-N)、磷酸盐(PO4-P)和叶绿素a(Chl a)的水样,经过滤后带回实验室-20℃保存至分析.
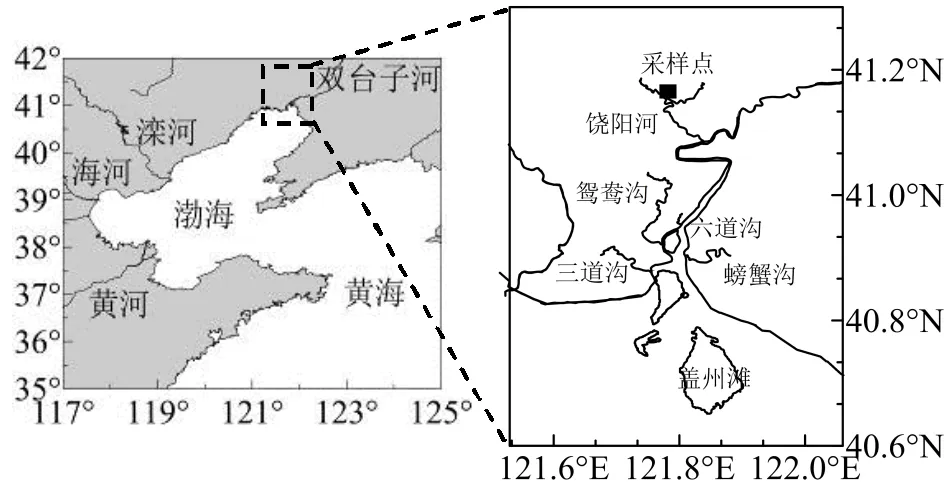
图1 辽河口芦苇湿地积水样品采样点
1.2 样品分析
1.2.1 环境参数的测定 积水样品的T、pH值、DO和Sal采用HQ40d (HACH, USA)现场采样测定.DOC、Chl a和营养盐的测定详见已有研究[14].
1.2.3 定量PCR 使用细菌16S rRNA基因引物338F和806R[15]和古菌16S rRNA基因引物U519F/806R[16],扩增16S rRNA基因.退火温度为58 ℃,扩增产物长度约为470bp和300bp.1%琼脂糖凝胶电泳分离PCR产物,纯化目的片段.pMD18-T载体连接纯化产物进行转化反应后,选取阳性克隆提取质粒.以梯度稀释质粒标准品作为模板,采用Applied Biosystems 7500 Real Time PCR System (Life Technologies, Forster City, CA, USA)进行qPCR.反应体系均为:10μL FastStart Universal SYBR Green Master (ROX)(Roche, 德国),0.6μL 10μM正向引物,0.6μL 10μM反向引物(表1),2μL模板DNA,6.6μL去离子水和0.2μL mg/mL牛血清蛋白.每个样品设置3个平行样,每组实验设置阴性对照.扩增条件均为:50℃ 2min;40个循环,95℃ 10min, 95 ℃ 15s,58℃ 2min.扩增完毕后分析熔解曲线,确保模板DNA的特异性扩增.以质粒拷贝数对数值为横坐标,平均Ct值为纵坐标,绘制细菌和古菌16S rRNA基因质粒标准曲线.
样品中细菌和古菌16S rRNA基因qPCR步骤与上述步骤相同,每个样品设置3个平行样,并添加阳性对照和阴性对照.

表1 细菌和古菌16S rRNA基因引物
1.2.4 高通量测序 以样品DNA为PCR反应模板,扩增细菌16S rRNA基因V3-V4区和古菌16S rRNA基因V4-V5区.细菌和古菌PCR反应引物分别为343F/798R[17]和Arch344F/Arch915R[18](表1).纯化后质量合格的PCR产物构建DNA文库,合格后使用Illumina Sequencer Miseq平台进行高通量测序.测序所得原始数据已上传NCBI数据库,序列号为SRP161823.
1.3 数据分析
利用Trimmomatic(v 0.35)[19]软件对高通量测序得到的双端序列(raw reads)去杂后,使用FLASH (v 1.2.11)[20]软件进行拼接.拼接后的序列使用QIIME (v 8.0)[21]软件进行降噪和过滤,将含有模糊碱基、单碱基高重复区或长度过短的序列去除(clean reads),保留75%以上的碱基质量高于Q20的序列.之后对clean reads进行去嵌合体序列处理,得到优质序列(valid reads)用于下游分析.使用VSERCH (v 2.4.2)[22]软件对所有优质序列以97%相似度进行OTU聚类划分,并用RDP classifier (v 2.2)[23]将代表序列与Silva (v 123)[24]数据库进行比对,得到系统发育树和OTU分类表格.保证足够测序深度的同时,对所有样品进行均一化处理,以避免不同数据量大小造成的分析偏差.细菌和古菌群落16S rRNA基因拷贝数根据qPCR结果和质粒标准曲线计算.利用QIIME软件计算Alpha多样性指数及稀释曲线相关点的抽取.
2 结果与分析
2.1 理化参数
湿地积水样品理化参数统计结果见表2.中午水样温度最高,夜晚水样温度最低,且DO、TN和NH4-N与温度具有同样的变化趋势,而NO2-N具有与温度相反的变化趋势.pH值和DOC在样品中的浓度在早上、夜晚和中午依次递减,Chl a则依次递增,Chl a在中午的浓度为46.16μg/L,远高于早晨和夜晚.

表2 辽河口芦苇湿地积水样品理化参数
2.2 细菌和古菌16S rRNA基因丰度

图2 辽河口芦苇湿地不同时间积水中细菌和古菌16S rRNA基因丰度
定量PCR结果表明,辽河口芦苇湿地积水中细菌和古菌16S rRNA基因丰度分别为3.30×104~ 7.62×107copy/L和8.40×106~6.56×107copy/L(图2).从8:00~23:00,细菌和古菌16S rRNA 基因丰度均呈现出降低趋势,且早上和中午细菌和古菌16S rRNA基因丰度远高于夜晚.总体来看,古菌16S rRNA 基因丰度略高于细菌,占细菌和古菌总16S rRNA 基因丰度的52.54%.
2.3 细菌和古菌群落多样性
辽河口芦苇湿地积水中不同时间样品通过高通量测序并质控后,共得到52802条高质量细菌序列.样品序列条数范围为15999~18502条,根据稀释曲线随机抽取15998条序列对所有样品做均一化处理(图3).以97%的相似性水平聚类,共得到530个OTU,OTU数目最多的时间点是早上8:00的水样,最低的是夜晚23:00的水样.早上、中午和夜晚共有203个OTU(图4),占总OTU数的38.30%.早上独有74个OTU,中午和夜晚则分别独有61和44个OTU,表明从早上到夜晚的过程中细菌种类随环境条件的改变而减少.早上与中午共有79个OTU,与夜晚共有34个OTU,而中午与夜晚共有35个OTU,可见早上与中午细菌种类差别较小,而夜晚与早上、中午细菌种类差别较大.
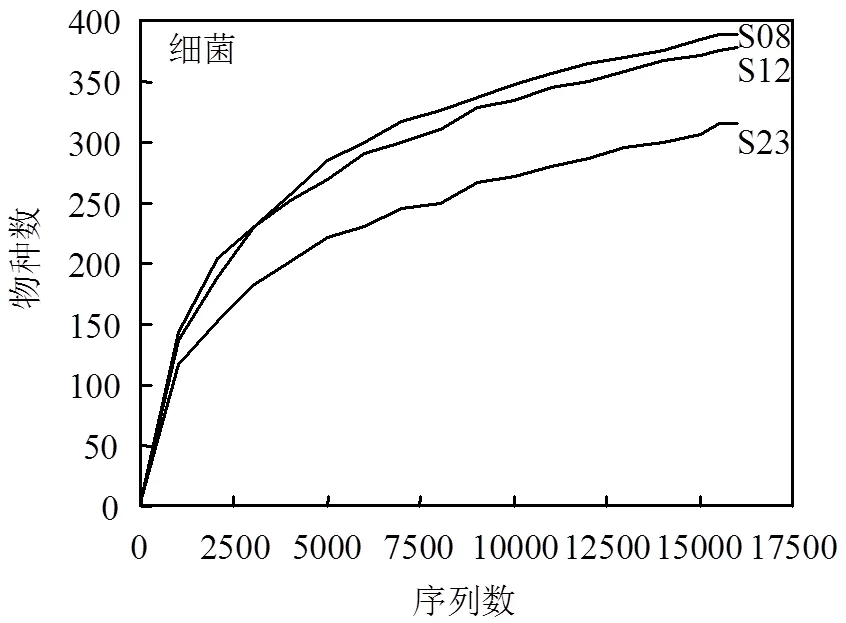
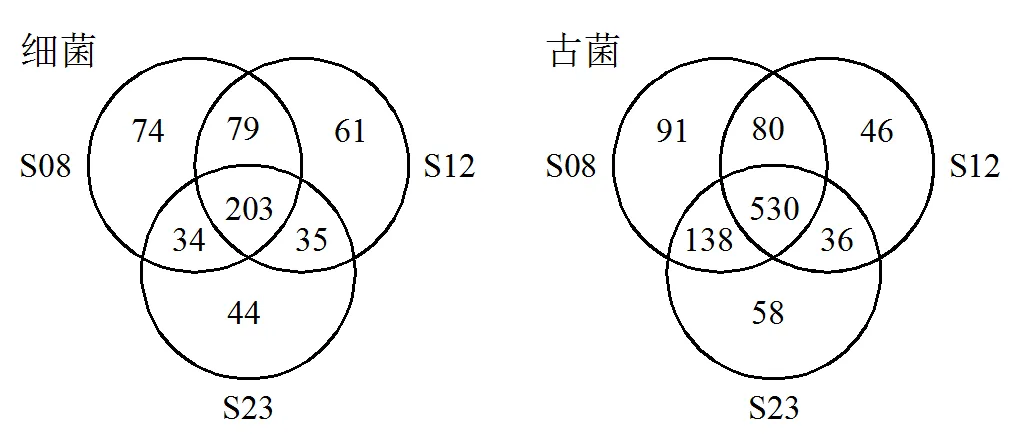
图4 辽河口芦苇湿地不同时间积水中细菌和古菌OTU韦恩
所有水样中细菌测序覆盖度均在99.36%以上,测序深度能够基本覆盖全部物种(表3).Shannon指数表明早上细菌群落多样性最高,夜晚最低,与NO3-N具有相反的变化趋势.而Chao 1指数表明中午细菌群落丰富度略高于早上,夜晚则远低于早上和中午,其变化趋势与T、DO、TN和NH4-N一致,与NO2-N相反.此外,早上水样中的细菌群落均匀度最高(Pielou指数).
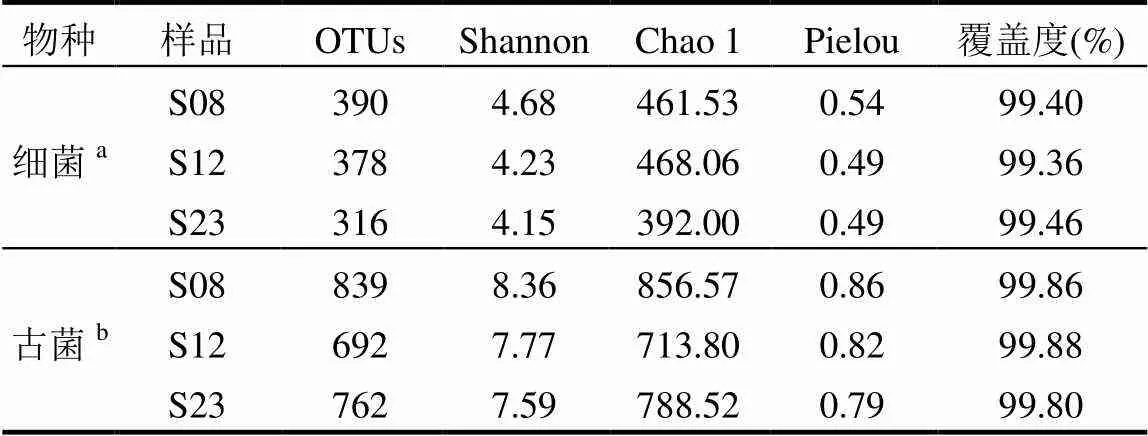
表3 辽河口芦苇湿地不同时间积水中细菌和古菌的α-多样性指数
注:a序列条数抽平为15998,b序列条数抽平为29352.
不同时间积水中古菌测序并质控后,共得到106091条高质量序列,平均序列条数为35363±5708.对所有样品抽平后序列数量足够,测序深度选取比较合理(29352条序列,图3),共得到979个OTU(97%的相似性聚类).早上8:00OTU数量最高,中午12:00OTU数量最低,分别为839和692个,表明古菌群落对环境的适应性与细菌不同.3个时间点共有530个OTU(图4),占所有OTU数的54.14%.早上独有91个OTU,中午独有46个OTU,夜晚独有58个OTU,表明从早上到夜晚的过程中古菌种类随环境条件的改变先减少后升高.早上与中午共有80个OTU,与夜晚共有138个OTU,而中午与夜晚共有36个OTU,可见早上与夜晚古菌种类差别较小,而中午与两者古菌种类差别较大.
古菌测序覆盖度在所有样品中均在99.80%以上,测序深度能够基本覆盖全部物种(表3).早上、中午和夜晚古菌群落多样性和均匀度逐渐降低,与NO3-N具有相反的变化趋势,而丰富度最高值出现在早上,最低值出现在中午,变化趋势与DOC相同,与Chl a相反.
2.4 细菌和古菌群落的组成
通过与Silva (v123)数据库进行比对,对OTU进行物种注释和分类.结果表明,99.92%的细菌序列被注释到门分类水平,31.34%的细菌序列被注释到属水平(图5),河口湿地还蕴含着大量的未知细菌资源.分别在门、纲、目、科、属水平上对相对丰度较高的细菌类群做相对丰度图(图6).在门分类水平上(图6),相对丰度大于1%的优势门[25]包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes).其中,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)的相对丰度总和为96.60%~97.65%.从早上~夜晚,变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和Saccharibacteria相对丰度逐渐增大,而拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia)相对丰度逐渐减少,且早上变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)相对丰度与中午和夜晚差别较大.在纲、目和科水平上(图6),优势细菌菌群具有和门水平相似的时间变化规律,例如α变形菌纲(Alphaproteobacteria)相对丰度从早上到夜晚逐渐增大,Flavobacteriia相对丰度逐渐减少,且两者在早上的相对丰度与中午和夜晚差别较大.在属分类水平上,早上、中午和夜晚积水样品中细菌群落组成差别较大(图6).早上水样中相对丰度大于1%的优势菌属[25]为(相对丰度8.65%)、黄杆菌属(,3.84%)、(1.81%)和(1.66%),中午为嗜甲基菌属(,2.18%)、(1.34%)、(1.27%)和(1.18%),夜晚为莱茵海默氏菌(, 5.66%)、(2.31%)、交替赤杆菌属(,2.16%)、交替单胞菌(,1.69%)、(1.51%)、(1.36%)和(1.14%).

图5 辽河口芦苇湿地不同时间积水中细菌和古菌注释序列条数占总条数的百分比
Fig.5 The proportion of annotated sequences in all sequences of bacterial and archaeal communities in the reed wetland waters of Liaohe estuary
所有样品共有95.80%古菌序列注释到门水平,23.95%注释到属水平,河口湿地蕴含的未知古菌资源更为丰富(图5).在门分类水平上,所有序列一共注释了9个古菌门,其中Woesearchaeota_ (DHVEG_6)、广古菌门(Euryarchaeota)和Miscellaneous_Euryarchaeotic_Group(MEG)占所有菌群的93.80~94.72%.早上和中午Woesearchaeota_ (DHVEG_6)相对丰度高于夜晚,广古菌门(Euryarchaeota)则低于夜晚.热源体纲(Thermoplasmata)、甲烷杆菌纲(Methanobacteria)和甲烷微菌纲(Methanomicrobia)为主要古菌纲类群(图7),20a_9、热原体目(Thermoplasmatales)、甲烷杆菌目(Methanobacteriales)、Methanocellales和球菌目(Methanosarcinales)为主要古菌目类群(图7),甲烷杆菌科(Methanobacteriaceae)、Methanocellaceae和球菌科(Methanosarcinaceae)为主要古菌科类群(图7),甲烷细菌属()、和甲烷八叠球菌属()为主要古菌属类群(图7d).辽河口湿地积水中检出了7个产甲烷古菌目中的5个,即热原体目、甲烷杆菌目、甲烷胞菌目、甲烷八叠球菌目和甲烷微菌目,且具有较高的相对丰度.样品中古菌群落组成特征表明了辽河口芦苇湿地活跃的甲烷相关生物地球化学过程.
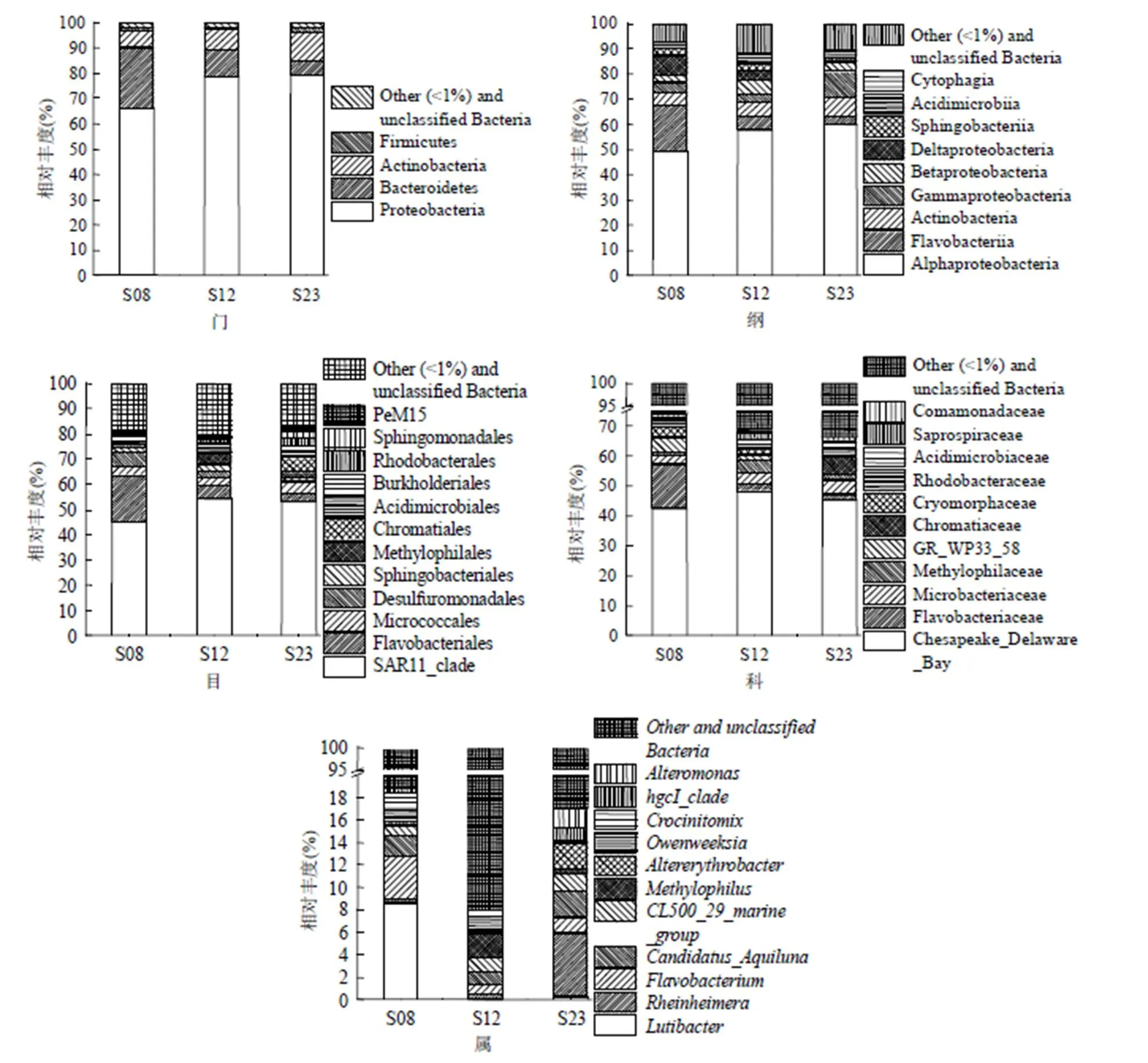
图6 辽河口芦苇湿地不同时间积水中细菌门、纲、目、科和属水平相对丰度

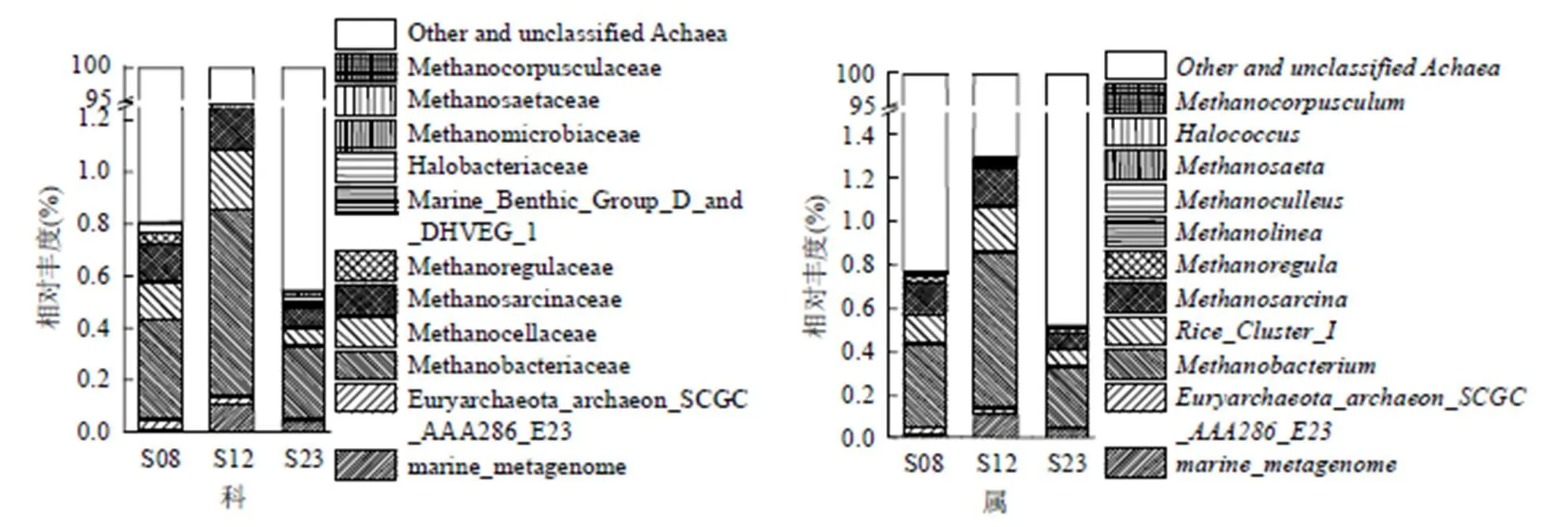
图7 辽河口芦苇湿地不同时间积水中古菌纲、目、科、属水平相对丰度
2.5 细菌和古菌群落结构
挑选细菌和古菌相对丰度最高的前30个OTU做OTU丰度热图(图8).不同时间积水样品中细菌和古菌群落结构明显不同,且中午和夜晚细菌群落结构较为相似,聚为一类,早上和夜晚古菌群落结构较为相似,聚为一类.此外,早上、中午和夜晚水样中细菌优势OTU明显各不相同,古菌优势OTU也有一定的差异,表明不同时间点辽河口芦苇湿地细菌群落结构有一定的差异.从属分类水平挑选丰度最高的前15个物种做物种丰度热图(图8),细菌和古菌群落均是早上和中午聚为一类.不同时间点细菌和古菌优势种群具有明显差异,例如,早上古菌优势属为、甲烷粒菌属()和盐球菌属()等,中午为甲烷杆菌属()等,而夜晚为甲烷囊菌属()等.

图8 辽河口芦苇湿地不同时间积水中细菌OTU、古菌OTU、细菌属和古菌属水平丰度热图
3 讨论
采用qPCR技术和Illumina高通量测序技术,结合环境因子,对辽河口芦苇湿地羊圈子苇场内积水中细菌和古菌群落的丰度、多样性、组成和结构进行了分析研究.结果表明,研究区域内营养盐浓度整体低于辽河口芦苇湿地河蟹养殖区[26]和其他区域[27],且远低于其他湿地水体[28].DOC含量为13m/L左右,与其他湿地相比略低[29]. Chl a表现出显著的周期日变化特征,与已有报道一致,最高值出现在正午前后[30].
辽河口芦苇湿地中细菌丰度与湖泊湿地浮游细菌丰度相当,但低于其他水体环境[31-32](细胞个数与16S rRNA基因拷贝数进行3.45倍换算[33]).而古菌丰度与其他水体环境相当[34],且古菌丰度高于细菌,这与其他研究结果不同[35].本研究位点中细菌和古菌丰度均存在周期日变化特征(图2),在温度最低的夜晚,丰度最低,且显著低于其他时间点.有研究表明湿地浮游细菌丰度与温度和Chl a等具有相关性[36],异养细菌丰度周期日变化与微微型真核浮游植物之间具有相关性,且受到浊度的影响[37].因此,白天水温升高刺激微生物生长繁殖加速,同时光合作用增强引起的湿地植物生长繁殖加快间接促进微生物的生长,都是细菌和古菌丰度在早上出现最高值的原因[38].但是,中午细菌和古菌丰度略低于早上,这则与中午的强光照和紫外线辐射有密切的联系[30].本研究中细菌和古菌丰度的周期日变化趋势与Chl a变化趋势不一致,其丰度可能受到温度、Chl a和早晚透光度等共同作用的影响.
细菌和古菌群落Alpha多样性分析包括Shannon指数、Chao 1指数和Pielou指数,分别代表多样性、丰富度和均匀度.辽河口芦苇湿地积水中细菌和古菌群落多样性和丰富度都很高(表3),且古菌群落多样性、丰富度和均匀度均高于细菌,这与16S rRNA基因丰度定量结果一致.此外,辽河口湿地积水中细菌和古菌群落生物多样性均具有周期日变化特征,整体表现为早上生物多样性较高,夜晚较低. 由于对湿地水体中微生物群落的研究较少,具体原因目前还无法判读,但有报道发现海水中细菌在白天的多样性高于夜晚,具有明显的昼夜之分[30]. Zhang等[39]发现近岸海域中浮游细菌的丰度、生长和多样性受到病毒和鞭毛虫的共同作用影响,而Christian等[40]指出病毒性的细菌裂解通常发生在中午~下午,且大都在夜间发生感染,导致夜间细菌数量减少,多样性也随之降低.
细菌主要优势菌群为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria),此结论与其他湿地浮游细菌优势菌群一致[41-45].辽河口芦苇湿地积水中检测到了相对丰度较高的海洋菌属,说明湿地系统可能受到海水的影响.然而,氮代谢相关的菌属相对丰度较小,可能是因为该湿地系统氮代谢不活跃.研究表明,辽河口湿地典型的寒冷气候会大幅度影响参与氨转化的酶的活性,从而进一步影响硝化效率[46],且低温和高盐度是辽河口硝化作用的主要限制因子[47].
关于湿地水体中古菌群落组成的研究较少,湿地土壤或沉积物中古菌优势种群为Euyarchaeota和Thaumarchaeota等[48-49],与本研究湿地水体中主要种群存在一定的差异.湿地是大气甲烷最大的自然源,辽河口湿地积水中古菌群落组成以产甲烷菌为主,其中极端厌氧甲烷杆菌属和甲烷八叠菌属等厌氧菌有较高的相对丰度.但是辽河口湿地积水中DO较为丰富,且有研究发现在芦苇湿地土壤中产甲烷菌仅存在于地表下10cm深处[12],这说明湿地积水与沉积物之间可能具有活跃的交换作用.
序列分析表明,不同时间点积水样品中细菌和古菌群落组成和结构表现出了明显的时间分布差异.变形菌门和放线菌门昼夜变化表现出相反的趋势,在海水中也有同样的发现[30].-变形菌纲相对丰度在夜间明显高于白天.有研究发现,由于该类群的光捕获色素基因在光照下激发细胞分裂,其数量在白天明显高于夜间[50].甲烷八叠球菌属在中午的相对丰度最高、夜晚最低,表明湿地中甲基营养型产甲烷途径在中午最为活跃[51].
微生物作用是湿地系统生态功能的重要组成部分,生态环境的改变与微生物群落的变化息息相关[52].研究区域位于羊圈子苇场芦苇覆盖密集区,根据芦苇不同生长期的需求,积水每年秋季排入辽河.积水微生物群落周期日特征主要受昼夜变化的影响,无潮汐等其他因素的影响.微生物群落丰度、多样性等均在夜晚较低,若能在夜间排水,芦苇湿地微生物资源损失较小,芦苇湿地功能可维持相对稳定的状态.而且,积水中微生物群落对辽河微生物群落也会有较小的冲击.辽河口芦苇湿地位于重要的工业区和农业区,受人类活动影响较大,微生物群落组成及其多样性等特性随之受到影响.对辽河口芦苇湿地微生物群落的研究不仅有利于了解微生物群落整体特征,而且有助于发现功能微生物,为辽河口污染物降解和湿地管理等提供理论基础.因此,进一步考察辽河口芦苇湿地中微生物群落的时空分布特征具有重要意义.由于受各种条件的限制,我们只进行了一个采样点不同时段的一次采样,对湿地积水中微生物群落的周期日变化进行了初步探讨.在后期研究中,需要进一步在不同采样站位进行采样,以获得更为全面的微生物群落信息.
4 结论
4.1 对辽河口芦苇湿地一昼夜细菌和古菌群落的实时荧光定量PCR和Illumina高通量测序分析结果表明,细菌和古菌群落丰度、多样性、组成和结构具有显著的周期日变化.早上和中午积水中细菌和古菌群落丰度差异较小,且远高于夜间.早上细菌和古菌多样性最高,夜晚最低.
4.2 古菌群落组成特征表明辽河口芦苇湿地具有较高的潜在甲烷生成能力.
[1] 杨朝飞.中国湿地现状及其保护对策 [J]. 中国环境科学, 1995, 15(6):407-412.Yang C F. Current status and conservation strategy of China’s wetland [J]. China Environmental Science, 1995,15(6):407-412.
[2] 黄桂林,何 平,侯 盟.中国河口湿地研究现状及展望 [J]. 应用生态学报, 2006,17(9):1751-1756. Huang G L, He P, Hou M. Present status and prospects of estuarine wetland research in China [J]. Chinese Journal of Applied Ecology, 2006,17(9):1751-1756.
[3] 车玉伶,王 慧,胡洪营,等.微生物群落结构和多样性解析技术研究进展 [J]. 生态环境学报, 2005,14(1):127-133. Che Y L, Wang H, Hu H Y, et al. Research progresses on analytical technologies used in microbial community structure and diversity [J]. Ecology and Environment, 2005,14(1):127-133.
[4] Franklin R B, Morrissey E Mand Morina J C. Changes in abundance and community structure of nitrate-reducing bacteria along a salinity gradient in tidal wetlands [J]. Pedobiologia, 2017,60:21-26.
[5] Zhang H, Zheng S, Ding J, et al. Spatial variation in bacterial community in natural wetland-river-sea ecosystems [J]. Journal of Basic Microbiology, 2017,57(6):536-546.
[6] Li X, Hou L, Liu M, et al. Primary effects of extracellular enzyme activity and microbial community on carbon and nitrogen mineralization in estuarine and tidal wetlands [J]. Applied Microbiology & Biotechnology, 2015,99(6):2895-2909.
[7] Fei X X, Wang L, Jun H J, et al. Salinity influence on soil microbial respiration rate of wetland in the Yangtze River estuary through changing microbial community [J]. Journal of Environmental Science, 2014,26(12):2562-2570.
[8] Zhang Y, Wang L, Hu Y, et al. Water Organic Pollution and Eutrophication Influence Soil Microbial Processes, Increasing Soil Respiration of Estuarine Wetlands: Site Study in Jiuduansha Wetland [J]. Plos One, 2015,10(5):e0126951.
[9] Lin T, Ye S, Ma C, et al. Sources and preservation of organic matter in soils of the wetlands in the Liaohe (Liao River) Delta, North China [J]. Marine Pollution Bulletin, 2013,71(1/2):276-285.
[10] 关晓燕,韩家波,王 摆,等.辽东湾大凌河口湿地土壤微生物群落分析 [J]. 生态环境学报, 2012,21(6):1063-1070. Guan X Y, Han J P, Wang B, et al. Analysis of bacterial communities in Liaodong Bay Dalinghe estuarine wetland [J]. Ecology and Environmental Sciences, 2012,21(6):1063-1070.
[11] 赵先丽,周广胜,周 莉,等.盘锦芦苇湿地土壤微生物数量研究 [J]. 土壤通报, 2008,39(6):1376-1379.Zhao X L, Zhou G S, Zhou L, et al. Characteristics of soil microbial community in bulrush wetlands of Panjin, Northeast China [J]. Chinese Journal of Soil Science, 2008,39(6):1376-1379.
[12] 鲁青原.辽河三角洲滨海湿地微生物群落组成及其环境意义 [D]. 北京:中国地质大学, 2016. Lu Q Y. The microbial community structure in Liaohe Delta wetlands and its environmental significances [D]. Beijing: China University of Geosciences, 2016.
[13] 田伟君,王勇梅,孙会梅,等.石油输入对河口芦苇湿地根际微生物的影响 [J]. 中国环境科学, 2014,34(10):2676-2683. Tian W J, Wang Y M, Sun H M, et al. Effect of oil pollution on community structure of plants rhizospheric microorganisms in estuarine wetland [J]. China Environmental Science, 2014,34(10): 2676-2683.
[14] 李 璐,邹 立,孟泰舟,等.辽河口芦苇湿地有色溶解有机物的光谱特征研究 [J]. 中国海洋大学学报:自然科学版, 2017,(12):27-36. Li L, Zou L, Meng T Z, et al. Absorbance and fluorescence spectrums of chromophoric dissolved organic matter in the reed wetland of Liaohe estuary [J]. Periodical of Ocean University of China, 2017, (12):27-36.
[15] Peiffer J A, Spor A, Koren O, et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions [J]. Proceedings of the National Academy of Sciences, 2013,110(16):6548-6553.
[16] Porat I, Vishnivetskaya T A, Mosher J J, et al. Characterization of archaeal community in contaminated and uncontaminated surface stream sediments [J]. Microbial ecology, 2010,60(4):784-795.
[17] Carlos, Nossa, William, et al. Design of 16S rRNA gene primers for 454pyrosequencing of the human foregut microbiome [J]. World Journal of Gastroenterology, 2010,16(33):4135-4144.
[18] Ohene-Adjei S, Teather R M, Ivan M, et al. Postinoculation protozoan establishment and association patterns of methanogenic archaea in the ovine rumen [J]. Applied and Environmental Microbiology, 2007, 73(14):4609-4618.
[19] Bolger A M, Lohse Mand Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data [J]. Bioinformatics, 2014,30(15):2114- 2120.
[20] Reyon D, Tsai S Q, Khayter C, et al. FLASH assembly of TALENs for high-throughput genome editing [J]. Nature Biotechnology, 2012, 30(5):460-465.
[21] Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data [J]. Nature Methods, 2010,7(5):335-336.
[22] Rognes T, Flouri T, Nichols B, et al. VSEARCH: a versatile open source tool for metagenomics [J]. Peerj, 2016,4(10):1-22.
[23] Wang Q, Garrity G M, Tiedje J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy [J]. Applied & Environmental Microbiology, 2007,73(16):5261-5267.
[24] Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools [J]. Nucleic Acids Research, 2013,41(Database issue):590-596.
[25] Galand P E, Casamayor E O, Kirchman D L, et al. Ecology of the rare microbial biosphere of the Arctic Ocean [J]. Proceedings of the National Academy of Sciences, 2009,106(52):22427-22432.
[26] 罗西玲,邢 磊,徐宾铎,等.辽河口芦苇湿地河蟹养殖区水体N、P营养盐和COD的变化 [J]. 海洋湖沼通报, 2016,(3):20-27. Luo X L, Xing L, Xu B Y, et al. Variations in nitrogen and phosphorous nutrients and COD in the river crab aquaculture areas in reed wetland of Liaohe estuary [J]. Transactions of Oceanology and Limnology, 2016,(3):20-27.
[27] 徐微雪,段 亮,宋永会,等.辽河保护区七星湿地表层水与间隙水中氮的时空分布 [J]. 环境工程技术学报, 2014,4(1):40-45. Xu W X, Duan L, Song Y H, et al. Spatio-temporal distributions of nitrogen in surface water and pore water in Qixing Wetland of Liaohe conservation area [J]. Journal of Environmental Engineering Technology, 2014,4(1):40-45.
[28] 吴 明,邵学新,蒋科毅.西溪国家湿地公园水体和底泥N、P营养盐分布特征及评价 [J]. 林业科学研究, 2008,21(4):587-591. Wu M, Shao X X, Jiang K Y. Characteristics and assessment on nutrient distribution in water and sediments of Xixi National Wetland Park in Hangzhou [J]. Forest Research, 2008,21(4):587-591.
[29] 唐玉姝,王 磊,席雪飞,等.九段沙湿地潮间带水域有机碳与无机碳含量的空间分布特征 [J]. 农业环境科学学报, 2012,31(12):2459- 2465. Tang Y S, Wang L, Xi X F, et al. The spatial distribution characteristics of organic and inorganic carbon content of intertidal water in Jiuduansha Wetland [J]. Journal of Agro-Environment Science, 2012, 31(12):2459-2465.
[30] 刘晓辉.长江口及邻近海域海水细菌多样性及时空分布规律研究 [D]. 舟山:浙江海洋大学, 2017. Liu X H. Temporal and spatial distribution of bacterial diversity in seawater of Changjiang estuary and adjacent areas [D]. Zhoushan: Zhejiang Ocean University, 2017.
[31] 宋玉芝,赵淑颖,黄 瑾,等.太湖水体附着细菌和浮游细菌的丰度与分布特征 [J]. 环境工程学报, 2013,7(8):2825-2831. Song Y Z, Zhao S Y, Huang J, et al. Abundance and distribution of particle-attached bacteria and free-living bacteria in Lake Taihu [J]. Chinese Journal of Environmental Engineering, 2013,7(8):2825-2831.
[32] Wang L, Zhang J, Li H, et al. Shift in the microbial community composition of surface water and sediment along an urban river [J]. Science of the Total Environment, 2018,627:600-612.
[33] Li J, Zhou H, Fang J, et al. Microbial distribution in different spatial positions within the walls of a black sulfide hydrothermal chimney [J]. Marine Ecology Progress Series, 2014,508:67-85.
[34] Yang Y, Dai Y, Wu Z, et al. Temporal and spatial dynamics of archaeal communities in two freshwater lakes at different trophic status [J]. Frontiers in Microbiology, 2016,7:451.
[35] Hu A, Hou Land Yu C-P. Biogeography of planktonic and benthic archaeal communities in a subtropical eutrophic estuary of China [J]. Microbial Ecology, 2015,70(2):322-335.
[36] 崔尹赡,李 珊,魏云林,等.纳帕海湿地浮游细菌的丰度及可培养低温细菌多样性研究 [J]. 上海环境科学, 2017,36(3):93-138.Cui Y S, Li S, Wei Y L, et al. Studies on the abundance of bacterioplankton and diversity of culturable low-temperature bacteria in Napahai Wetland [J]. Shanghai Environmental Science, 2017,36(3): 93-138.
[37] 李 云,李道季,张利华,等.长江口极微型和微微型浮游生物的垂向变化与周日波动 [J]. 海洋科学, 2011,35(9):24-30. Li Y, Li D J, Zhang L H, et al. Vertical characteristics and diel variations of femtoplankton and picoplankton at the Changjiang estuary [J]. Marine Science, 2011,35(9):24-30.
[38] Barbosa A B, Galva˜o H M, Mendes P A, et al. Short-term variability of heterotrophic bacterioplankton during upwelling off the NW Iberian margin [J]. Progress in Oceanography, 2001,51:339-359.
[39] Zhang R, Weinbauer M G, Qian P Y. Viruses and flagellates sustain apparent richness and reduce biomass accumulation of bacterioplankton in coastal marine waters [J]. Environmental Microbiology, 2007,9(12):3008-3018.
[40] Winter C, Herndl G J, Weinbauer M G. Diel cycles in viral infection of bacterioplankton in the North Sea [J]. Aquatic Microbial Ecology, 2004,35:207-216.
[41] Zhang J, Zhang X, Liu Y, et al. Bacterioplankton communities in a high-altitude freshwater wetland [J]. Annals of Microbiology, 2014, 64(3):1405-1411.
[42] Elsayed O, Maillard E, Vuilleumier S, et al. Bacterial communities in batch and continuous-flow wetlands treating the herbicide S-metolachlor [J]. Science of the Total Environment, 2014,499:327- 335.
[43] Zhang L, Gao G, Tang X, et al. Bacterial community changes along a salinity gradient in a Chinese wetland [J]. Canadian Journal of Microbiology, 2013,59(9):611-619.
[44] Baik K S, Park S C, Kim E M, et al. Diversity of bacterial community in freshwater of Woopo wetland [J]. The Journal of Microbiology, 2008,46(6):647-655.
[45] Buesing N, Filippini M, Bürgmann H, et al. Microbial communities in contrasting freshwater marsh microhabitats [J]. FEMS Microbiology Ecology, 2009,69(1):84-97.
[46] Wan T, Zhang G, Du F, et al. Combined biologic aerated filter and sulfur/ceramisite autotrophic denitrification for advanced wastewater nitrogen removal at low temperatures [J]. Frontiers of Environmental Science & Engineering, 2014,8(6):967-972.
[47] Huang X, Bai J, Li K R, et al. Characteristics of two novel cold- and salt-tolerant ammonia-oxidizing bacteria from Liaohe Estuarine Wetland [J]. Marine Pollution Bulletin, 2017,114(1):192-200.
[48] Li B, Chen H, Li N, et al. Spatio-temporal shifts in the archaeal community of a constructed wetland treating river water [J]. Science of the Total Environment, 2017,605:269-275.
[49] Long Y, Yi H, Chen S, et al. Influences of plant type on bacterial and archaeal communities in constructed wetland treating polluted river water [J]. Environmental Science and Pollution Research, 2016,23(19): 19570-19579.
[50] Giovannoni S J, Stinql U. Molecular diversity and ecology of microbial plankton [J]. Nature, 2005,437(7057):343-348.
[51] Liu Y, Whitman W B. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea [J]. Annals of the New York Academy of Sciences, 2008,1125(1):171-189.
[52] 张 健,赵阳国,李海艳,等.黄海西北近岸沉积物中细菌群落空间分布特征 [J]. 海洋学报, 2010,32(2):118-127.Zhang J, Zhao Y G, Li H Y, et al. Temporal and spatial distribution characterization of bacterial community in sediments from the inshore of the northwest Huanghai Sea [J]. Acta Oceanologica Sinica, 2010, 32(2):118-127.
致谢:感谢中国海洋大学环境科学与工程学院邹立副教授、李璐同学在样品采集和环境因子分析中提供的大力支持与辛勤工作.
Diurnal variations of bacterial and archaeal communities in the reed wetland of Liaohe estuary.
LI Ming-yue1,2,3, MI Tie-zhu1,2,3*, ZHEN Yu1,2,3, WANG Xun-gong2,3
(1.Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;2.Key Laboratory of Marine Environment and Ecology, Ministry of Education, Ocean University of China, Qingdao 266100, China;3.College of Environmental Science and Engineering, Ocean University of China, Qingdao 266100, China)., 2019,39(2):849~858
The present study investigated the diurnal variations of bacterial and archaeal communities in Liaohe estuarine wetland in autumn, using the real-time fluorescence quantitative PCR method and Illumina high-throughput sequencing. The results showed that 52802 bacterial and 106091 archaeal sequences were obtained, which could be classified into 530 and 979 OTUs in 97% similarity level, respectively. The results showed that bacterial and archaeal 16S rRNA gene abundances both had the highest values in the morning, and the lowest ones at midnight. There were diurnal variations in the bacterial and archaeal community α-diversity indices, and obvious temporal changes were also observed in the bacterial and archaeal community compositions and structures. In addition, Liaohe estuarine wetland had high potential ability of methanogenesis which could be reflected by the composition of archaeal community. This study provides the basic information and knowledge to further understand the characteristics and functions of bacterial and archaeal communities in estuarine wetlands.
wetland;16S rRNA;bacteria;archaea;community structure
X172
A
1000-6923(2019)02-0849-10
李明月(1990-),女,河南周口人,博士,主要从事海洋生态学研究.发表论文4篇.
2018-06-21
国家重点研发计划课题(2017YFC1404402);鳌山科技创新计划项目(2016ASKJ02);国家自然科学基金资助项目(41620104001)
* 责任作者, 副教授, mitiezhu@ouc.edu.cn

