近红外特征光谱定量检测羊肉卷中猪肉掺假比例
白 京,李家鹏,邹 昊,田寒友,刘 飞,王 辉,李文采,张振琪,王守伟
(中国肉类食品综合研究中心,北京食品科学研究院,肉类加工技术北京市重点实验室,北京 100068)
羊肉作为一种食药两用、营养丰富的肉类食品[1],深受消费者的喜爱。羊肉卷作为火锅等的必备食材,市场需求量极大。但近年来,随着肉品加工业的迅速发展,受羊肉卷冷冻的制作过程启发,很多商人受巨大利益驱使,使用鸭肉、猪肉等低价位品种肉进行掺假,靠羊尾等羊肥肉的膻味以假乱真,对消费者造成严重利益损失,构成严重食品健康隐患[2]。而由于肉品种类繁多,成分复杂,掺杂物质种类很多,外观成分组成性质比较接近,传统的掺假化学检测一般通过化学分析、仪器分析、感官评定等时间较长的检测手段来完成,且操作步骤较为复杂,不能满足大批量快速检测的需求,因此快速鉴别检测掺假肉具有十分重要的意义[3-5]。
近红外光谱作为一种快速检测手段,分析速度快、操作简单并且效率较高[6-8]。该技术主要在肉类方面的应用有:肉品品质的多指标综合评价[9-12],肉类化学成分的定量分析[13-14],肉品产地[15]、部位[16]、掺假[17-19]等方面的鉴定。如牛晓颖等[20]比较了马氏距离判别分析、簇类独立软模式分类法、最小二乘-支持向量机方法分别结合平滑(5、15 点及25 点)、一阶和二阶微分、多元散射校正(multiplicative scatter correction,MSC)和标准归一化的光谱预处理方法对肉块样品及大中小3 个不同粉碎粒径建立近红外光谱鉴别驴肉模型,不同粉碎粒径的肉类样品分类中,驴肉样品均得到了100%的判别正确率。但目前针对肉的掺假问题,快速定量检测研究较少,且研究对象主要为鲜瘦肉,无法应用于鲜瘦肉和肥肉相间的羊肉卷快速定量检测。本研究为填补现有研究的空白,以不同肥肉占比的猪肉掺假解冻羊肉卷为检测对象,在进行简单前处理情况下,采集样品近红外光谱信息,选取合适的预处理方法建立全波段偏最小二乘回归(partial least square regression,PLSR)模型,并基于竞争性自适应加权采样(competitive adaptive reweighted sampling,CARS)算法提取特征波长建立CARS-PLSR模型,进一步简化预测模型,提出一种快速检测不同肥肉占比的解冻羊肉卷中猪肉掺假比例的定量分析方法,为掺假羊肉卷的定量检测提供技术支持。
1 材料与方法
1.1 材料与仪器
肉样品均为冷鲜肉,按照市场羊肉卷制作实际情况,选择以下样品:猪瘦肉(北京永辉商业有限公司),品种为北京三元猪;羊瘦肉(羊腿肉)、羊肥肉(羊尾)均购自北京穆森伟业清真食品有限责任公司,品种为北京土种羊。样品1 h内运至实验室后密封保存于0~4 ℃冷库中。
MPA近红外光谱仪 德国布鲁克光学仪器公司;PGC 6002i天平 艾德姆衡器(武汉)有限公司;RS-JR80A绞肉机 合肥荣电实业股份有限公司。
1.2 方法
1.2.1 样品制备
为建立准确定量检测模型,将原料肉的表皮、筋膜、肥肉、血块等剔除,切成片状,按照比例混合掺杂成卷状。其中猪肉掺加量占瘦肉总量的比例分别为0%、20%、40%、60%、80%和100%,综合羊肉中脂肪占比以及市场因素[1],选择肥肉占比分别为15%、25%和35%,每个配比18 个样本平行,共324 个样本。将掺杂好的样品分别置于-18 ℃冷库中保存。实验开始前,将样品取出置于0~4 ℃冷库中24 h解冻。对解冻完的样品使用绞肉机,以直径为3 mm的板孔绞成小颗粒肉糜,绞2 次,保证样品掺杂均匀。每个样本称取(180±1)g,使用直径为8 cm的模具压实压平,以便进行近红外光谱的采集。
1.2.2 光谱采集
利用近红外光谱仪样品旋转器采集,光谱采集软件为OPUS 7.0。将样品置于样品杯中压实,旋转采集,光谱仪扫描范围为12 500~3 500 cm-1,扫描次数为64 次,分辨率为16 cm-1。实验时环境温度为(25±1)℃,确保无环境可见光干扰,每个样品采集3 次光谱数据,取其平均值作为该样品的光谱数据。
所有光谱采集实验在1 个月内完成,保证肉品冷冻期间化学成分无明显变化[21]。
1.2.3 分析方法
将324 个样本按照Kennard Stone法[22]以3∶1分为校正集和验证集,数据处理采用MATLAB2017a软件编程实现。
为去除光谱噪声提高信噪比,选取SG(Savitzky-Golay)卷积平滑(7 点)、SG平滑结合一阶导(SG-first derivation,SG-1st)、SG平滑结合二阶导(SG-second derivation,SG-2st)、MSC、中心化、标准正态变量校正(standard normal variate,SNV)等预处理方法,并结合PLSR进行定量分析建立判别解冻羊肉卷中猪肉掺假比例的预测模型。选取模型最优的预处理方法,采用CARS进行波长优选,简化模型和提高模型预测精确度和稳定性,根据所筛选波长用PLSR方法再对猪肉掺假比例进行建模分析。以校正均方根误差(root mean square error of calibration,RMSEC)、预测均方根误差(root mean square error of prediction,RMSEP)、校正集决定系数()、验证集决定系数()、范围误差比(ratio of performance to standard deviate,RPD)作为模型的评价指标[23]。寻找具有较低且相近的RMSEC和RMSEP,拥有较高和且RPD大于3[23]的组合处理方法,使模型的准确度、稳定性、分辨能力达到最佳。
1.2.4 模型验证
实验采用外部验证[24]的方法对所建立的模型预测效果进行验证。按照上述样品制备方法,取不同掺杂比例的样品为检验集(test set,TS)进行实验验证,具体掺杂比例如表1所示,共20 个样本,分为4 组。其中第1、2两组样品肥肉占比与建模集样品肥肉占比相同,但猪肉掺假比例不相同,而第3组样品的肥肉占比和猪肉掺假比例均与建模集样品相同,第4组样品的肥肉占比和猪肉掺假比例均与建模集样品不同,以便分别对模型的重复性和适用性作验证。

表1 检验集样品组成Table1 Sample composition of test set
2 结果与分析
2.1 原始光谱的测定

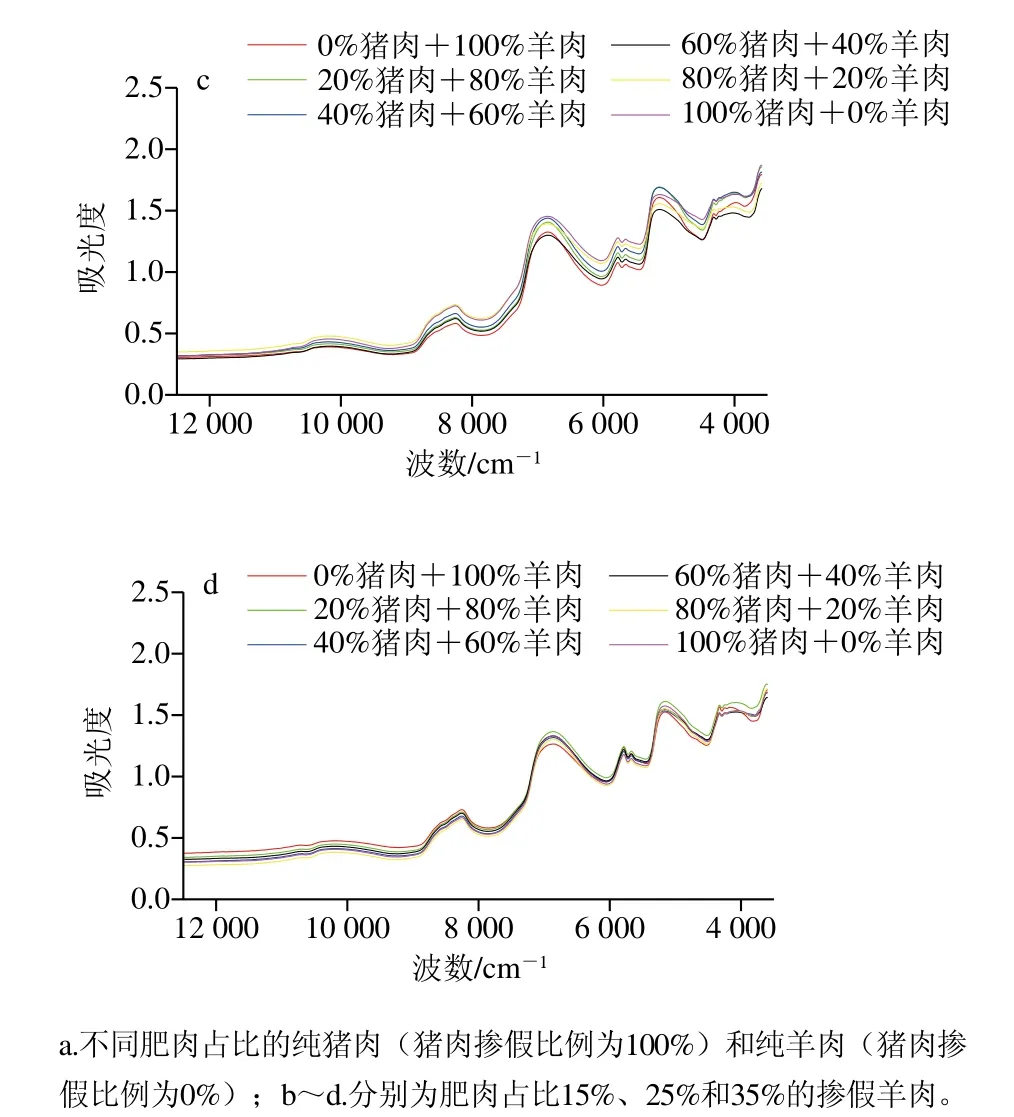
图1 原始光谱Fig.1 Original spectra of samples
从图1a可以看出,不同肥肉占比的纯猪肉与纯羊肉的吸光度曲线非常相似,在某些波数范围内可明显区分,如9 000~7 500 cm-1范围内,吸光度从高到低依次是猪肉(肥肉占比15%)、羊肉(肥肉占比15%)、猪肉(肥肉占比25%)、羊肉(肥肉占比35%)、猪肉(肥肉占比35%)、羊肉(肥肉占比25%),同一肉种不同肥肉占比的吸光度变化规律不同。不同种类的肉化学成分不同,肥肉与瘦肉化学成分相差较大[25],因此近红外光谱的吸光度也有所变化。
不同掺假比例的样品光谱图趋势相同,波峰波谷处基本相同,但吸光度呈现区别。而比较图1b~d可以看出,不同肥肉占比的掺假肉光谱趋势基本相同,但肥肉占比越高,肥肉占比对光谱曲线的影响越大,掺假比例对光谱曲线影响越小,肥肉占比35%的样品吸光度差别较小。
2.2 全波段建模

表2 不同预处理方法建立羊肉中猪肉掺假模型结果Table2 Results of calibration and prediction with different preprocessing methods for adulterated mutton rolls
在原始光谱12 000~3 500 cm-1波数范围内,分别运用SG(7 点)、SG-1st、SG-2st、MSC、中心化、SNV等预处理方法进行PLSR定量建模,如表2所示。各种预处理方法得到的PLSR模型预测集的决定系数均达到0.9以上,且RPD都在3以上,表明该方法对制备的掺假羊肉样品中猪肉掺假比例定量预测是可行且稳定的,其中采用SG-1st作为预处理方法,预测的精确度和模型的稳定性高于其他方法,因此采用SG-1st处理之后的光谱数据作进一步分析。
2.3 特征波长建模
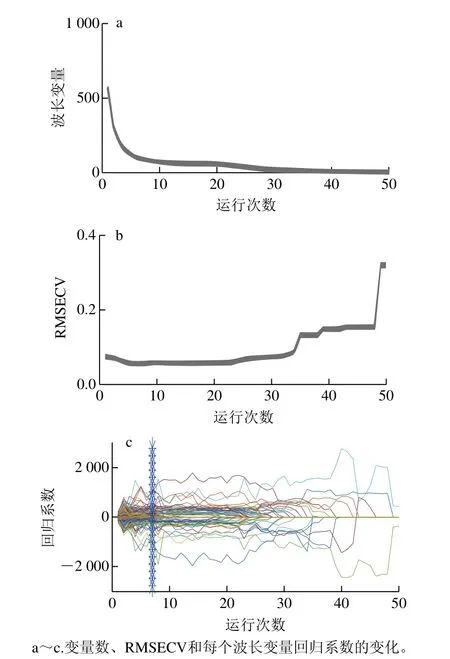
图2 特征波长提取过程Fig.2 Extraction of feature wavebands

图3 选择特征波长变量Fig.3 Selection of characteristic wavelength variates
CARS是通过自适应重加权采样技术选择出PLSR模型中回归系数绝对值大的波长点,去掉权重小的波长点,利用交叉验证优选出多个PLSR模型中交互验证均方根误差(root mean square error of cross validation,RMSECV)最低的子集,该子集所包含的变量即为最优变量组合[26-27]。对SG-1st预处理后的光谱数据应用CARS算法,选取特征波长,建立CARS-PLSR定量检测模型。图2表示算法运行过程中,变量数、RMSECV和每个波长变量回归系数的变化情况。如图2a所示,随着采样次数的增加,保留的相关波长变量数逐渐减少,且减少的速度由高变低,表明筛选过程是由粗选到精选。如图2b所示,随着样品运行次数的增加,单个PLSR模型RMSECV先降低后升高,其中运行到第7次时RMSECV最小,表明运行1~7 次之间,大量的无关变量被剔除,而运行7 次之后,与掺假定量预测相关的波长变量被剔除导致RMSECV升高。基于选取RMSECV最小的原则,选取采样运行7 次获取的变量子集为与掺假定量预测相关的关键特征变量,共93 个波长变量,如图3所示。选择的特征波长变量主要集中在6 600~5 500 cm-1和12 000~7 000 cm-1谱段之间,大多在中波近红外谱区范围内,特征谱段主要记录的是C—H基团的一级倍频和二级倍频的振动吸收[28],表明C—H基团振动的信息对于定量分析具有更大的意义。
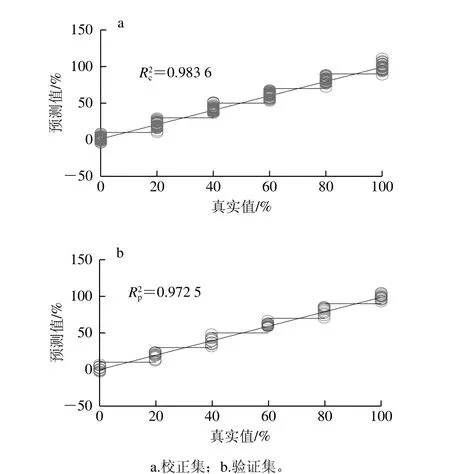
图4 模型预测结果Fig.4 Model prediction results
应用提取的特征波长变量建立PLSR定量检测模型,模型校正集和验证集的决定系数分别为0.983 6和0.972 5,RMSEC和RMSEP分别为0.043 7和0.057 7,RPD为7.62,如图4所示。选定相连2 个比例值的均值即10%、30%、50%、70%、90%为阈值,可以看出基本每个掺假比例均与前后掺假比例无重叠,说明该模型的定量检测针对建模集猪肉掺假比例的±10%范围内是准确的。CARS-PLSR模型的效果明显优于全波段建模结果,模型也更加稳定,表明CARS算法有效筛选的特征波长主要含有猪肉掺假比例定量预测的相关信息,其他无关光谱特征变量被尽可能删除。
2.4 模型验证
为验证模型在各肥肉占比区间内对样品种猪肉掺假比例预测的准确性、重复性和适用性,应用上述最优猪肉掺假比例定量分析模型对TS样品掺假比例进行定量预测。4 组TS样品总体的真实值与预测值的相关系数为0.913 8,均方根误差为0.238 7,预测结果如表3所示。1、2、3组的预测误差绝对值均低于5%,表明当检验样品与样品集肥肉占比相同时,该模型对样品中猪肉掺假比例有良好的预测效果,重复性较好。而第3组预测并未明显优于1、2组,表明该模型对相同肥肉占比样品检测具有较好得适用性。而第4组的误差绝对值中10%掺假、30%掺假和90%掺假均大于5%,表明该模型对与建模集肥肉占比不同的样品定量预测效果稍差,但总体差异不大。4 组样品预测结果可以看出,模型整体对待测样品的重复性和适用性较好,若进一步提高模型的适用性,需要增加不同肥肉占比的样品。

表3 TS样品预测结果Table3 Prediction results for test set
3 结 论
本实验结合近红外光谱技术对不同肥肉占比的解冻羊肉卷中的猪肉掺假比例进行研究对全波段光谱,经SG-1st预处理后的PLSR模型效果最好,校正集和验证集相关系数分别为0.969和0.953,RPD为4.595大于3,即表明基于近红外光谱技术对不同肥肉占比的羊肉卷中猪肉掺假比例的测定方法可行。CARS-PLSR模型仅使用16.1%的波长变量,校正集和验证集决定系数分别为0.983 6和0.972 5,RPD为7.62,预测集TS预测值和真实值的相关系数为0.913 8,剔除了不相关波长变量,模型效果得到提高。
本研究中需进一步补充样本数量,扩大不同肥肉占比下的猪肉掺假比例样品数量,并增加不同部位肉品掺假的样品数量,扩大模型的使用范围。

