抗稻瘟病位点LABR_64的起源及其分布和序列多样性
邓雨飞 刘明浩 王丹 左示敏 康厚祥,* 王国梁,4,*
抗稻瘟病位点LABR_64的起源及其分布和序列多样性
邓雨飞1,2刘明浩2王丹1左示敏3康厚祥2,*王国梁2,4,*
(1湖南农业大学 农学院, 长沙 410128;2中国农业科学院 植物保护研究所/植物病虫害生物学国家重点实验室, 北京 100193;3扬州大学 植物功能基因组学教育部重点实验室/江苏省粮食作物现代产业技术协同创新中心, 江苏 扬州 225009;4俄亥俄州立大学 植物病理学系, 美国 俄亥俄州 哥伦布 43210;*通讯联系人, E-mail: kanghouxiangcaas@163.com;wang.620@osu.edu)
【目的】研究抗病基因在水稻种群中的分布特征是培育抗病品种的基础。【方法】以水稻第9染色体上与等位的抗病位点LABR_64为研究对象,结合稻瘟病抗性鉴定,对该位点中包含的两个抗病基因和在水稻群体中的分布特征进行了研究,并对其在单子叶植物中对应的直系同源序列进行了共线性分析。【结果】LABR_64位点在水稻中存在的频率约为16%,所有存在该位点的粳稻均表现高抗病性,缺失该位点的粳稻品种均表现出高感病性,而籼稻品种中LABR_64位点存在频率低于5%,虽然其存在情况下均表现出抗病性,但大部分缺失该位点品种也表现出抗性;存在LABR_64位点的品种中,基因编码序列高度保守;LABR_64起源于单子叶与双子叶植物完全分离后及单子叶植物分化早期,其在不同的单子叶植物的分化过程中一直保持着良好的共线性。【结论】粳稻抗稻瘟病表型与和存在与否紧密关联,而籼稻的相关性不大,表明籼稻中其他抗性基因在稻瘟病抗性过程中起主导作用;鉴于的高度保守性,可根据其编码序列设计分子标记,用于高效筛选抗稻瘟病粳稻品种;LABR_64可能在不同单子叶植物抗病过程中均具有重要作用。
水稻;抗稻瘟病基因;序列多样性;基因起源
稻瘟病是由稻瘟病菌()引起的一种真菌病害,主要通过气流传播,是水稻的三大病害之一,每年造成水稻减产高达11%~13%,给粮食产业造成巨大经济损失。因此,有效防治稻瘟病是水稻安全生产的重要任务。目前,选育优良抗病品种和化学防治是防治稻瘟病的两种主要方法,其中,前者是控制稻瘟病最经济、有效和环保的方法[1]。同时,稻瘟菌菌株多样性丰富且变异速度快,导致水稻抗稻瘟病品种在大面积推广2~3年后容易丧失抗病性,这对稻瘟病抗病育种和抗病品种田间布局带来极大困难,也成为制约我国乃至世界水稻持续高产、稳产的主要因素之一[2]。因此,发掘自然群体中的抗稻瘟病新基因及建立抗病品种在不同地区的合理布局对水稻抗病育种具有重要意义。
根据植物抗性基因编码蛋白产物的类型,可将抗病基因分为五大类,包括NBS-LRR、eLRR-TM、eLRR-TM-pkinase、STK以及除这四类以外的其他类型[3-4]。NBS-LRR类基因编码具有核苷酸结合位点(NBS)和富亮氨酸重复(LRR)的胞内受体蛋白。根据NBS的N端结构不同,又可分为TIR-NBS-LRR和CC-NBS-LRR两大类。其中,TIR-NBS-LRR类型只存在于双子叶植物中,单子叶植物抗病基因主要类型为CC-NBS-LRR[5-7]。目前,共鉴定和报道了超过100个水稻抗稻瘟病基因位点,已经克隆的水稻抗稻瘟病基因有等28个[8-18],其中只有一个隐性基因,为,其余全部为显性抗稻瘟基因[13]。在这20多个已被克隆的抗病基因中,除编码B-lectin激酶[10]和编码富含脯氨酸蛋白[13]以外,其余均为NBS-LRR类型基因。抗病基因呈串联重复分布于同一遗传座位在水稻NBS-LRR基因家族中为普遍现象。在已克隆的20多个抗稻瘟病基因中,和都位于第6染色体近着丝粒区域,属于同一遗传座位。此外,和也呈串联重复分布于第11染色体同一遗传座位。和是位于第9染色体同一遗传座位的等位基因,对序列和功能分析表明,其中包含两个抗病基因和,需二者同时存在时才具有抗性[19-21]。Kang等[22]利用RDP1(rice diversity panel 1)群体接种5个不同的稻瘟菌菌株,结合高通量测序获得的高密度SNP标记进行全基因组关联分析,鉴定了97个与稻瘟病相关的位点LABR(loci associated with blast resistance)。这些抗病位点中,有且只有64号位点LABR_64对所有5个测试稻瘟菌的抗性均关联,通过基因沉默方法对LABR_64位点中潜在的候选基因进行了功能分析,鉴定到两个新的抗性等位基因和。本研究系统分析了其在水稻群体中的分布情况,并通过进一步序列比对分析,发现此位点在单子叶植物中非常保守,是一个古老的抗病位点。对该基因区域的进一步研究将有助于明确水稻抗稻瘟基因的起源与演化,为挖掘新的抗稻瘟病基因以及充分利用抗病等位基因来提高水稻抗性提供新思路。
1 材料与方法
1.1 水稻材料、稻瘟菌材料以及试剂
本研究选用227份水稻核心材料,其中籼稻68份,粳稻159份,日本晴为对照感病材料。所用稻瘟菌菌株为RB22。聚合酶等分子试剂均购自全式金公司。
1.2 实验方法
1.2.1 水稻种植方法
将水稻种子去壳,按照Park等[23]的方法用酒精和次氯酸钠消毒后铺于1/2MS培养基上,置于光照培养箱中(12 h光照/12 h黑暗)培养,保证水稻起始状态一致。一周后,将水稻幼苗移栽于中国农业科学院廊坊基地温室苗钵,三周后进行接种。
1.2.2 稻瘟菌培养
参照Wang等[24]的方法,将稻瘟菌纸片转接到燕麦培养基上;于25℃黑暗培养箱中培养5~7 d,待菌丝扩散后,转移至24 h光照培养箱中,继续培养一周,即可产生分生孢子。
1.2.3 稻瘟病接种方法
用0.05%的吐温水溶液刮洗孢子,过滤至50 mL离心管中;在光学显微镜下调节孢子浓度约为1×105个/mL;用喷壶将孢子悬浮液均匀喷于3叶1心的水稻苗,用封口膜轻轻封住接种箱,置于25℃、相对湿度95%条件下,避光24 h后,在光照12 h/黑暗12 h条件下交替培养一周,调查病情。
1.2.4 DNA提取、PCR和测序分析
采用CTAB法提取水稻叶片DNA。使用Primer Premier 5.0设计引物(表1),长度约为18~25 bp,GC含量为40%~60%,退火温度为55℃~60℃。DNA测序等由北京华大基因研究中心有限公司完成。
1.3 数据分析
不同单子叶植物的参考基因组序列从NCBI网站(ftp://ftp.ncbi.nlm.nih.gov/genomes/)下载,利用BLAST、ClustalW2和DNAMAN等对序列进行比对和分析,Perl SVG模块完成共线性作图。

表1 本研究所用的引物序列
2 结果与分析
2.1 LABR_64位点稻瘟病抗性鉴定
康厚祥等[22]通过基因敲除技术敲除LABR_64位点中两个抗病基因中的任意一个后,不同遗传背景的水稻品种均表现出感病性。本研究利用强致病稻瘟菌菌株RB22喷雾接种籼稻和粳稻材料后,发现对照日本晴(缺失LABR_64区域两个抗病基因)叶片具有典型病斑,表现为感病,而携带LABR_64位点的材料叶片无典型病斑,表现为抗病(图1)。进一步表明LABR_64位点在供试水稻品种中与抗RB22紧密关联。
2.2 LABR_64在籼粳稻中的分布
用RB22接种不同遗传背景的品种,分别筛选出籼稻和粳稻的抗、感病品种。针对LABR_64位点包含的两个抗病功能基因和进行PCR检测。接种结果表明,在感病粳稻品种中两个基因均缺失,而在抗病品种中扩增条带清晰一致。在所测试粳稻品种中,LABR_64的存在与否与抗/感RB22菌株高度关联;籼稻抗性品种中,只有部分品种包含LABR_64位点,包含LABR_64的籼稻品种均对RB22菌株高抗,同时,籼稻抗病品种中还有很大一部分不包含LABR_64,表明这些籼稻品种中存在其他未知的抗病基因对RB22起抗性作用(图2)。
2.3 LABR_64位点的起源与演化
为了进一步明确抗稻瘟位点LABR_64的起源及在水稻不同亚群中的演化。选取12个包含LABR_64位点的抗病水稻品种进行DNA提取和PCR测序。通过对和基因序列比对发现,两个基因序列在不同抗稻瘟病水稻品种中相对保守,同时序列之间也具有多态性位点,这些多态性位点分布在第175-458位氨基酸(NBS结构域),第583-602位和第796-818位氨基酸区域(LRR结构域)(图3~4)。这些NBS和LRR结构区域的序列多样性可能对其抗稻瘟病功能的分化具有重要作用。

M-5000 bp标记; 1-Binulawan; 2-IR36; 3-IR8; 4-Carolina Gold; 5-Iguape Cateto; N-日本晴。其中Binulawan、IR36和IR8为籼稻,抗性级别为0,Carolina Gold和Iguape Cateto为粳稻,抗性级别为1。
Fig. 1. Blast resistance evaluation of the rice varieties which contain LABR_64 locus.
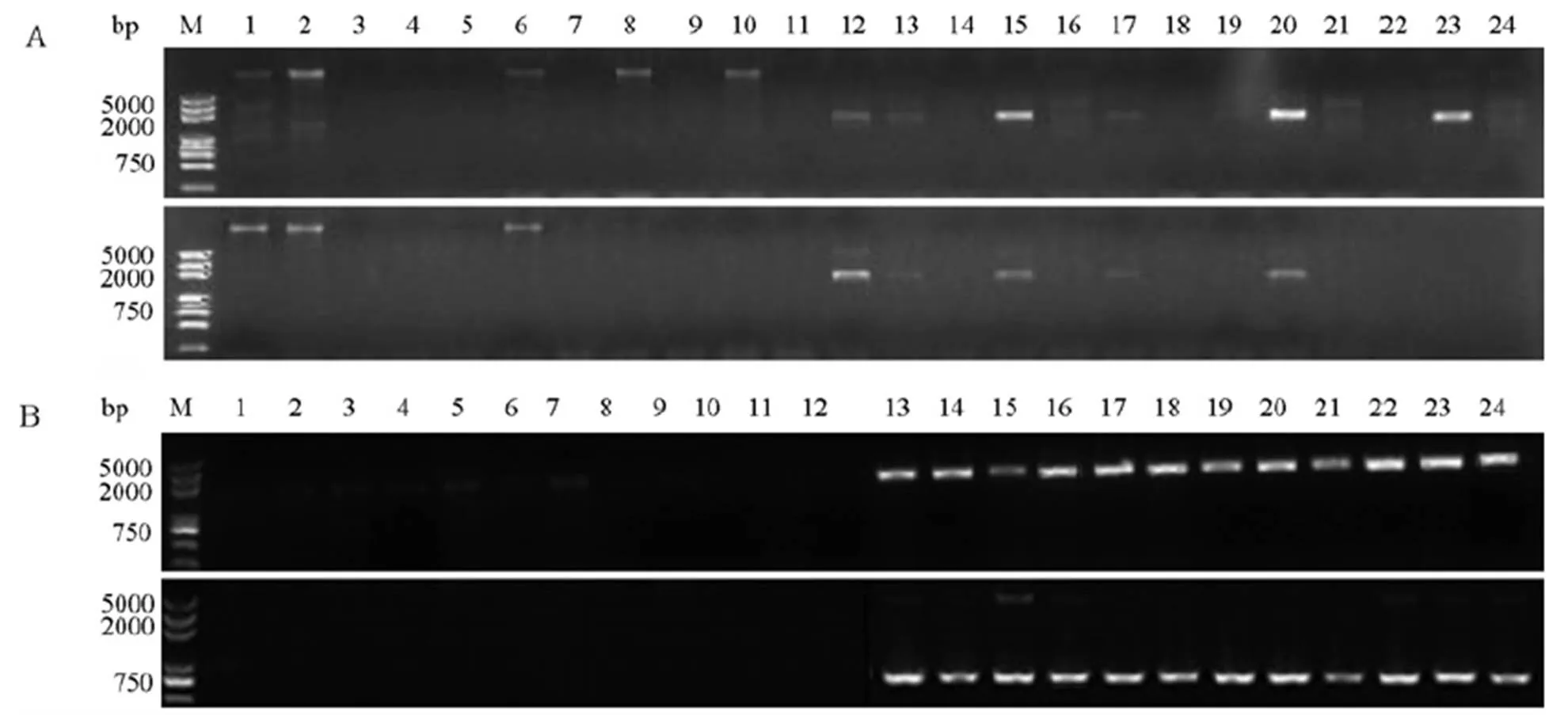
A: LABR_64-1(上)、LABR_64-2(下)在籼稻感病和抗病品种中的分布,M-5000 bp标记;1~12为感病品种;13~24为抗病品种。B: LABR_64-1(上)、LABR_64-2(下)在粳稻感病和抗病品种中的分布,M-5000 bp标记;1~12为感病品种;13~24为抗病品种。
Fig. 2. Distribution of LABR_64 inandrice.

图3 抗稻瘟病基因LABR_64-1序列多样性分析
Fig. 3. Sequences diversity of rice blast resistance alleles of

Fig. 4. Sequences diversity of rice blast resistance alleles of
2.4 不同单子叶植物中LABR_64直系同源区段的序列共线性分析
在抗病粳稻和籼稻中,基因与序列一致性高且所在区域共线性关系一致(图5-A)。其中二穗短柄草LABR_64直系同源区域与水稻比较,发现直系同源基因差异相对于的直系同源差异更大,侧翼序列呈共线性排列。我们通过对基因和所在的200 kb DNA区段在几种单子叶植物(包括二穗短柄草、狗尾草、高粱、玉米)中的直系同源区段的分析,发现均存在明显共线性关系。表明在单子叶植物分化过程中LABR_64直系同源区域一直保持着共线性,此区域在不同的单子叶植物中可能均起重要作用。此外,序列比对分析发现双子叶植物(如拟南芥、大豆等)均不存在LABR_64直系同源区域,表明LABR_64区域为单子叶植物所特有。
2.5 LABR_64位点的起源
单双子叶植物分化的时间节点在7300万年前,单子叶植物后续分化出不同的物种[25-26](图6)。其中,玉米、高粱祖先大概在4500万~6000万年与其他单子叶植物(如小麦、二穗短柄草、水稻等)物种分离,水稻在4000万~5400万年前与小麦和二穗短柄草等物种分离。水稻和其他测试单子叶植物均存在一个与LABR_64直系同源的共线性区域(图5),因此,LABR_64抗病位点的起源可追溯到单子叶植物分化早期,即5600万~7300万年,是一个古老的抗病位点。
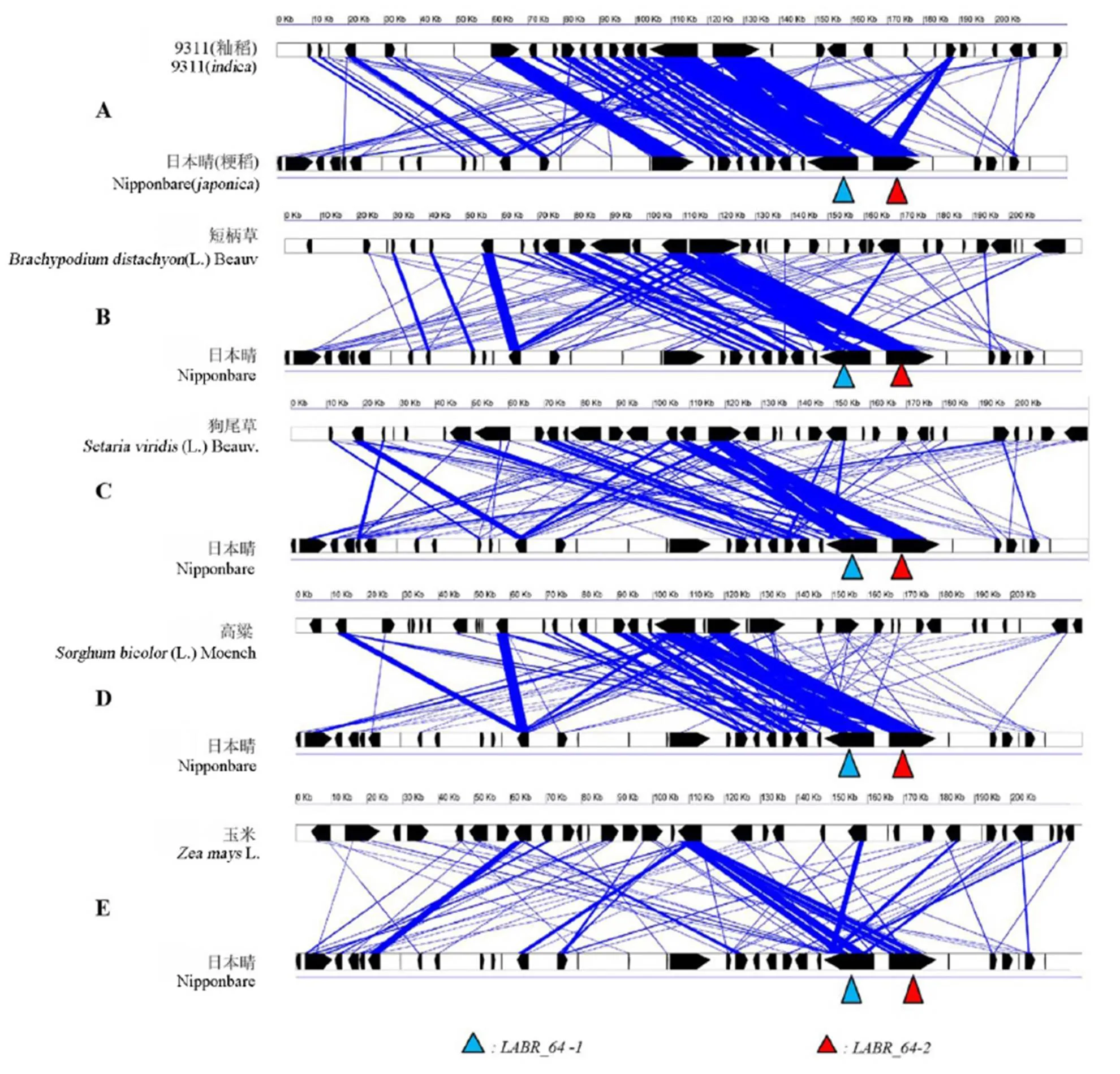
A-日本晴和9311(LABR_64位点在不同粳稻品种中表现为存在或缺失两种主要类型,而日本晴中缺失LABR_64位点,为了便于共线性分析,图中三角形区域表示来自抗病粳稻的LABR_64序列插入日本晴等位区域);B-二穗短柄草与水稻;C-粟米与水稻;D-高粱与水稻;E-玉米与水稻。
A, Nipponbare and 9311 (Nipponbare does not contain LABR_64, the triangles represent theandgenes from the resistantvarieties). B, Nipponbare and; C, Nipponbare and; D, Nipponbare and; E, Nipponbare and.
图5 不同单子叶植物中LABR_64直系同源区域的序列共线性分析
Fig. 5. Microsynteny of the LABR_64 orthologous regions in different monocotyledons.
3 讨论
水稻是世界上最重要的粮食作物之一,其品种繁多且分布广泛,同时水稻也是植物生物学研究的模式植物之一。相对水稻的起源与驯化、生长与发育、产量与品质等研究[27-29]而言,水稻抗病性研究相对滞后,为水稻的分子设计育种带来了潜在风险。本研究关于抗稻瘟病基因的起源和演化研究可为分子设计育种提供理论基础,同时可为其他非模式作物的相关研究提供重要参考价值。
在水稻抗稻瘟病研究中,通过全基因组关联分析(Genome-wide association study, GWAS)挖掘出与抗稻瘟病相关的位点并进一步结合分子水平功能验证已成为定位和克隆抗稻瘟病基因可靠的新方法。本研究对通过GWAS分析获得的LABR_64抗病位点,在227份核心水稻品种资源中进行抗性验证,发现LABR_64与抗稻瘟病具有高度相关性。稻瘟菌接种发现携带这个位点的水稻品种具有良好的抗性表型,进一步从群体水平说明了LABR_64位点与水稻抗性相关。

图6 单子叶植物分化的时间节点(单位:百万年)
Fig. 6. Timeline of monocotyledon differentiation (Unit: million years).
我国的地理环境复杂,南北气候差异大,栽培的水稻品种也不同,南方以籼稻为主,北方以粳稻为主[30]。因而南北方稻瘟菌菌株也不尽相同[31]。因此,挖掘并有效利用更多广谱抗稻瘟病基因是有效防治水稻稻瘟病的途径。是已克隆的广谱抗稻瘟病基因之一[21],LABR_64位于水稻第9染色体等位区域[32]。LABR_64位点在不同粳稻抗病品种都存在,且不同品种间具有序列多样性,这些序列多样性可能对LABR_64的广谱抗病功能具有重要作用,LABR_64区域序列多样性和其广谱抗病性之间的关系和机制还有待进一步研究。
植物中NBS-LRR类型基因家族是抗病基因中最大的家族,其旁系同源基因序列之间差异大、多样性丰富,通常认为其与病原菌的效应因子等存在协同演化关系[9]。通常情况下,NBS-LRR类型基因在基因组中存在非常高频率的缺失、加倍、异位插入、同源序列间彼此交换进而形成新基因[11]。因此,对亲缘关系稍远的物种,随演化时间尺度的延长,NBS-LRR基因及其侧翼序列的共线性极易被破坏。然而,本研究发现,抗稻瘟病位点LABR_64不仅古老,且其包含的基因和及其侧翼基因所在的200 kb区域在不同的单子叶植物中均保守且呈典型的共线性,虽然它在其他单子叶植物中的直系同源基因的功能尚不明确,但此研究结果表明,LABR_64直系同源区段在不同的单子叶植物抗病过程中均可能扮演重要作用。
总之,对抗稻瘟病位点LABR_64的起源及演化的研究为水稻其他抗病基因的起源与演化分析奠定了基础,也为将来可能利用近缘物种的优良直系同源基因进行作物抗病性改良提供了科学依据。
[1] Jiang N, Liu X L,Dai L Y, Wang G L. Advances on the mapping and cloning of blast resistance gene in rice., 2010, 26(10): 270-275.
[2] He X Y, Wang L, Wu W H, Chen Z M, Lin F, Chen Y S, Liu W, Chen Y H, Liao Y P. The progress of mapping, isolation of the genes resistance to blast and their breeding application in rice., 2014, 30(6): 1-12.
[3] Dangl J L, Jones J D G. Plant pathogens and integrated defense responses to infection., 2001, 411(6839): 826-333.
[4] Iyer A S, Mccouch S R. The rice bacterial blight resistance geneencodes a novel form of disease resistance., 2004, 17(12): 1348-1354.
[5] Kobe B, Deisenhofer J. A structural basis of the interactions between leucine-rich repeats and protein ligands., 1995, 374(6518): 183-186.
[6] Tameling W I, Elzinga S D, Darmin P S, Vossen J H, Takken F L, Haring M A, Cornelissen B J. The tomato R gene products I-2 and MI-1 are functional ATP binding proteins with ATPase activity., 2002, 14(11): 2929.
[7] Zhang X, Yang S, Wang J, Jia Y, Huang J, Tan S, Zhong Y, Wang L, Gu L, Chen J Q, Pan Q, Bergrelson J, Tian D. A genome-wide survey reveals abundant rice blastgenes in resistant cultivars., 2015, 84(1): 20-28.
[8] Wang Z X, Yano M, Yamanouchi U, Iwamoto M, Hayasaka H, Katayose Y, Sasaki T. Thegene for rice blast resistance belongs to the nucleotide binding and leucine-rich repeat class of plant disease resistance genes., 1999, 19(1): 55-64.
[9] Bryan G T, Wu K S, Farrall L, Jia Y, Hershey H P, McAdams S A, Faulk K N, Donaldson G K, Tarchini R, Valent B. A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistance gene., 2000, 12(11): 2033-2046.
[10] Zhou B, Qu S, Liu G, Dolan M, Sakai H, Lu G, Bellizzi M, Wang G L. The eight amino-acid differences within three leucine-rich repeats betweenandresistance proteins determine the resistance specificity to., 2006, 19(11): 1216-1228.
[11] Qu S H, Liu G F, Zhou B, Bellizzi M, Zeng L R, Dai L Y, Han B, Wang G L. The broad-spectrum blast resistance geneencodes a nucleotide-binding site–leucine-rich repeat protein and is a member of a multigene family in rice., 2006, 172(3): 1901-1914.
[12] Lee S K, Song M Y, Seo Y S, Kim H K, Ko S, Cao P J, Suh J P, Yi G, Roh J H, Lee S, An G, Hahn T R, Wang G L, Ronald P, Jeon J S. Rice-mediated resistance torequires the presence of two coiled-coil–nucleotide-binding–leucine-rich repeat genes., 2009, 181(4): 1627-1638.
[13] Fukuoka S, Saka N, Koga H, Ono K, Shimizu T, Ebana K, Hayashi N, Takahashi A, Hirochika H, Okuno K, Masahiro Yano. Loss of function of a proline-containing protein confers durable disease resistance in rice., 2009, 325(5943): 998-1001.
[14] Lin F, Chen S, Que Z Q, Wang L, Liu X Q, Pan Q H. The blast resistance geneencodes a nucleotide binding site–leucine-rich repeat protein and is a member of a resistance gene cluster on rice chromosome 1., 2007, 177(3): 1871-1880.
[15] Ashikawa I, Hayashi N, Yamane H, Kanamori H, Wu J Z, Matsumoto T, Ono K, Yano M. Two adjacent nucleotide-binding site–leucine-rich repeat class genes are required to confer-specific rice blast resistance., 2008, 180(4): 2267-2276.
[16] Takahashi A, Hayashi N, Miyao A, Hirochika H. Unique features of the rice blast resistancelocus revealed by large scale retrotransposon-tagging., 2010, 10(1): 175-186.
[17] Zhai C, Lin F, Dong Z Q, He X Y, Yuan B, Zeng X S, Wang L, Pan Q H. The isolation and characterization of, a rice blast resistance gene which emerged after rice domestication., 2011, 189(1): 321-334.
[18] Deng Y, Zhai K, Xie Z, Yang D, Zhu X, Liu J, Wang X, Qin P, Yang Y, Zhang G, Li Q, Zhang J, Wu S, Millazzo J, Mao B, Wang E, Xie H, Tharreau D, He Z. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance., 2017, 355(6328): 962-966.
[19] Yi G, Lee S K, Hong Y K, Cho Y C, Nam M H, Kim S C, Han S S, Wang G L, Hahn T R, Ronald P C, Jeon J S. Use of(t) markers in marker-assisted selection to screen for cultivars with resistance to., 2004, 109(5): 978-985.
[20] Jeon J S, Chen D, Yi G H, Wang G L, Ronald P C. Genetic and physical mapping of(t), a locus associated with broad-spectrum resistance to rice blast., 2003, 269(2): 280-289.
[21] Inukai T, Zeigler R S, Sarkarung S, Bronson M, Dung L V, Kinoshita T, Nelson R J. Development of pre-isogenic lines for rice blast-resistance by marker-aided selection from a recombinant inbred population., 1996, 93(4): 560-567.
[22] Kang H X, Wang Y, Peng S S, Zhang Y L, Xiao Y H, Wang D, Qu S H, Li Z Q, Yan S Y, Wang Z L, Liu W D, Ning Y S, Korniliev P, Leung H, Mezey J, McCouch S R, Wang G L. Dissection of the genetic architecture of rice resistance to the blastfungus., 2015, 17(6): 959-972.
[23] ParK C H, Wang G L. TheeffectorTargets the RING E3 ubiquitin ligase APIP6 to suppress pathogen-associated molecular pattern-triggered immunity in rice., 2012, 24(11): 4748-4762.
[24] Wang B H, Zhen W, Lu G D, Zhang X B, Wang Z H. Screening on the spore-producing media of., 2000(2): 1-2.
[25] Huang X H, Yang S H, Gong J Y, Zhao Q, Feng Q, Zhan Q L, Zhao Y, Li W J, Cheng B Y, Xia J H, Chen N, Huang T, Zhang L, Fan D L, Chen J Y, Zhou C C, Lu Y Q, Weng Q J, Han B. Genomic architecture of heterosis for yield traits in rice., 2016, 537(7622): 629-633.
[26] Vogel J P, Garvin D F, Mockler T C. Genome sequencing and analysis of the model grass., 2010, 463(7282):763-768.
[27] Zhang L, Yu H, Ma B, Liu G F, Wang J J, Wang J M, Gao R C, Li J J, Liu J Y, Xu J, Zhang Y Y, Li Q, Huang X H, Xu J L, Li J M, Qian Q, Han B, He Z H, Li J Y. A natural tandem array alleviates epigenetic repression of IPA1 and leads to superior yielding rice., 2017, 8(14789): 1-14.
[28] Marschalek R, Silva M C, Santos S B D, Manke J R, Bieging C, Porto G, Wickert E, Andrade A D. Image-Rice Grain Scanner: A three-dimensional fully automated assessment of grain size and quality traits., 2017, 17: 89-97.
[29] Luo C P, Ni L, Chen Z Y, Liu Y F, Liu Y Z, Nie Y F. Inoculation technology of rice blast and rice resistance to it in Jiangsu regional tests in 2009., 2009(6): 178-179.
[30] Zhu Y Y, Chen H, Wang Y Y, Li Z S, Li Y, Fan J H, Chen J B, Yang S S, Ma G L, Hu L P, Zou J Y, Mundt C C, Borromeo E, Leung H, Mew T W. Diversifying variety for the control of rice blast in China., 2001, 2(1): 10-14.
[31] Lei C L, Wang J L, Jiang W R, Ling Z Z, George M L. Population structure and genetic variation of rice blast fungus in some rice-growing regions in northern China., 2002, 32(3): 219-226.
[32] Wang G L, Mackill D J, Bonman J M, McCouch S, Champoux M C, Nelson R J. RFLP Mapping of genes conferring complete and partial resistance to blast in a durably resistant rice cultivar., 1994, 136(4): 1421-1434.
Origin, Distribution and Sequence Diversity of Rice Blast Resistance Locus LABR_64in Rice
DENG Yufei1,2, LIU Minghao2, WANG Dan1, ZUO Shimin3, KANG Houxiang2,*, WANG Guoliang2,*
(Agricultural College,,,;State Key Laboratory for Biology of Plant Diseases and Insect Pest /,,,;,,,,;,,,;,:,)
【Objective】A better understanding of the origin and distribution of disease resistance genes in rice germplasm is useful for breeding highly resistant varieties.【Method】We analyzed sequence diversity of the rice blast resistance locus LABR_64, which contains two homologous genes,and, is located in the allelic region ofon rice chromosome 9. In addition, we analyzed the microsynteny of the LABR_64 orthologous region in different monocotyledons.【Result】The presence frequency (PF) of LABR_64 is 16%. All of therice cultivars carrying LABR_64 are highly resistant to rice blast and all of those without the locus are susceptible. In addition, the PF of LABR_64 in thesubpopulation is lower than 5%. Although LABR_64 is correlated with the resistance to rice blast, manyrice cultivars without LABR_64 are also resistant to rice blast. We also found that thesequence is much conserved. Moreover, the microsynteny analysis of the LABR_64 orthologous region in different monocotyledons indicated that LABR_64 originates after the separation of the monocotyledonous and dicotyledonous species, and at the early differentiation stage of monocotyledons.【Conclusion】The rice blast resistance phenotypes are closely correlated with the presence of the LABR_64 locus insubpopulation, but not in, indicating that there are many other resistance loci in thesubpopulation. Thesequence can be used for developing molecular markers in marker-assisted rice blast resistance breeding. It also indicated that LABR_64 may play a role in disease resistance in different monocotyledons.
rice; rice blast resistance gene; sequence diversity; gene origin
10.16819/j.1001-7216.2019.8017
S435.111.4+1; S511.034
A
1001-7216(2019)01-0020-08
2018-02-08;
2018-06-27。
国家自然科学基金国际合作重点项目(31461143019);江苏省农业重大新品种创制项目(PZCZ201703)。

