取代三唑-噻二唑类化合物生物活性的QSAR研究
冯长君
(徐州工程学院 化学化工学院,江苏 徐州 221018)
定量构效关系(quantitative structure-activity relationship,QSAR)[1-4]研究主要通过理论计算和各种统计分析工具来建立若干化合物的结构与其生物活性之间的数学关系,以此预测其它化合物的相关性质(例如抑菌活性、生物毒性、致癌性等);并在分子水平上揭示微观结构对化合物各种生物活性的影响,推测其可能的作用机理.QSAR研究的关键是建立分子结构的描述符.拓扑指数因可直接从分子结构衍生出来,不依赖于实验测量而备受关注.Randic[5]提出并由Kier和Hall[6]扩展的分子连通性指标是最广泛应用的拓扑指标之一.最近报道了一种基于2D拓扑结构和13种原子类型的分子电性距离矢量(Mt)[7-9],成功建立有机化合物多种生物活性与Mt之间的QSAR模型.
杂环类化合物结构变化多样、活性高且污染小,日益受到人们的普遍重视.如均三唑并噻二唑类化合物具有抗肿瘤、抗菌、杀虫以及调节植物生长等生物活性.姚明星等[10]根据生物活性叠加原理,把吡唑引入到均三唑并噻二唑中,设计合成 21个吡唑取代基的 1,2,4-三唑[3,4-b]-1,3,4-噻二唑衍生物,简称“取代三唑-噻二唑衍生物(Substituted-triazolo-benzothiazole,STB)”.他们采用不同方法测试了这些化合物的多种生物活性.本文基于电性距离矢量[7-9],通过最佳变量子集回归建立STB生物活性的QSAR模型,估算与预测STB生物活性,并在分子水平上揭示影响其生物活性的微观结构,为合理设计新颖、高效STB的先导化合物提供理论参考.
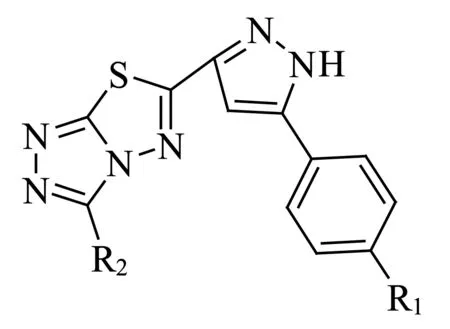
图1 取代三唑-噻二唑衍生物的母体结构
1 材料与方法
1.1 STB及其生物活性
姚明星等[10]合成的21个取代三唑-噻二唑类化合物的母体结构如图1所示,相应取代基R1、R2见表1.
采用小麦芽鞘法测试上述化合物的麦芽生长活性(wheat gemma activities,Wg),Wg越大,表示对小麦芽鞘生长具有良好促进作用.采用打孔法测试这些化合物对大肠杆菌(E.coli) 的抑菌活性,即抑菌率(%),并根据生物统计概率值换算表换算成抑制概率值,以“Ec”表示.Ec越大,表示对大肠杆菌(E.coli) 的抑菌活性越高.Wg、Ec的值[10]见表1、表2.

表1 取代三唑-噻二唑衍生物的生长素活性(Wg)与电性距离矢量 (Mt) 的相关性

表2 取代三唑-噻二唑衍生物的抑菌活性(Ec)与电性距离矢量(Mt)的相关性
续表2
1.2 分子描述符
化合物的分子结构信息是建立良好构效关系的前提,目前QSAR方法中用于分子结构表征的二维描述子主要有分子全息、拓扑指数等,它们已在环境科学、生命科学、药物设计等方面已经取得广泛的应用.本文基于13种原子类型的分子电性距离矢量描述子(Mt)[7-9]表征不同类别有机物的分子结构,其计算过程参见文献[7-9].
本文化合物中只有第1、2、3、6、7、9、10共7种原子类型,它们两两相互作用(包含自身相互作用),理论上可以形成38种电性距离矢量.由于某些原子类型在分子中不存在,导致某些相互作用类型不存在,因此,实际只有33个不全为零的电性距离矢量.部分电性距离矢量见表1、表2.
1.3 统计回归分析
以生物活性作为因变量,电性距离矢量作为独立变量建立QSAR模型.通常采用最佳变量子集回归(LBR)等程序进行.验证是建立具有可靠性、准确性模型的重要一步.常用统计验证指标:
1) 方差膨胀因子(VIF)[11]:检验模型中各变量之间的相关性,定义式
VIF= 1 /(1-β2),
(1)
式中β是方程中一个自变量与其他自变量之间的多元回归的相关系数.其评判标准是VIF<5.0,表示变量间没有明显的自相关性,所建模型是稳定的;当VIF大于5.0时,回归方程不稳定,必须重新检查变量的相关系数.

3)t-检验:如果在某个置信水平α下,验证模型中所有变量t的绝对值大于标准t值(tα/2),则表明模型通过t-检验并具有明显的统计学意义.
4) Akaike信息准则(AIC)[14-15]:定义方程为式(2),产生最小AIC值的模型被认为是最有用的.
(2)
5) Kubinyi函数(FIT)[14-15]:定义式为方程(3),即最好的模型将呈现这个函数的最高值来确定模型中是否应包含某个变量.
(3)
在上述式中:RSS是残差平方和,f是模型中包含的化合物数,b是模型中包含的变量数.在添加额外的变量时,如果Akaike信息准则值降低了,并且Kubinyi函数的值增加,那么此新变量的引入是合理的.
2 结果与讨论
2.1 建立QSAR模型
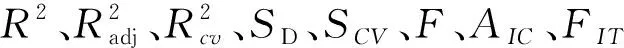
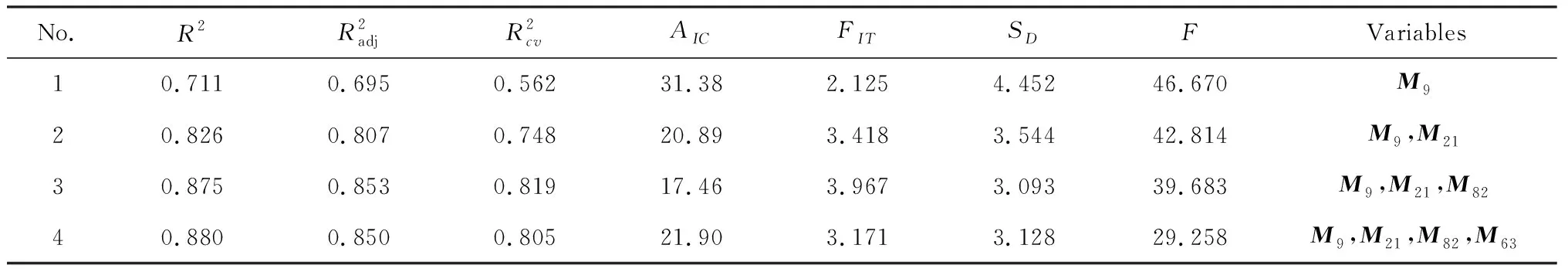
表3 取代三唑-噻二唑衍生物的Wg与Mt逐步回归结果
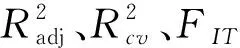
Wg=6.034(±1.176)-39.849(±15.474)M82+0.816(±0.244)M21+136.465(±24.165)M9
(4)
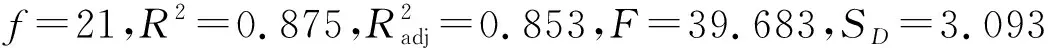
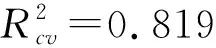
取代三唑-噻二唑衍生物对大肠杆菌的抑菌活性(Ec)与电性距离矢量(Mt)的各元QSAR模型见表4.
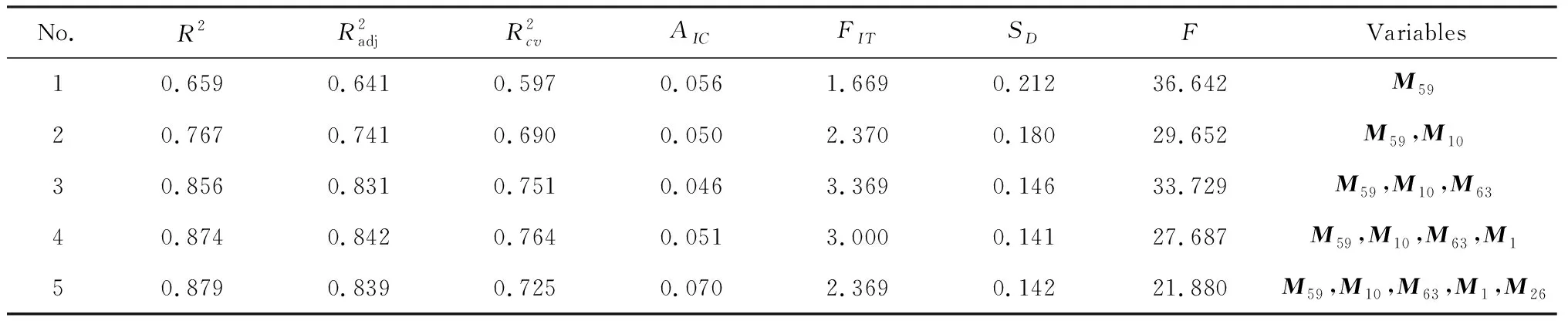
表4 取代三唑-噻二唑衍生物的Ec与Mt逐步回归结果
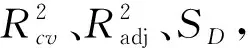
Ec=4.644(±0.058)+0.604(±0.169)M63-0.350(±0.058)M59+0.048(±0.017)M10+
5.783(±3.932)M1,
(5)


方程(5)中括号“±”后的值是指与回归系数相对应的标准偏差,其所有标准偏差均小于回归系数的1/2,表明该模型是稳定的.但是方程(5)中M1的标准偏差大于其回归系数的1/2,表明该模型是不稳定的,应予以剔除,建立最佳的三元方程:
Ec=4.654(±0.059)+0.559(±0.172)M63-0.362(±0.059)M59+0.060(±0.015)M10,
(6)
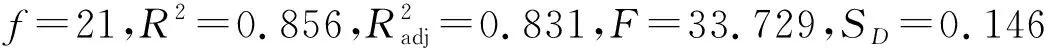
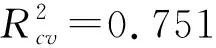
使用QSAR方程(4)、(6)得到的预测值见表1中的Wg.cal和表2中的Ec.cal,与相应实验值基本吻合.
2.2 模型质量检验
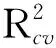
方程(4)中三个独立变量的标准回归系数(SR)和t值见表3.当置信水平为95%时,模型的标准t值(tα/2)为2.120.从表5可以看出,模型中各自变量的t绝对值均大于标准tα/2值,证明模型具有可信度.同时,M9、M21、M82的t的绝对值依次减小,此与SR的递变规律一致,这表明M9对Wg有最强影响.对于模型(6),可得出类似的规律:M59是影响Ec的最强因素.表5显示模型(4)、(6)中变量的VIF值均小于5.0,且非常接近1,表明它们均具有统计学意义,各个自变量之间几乎没有共线性.

表5 模型(4)、(6)质量的检验指标(SR、tα/2和VIF)
判定系数R2又称削减误差比例.模型(4)的R2=0.875,表明M9、M21、M82和常数项共同揭示了影响小麦芽鞘生长活性(Wg)的87.5%因素,仅有12.5%属于随机因素;模型(6)的R2=0.856,表明M10、M59、M63和常数项共同揭示了影响大肠杆菌抑菌活性(Ec)的85.6%因素,仅有14.4%属于随机因素.进一步表明上述模型的正确性.由此表明模型(4)、(6)均为高度鲁棒,显著相关模型,不仅可用于标题化合物生物活性的估算与预测,也可用于生物机理的解释.
2.3 模型分析
根据分子电性距离矢量理论可知,进入模型(4)中的电性距离矢量:M9反映第一类碳原子(—CH3)与第九类氧原子(=O、—OH)相互作用,M21反映第二类碳原子与第九类氧原子(=O、—OH)相互作用,M82反映第十类醚氧原子或者硫醚硫原子(—O—、—S—)之间相互作用.进入模型(6)中的电性距离矢量:M10反映第一类碳原子(—CH3)与第十类醚氧原子或者硫醚中硫原子相互作用,M59反映第六类氮原子(—NH—、—N=)与第九类氧原子相互作用,M63反映第六类氮原子与第十三类卤素原子相互作用.即这6个电性距离矢量分别隐含了6类非氢原子的结构信息.其中第一、二类碳原子为非极性基团,具有疏水性;余下4类为高电负性的极性基团,能够与电负性大的基团形成氢键.因此,它们都是影响取代三唑-噻二唑衍生物生物活性的结构基团.从表5的SR和t值可见,M9对小麦芽鞘生长活性(Wg)、M59对大肠杆菌抑菌活性(Ec)起主要影响作用.它们都对应高电负性的羰基氧、羟基氧基团,能与生物体内蛋白质形成氢键,发挥促进或抑制作用.由此推论:1) 取代三唑-噻二唑衍生物对小麦芽鞘生长活性(Wg)影响的结构基团与对大肠杆菌抑菌活性(Ec)的影响是不同的.2) 它们的结构基团不同,说明二者的生物作用机理不同.
3 结论
1) 分子电性距离矢量可对21种取代三唑-噻二唑衍生物的抽象分子结构实现数值化表征,呈现良好的结构选择性.

3) 药物的生物活性通常是由分子的多结构因素协同发挥作用的结果,而不是由单一参数决定.因此,根据进入模型(4)、(6)的电性距离矢量可知,它们对生物活性(Wg、Ec)发挥影响的分子结构单元有:—CH3、—C—、—NH—、—N=、—OH、O=、—O—、—S—等,其中=O、—OH起主要作用.

