不同管理模式下设施番茄土壤微生物变化
任春晖,王远宏,常若葵,刘慧芹,张斌,池明,于玮玮
(天津农学院 园艺系,天津 300384)
土壤是覆盖在地球表面的一层可以生长植物的疏松物质,由各种有机物、无机物、微生物等共同组成。土壤是微生物、农作物、动物乃至人类赖以生存的基础。随着科学技术的发展,以及人口数量的日益增加,对有限的土壤资源进行长远高效的利用成了当下研究的热点。微生物群体几乎存在于世界的每一个生态群落之中,从动物到人类,从土壤到植被,从树木到森林,都扮演者不可或缺的角色,但是土壤中数以万亿计的微生物种类中,仅0.1%~1%的物种可培养,极大地限制了人们对于微生物群体结构及功能多样性的研究。
DNA测序技术从以Sanger法为代表的第一代测序技术发展到如今的高通量测序(HTS)技术,经历了久远而艰难的过程。高通量测序技术以成本低廉[1]、产出量高的特点,成为现代测序领域应用最广泛的方法。科研人员利用高通量测序技术在病毒分子的进化、基因组变异、微生物多样性研究与临床诊断等众多方面进行了深层次的研究。目前,高通量测序中最为成熟的组学就是宏基因组学。宏基因组学[2]诞生于20世纪,规避了分离培养,可将环境中所有的DNA提取出来,在宏观上对基因组进行整体研究,为人类了解自然界中不能被培养的微生物提供了客观全面认知的途径。宏基因组技术目前主要应用在两个方面:筛选所需功能基因和研究微生物与环境的互作。本试验以施用枯草芽孢杆菌、地特灵和常规施肥3种管理模式下的番茄土壤微生物为研究对象,利用宏基因组学研究样本中的微生物组成和互作情况,同时在分子水平对其代谢通路、基因功能进行研究[3],以期为优化、明确大棚内设施番茄施肥量及施肥种类提供一定的试验基础。
1 材料与方法
1.1 处理设计
试验于2017年3—6月在天津市武清区番茄种植大棚内进行。供试番茄品种为以色列选育的海泽拉。枯草芽孢杆菌来自保定市科绿丰生化科技有限公司,地特灵系山东农夫生物科技有限公司所产。
试验共设3个处理:对照(CK),土壤加有机肥(667 m210 m3)后直接旋耕作畦;处理1,土壤加有机肥(667 m210 m3)后,用枯草芽孢杆菌对水(667 m2100 g)喷雾土壤表面;处理2,土壤加有机肥(667 m210 m3)后,用地特灵拌细砂(667 m21 kg)均匀撒施在土壤表面,后旋耕作畦。每小区面积9.8 m2,每个处理重复3次。
1.2 测序数据预处理与分析
1.2.1 数据质控
从9个小区内番茄根际土壤取样,将样本低温运输(0 ℃以下)送往诺合致源公司。由该公司对试验样本进行DNA提取、文库构建,以及测序等操作[4]。
1.2.2 Metagonomic组装
经过预处理得到“clean data”,使用SOAP组装软件进行组装分析,将个体组装与混合组装所得到的“scaffolds”从N连接处打断,得到的序列片段称为“scaftigs”[5]。将500 bp以下的片段剔除,得到用于后续分析的基因片段(unigenes)[6]。
1.2.3 基因预测
将scaftigs进行基因预测去冗余后获得初始“gene catalogue”[7],然后将9个样品的clean data与gene catalogue比对,得出各基因片段在各样品中的数目,从数目及基因长度出发,计算出各基因在不同样本中的丰度信息[8]。
1.3 物种注释
首先,使用Diamond软件将unigenes与从美国国立生物技术信息中心(NCBI)的NR数据库中抽提出的细菌、真菌、古菌和病毒序列进行比对[9];然后,从注释结果及基因丰度出发,获得各个样品在门分类层级上的丰度信息[10]。某个物种在某个样品中的丰度,等于注释为该物种的基因丰度的加和。
1.4 常用功能数据库注释
使用Diamond软件将各样本的unigenes与KEGG数据库进行比对。对于每一条序列的比对结果,选取得分(score)最高的比对结果进行后续分析[11]。从比对结果出发,得出各样本在第一层级上的6大类功能基因相对丰度。
2 结果与分析
2.1 测序数据预处理与分析
2.1.1 数据质控
经分析,3个处理的测序有效性分别为99.61%、98.89%、99.37%。结果均已达标,可以用作后续分析[12]。
2.1.2 组装结果
CK样本组装得到的scaftigs的总长度为175 344 050 bp,条数是218 091条,平均每条scaftigs的长度为803 bp。将scaftigs按照长度进行排序,由长到短加和,当加和值达到scaftigs总长的50%时,对应的scaftig的长度为759 bp,加和值达到总长的90%时,对应的scaftig的长度为531 bp。在CK样本中,组装得到的最长scaftig的长度为97 391 bp。
处理1样本组装得到的scaftigs的总长度为162 840 829 bp,条数是193 945条,平均每条scaftigs的长度为840 bp。将scaftigs按照长度进行排序,由长到短加和,当加和值达到scaftigs总长的50%时,对应的scaftig的长度为811 bp,加和值达到总长的90%时,对应的scaftig的长度为535 bp。在处理1样本中,组装得到的最长scaftig的长度为56 220 bp。
处理2样本组装得到的scaftigs的总长度为167 676 448 bp,条数是201 937条,平均每条scaftigs的长度为822 bp。将scaftigs按照长度进行排序,由长到短加和,当加和值达到scaftigs总长的50%时,对应的scaftig的长度为765 bp,加和值达到总长的90%时,对应的scaftig的长度为531 bp。在处理2样本中,组装得到的最长scaftig的长度为110 747 bp。
2.1.3 基因预测
经分析,gene catalogue中基因数为2 019 168,其中:只含有起始密码子的基因数为411 103(占比20.36%),只含有终止密码子的基因数为504 186(占比24.97%),没有起始密码子也没有终止密码子的基因数为521 349(占比25.82%);完整基因(既有起始密码子也有终止密码子)数为582 530(占比28.85%)。gene catalogue中基因的总长为917.19 Mbp,平均长度为226.54 bp,基因的整体GC含量为61.3%。
2.2 不同管理模式下土壤微生物的群落变化
如表1所示,3个处理中土壤微生物总相对丰度排在前10位的细菌包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、酸杆菌门(Acidobacteria)、芽单胞菌门(Gemmatimonadetes)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、蓝细菌门(Cyanobacteria)、毛霉菌门(Mucoromycota)、浮霉菌门(Planctomycetes)、硝化螺旋菌门(Nitrospirae)。不同处理的优势菌分布及相对丰度存在一定差异。处理1和处理2中相对丰度排在前10位的优势种群相对丰度高于CK,且芽单胞菌和放线菌所占比例均高于CK,说明施用枯草芽孢杆菌或者迪特灵可以在一定程度上提高这两种菌的丰度。

表1 各处理的优势菌群占比 %
2.3 组间差异物种的LEfSe分析
为了进一步探究施用枯草芽孢杆菌和迪特灵后土壤微生物群落的变化,采用LEfSe分析对3个处理的菌群相对丰度进行比较[13],以找出显著性变化的菌群。对照组与处理1相比,具有显著性差异的种群共15种,由高到低依次为芽单胞菌纲、Gemmatirosa属、未分类的芽单胞菌目、未分类的芽单胞菌科、未分类的芽单胞菌纲、未分类的绿弯菌纲、未分类的绿弯菌目、Gemmatirosa_kalamazoonesis种、芽单胞菌杆菌属SCN_70_22种、芽单胞菌sp_SG8_23种、β变形菌杆菌属_RIFCSPLOWO2_12_FULL_68_19种、芽单胞菌属、未分类的绿弯菌属、芽单胞菌_sp_SM23_52属、类诺卡氏属。对照组与处理2相比,具有显著性差异的种共8种,分别为芽单胞菌种、β变形菌_杆菌属_RUFCPLOWO2_12_FULL_65_14、变形菌RUFCPLOWO2_12_FULL_68_19种、变形菌SCGC_AG_212_123种、未分类的绿弯菌科、未分类的绿弯菌目、未分类的绿弯菌纲、绿弯菌_OLB7种。
2.4 不同管理模式下KEGG代谢通路分析
将unigenes与KEGG数据库进行比对,得出在第一层级上的各处理相对丰度分析结果[14]。由表2可知, CK中第一层级上的代谢相关基因最多,占13.13%,遗传信息处理相关基因占4.45%,环境信息处理相关基因占3.81%,细胞过程相关基因占2.22%,人类疾病相关基因占1.79%,生物体系统相关基因占比最少,为0.94%。在处理1中,同样以代谢基因占比最大,为16.26%,其次为环境信息处理基因,占5.99%,再次为人类疾病相关基因,占5.52%,之后依次为遗传信息处理基因(4.48%)、细胞过程相关基因(4.17%)、生物体系统相关基因(3.69%)。在处理2中,各类基因占比由高到低依次为代谢相关基因(15.63%)、环境信息处理基因(4.57%)、遗传信息处理相关基因(4.48%)、细胞过程相关基因(2.80%)、人类疾病相关基因(2.71%)、生物体系统相关基因(1.57%)。
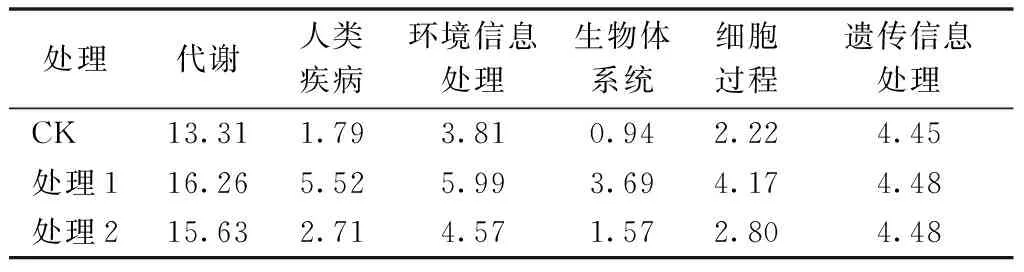
表2 各处理代谢通路相关基因占比 %
KEGG模块是一个由人工定义的功能单元集合,由M号识别,用于对序列基因组进行注释和生物学解释。下面选取了10种在KEGG代谢路径图中的致密功能单元(M00360、M00359、M00009、M00173、M00178、M00011、M00144、M00237、M00179、M00374),以期发现各处理间的功能相似性。如表3所示,M00360、M00359、M00009、M00173模块在番茄3组处理中的变化情况相同,都是在CK中最多,处理2次之,处理1最低。M00178模块在处理2中含量最高,其次为处理1,在CK中最低。M00011模块以CK中含量最高,其次为处理2,含量最低的为处理1。M00144模块以处理2含量最高,CK次之,以处理1最低。M00237模块以CK含量最高,其次为处理2,最低的为处理1。M00179模块以处理2中含量最高,其次为处理1,含量最低的为CK。M00374模块在CK中含量最高,其次为处理2,含量最低的为处理1。

表3 各处理KEGG模块分布
3 小结
组装结果显示,3个处理中组装得到的最长片段出现在施用地特灵的处理中。从平均长度来看,施用枯草芽孢杆菌处理的高于对照组和施用地特灵的处理。表明施加生物肥料,可以在一定程度上保持土壤细菌基因片段的完整性。从各处理结果来看,CK测序有效性最高,施用地特灵处理的次之,最低的为施用枯草芽孢杆菌的处理,说明番茄传统耕作模式下根基土壤微生物宏基因组中群落稳定性更高。基因数目差异分析显示,以施用枯草芽孢杆菌处理的差异最大,而施用地特灵处理的最小,说明在番茄土壤中施加枯草芽孢杆菌可以在一定程度上增加群落多样性。土壤微生物群落组成影响土壤养分循环,对于维持农田生产力与保护环境有重要意义。枯草芽孢杆菌菌体生长过程中产生的枯草菌素、多黏菌素、制霉菌素、短杆菌肽等活性物质,对致病菌或内源性感染的条件致病菌有明显的抑制作用,在农业生产中具有重要作用。本研究条件下,施加枯草芽孢杆菌与地特灵可以有效增加番茄土壤中变形菌、放线菌和蓝细菌等有益微生物的含量,由LEfSe分析结果可知,施用枯草芽孢杆菌或地特灵后,芽单胞菌、绿弯菌、变形菌等有益微生物类群显著提高,有助于提高土壤微生物活性,改善微生物结构和功能,从而实现土壤微生物生态平衡。KEGG分析发现,在番茄土壤中施用地特灵或枯草芽孢杆菌可以大幅提高代谢类基因所占比例,同时人类疾病相关基因、环境信息处理、生物体系统、细胞过程,以及遗传信息处理的基因占比均有所提高,表明番茄土壤中施用地特灵或枯草芽孢杆菌可以增强微生物代谢活性,从而提高土壤微生物功能多样性。本研究显示,施加生物肥料有利于维持微生物群落多样性和功能多样性,改善土壤理化性质,从而更好地维持土壤微生物生态平衡。

