不同天然奶酪发酵剂和非发酵剂微生物多样性的高通量测序研究
曾椿淋,朱琳,高凤,俞冰倩,徐琴,杨赛,魏巍
(1.江苏大学食品与生物工程学院,江苏镇江 212013)(2.江苏大学农业装备工程学院,江苏镇江 212013)
奶酪又称干酪,是动物乳经发酵产生的有机酸或不同风味的乳制品[1]。奶酪中的微生物分为发酵剂微生物和非发酵剂微生物两种,这些微生物在蛋白质凝固和独特风味形成过程中发挥着决定性的作用[2]。发酵剂微生物包括标注于商品标签上的人为添加已知微生物和源于原料奶及生产环境的未知微生物,它们将糖类等物质转化为有机酸或分泌凝乳酶使蛋白质凝固,也为后续微生物的生长及代谢创造适宜的环境[3]。非发酵剂微生物是除发酵剂微生物之外的微生物类群的统称,其可以分泌种类繁多的蛋白酶降解酪蛋白和多肽,产生大量氨基酸,在奶酪成熟期独特风味和质构的形成过程中发挥重要作用[4~6]。目前,人们对奶酪中微生物的研究主要集中在占据优势丰度的发酵剂微生物[7],但对相对丰度较低的非发酵剂微生物的认知不足。
因此,全面解析奶酪样品中微生物群落多样性,比较不同种类奶酪样品发酵剂和非发酵剂微生物群落结构差异,对解析奶酪蛋白质凝固和风味质构形成具有重要的参考价值。
我国市场目前销售的主要天然奶酪均为进口原酪,其商品标签上均注明了人为添加的发酵剂微生物种类。由于天然奶酪存在成熟过程,造成奶酪表皮和内部性质不同[8]。尽管人们已经明确了乳酸菌(LAB)类发酵剂微生物是天然奶酪中的主要微生物类群,但不同种类、不同部位天然奶酪微生物群落多样性是否存在差异,天然奶酪中人为添加发酵剂微生物类群是否为该奶酪中的优势微生物类群,天然奶酪中是否还存在其它发酵剂微生物类群,以及天然奶酪中非发酵剂微生物类群的多样性特征等问题,由于奶酪微生物研究方法的限制,仍尚未明确。
目前奶酪中微生物群落多样性研究主要依靠的培养法和非培养法(PLFA、PCR-DGGE和16SrRNA克隆文库等)[9],均难以确定奶酪微生物群落全貌,造成对奶酪内微生物群落多样性评价的偏差。近年来迅速发展的高通量测序技术,可以快速且准确获得样品中大多数微生物的多样性信息[10],为深度解析上述奶酪微生物群落的多样性特征提供了可行的方法。
本研究应用高通量测序技术,对不同天然软质奶酪样品进行微生物群落结构特征解析。通过对不同奶酪样品中测序文库的分析和对应微生物物种的注释,解析不同天然奶酪不同部位(表皮和内部)的发酵剂和非发酵剂微生物群落构成和特征差异、探讨天然奶酪样品中发酵剂微生物可能的来源和非发酵剂微生物中发挥成熟作用的潜在类群。为进一步解析微生物与奶酪风味质构之间的关系提供有效的基础数据。同时,凝乳酶使蛋白质凝固,并经进一步成熟而形成的具有也为发掘和分离奶酪中的微生物菌株资源奠定基础。
1 材料与方法
1.1 材料与试剂
本研究共采集江苏地区市面主要销售的5种不同进口天然软质成熟奶酪作为供试样品,5种奶酪的生产工艺包括原料乳杀菌、接种发酵、凝乳、排除乳清、盐浸、接种霉菌发酵剂和成熟等工序[11~13],各奶酪成熟的环境根据制作工厂的不同而各不相同。所有样品均购自镇江市欧尚超市,未开封样品购回后置于4 ℃冰箱保存,以用于后续试验分析。样品具体信息如下:
M牌卡门培尔奶酪,已标注的人为添加发酵剂微生物为:乳酸乳球菌乳脂亚种(Lactococcus.lactis subsp.lactis)和乳酸乳球菌双乙酰亚种(Lc.lactis subsp.cremoris)。该奶酪样品设置表皮(MCr)和内部(MC)两个处理;
M牌布里奶酪,已标注的人为添加发酵剂微生物为:乳酸乳球菌乳脂亚种(Lc.lactis subsp.lactis)和乳酸乳球菌双乙酰亚种(Lc.lactis subsp.cremoris)。该奶酪样品设置表皮(MBr)和内部(MB)两个处理;
B牌小布里奶酪,已标注的人为添加发酵剂微生物为:嗜热链球菌(Streptococcus thermophilus)、嗜酸乳杆菌(Lb.acidophilus)和白青霉菌(Penicillium candidum)。该奶酪样品设置表皮(BPr)和内部(BP)两个处理;
B牌布里布兰奶酪,已标注的人为添加发酵剂微生物为:嗜热链球菌(S.thermophilus)、嗜酸乳杆菌(Lb.acidophilus)、白青霉菌(P.candidum)和洛克福青霉(P.roqueforti)。该奶酪样品设置表皮(BBr)和内部(BB)两个处理;
P牌卡门培尔奶酪,已标注的人为添加发酵剂微生物为:乳酸乳球菌乳脂亚种(S.thermophilus)、乳酸乳球菌双乙酰亚种(Lc.lactis subsp.cremoris)和白青霉菌(P.candidum)。该奶酪样品设置表皮(PCr)和内部(PC)两个处理。研究用的试剂和试剂盒包括:Power Food Microbial DNA Isolation Kit试剂盒,美国Mobio公司;HS Taq酶,Takara试剂公司;KOD SYBR qPCR Mix试剂盒,东洋纺公司。
1.2 仪器与设备
MP*FastPrep-24样品处理系统,美国安倍医疗(MP Biomedicals)公司;电子天平,上海精科天平仪器厂;高速冷冻离心机,德国艾本德(Eppendorf)公司;PCR仪,美国Thermo公司;DYY-6C型电泳仪,北京市六一仪器厂。
1.3 方法
1.3.1 奶酪样品微生物总DNA提取及PCR扩增
奶酪样品总DNA提取参考Power Food Microbial DNA Isolation Kit试剂盒说明书。取样方式参考郭善广等[12]的方法,以天然奶酪最外缘向内7 mm作为表皮,其余部分作为内部。
以提取到的样品总 DNA为模板,对细菌 16S rDNA的V4区对应的DNA序列进行PCR扩增。以通用引物 515F (5′-GTGCCAGCMGCCGCGGTAA-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′)对细菌 16S rDNA的V4区域进行扩增[14]。采用Takara试剂公司的HS Taq酶进行PCR扩增,PCR扩增体系为:Buffer 2.5 μL、Mg2+(25 mmol/L) 2 μL、dNTP 2 μL、Primer F (515F)0.5 μL、Primer R (806R) 0.5 μL、BSA (5 mg/mL) 2.5 μL、Taq酶0.1 μL、DNA模板1 μL、加ddH2O至25 μL。反应条件为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸40 s,30个循环;72 ℃延伸10 min[14]。PCR扩增反应完成后,取5 μL扩增产物用2%琼脂糖电泳检测,若在300 bp左右处出现清晰无拖尾无弥散现象的条带,则说明扩增成功。
1.3.2 高通量测序及数据分析
将PCR扩增到的奶酪样品细菌16S rDNA委托北京诺禾致源生物信息科技有限公司在 Illumina-HiSeq平台上进行高通量测序,分析奶酪样品细菌群落多样性。对高通量测序初始数据进行质量控制,以获得更为精准、高质量的DNA序列信息,采用Mothur软件将得到的 16S rDNA基因序列在 RDP (ribosomal database project)数据库中进行嵌合体检验,充分去除嵌合体序列。为了得到每个 OTU(Operational Taxonomic Unit)对应的物种分类信息,采用 RDP classifier 贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,用 Mothur软件构建稀释性曲线[15]。然后基于有效数据进行OTUs聚类和物种分类分析,并将 OTU和物种注释结合,从而得到每个样品的OTUs 和分类谱系的基本分析结果。再对OTUs进行丰度、多样性指数等分析,利用QIIME软件计算样品Chao1指数、Shannon指数和Simpson指数[15~17]。在以上分析的基础上,进行一系列 OTUs、物种组成聚类分析与PCoA分析,挖掘样品之间物种组成差异。
2 结果与分析
2.1 不同奶酪样品细菌群落的高通量测序文库评价
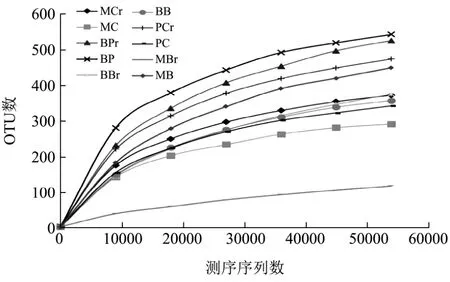
图1 细菌群落高通量测序文库稀释曲线Fig.1 Rarefaction curve of bacterial high-throughput sequencing library
根据细菌16S rDNA高通量测序文库的稀释曲线(图1),当测序序列超过50000条时,虽然仍然有新的OTU被发现,但是稀释曲线趋于平缓,达到平台期,说明测序数据量渐进合理,更多的数据量只会产生少量新的 OTU以及新得注释物种,该文库的测序结果已经基本能够代表样品细菌群落的组成。计算所有10个测序文库的覆盖率均超过99%(表1)。因此,10个样品的测序文库趋于饱和,测序结果可以用于后续的生物信息学分析。
根据奶酪样品高通量测序文库质量汇总表(表1),10个样品获得有效序列数量均超过50000条,可操纵分类单元(OTU)数量为118~646之间。Chao1指数是群落微生物的丰度指数,主要反映生态系统中物种的数目,其值越高表明群落物种的丰度越高;Shannon指数主要反映群落种数和各种间个体分配的均匀性,各种之间,个体分配越均匀,该指数就越大;Simpson指数具体表示的是在一个群落中随机取样的两个个体属于不同种的概率,该指数不会超过 1,且越大,表示该群落物种多样性越大。综合比较各个样品 OTU数量、Chao 1指数、Simpson指数和Shannon指数,发现除M牌布里奶酪和B牌布里布兰奶酪样品外,其它样品表皮比内部均具有更高的OTU数量、Chao 1指数、Simpson指数和Shannon指数,说明天然奶酪表皮的细菌种丰富度普遍高于内部。
2.2 OTU水平上天然奶酪不同部位细菌群落特征差异
韦恩图用于表示多个组共有和特有OTU的情况。从图2可以看出,5种奶酪样品表皮的OTU总数为2251,内部的OTU总数为2289,对5种奶酪的表皮和内部分别进行基于OTU的韦恩分析,发现5种奶酪表皮的具有51个共有OTU,占表皮OTU总数的2.3%;内部具有 121个共有 OTU,占 OTU总数的5.3%。各个样品表皮特有OTU数为21、54、62、142、148,P牌卡门培尔奶酪表皮的特有OTU数最多。各个样品内部特有OTU数为38、45、63、76、137,B牌小布里奶酪内部的特有OTU数最多。

表1 奶酪样品高通量测序文库信息汇总Table 1 Summary of the bacterial high-throughput sequencing library of all cheese samples

图2 不同天然奶酪表皮细菌OTU韦恩图(a),内部细菌OTU韦恩图(b)Fig.2 Venn figure of bacterial OTU from the rind (a) and core(b) of all cheese samples
为进一步对每个样品的群落结构差异进行分析,对样品进行基于UniFrac的加权的主坐标分析。即应用Unifrac方法将所有处理细菌OTU进行加权的聚类分析,即在考虑 OTU序列的数量和进化关系的基础上,计算各个样品间的距离矩阵,再对上述距离矩阵进行降维分析,结果如图3所示。基于UniFrac的加权的主坐标分析其第一主成分和第二主成分的贡献率分别为52.79%和29.18%,除PC样品外,各个样品表皮和内部之间主成分距离较大,而所有样品内部聚成一组,表皮各自分开。该结果说明天然奶酪表皮和内部之间细菌群落构成差异较大。同时,不同天然奶酪的表皮之间细菌群落结构组成差异较大,不同天然奶酪内部之间细菌群落组成结构相似。
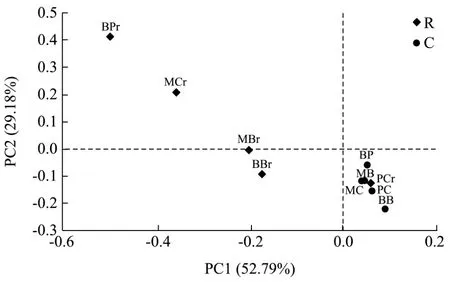
图3 不同奶酪样品细菌群落结构的主坐标分析Fig.3 Principal Co-ordinates Analysis of bacterial community of all cheese samples
2.3 不同奶酪样品发酵剂细菌和非发酵剂细菌的物种注释及分布特征
对所有OTU序列进行物种注释,共得到414个不同的细菌属。其中,各个处理中相对丰度所占比例排名前十(TOP10)细菌属的相对丰度总和超过了总数的90%。对各处理中的TOP10的细菌属组成(图4)进行统计发现,5种天然奶酪的表皮和内部最优势的细菌菌属均为发酵剂微生物所在的属,即乳球菌属(Lactococcus)、链球菌属(Streptococcus)和乳杆菌属(Lactobacillus),相对丰度占细菌总丰度的 51~97%之间。M牌不同种类奶酪的表皮和内部最优势细菌属均为乳球菌属(Lactococcus)。其中,MC奶酪的表皮和内部乳球菌属相对丰度分别为 63.55%和 91.94%,MB奶酪的表皮和内部乳球菌属相对丰度分别为 84.48%和91.20%。B牌不同种类奶酪的最优势细菌属并不相同,BP奶酪表皮和内部最优势细菌属均为乳球菌属(Lactococcus),相对丰度分别为48.30%和75.82%;BB奶酪表皮和内部最优势细菌属均为乳杆菌属(Lactobacillus),相对丰度分别为77.23%和68.83%。P牌奶酪的表皮和内部最优势细菌属均为链球菌属(Streptococcus),PC奶酪表皮和内部链球菌属的相对丰度分别为82.24%和87.40%。所有样品表皮和内部最优势的菌属均为同一细菌属。
除人为添加发酵剂细菌属之外,所有的样品中均存在的其它发酵剂细菌属。MC奶酪中人为添加乳球菌属,还存在链球菌属(表皮1.71%,内部1.24%)和乳杆菌属(表皮1.38%,内部0.97%)。MB奶酪中人为添加乳球菌属,还存在链球菌属(表皮0.42%,内部1.52%)和乳杆菌属(表皮0.09%,内部0.74%)。BP奶酪中人为添加链球菌属和乳杆菌属,还存在乳球菌属(表皮48.28%,内部75.74%)。BB奶酪中人为添加链球菌属和乳杆菌属,还存在乳球菌属(表皮 1.59%,内部2.75%)。PC奶酪中人为添加乳球菌属,还存在链球菌属(表皮 82.19%,内部 87.40%)和乳杆菌属(表皮0.91%,内部 0.54%)。这些发酵剂细菌可能来自原料奶或者制作奶酪的环境。同时,样品中最优势的并不一定是商品标注的人为添加的微生物,例如BP处理(B牌小布里奶酪)中添加了嗜热链球菌、嗜酸乳杆菌和白青霉菌,但其最优势的细菌属为乳球菌属;PC处理(P牌卡门培尔奶酪)中添加了乳酸乳球菌乳脂亚种、乳酸乳球菌双乙酰亚种和白青霉菌,但其最优势的细菌属为链球菌属。
10个样品中排名前 10的非发酵剂细菌属有Helicobacter属、Actinobacillus属、Sphingomonas属、Thermus属、Acinetobacter属、Campylobacter属。MC奶酪中最优势的非发酵剂细菌属为Thermus属(表皮0.30%,内部0.36%);MB奶酪中最优势的非发酵剂细菌属为Thermus属(表皮0.17%,内部0.99%);BP奶酪表皮中最优势的非发酵剂属为 Thermus属(0.46%),内部最优势的非发酵剂属为Sphingomonas属(4.11%);BB奶酪中最优势的非发酵剂属为Acinetobacter属(表皮0.03%,内部0.04%);PC奶酪中最优势的非发酵剂属为Thermus属(表皮0.69%,内部2.22%)。除BP奶酪以外,其余奶酪样品表皮和内部最优势的非发酵剂细菌属为同一菌属。
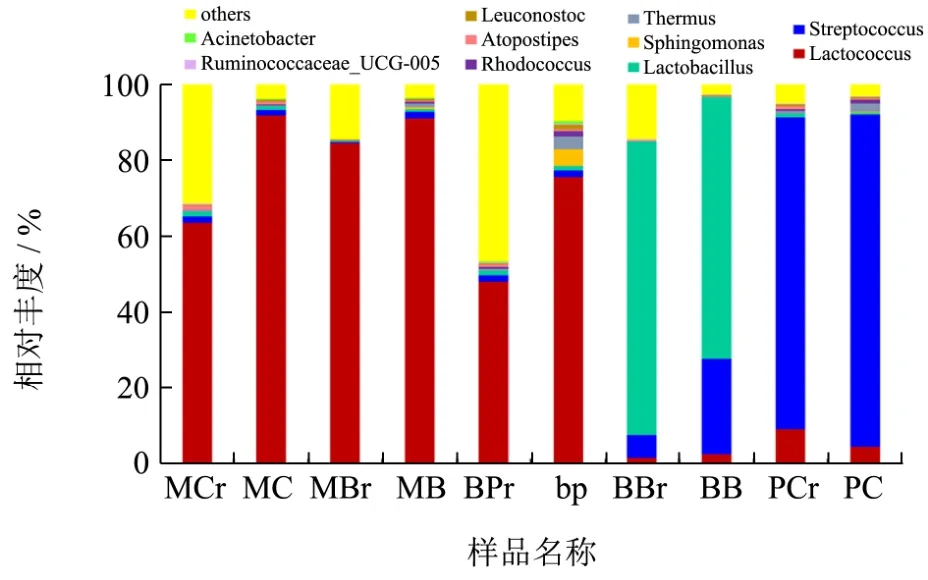
图4 不同奶酪样品TOP10的优势细菌属相对丰度Fig.4 Relative abundance of bacterial genus at TOP10 level from all cheese samples
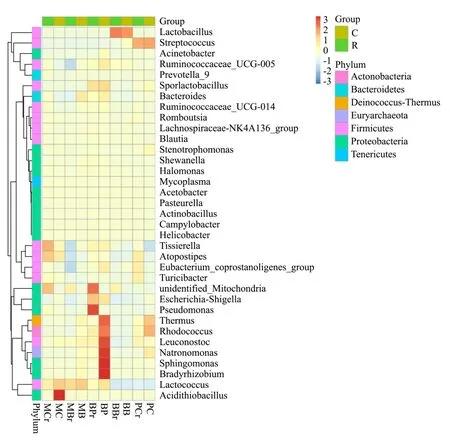
图5 不同奶酪样品TOP35的细菌属相对丰度及聚类分布热图Fig.5 Heatmap of bacterial genus at TOP35 level from all cheese samples
进一步对各处理的TOP35细菌属进行研究,绘制其相对丰度热图(图5)。结果发现除了少数分布在厚壁菌门(Firmicutes)的几个发酵剂微生物细菌所在属外,其余细菌属均为非发酵剂微生物所在的属,分布于细菌的厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、异常球菌-栖热菌门(Deinococcus-Thermus)和软壁菌门(Tenericutes)。热图可以通过颜色梯度及相似程度来反映多个样品在各分类水平上同一门类的相对丰度差异,从而体现各分类水平上群落组成的相似性和差异性。通过比较10个奶酪样品各个属相对丰度热图,发现不同进口奶酪之间发酵剂细菌所在的属相对丰度存在差异,如BBr和BB样品中Lactobacillus属相对丰度明显高于该属在其他样品中的相对丰度,PCr和PC样品中链球菌Streptococcus属相对丰度明显高于该属在其他样品中的相对丰度。有一些非发酵剂细菌所在的属相对丰度也存在明显差异,如MC样品中的非发酵剂细菌属 Acidithiobacillus属,BPr样品中Pseudomonas属,BP样品中Thermus属、Rhodococcus属、Leuconostoc属、Natronomonas属、Sphingomonas属、Bradyrhizobium属的相对丰度明显不同于这些属在其他样品中的相对丰度。
3 讨论
3.1 本研究较已报道的分离培养的方法获得了更多关于奶酪中细菌群落结构信息。传统的培养法的工作量大耗时长,又因培养基和培养条件的限制,使得检出的奶酪中微生物种类受限且无法获得种群结构,造成对奶酪内微生态系统评价的偏差。目前,通过培养法从奶酪中分离出的细菌主要集中在优势性较强的乳杆菌、嗜热链球菌、乳球菌、明串珠菌、肠球菌、片球菌等发酵剂与非发酵剂细菌属[18~22]。而本研究利用高通量测序的方法,亦获得了上述已分离到的微生物属,这些属在各个样品中的相对比例为5.5%~93.3%,同时还获得了分属于厚壁菌门、变形菌门、拟杆菌门、放线菌门、异常球菌-栖热菌门和软壁菌门等6个门的60个纲、112个目、196个科的407个不同的非发酵剂细菌属,相对丰度为2.7%~47.5%。这些详细而全面的数据为后期研究微生物与奶酪风味质构的关系提供了有效的基础信息。
3.2 在本研究中,主坐标分析结果显示同一种类天然奶酪的不同部位、不同种类天然奶酪表皮之间细菌群落结构存在明显差异,而不同品牌天然内部之间细菌群落结构相似。影响食品中微生物生长繁殖的因素包括温度、pH、水分、渗透压、氧气等理化因素,以及原料奶的动物来源、添加的原料、原料奶是否采用巴氏杀菌技术、奶酪中原料奶中自带的微生物和奶酪的酸化过程等加工工艺因素[23~25]。同一种奶酪的加工工艺条件相同,但是其不同部位的水分、渗透压等理化因素存在差异,所以同一天然奶酪表皮和内部细菌群落结构不同,很有可能是理化性质不同造成的。此外,奶酪皮暴露在外部环境中接触到了内部接触不到的菌,也有可能造成表皮和内部的细菌群落结构不一致[26]。但是不同品牌天然内部之间细菌群落结构相似,说明了更有可能是外界环境造成了天然奶酪表皮和内部细菌群落结构不一致。这一推测正确与否,还需要后续更深入的研究加以验证。
3.3 物种注释的结果表明,天然奶酪中存在的发酵剂细菌属为乳球菌属(Lactococcus)、链球菌属(Streptococcus)、乳杆菌属(Lactobacillus)等,这些细菌属也常常出现在其他奶酪和酸奶等发酵乳制品中[18,27,28]。通过分离培养的方法,就可以鉴定这些发酵剂细菌属。但高通量测序技术在鉴定出这些细菌属的同时,还可以对这些属的丰度进行比较,从而获得一些只进行物种注释研究不能得到的发现。通过对发酵剂细菌属的丰度进行比较,我们发现,即使天然奶酪中人工添加了大量发酵剂细菌,但是每个奶酪样品中除了有人工添加发酵剂细菌外,还有来自原料或者奶酪制作环境的发酵剂细菌,且在最后的奶酪商品中人工添加发酵剂细菌不一定是最优势菌属。如PC奶酪样品中的最优势细菌属是链球菌属而不是人工加入的乳球菌属,BP样品中人工加入的细菌属是链球菌属和乳杆菌属,但其最优势细菌属却是乳球菌属。出现这种现象的原因可能是奶酪在制作过程中,pH值和含水率均下降,理化性质发生了变化,食物环境发生了转变,导致奶酪中存在的微生物群体的选择[29]。外界环境因素如温度和相对湿度也影响奶酪生产和成熟期间微生物多样性的演变[30]。此外,加入的微生物同原料奶以及奶酪制作环境中的微生物相互影响,也有可能造成奶酪成品中人工加入的细菌不是最后奶酪商品中最优势的细菌。奶酪中微生物的变化是一个动态过程[31],把不同时期奶酪的微生物群落结构与理化性质及环境因素结合起来研究,将更好地揭示奶酪制作过程中微生物群落结构的演替方向。
3.4 除了发酵剂细菌属之外,天然奶酪样品中还存在大量的其他非发酵剂细菌属。TOP35属中的Halomonas属是一类能在绝对盐度(NaCl)为(0~32)%条件下生长的耐盐细菌,该属为亮氨酸氨肽酶阳性且能够利用 DL-乳酸[32];Rhodococcus属中的不透明红球菌可以产谷氨酸氧化酶,能氧化 L-谷氨酸生成 α-酮戊二酸(α-KG)、NH3和 H2O2[33];Pseudomonas属含有mgl序列,mgl序列编码甲硫氨酸-γ-裂合酶(MGL)将甲硫氨酸转化为甲硫醇,这一反应是奶酪中硫化物形成的关键步骤[34];Acetobacter属可以发酵糖类产生醋酸;Atopostipes属可以产生半胱氨酸蛋白酶;Turicibacter属可以产生Lon蛋白酶; Thermus属、Acidithiobacillus属可以产丝氨酸蛋白酶。这些微生物类群多属于目前尚未被广泛明确认知的微生物类群,都可能对奶酪的成熟、风味质构形成具有至关重要的作用。通过比较各个属相对丰度,分析非发酵剂细菌属在不同样品中的存在情况,结果发现MC样品中的非发酵剂细菌属 Acidithiobacillus属,BPr样品中Pseudomonas属,BP样品中Thermus属、Rhodococcus属、Leuconostoc属、Natronomonas属、Sphingomonas属、Bradyrhizobium属的的相对丰度明显高于这些属在其他样品中的相对丰度。猜测上述细菌属可能与MC、BP奶酪样品的某种特异性风味的形成有关。在TOP35的属中出现了不可分类微生物unidentified_Mitochondria,其相对丰度在0.1%~40.4%,B牌小布里奶酪中该序列相对丰度最高。由于测序结果与数据库中序列不匹配或相似度不够阈值,这些序列被归为不可分类的微生物。但QUIGLEY[23]等人在爱尔兰手工奶酪中亦检测到了unidentified_Mitochondria的存在,经过他们对该序列的进一步检查,发现这段序列对应于青霉属的线粒体16S RNA基因。鉴于unidentified_Mitochondria较高的相对丰度以及B牌小布里奶酪加工过程中添加了青霉属,我们推测这段序列很有可能也是来自青霉属的线粒体16S RNA基因。
3.5 此外,利用高通量测序技术也得到了更多的、之前技术不能检测但具有潜在功能的微生物类群。比如,在 10个样品中发现了之前未报道过在奶酪中存在的栖热菌属(Thermus),且该属在 10个样品的相对丰度均超过了0.02%。栖热菌是耐高温细菌,主要分布在水热环境如温泉、海底火山口、热水管道以及自热堆肥等地[35],其最适生长温度为 70 ℃~72 ℃,最高生长温度为 79 ℃,最低生长温度为 40 ℃[36],有些奶酪加工时温度能达到 50 ℃,达到该菌生长要求。张敏爱等[37]用16S rDNA克隆文库法分析生鲜牛乳中细菌种群的多样性,在鲜牛乳中也发现了栖热菌属的存在。因此,可以推测奶酪商品中的栖热菌属可能来自制作奶酪的原料乳。有研究表明栖热菌属可以产生耐热的乳糖酶[38],该属极有可能是一种可以应用于奶酪加工或者高温条件下发酵乳糖相关工业的微生物资源。由此可见,拥有庞大的数据量以及测序深度的高通量测序是分析奶酪中占少数的难以分离检测的非发酵剂微生物多样性,以及挖掘具有潜在功能微生物的有力工具。
4 结论
本研究应用Illumina-HiSeq高通量测序技术,解析了不同进口天然奶酪表皮和内部的发酵剂和非发酵剂细菌的群落结构差异和多样性组成。实验结果表明,天然奶酪表皮普遍较其内部具有更高的细菌群落丰富度,且天然奶酪表皮和内部细菌群落具有明显不同的结构组成,说明外界环境可能对天然奶酪表皮的微生物群落产生了影响。天然奶酪的发酵剂细菌属为乳球菌属、乳杆菌属和链球菌属,相对丰度在51%~97%,且奶酪制作过程中人为大量加入的发酵剂细菌属不一定是奶酪商品中最优势的细菌属;非发酵剂微生物分布于细菌厚壁菌门、变形菌门、拟杆菌门、放线菌门、异常球菌-栖热菌门和软壁菌门,相对丰度在(2~16)%,许多非发酵剂细菌具有发酵糖类、利用乳酸以及产蛋白酶等功能,对奶酪风味质构的形成有重要作用。之前未在奶酪中被报道的Thermus属的发现,也为发掘和分离奶酪中的食源性微生物资源奠定了基础。

