基于MiSeq高通量测序技术 内蒙古地区酸粥细菌多样性研究
王玉荣,折米娜,刘康玲,张振东,双 全,*
(1.内蒙古农业大学食品科学与工程学院,内蒙古呼和浩特 010018) 2.湖北文理学院食品科学技术学院,鄂西北传统发酵食品研究所,湖北襄阳 441053)
作为一种传统地方发酵食品,酸粥在我国已有上千年的制作和食用历史,因其风味独特、酸滑适口、营养丰富,而深受内蒙古中西部、陕西北部和山西西北部等地人们的青睐[1-2]。酸粥主要以糜米和大米等谷物为原料,经自然发酵制作而成,这种传统的制作方法较好地保存了当地环境中的微生物[3]。近年来,研究人员采用传统微生物学手段,对酸粥微生物多样性进行了卓有成效的研究。白梅等[4]采用传统微生物学手段,从内蒙古西部地区28份酸粥样品中分离出40株酵母菌;王炜宏等[5]对从内蒙古鄂尔多斯地区采集的酸粥样品分离的乳酸菌进行了分离鉴定,认为该地区酸粥发酵液中的优势菌群为干酪乳杆菌(Lactobacilluscasei);李文亚等[6]通过个体形态特征观察和生理生化试验等方法,对晋西北酸粥中酵母菌进行了分离鉴定,得出酿酒酵母(Saccharomycescerevisiae)、东方伊萨酵母(Issatchenkiaorientalis)和库德毕赤酵母(Pichiakudriavzevii)存在于整个酸粥发酵过程中。上述研究的开展,为明确酸粥中微生物的多样性提供了借鉴,然而这些研究多数是以微生物纯培养技术为主要研究手段,因而在研究酸粥中微生物群体的物种组成方面显示出一定的片面性和局限性,因而采用分子生物学手段,对内蒙地区酸粥中细菌多样性进行解析是必要的[7]。
Illumina MiSeq高通量测序技术因具备灵敏度高、读数长和效率高的特点,比传统微生物学手段能更加真实的反映发酵食品中微生物群落结构信息[8]。近年来,该技术已广泛应用于植物内生真菌多样性分析[9]、环境微生物群落结构监测[10]以及发酵食品中微生物多样性研究[11-12]等领域。
本文以内蒙古地区酸粥为研究对象,采用高通量测序技术与传统微生物学方法相结合的手段,对酸粥样品中细菌进行分离鉴定和多样性研究,同时使用多元统计学手段对其群落结构信息进行解析,以期为传统发酵食品中菌种发掘提供一定的理论依据。
1 材料与方法
1.1 材料与仪器
酸粥 采集自内蒙古乌拉特前旗、杭锦后旗和准格尔旗各农户家中,编号分别为A1、A2和A3,样品采集后置于含有干冰的采样箱中,运回实验室分装备用;MRS培养基 青岛海博生物技术有限公司;碳酸钙、过氧化氢、三氯甲烷、氯化钠、氢氧化钠、三羟甲基氨基甲烷 国药集团化学试剂有限公司;Axygen清洁试剂盒 康宁生命科学(吴江)有限公司;QIAGEN DNeasymericon Food Kit试剂盒 德国QIAGEN公司;FastPfu Buffer、dNTPs Mix、FastPfu Fly DNA Polymerase、5×TransStartTM、蛋白酶K 宝生物工程(大连)有限公司;6×Loading buffer、DL500 DNA Marker 宝日医生物技术(北京)有限公司;引物338F/806R(正向引物中加入7个核苷酸barcodes)、27F/1495R 均由武汉天一辉远生物科技有限公司合成。
5810R台式高速冷冻离心机 德国Eppendorf公司;DYY-12电泳仪 北京市六一仪器厂;ND-2000C微量紫外分光光度计 美国Nano Drop公司;UVPCDS8000凝胶成像分析系统 美国BIO-RAD公司;Veriti梯度基因扩增仪 美国AB公司;Miseq PE300高通量测序平台 美国Illumina公司;R920机架式服务器 美国DELL公司;SX-700高压灭菌锅 宝生物工程(大连)有限公司;DG250厌氧工作站 英国DWS公司;ECLIPSE Ci生物显微镜 日本Nikon公司。
1.2 实验方法
1.2.1 样品宏基因组提取 参照QIAGEN DNeasymericon Food Kit试剂盒中的使用方法,提取酸粥样品总DNA。
1.2.2 细菌16S rRNA PCR扩增及检测 扩增体系为4 μL 5×PCR Buffer,2 μL 2.5 mmol/L dNTPs mix,5 μmol/L 338F和806R各0.8 μL,0.4 μL 5 U/μL Taq酶,模板10 ng,用无菌ddH2O补齐至20 μL。PCR扩增程序为:95 ℃预变性5 min;95 ℃变性1 min,55 ℃退火45 s,72 ℃延伸90 s,将变性、退化和延伸三步循环次数设定为30,然后72 ℃完全延伸10 min,维持4 ℃[13-14]。扩增产物用1.0%的琼脂糖凝胶电泳(120 V,30 min)检测是否扩增出目的条带。
1.2.3 扩增产物纯化及定量 用清洁试剂盒清洁PCR产物,微量紫外分光光度计检测OD260/OD280在1.8~2.0之间的产物,用无菌ddH2O将浓度稀释至100 nmol/L后寄至上海美吉生物医药科技有限公司进行Miseq高通量测序。
1.2.4 双端序列拼接及质量控制 将双端序列拼接成一条序列,拼接过程中将同时满足以下条件的序列予以保留:重叠区域碱基数≥10 bp,最大错配率≤0.2,引物或便签的错配碱基数≤2 bp和切掉的碱基数≥50 bp[15]。
1.2.5 生物信息学分析 参照郭壮等[16]、Edgar[17]和Caporaso等[18]的方法,利用QIIME分析平台分析质控后的高质量序列:首先进行100%序列鉴定聚类分析,再根据97%相似性对这些序列进行操作分类单元(operational taxonomic units,OTU)划分;从每个OTU中选择一条代表性序列,利用RDP软件[19]和Greenbank数据库对其进行同源性比对分析,分别在门、纲、目、科和属水平对各样品的细菌多样性进行统计。
1.2.6 核酸登录号 本研究中所有序列数据已提交至MG-RAST数据库,ID号为mgp83829。
1.2.7 乳酸菌的分离鉴定 将样品摇匀后用移液枪吸取0.5 mL进行倍比稀释,取10-3、10-4和10-53个稀释度,分别涂布到含1.0%~1.2% CaCO3的MRS琼脂培养基上,37 ℃厌氧培养48 h后进行菌株纯化、生理生化鉴定及保藏。采用CTAB法[20]对纯化菌株进行DNA提取,然后进行PCR扩增、琼脂糖凝胶电泳检测、扩增产物清洁、连接pMD18-T载体并用连接产物转化大肠杆菌Top10,挑取阳性克隆送至金斯瑞生物科技有限公司测序。在NCBI(https://www.ncbi.nlm.nih.gov/)上对测序结果进行同源性比对,并与从LPSN(http://www.bacterio.net/)上下载的标准菌株一起构建系统发育树。
1.3 数据分析
使用维恩(Venn)图展示不同样品中共有或特有的OTU数,使用皮尔森(Person)相关性分析法分析各核心OTU之间的相关性。Venn图由Venny 2.1.0(http://bioinfogp.cnb.csic.es/tools/venny/index.html)在线绘制,热图由Matlab 2010b软件绘制,系统发育树由BioEdit 7.1.3、DNAMAN8和Mega 5.05绘制,其他图使用Origin 8.5软件绘制。
2 结果与分析
2.1 序列丰富度和多样性分析
本研究采集的3个酸粥样品通过MiSeq高通量测序,共产生84985条序列,经过100%序列鉴定聚类分析后,有25323条代表性序列被发现,再根据97%相似性对这些序列进行分类操作单元划分后得到334个OTU。酸粥样品16S rRNA测序结果及各分类水平数量如表1所示。
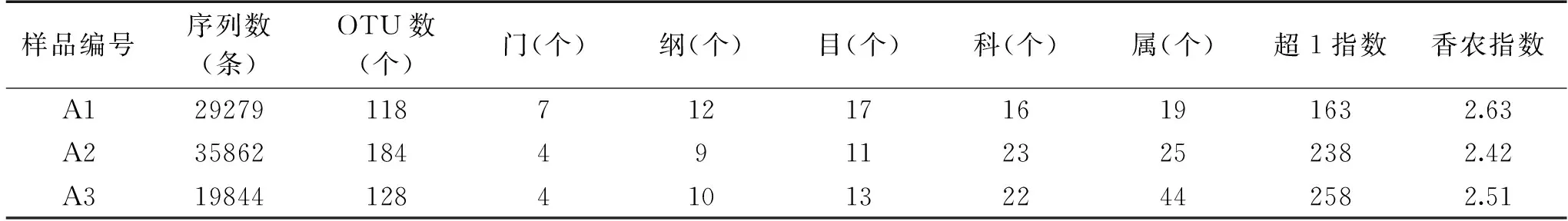
表1 样品16S rRNA测序结果及各分类水平数量Table 1 Results of 16S rRNA reads and the number of different classification
作为α多样性指数,超1指数和香农指数可以从丰度和多样性两个维度,对样品中微生物群落结构进行评价。由表1可知,样品A3的超1指数最大,而样品A1的香农指数最大,这说明A3样品具有最高的细菌物种多样性,而A1样品细菌物种丰度最大。本研究进一步采用稀疏性分析和α多样性指数分析,对取样深度和物种组成进行了综合评价[21],在97%相似性水平下,划分OTU并制作的稀疏性曲线和香农指数曲线如图1所示。
由图1可知,尽管依据当前的测序量每个样品的稀疏曲线依旧不能进入平台期(A),但是香农指数曲线已经达到饱和状态(B),这说明,当继续增加测序量时可能会有少量新的OTU被发现,但样品微生物多样性不会再有明显变化,因而本研究测序深度合适。
2.2 酸粥样品中细菌构成研究
对测得的序列进行质控后,使用RDP和Greengenes数据库对合格序列进行同源性比对,比对结果显示,84985条序列可划分为7个门、16个纲、22个目和37个属。酸粥中所有序列在门水平上可鉴定为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、蓝细菌门(Cyanobacteria)和异常球菌-栖热菌门(Deinococcus-Thermus)。3个酸粥样品中相对含量大于1.0%的优势门和属如图2所示。

图2 酸粥中优势细菌门(A)和属(B)相对含量Fig.2 Relative content of the dominant bacteria phyla(A) andgenera(B)in acidic-gruelsamples
由图2(A)可知,其中3个样品中共有且平均相对含量大于1.0%的细菌门有厚壁菌门(Firmicutes,87.45%)和变形菌门(Proteobacteria,11.84%),二者累积平均相对含量为99.28%。由图2(B)可知,在属水平上,3个样品中共有且平均相对含量大于1.0%的细菌属有乳酸杆菌属(Lactobacillus,87.23%)和醋酸杆菌属(Acetobacter,11.67%),在这一水平上仅有0.64%的序列不能被鉴定出,由此可见,酸粥中细菌主要由乳酸杆菌和醋酸杆菌构成。薛建岗等[22]分析发现了内蒙古西部地区28份酸粥样品中乳酸菌的平均含量为(7.51±1.23)lg CFU/mL;张春林等[7]采用PCR-DGGE相结合的手段,对内蒙古地区28份自然发酵酸粥样品进行研究,发现样品微生物多样性均较高,且细菌图谱较真菌图谱复杂,并得出瑞士乳杆菌(Lactobacillushelveticus)是酸粥样品中的最优势菌的结论,这些研究均与本文结果一致。
进一步对每个样品中的OTU数进行统计,由图3可知,3个样品共测得OTU 334个,仅存在于1个样品中的OTU有251个,占总OTU数的75.15%,出现2次的OTU有70个,占总OTU数的20.96%,所有样品中都存在的OTU即核心OTU有13个,占OTU总数的3.89%。基于各样品中OTU数和序列数绘制的Veen图如图4所示。

图3 OTU出现次数统计Fig.3 Occurrence number of OTU
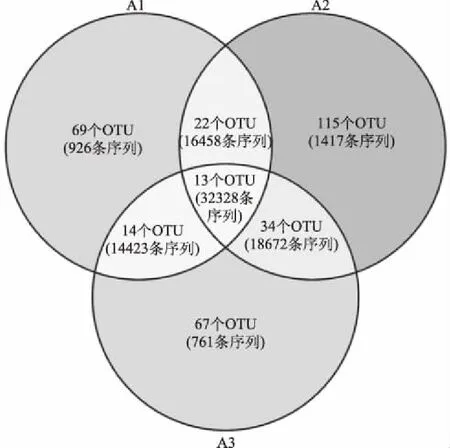
图4 基于OTU水平的Veen图Fig.4 Veen diagram based on OTU level
由图4可看出,A1、A2和A3中特有的OTU分别为69个、115个和67个,分别占总OTU数的20.66%、34.43%和20.06%,特有的序列条数及占总序列数的比例分别为926(1.09%)、1417(1.67%)和761(0.90%)。A1和A2共有22个OTU,序列16458条;A2和A3共有34个OTU,序列18672条;A1和A3共有14个OTU,序列14423条;13个核心OTU含有32328条序列,占总序列数的比例高达38.04%。以上研究表明,各酸粥样品中虽含有特有种系但含量较少,多数为共有菌群。进一步对核心OTU相对含量进行了分析,结果如图5所示。
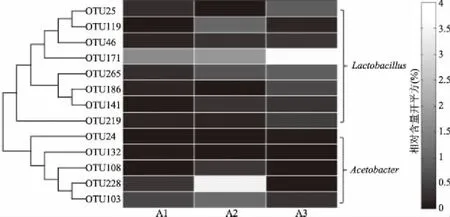
图5 核心OTU在各酸粥样品中的相对含量Fig.5 Relative content of core OTUs in each acidic-gruel sample
由图5可知,除OTU24、OTU132、OTU108、OTU228和OTU103隶属于醋酸杆菌属(Acetobacter)外其余的均属乳酸杆菌属(Lactobacillus)。各核心优势OTU所包含的序列数在各样品中存在较大差异,尤以OTU171和OTU228。OTU171在A3中的相对含量为13.57%,而在A1中的相对含量仅为2.75%;A2中OTU228的相对含量为11.62%,而A1和A3中,OTU228的相对含量仅为0.15%和0.004%。值得一提的是,OTU119(隶属于乳酸杆菌属)和OTU103(隶属于醋酸杆菌属)仅在A2中的相对含量大于1.0%。核心OTU相关性热图如图6所示。

图6 核心OTU相关性热图Fig.6 Correlation heat map of the different OTUs注:*表示差异显著(p<0.05), **表示差异极显著(p<0.01)。
由图6可看出,OTU219(属于Lactobacillus)与OTU25(属于Lactobacillus)相关性显著(p<0.05)而与OTU186(属于Lactobacillus)呈极显著正相关(p<0.01),OTU25与OTU186呈显著正相关(p<0.05);OTU119(属于Lactobacillus)与OTU228(属于Acetobacter)呈显著正相关(p<0.05),而与OTU108(属于Acetobacter)相关性极显著(p<0.01),OTU141(属于Lactobacillus)和OTU132(属于Acetobacter)呈显著负相关(p<0.05)。根据核心OTU序列进一步构建系统发育树,结果如图7所示。

图7 核心OTU系统发育树Fig.7 The phylogenetic tree of core OTUs
由图7可知,13个核心OTU大致可分成乳酸杆菌属和醋酸杆菌属两类,这与核心OTU在各酸粥样品中的相对含量分析结果一致。分析系统发育树可发现OTU25与解淀粉乳杆菌(Lactobacillusamylolyticus)较相近,OTU119与鼠李糖乳杆菌(Lactobacillushamster)较相近,OTU265与面包乳杆菌(Lactobacilluspanis)较相近,OTU171与桥乳杆菌(Lactobacilluspontis)较相近,OTU228与过氧化醋酸杆菌(Acetobacterperoxydans)较相近,OTU132与罗旺醋杆菌(Acetobacterlovaniensis)较相近;OTU103与热带醋杆菌(Acetobactertropicalis)较相近。
2.3 酸粥中乳酸菌的分离鉴定
从3个酸粥样品中共分离出9株疑似乳酸菌,生理生化实验结果显示,所有菌株过氧化氢酶实验呈现阴性,而革兰氏染色为阳性。各菌株鉴定后与标准菌株一起构建系统发育树如图8所示。
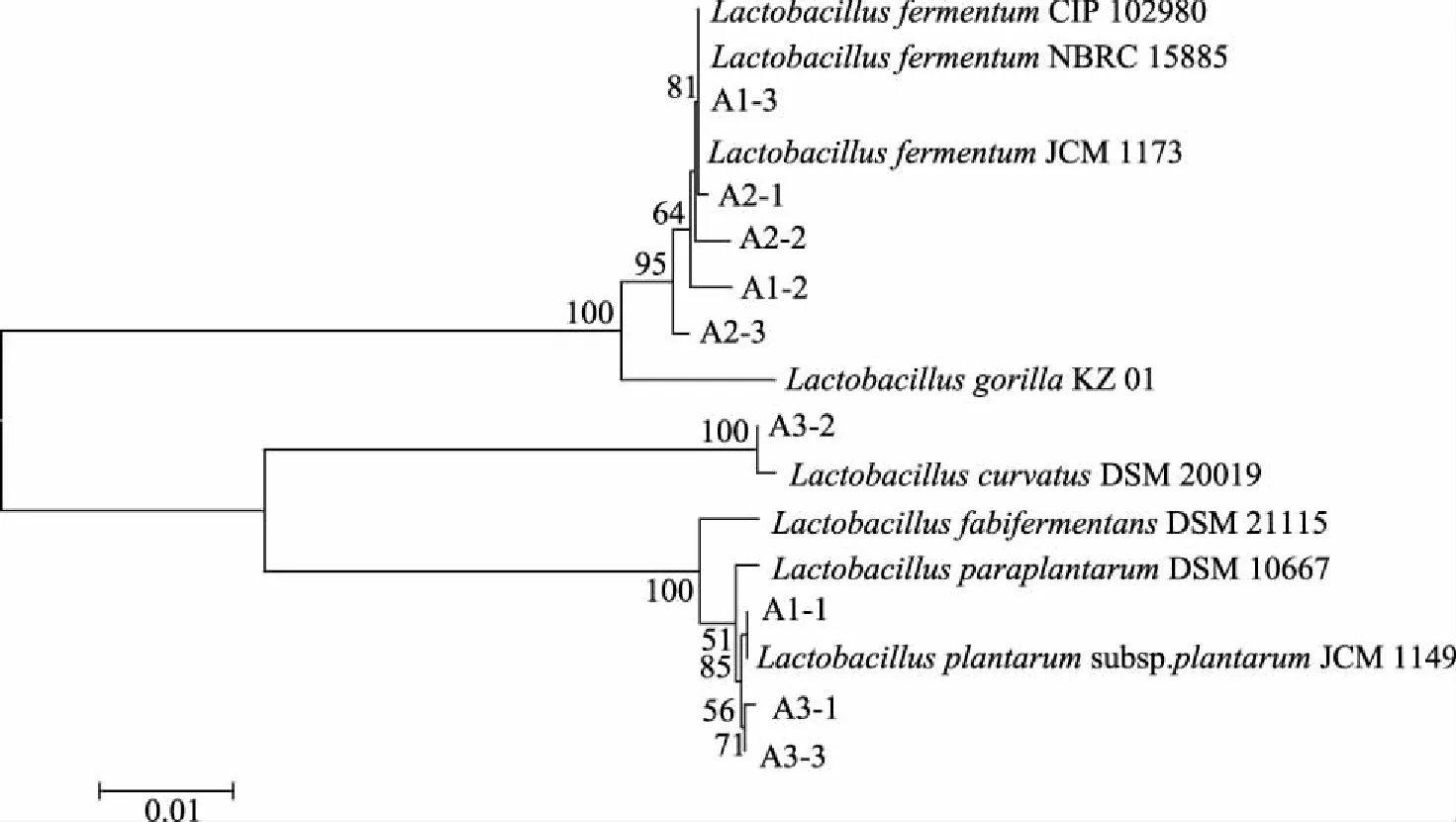
图8 乳酸菌系统发育树Fig.8 The phylogenetic tree of different strains
由图8可知,A1-2、A1-3、A2-1、A2-2和A2-3属于发酵乳杆菌(Lactobacillusfermentum),A3-2属于弯曲乳杆菌(Lactobacilluscurvatus),A1-1、A3-1和A3-3与植物乳杆菌Lactobacillusplantarumsubsp.plantarum JCM 1149的系统发育关系最近,故将其鉴定为植物乳杆菌。
3 结论
本文使用第二代高通量测序技术Illumina MiSeq与传统微生物学方法相结合的手段,对内蒙古地区酸粥样品中细菌多样性进行了研究。结果发现,所有细菌可划分为7个门和37个属,其中隶属于厚壁菌门(Firmicutes)的乳酸杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)为样品中共有且相对含量大于1.0%的细菌属,其平均相对含量分别为87.23%和11.67%。同时采用传统微生物手段,从3个样品中分离出9株潜在乳酸菌菌株,经鉴定发现有5株为发酵乳杆菌,3株为植物乳杆菌,1株为卷曲乳杆菌。由此可见,该地区酸粥中的细菌主要为乳酸杆菌(Lactobacillus)和醋酸杆菌(Acetobacter)。
一套《食品工业科技》在手, 纵观食品工业发展全貌

