尼罗罗非鱼β2m基因SNP位点和单倍型与无乳链球菌抗性的关联分析
张 栋 高风英 卢迈新 曹建萌 刘志刚 可小丽 王 淼
(1. 中国水产科学研究院珠江水产研究所,农业部热带亚热带水产资源利用与养殖重点实验室,广州 510300;2. 上海海洋大学,水产科学国家级实验教学示范中心,上海 201306)
罗非鱼(Oreochromisspp.)是我国重要的淡水养殖品种, 在国民经济中占有重要地位。尽管我国罗非鱼产业发展迅速, 但是近年来链球菌病的暴发引起的高死亡率(可达70%), 已严重威胁到罗非鱼养殖产业的健康发展[1,2]。因此, 培育罗非鱼的抗病品系是预防罗非鱼感染链球菌的重要途径之一。然而传统的育种方法存在选育周期长和优良性状遗传不稳定等缺点。随着分子标记辅助育种的不断发展, 利用分子标记辅助选育, 可以有效地缩短育种周期, 保证选育出的优良性状可以稳定遗传, 从而弥补传统选育的不足[3]。单核苷酸多态性(Single nucleotide polymorphism, SNP)是第三代DNA分子标记, 是目前最具发展潜力的分子标记。SNP标记富有代表性, 遗传稳定性高, 位点丰富且分布广泛, 检测快速, 易实现自动化分析等特点决定了其将在分子辅助育种中发挥着重要作用[4]。目前, 在水产养殖的很多物种中利用免疫相关基因进行抗病相关SNPs标记的筛选[5—12]。
鱼类中的免疫系统是防御病原体入侵的有效武器, 免疫系统分为非特异性和特异性免疫。MHC基因作为特异性免疫与非特异性免疫之间的桥梁, 在鱼类的免疫系统中发挥重要作用[13]。β2-微球蛋白是一种大约12 kD的可溶性小蛋白, 广泛存在细胞核表面, 在机体中含量稳定, 是一种含有免疫球蛋白基序的免疫球蛋白超家族成员[14]。其作为MHCⅠ类分子的重要组成部分, 是主要组织相容性复合体(Major histocompatibility complex,MHC)Ⅰ分子的亚基之一[14,15]。轻链分子β2m与重链分子MHCⅠ类(α链)通过非共价键形成稳定的异二聚体[16]。β2m还能够影响T-淋巴细胞受体(T-lymphocyte receptors, TCR)与MHC抗原肽复合物结合[17], 并且参与T细胞激活[18]。此外,β2m通过直接接触NK细胞抑制受体来进行调节NK细胞功能[19,20]。在人类和小鼠中β2m基因含有4个外显子和3个内含子[21], 然而在硬骨鱼类中含有3个外显子和2个内含子并且相对于哺乳动物和鸟类减少了2—3氨基酸残基[22]。目前草鱼(Ctenophayngodon idellus)、半滑舌鳎(Cynoglossus semilaevis)、斑点叉尾鮰(Ictalurus punctatus)、虹鳟(Oncorhynchus mykiss)、大西洋鲑(Salmo salar)和尼罗罗非鱼(Oreochromis niloticus)的β2m基因都已有研究报道[23—27]。
鉴于上述研究背景, 利用现代分子生物学技术,研究罗非鱼免疫相关基因的作用机制, 筛选出与罗非鱼抗病相关的分子标记, 实现罗非鱼抗性优良品种的培育, 对于罗非鱼养殖产业的健康的持续发展具有重要意义。本团队前期成功分离了尼罗罗非鱼β2m的cDNA及基因组序列, 并利用定量PCR检测了该基因在健康个体中的组织分布和感染链球菌以后该基因在各组织中表达的变化, 发现β2m基因在尼罗罗非鱼抗链球菌感染过程中发挥重要作用[27]。因此, 本研究以尼罗罗非鱼β2m基因为研究对象, 筛查其具有的SNP位点, 并通过SNPs与链球菌抗性及易感性状的关联分析, 筛选出与尼罗罗非鱼抗链球菌病相关SNP位点及单倍型, 为利用该分子标记进行罗非鱼抗病辅助育种奠定基础。
1 材料与方法
1.1 实验材料
实验样品采集实验中所用尼罗罗非鱼的抗病群体和易感群体均来源于中国水产科学研究院珠江水产研究所高要水产种质中心。
对2011年建立的20个家系的尼罗罗非鱼进行无乳链球菌感染, 感染后7d之内死亡的个体视为易感个体, 3个月之后仍存活的, 视为抗病个体。并将存活下来的鱼作为抗病选育基础群P0代繁殖构建F1代。
β2m基因与无乳链球菌抗性/敏感性状关联分析所用的抗性/易感尼罗罗非鱼的获得: 从F1代每个家系中均选90尾个体大小相近的健康尼罗罗非鱼平均分配到3个0.5 m3的充气水箱中, 20个家系共1800尾鱼。驯养2周后进行无乳链球菌人工感染实验(感染浓度为1×107cfu/mL, 每尾注射菌液200 μL),1周内死亡的尼罗罗非鱼视为易感群体, 而在人工感染无乳链球菌实验中始终存活的尼罗罗非鱼视为抗病群体。无乳链球菌菌株是WC1535, Ia型强毒株, 由本实验室分离鉴定并保存。
实验试剂常规PCR所用的TaqDNA polymerase、dNTP mixture、Buffer forTaqDNA Polymerase和MgCl2购于上海申能博彩生物科技有限公司; DNA提取试剂盒购于天根生化科技(北京)有限公司, 琼脂糖粉(西班牙, Biowest)。
1.2 实验方法
尼罗罗非鱼基因组DNA的提取使用TIANamp Marine Animals DNA Kit (天根)试剂盒提取尼罗罗非鱼的尾鳍基因组DNA, 参照试剂盒说明书进行提取, 1.5%琼脂糖凝胶电泳和超微量分光光度计检测其浓度和纯度, –20℃保存备用。
尼罗罗非鱼β2m基因序列SNP位点的筛查从抗病P0代20个家系中选取20尾抗病和20尾敏感个体(每个家系一尾)作为β2m基因序列SNP位点的筛查材料, 根据β2m基因序列(全长2064 bp)利用Primer Premier 5.0软件设计2对引物, 第1对引物β2m-sf1 (5′-AGTTTAGCTTCGGGAGCAGG-3′)和β2m-sr1 (5′-TGCAGCCGGTACCGGTCCGC-3′), 扩增长度931 bp; 第2对引物β2m-sf2 (5′-GCGGACCG GTACCGGCTGCA-3′)和β2m-sr2 (5′-CGCATA ATAGCCAAGAACCC-3′), 扩增长度1153 bp。PCR反应体系为50 μL: 基因组DNA 1 μL, 4× dNTP mixture 1 μL, 上下游引物(20 μmol/L) 1 μL,TaqDNA polymerase 1 μL, ddH2O 36.5 μL。PCR扩增条件: 94℃预变性3min; 94℃变性30s, 64℃退火30s,72℃延伸60s, 30个循环; 72℃延伸10min。PCR产物直接送广州立菲生物技术有限公司测序。测序结果导入DNAstar软件进行比对, 筛查SNP位点。
β2m基因序列SNP位点的基因分型根据已经筛选到SNP位点, 从F1代20个家系中随机选取102尾易感个体和102尾抗病个体尾鳍DNA, 采用snapshot法进行SNP位点基因型分型。基因分型由上海捷瑞生物工程有限公司测序完成。
数据统计分析使用PIC-CALC程序对SNP位点的分型结果计算其多态信息含量PIC(Polymorphism information content)。利用Popgen 32 软件计算其观测杂合度Ho(Observed heterozygosity)、期望杂合度He(Expected heterozygosity)、有效等位基因含量Ne(Effective number of alleles)以及哈迪-温伯格平衡(Hardy-Weinberg equilibrium,H-W)平衡(P>0.001)。利用SPSS 23.0软件对SNP位点的基因型和等位基因频率进行差异性显著检验。采用Haploview 4.2 软件对β2m序列的SNP位点进行连锁不平衡分析、标签SNP (Haplotype tag SNPs, htSNPs)的挑选以及单倍块的构建。
2 结果
2.1 β2m基因序列SNP位点及分析
利用设计的2对引物对P0代的20尾抗病群体和20尾易感群体进行扩增和测序分析, 从β2m基因序列中筛选到30个SNP位点(图1)。外显子区域的SNP2、SNP10-SNP16、SNP18-SNP24位点属于非同义突变, 其他在外显子区域的位点属于同义突变(表1)。30个SNPs位点中有4个T/C转化位点和10个A/G转化位点, 14个颠换位点和2个插入缺失位点。

图1 尼罗罗非鱼β2m基因组结构及SNP位点分布Fig. 1 Gene structure and polymorphism sites of β2m in O.niloticus

表1 尼罗罗非鱼β2m基因外显子中的SNP位点Tab. 1 The SNPs locus in the exon of β2m in O. niloticus
2.2 β2m基因SNP位点遗传参数分析
利用Popgen32软件对F1代的102尾抗病个体和102尾易感个体的β2m基因序列的30个SNPs基因分型结果进行多态性分析(表2)。易感群体的观测杂合度分布范围是0.1471—0.549; 期望杂合度的分布范围是0.1453—0.5023; 易感群体的多态信息含量的分布范围是0.1341—0.3749, 其中23个SNPs属于低度多态性(PIC<0.25), 7个SNPs属于中度多态性(0.25<PIC<0.5); 有效等位基因含量的分布范围是1.169—1.9992。抗病群体的观测杂合度分布范围是0.1275—0.4412; 期望杂合度的分布范围是0.1698—0.4943: 多态信息含量分布范围0.1546—0.3709, 其中5个SNPs属于低度多态性, 25个SNPs属于中度多态性; 有效等位基因含量的分布范围是1.2033—1.966。利用Popgen32软件分析表明易感群体和抗病群体的30个SNPs符合哈迪-温伯格平衡状态(P>0.001), 表明30个SNPs具有群体代表性可以进行下一步的连锁不平衡分析。
2.3 β2m基因SNP位点基因型和等位基因频率与无乳链球菌抗性关联分析
通过SPSS 23.0软件对β2m基因序列SNP位点的基因型和等位基因频率进行差异显著性检验, 发现24个SNP位点的基因型频率和等位基因频率与抗无乳链球菌病性状显著相关(表3), 其中SNP1的基因型GG和等位基因G、SNP5的AA和A及SNP6的II和I等24个SNPs的基因型频率和等位基因频率与无乳链球菌易感性状显著相关(表3中用下划线表示,P<0.05)。 SNP1的基因型CC和等位基因C、SNP5的GG和G及SNP6的DD和D等24个SNPs的基因型频率和等位基因频率与无乳链球菌抗性性状显著相关(表3中用波浪线表示,P<0.05)。其中有6个SNPs的基因型频率和等位基因频率差异达到极显著性水平(P<0.01)(表3)。
2.4 β2m基因SNP位点的单倍型分析及单倍型与无乳链球菌抗性关联分析
通过Haploview 4.2 软件对F1代β2m基因序列的30个SNP位点进行连锁不平衡 (linkage disequilibrium,LD)分析(图2)。根据Wang等四配子检验法(Four Gamete Rule)将β2m基因序列的30个SNP位点分为四个单倍块(图2)[28]。SNP1和SNP2两个位点构成单倍块1, SNP3-SNP12构成单倍块2, SNP14-SNP28构成单倍块3, SNP29和SNP30构成单倍块4。单倍块由几种单倍型构成及各个单倍型的频率见表4。标签SNP分析结果显示, 单倍块2中SNP8、SNP9、SNP11、SNP12这4个位点高度连锁(r2>0.9), 可选择其中一个代表另外3个位点; 单倍块3中的SNP14—SNP26这13个位点彼此之间完全连锁(r2=1,D=1), 每个位点均可作为单倍块3的标签SNP(htSNPs)。
利用Haploview 4.2软件分析得出单倍型H-1-1、H-2-1、H-3-2和H-4-2与无乳链球菌易感性状显著相关(P<0.05), 单倍型H-1-3、H-2-3、H-3-3和H-4-1与无乳链球菌的抗性性状显著相关(P<0.05)(表4)。

表2 30个SNP位点在尼罗罗非鱼易感群体和抗病群体的遗传信息Tab. 2 The genetic polymorphism information of 30 SNPs in the susceptible group and resistant group in O. niloticus
3 讨论
目前, 链球菌病是导致罗非鱼养殖产量降低的主要病害, 培育罗非鱼的抗病品系可以有效的防止链球菌病的侵害, 本团队在前期发现通过筛选与抗病相关的SNP位点可以加速选育抗病品系的进程。在本研究中从β2m基因序列中共筛选出30个SNP位点, 1个SNPs位于5′UTR, 17个SNPs位于外显子区域其中16个非同义突变位点, 一个同义突变位点, 9个SNPs位于内含子区域, 4个SNPs位于3′UTR。虽然位于内含子区域的SNP位点并不编码蛋白和直接进行表达, 但是可以影响基因的表达效率[29]。多项研究也表明内含子对多种哺乳动物中的基因表达有显著的积极作用[30,31]。因为内含子通过剪切从而影响mRNA代谢的几乎所有过程, 所以内含子对基因的表达调控具有重要作用[32]。非编码区的SNP位点可能在转录水平上影响转录因子结合位点、剪接位点等功能位点, 而编码区的SNP可引起氨基酸变化并改变翻译的蛋白质的功能或结构性质[33]。同义突变的SNP位点并不改变编码的氨基酸序列, 但是同义突变可能影响少数密码子共翻译折叠时间, 从而影响蛋白质的结构和功能[34,35]。本研究中外显子区域的同义突变位点和内含子区域中3个SNP位点与无乳链球菌的抗性显
著相关。多项研究证明, 内含子区域和外显子区域的同义突变的SNP位点和抗病相关。Fu等[6]在罗非鱼中发现MCP-8基因内含子区域的SNP1的基因型和等位基因频率与无乳链球菌的抗性显著相关, 在亚洲海鲈的LECT2基因发现内含子区域的SNP2位点等位基因在感染大腹部病(Big belly disease)的抗病群体和易感群体中的分布频率存在显著差异[10]。Pant等[36]发现牛(Bovine)的PGLYRP1基因外显子区域的同义突变位点SNP c.480G>A与感染MAP(Mycobacterium aviumssp.paratuberculosis)的易感性状显著相关。β2m基因序列外显子区域SNPs较多,这与Fu等[6]在罗非鱼的MCP-8基因中发现的SNPs类似。SNP位点的He、Ho、Ne和PIC反应的是该位点遗传多样性高低的一个指标。本研究发现易感群体中7个位点属于中度多态水平, 抗病群体中25个位点属于中度多态水平, 从而表明F1代尼罗罗非鱼的抗病群体保持了较高的遗传多样性, 可以用于后续的品种选育[37]。

表3 尼罗罗非鱼β2m基因在易感群体和抗病群体的SNPs统计分析Tab. 3 The SNPs distributions in both susceptible group and resistant group of β2m in O. niloticus
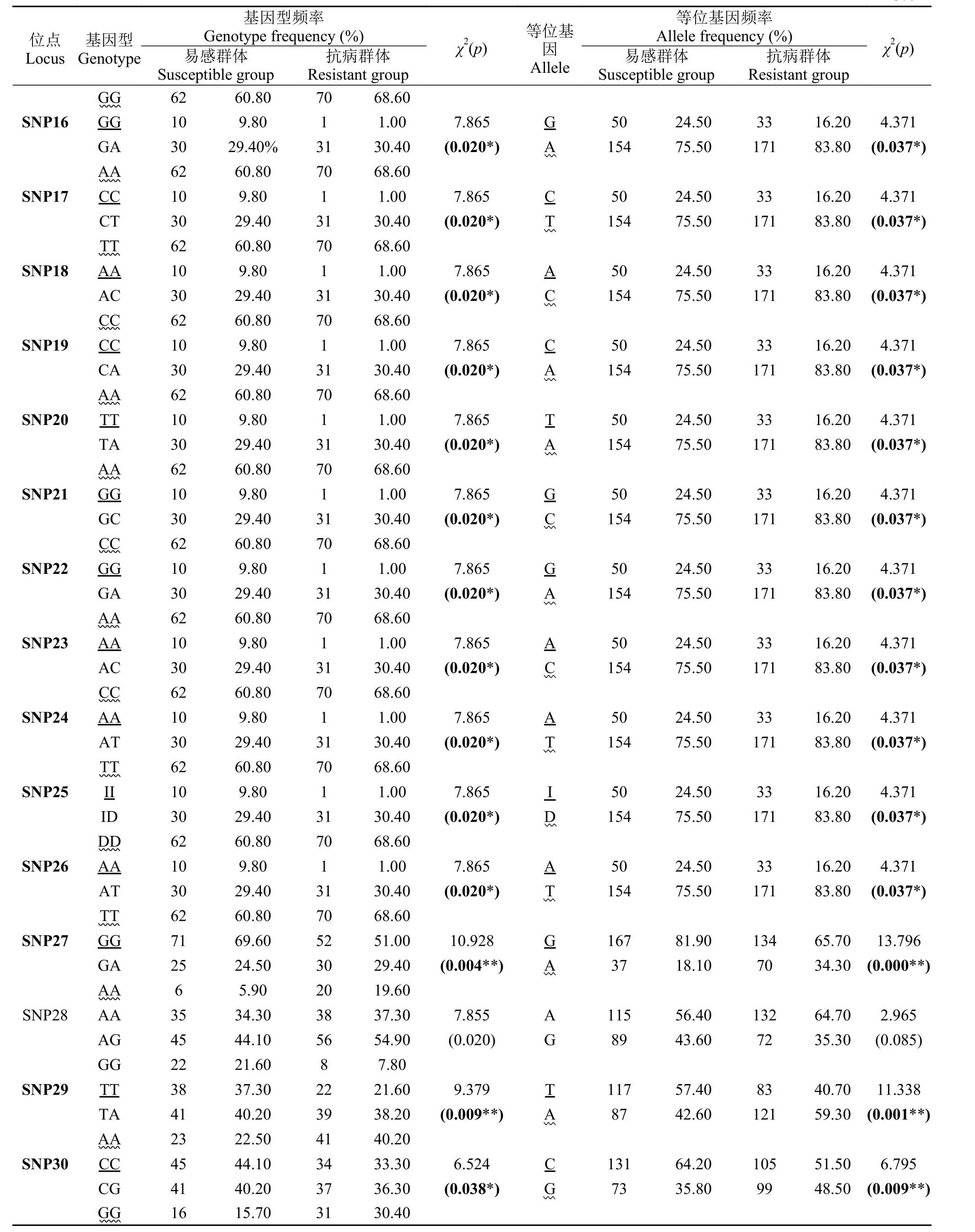
续表3

图2 尼罗罗非鱼β2m基因SNPs连锁不平衡分析Fig. 2 Linkage disequilibrium map of pair-wise LD between the SNPs of β2m in O. niloticus

表4 尼罗罗非鱼β2m基因单倍型与抗无乳链球菌感染的关联分析Tab. 4 Association of β2m haplotypes with the resistance to S. agalactiae in O. niloticus
本研究结果显示30个位点构成4个单倍块和14种单倍型, 其中单倍块2的4个SNP位点和单倍块3中的13个SNP位点各自彼此之间高度连锁(r2>0.9),单倍块2的4个SNP位点中的任何一个, 均可代表另外3个位点; 单倍块3中的任何一个SNP位点均可作为单倍块3的htSNPs, 发现的htSNPs使我们对30个SNPs的研究减少到15个, 本研究从F1代β2m基因序列中筛选到24个SNPs的基因型和等位基因频率与无乳链球菌抗性/易感性状显著相关, 这24个位点包括单倍块2和单倍块3的2个htSNPs。 htSNPs的出现不仅大大减少了我们后续检测与抗病群体显著相关的SNP位点, 而且使我们对整个基因组序列的单倍块和单倍型有了更深一步的了解[38]。到目前为止在罗非鱼的补体C9基因中发现2个SNPs[39],duodenase-1 基因中发现4个SNPs[7],MCP-8基因中发现3个SNPs与无乳链球菌无乳链球菌抗性/易感性状显著相关[6]。单倍型作为被染色体重组热点打断的区域, 相对与单个SNP分析提供更多的遗传关联依据, 为与疾病的关联分析研究提供了新的思路[40]。本研究从14个单倍型中发现其中4个单倍型与无乳链球菌抗性性状显著相关, 4个单倍型无乳链球菌易感性状显著相关。
4 结论
本研究通过对F1代尼罗罗非鱼β2m基因的SNPs筛选中发现24个SNPs的基因型和等位基因频率与无乳链球菌抗性/易感性状显著相关, 以及2个ht-SNPs。单倍型H-1-3、H-2-3、H-3-3和H-4-1 (与无乳链球菌抗性性状显著相关(P<0.05); 单倍型H-1-1、H-2-1、H-3-2和H-4-2与无乳链球菌易感性状显著相关(P<0.05)。上述β2m基因分子标记为辅助罗非鱼抗病选育提供了基础研究资料, 对加快罗非鱼抗病品系的培育具有重要意义。

