基于TCGA数据库建立的八基因预后模型在乳腺癌中的应用
贾晓晨,贾勇圣,孟文静,佟仲生
乳腺癌是全球女性最常见的恶性肿瘤之一,发病率正在逐年上升,死亡率居女性恶性肿瘤的第二位,因此建立准确预测乳腺癌患者预后的工具对于指导临床诊治至关重要[1−2]。目前临床上主要通过年龄、肿瘤大小、淋巴结转移情况以及组织学分级等临床病理特征评价乳腺癌患者预后[3]。乳腺癌是在分子水平上高度异质性的肿瘤,根据雌激素受体(ER)、孕激素受体(PR)、人表皮生长因子受体−2(HER−2)、Ki−67蛋白表达的结果将乳腺癌分为Luminal A型、Luminal B型、HER−2过表达型及Basal−like型,不同分子亚型的乳腺癌患者的预后差异明显[4−5]。癌症基因组图谱数据库(The Cancer Genome Atlas,TCGA)涵盖33个癌种,包含11 000例患者的肿瘤样本和与之对应的正常组织样本,是由美国国家癌症研究所(National Cancer Institute,NCI)及美国国家人类基因组研究所(National Human Genome Research Institute,NHGRI)启动并完成的对人类肿瘤基因组进行大规模测序得到的基因组变异图谱[6−7]。本研究利用TCGA数据库中的乳腺癌数据分析乳腺癌样本和正常样本中差异表达的基因,从中筛选并建立可以独立预测乳腺癌患者预后的多基因预后模型,并在总体乳腺癌患者和根据临床病理特征分组的各乳腺癌亚组中验证该模型的预测价值。
1 资料与方法
1.1 一般资料 从TCGA数据库中选取乳腺癌患者的mRNA数据,同时下载与患者相关的临床数据。提取的临床数据信息包括总生存期(overall survival,OS)、年龄、ER状态、HER−2表达情况、肿瘤大小、淋巴结转移情况和临床病理分期。OS定义为从患者诊断日期开始到死亡日期截止。本文所有下载数据均截止于2017年12月25日。
1.2 方法 应用R语言软件edgeR包对下载的数据进行表达值的标准化,采用DEGseq包分析和筛选出在乳腺癌组织样本和正常组织样本中的差异表达的基因,即差异基因(differentially expression genes,DEGs)。采用Survival包通过单因素Cox回归分析对DEGs进行筛选,筛选出影响患者总体生存期的DEGs,纳入多因素Cox回归分析建立多基因预后预测模型并计算预后评分(prognostic index,PI),根据评分中位值将患者分为高风险组及低风险组。临床特征分组及变量赋值如下:根据年龄分为高年龄组(≥60岁),低年龄组(<60岁);根据ER状态分为ER阳性组和ER阴性组;根据肿瘤大小分为T1组(肿瘤最大径≤2 cm),T2组(2 cm<肿瘤最大径≤5 cm),T3组(肿瘤最大径>5 cm),T4组(侵犯胸壁及皮肤);根据淋巴结转移状态分为淋巴结阳性组和淋巴结阴性组;根据美国癌症联合委员会(American Joint Committee on Cancer,AJCC)乳腺癌临床病理分期分为Ⅰ期组、Ⅱ期组、Ⅲ期组和Ⅳ期组。
1.3 统计学方法 采用R语言edgeR和DEGseq包筛选差异基因,差异倍数(Fold Change,FC)采取log2处理,筛选标准为:∣log2FC∣>1且P<0.05。通过R语言Survival包进行单因素和多因素Cox比例回归模型筛选,并建立多基因预后模型。通过SPSS 22.0软件将临床病理因素及预后评分纳入单因素和多因素Cox回归模型分析乳腺癌患者生存的影响因素,P<0.05为差异有统计学意义。生存分析采用Kaplan−Meier(K−M)法,根据年龄、ER状态、HER−2表达情况、淋巴结转移状态及病理分期,验证多基因预后模型在总体和各亚组乳腺癌患者中的预后预测价值;受试者工作特征(ROC)曲线用来评价多基因预后模型的有效性和敏感性,计算曲线下面积(AUC)。
2 结果
2.1 TCGA数据库中选取的乳腺癌患者的临床特征 TCGA数据库中共下载了1 109例乳腺癌组织样本和113例正常组织样本的mRNA表达信息,同时下载1 097例乳腺癌患者的临床信息,对可以提取到完整临床数据的709例乳腺癌患者的相关数据进行整理并分析,纳入患者的临床特征见表1。
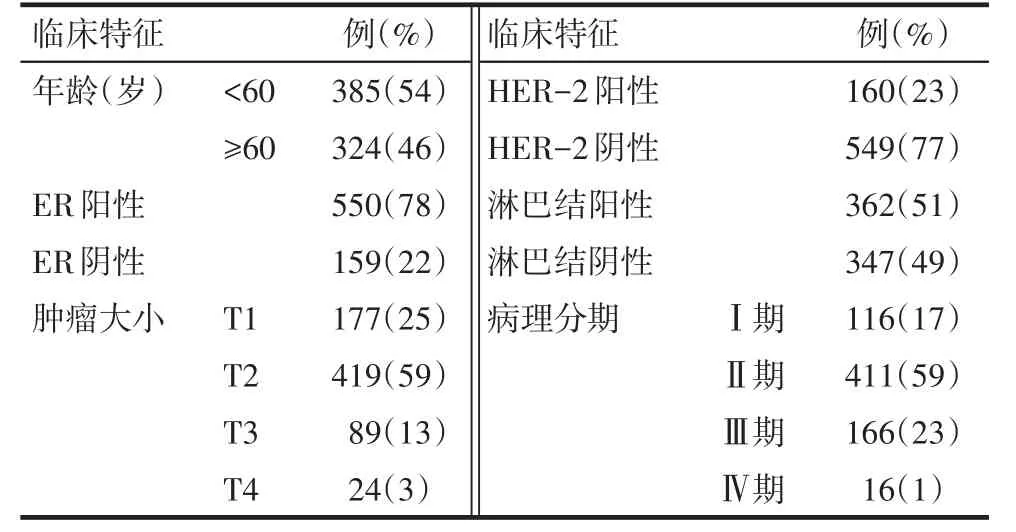
Tab.1 Clinical characteristics of 709 patients with breast cancer in TCGA database表1 TCGA数据库中709例乳腺癌患者的临床特征
2.2 差异基因的筛选以及多基因预后模型的建立 筛选出乳腺癌组织和正常组织中差异基因共2 142个,其中上调1 353个,下调789个。2 142个差异基因经过单因素Cox回归模型筛选后,共有18个差异基因可能影响患者的总体生存期,分别是CEL、POU3F2、CYP24A1、FABP7、LOC100190940、MURC、MYBPC1、TUBA3D、GCGR、NPY1R、LRP1B、FGFBP1、PRSS2、SLC25A47、ARPP21、LINC01854、DLGAP1-AS4及IGF2BP1。将上述18个基因进行多因素Cox分析后,共筛选出羧基酯脂肪酶(CEL)、POU区域转录因子(POU3F2)、维生素D−24羟化酶(CYP24A1)、脂肪酸结合蛋白7(FABP7)、MURC、G蛋白偶联受体(GCCR)、低密度脂蛋白受体相关蛋白−1B(LRP1B)及丝氨酸蛋白酶2(PRSS2)等8个基因并建立预后预测模型,见表2。预后评分公式为:PI=0.156×CEL的表达量+0.112×POU3F2的表达量-0.071×CYP24A1的表达量-0.065×FABP7的表达量+0.135×MURC的表达量-0.201×GCGR的表达量-0.063×LRP1B的表达量-0.090×PRSS2的表达量。根据预后评分公式计算709例患者的中位PI值为0.98,将评分<0.98的354例患者纳入低风险组,评分≥0.98的355例患者纳入高风险组。
2.3 乳腺癌患者生存影响因素的Cox回归分析 纳入Cox回归分析的自变量赋值如下:年龄(岁),ER状态(阴性=0,阳性=1),HER−2状态(阴性=0,阳性=1),肿瘤大小(T1期=1,T2期=2,T3期=3,T4期=4),淋巴结状态(阴性=0,阳性=1),病理分期(Ⅰ期=1,Ⅱ期=2,Ⅲ期=3,Ⅳ期=4),八基因模型(PI<0.98=0,PI≥0.98=1);因变量赋值如下:总体生存期(天)及生存状态(存活=0;死亡=1)。单因素Cox回归模型分析结果显示,年龄、肿瘤大小、淋巴结转移情况、病理分期及八基因预后模型可能是乳腺癌总体生存期的影响因素(P<0.05),见表3。将可能影响乳腺癌预后的因素纳入多因素Cox回归模型分析,结果显示年龄、病理分期、八基因预后模型是乳腺癌患者预后的独立影响因素(P<0.05),见表4。

Tab.3 Factors related with overall survival for 709 patients with breast cancer in univariate Cox proportional hazard analysis表3 单因素Cox回归分析影响709例患者总体生存期的因素
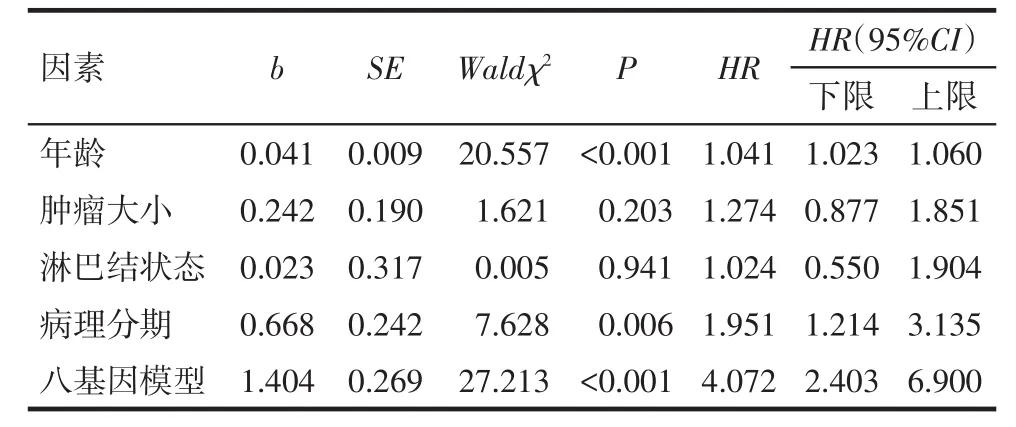
Tab.4 Factors related with overall survival for 709 patients with breast cancer in multivariate Cox proportional hazard analysis表4 多因素Cox回归分析影响患者总体生存期的因素
2.4 八基因预后模型与各临床病理特征关系 在共709例患者中,将低风险组和高风险组的乳腺癌患者的总生存期进行对比,结果显示低风险组的中位OS(2.75年)较高风险组(1.92年)明显延长,总生存率优于高风险组(Log rankχ2=63.700,P<0.01),见图1A。ROC曲线的AUC值为0.707(零假设:实面积=0.5,P<0.001),提示八基因模型预测预后能力较好,见图1B。除病理分期Ⅳ期外,在各亚组中,预后评分为低风险组患者的总生存率高于高风险组(P<0.01),见表5。
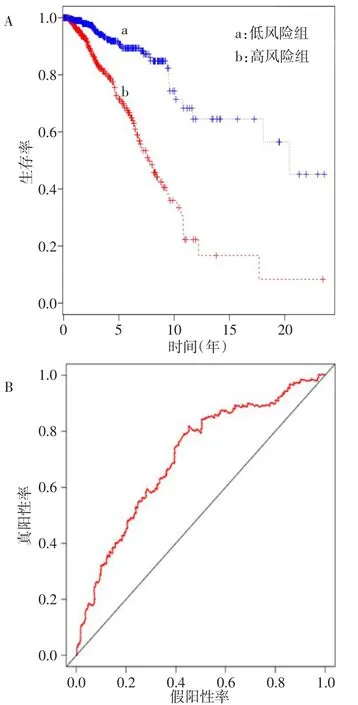
Fig.1 Kaplan−Meier plots(A)and ROC curves(B)of overall survival in 8−gene signature and prognosis in 709 patients with breast cancer图1 709例乳腺癌患者中八基因预后模型与预后关系的生存曲线Kaplan−Meier分析(A)及ROC曲线分析(B)
3 讨论
本研究建立了八基因预后预测模型,在总体乳腺癌患者及各乳腺癌亚组中进行了验证,证实八基因预后模型可以预测乳腺癌患者的预后。
CEL参与肝脏中胆固醇酯的摄取及降解过程,影响脂类的代谢,有研究证实CEL突变可能与非酒精性脂肪肝相关肝细胞癌和胰腺癌的发生有关[8−9]。POU3F2通过调节肿瘤相关NADH氧化酶的表达水平参与肿瘤形成的过程,POU3F2过表达可能会促进胃癌细胞的增殖、迁徙及侵袭[10]。CYP24A1被证实在多个癌种中过表达,目前已经证实CYP24A1的过表达与结直肠癌、乳腺癌的发生密切相关[11−12]。在胶质母细胞瘤、肾透明细胞肿瘤中,细胞的增殖、浸润都可能与FABP7的表达上调有关,也有报道证实,在基底样乳腺癌中FABP7基因高表达预示着较差的预后[13−15]。MURC/cavin−4与小凹蛋白一起参与调节心肌功能和小凹结构的形成和功能,有报道称MURC/cavin−4基因沉默阻止了横纹肌肉瘤细胞的分化[16−17]。GCGR 是 B 类 G 蛋白偶联受体(G protein coupled receptor,GPCR)的一员,参与维持机体血糖的稳定,是2型糖尿病病生理过程的重要影响因素[18]。LRP1B是低密度脂蛋白(LDL)家族的一员,有研究证实,LRP1B可能是非小细胞肺癌和胃癌的肿瘤抑制因子[19]。多个研究报道,PRSS2可以减轻胰腺内胰蛋白酶活性,对慢性胰腺炎起到保护作用[20]。
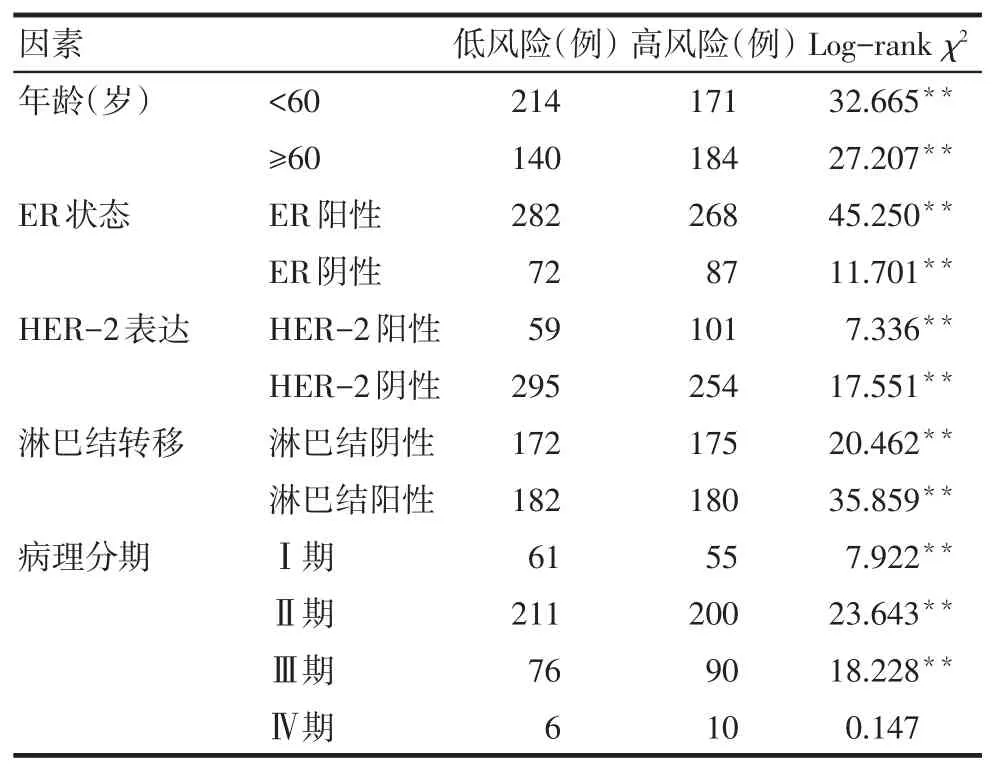
Tab.5 Association between eight-gene signature and prognosis in patients of subgroups(Kaplan-Meier method)表5 八基因模型与各亚组乳腺癌患者预后的关系(Kaplan-Meier方法)
目前乳腺癌预后预测工具包括第一代预后标志物(21基因检测,MammaPrint,Genomic Grade Index)[21]和第二代预后标志物(Prosigna,EndoPredict,乳腺癌指数)[22]。美国国家癌症网络(NCCN)指南推荐乳腺癌21基因检测适用对象为:Ⅰ期或Ⅱ期、ER阳性、淋巴结阴性的新确诊乳腺癌;淋巴结阳性(1~3个)、ER阳性的绝经后浸润性乳腺癌患者,可以评估是否需要化疗。目前现有的乳腺癌预后标志物仅适用于ER阳性的早期乳腺癌,对于ER阴性、淋巴结转移大于3个的乳腺癌患者仍缺乏有效的预后预测手段[23−24]。在本研究中,八基因模型在不同临床病理分组(Ⅳ期除外)的患者中均具有较好的预后预测价值,特别是对于淋巴结阳性,病理分期Ⅱ期和Ⅲ期以及HER−2阴性和阳性的患者。
本研究仍存在许多不足和问题亟待解决:(1)多基因预后模型的预测能力仍需大量多中心的循证医学证据证实。(2)多基因预后模型对于不同亚型乳腺癌患者的治疗方案选择及治疗周期的指导意义尚不明确。(3)由于经济、技术等局限性,多基因检测模型在未来临床应用的前景仍不明确。(4)被纳入的多基因模型的基因功能和参与的机制尚不明确,与乳腺癌的发生、发展的关系仍需要大量的研究进一步印证。
建立多基因预后模型比临床病理指标更能提供精确的预后评价指导,可以减少医疗资源的浪费,对个体化治疗方案的选择具有重要的参考价值,使患者从临床获益。

