串联重复序列在高粱基因组中的特征及分布
赵志新,李春鑫,李雨娇
(1.商洛学院 生物医药与食品工程学院,陕西 商洛 726000;2.河南省农业科学院 小麦研究所 分子育种研究室,河南 郑州 450002)
高粱[Sorghumbicolor(L) Moench]为禾本科高粱族高粱属植物,是一种综合利用价值高的粮食、饲料等多用途的农作物[1]。高粱适应能力强,具有较强的抗旱、耐寒、耐盐碱等特性,在干旱和半干旱农业生产中占有极其重要的地位,因此,从基因组角度研究重复序列在高粱中的特征及基因调控机制对高粱分子育种及抗性特征分析具有重要的借鉴意义[2]。
串联重复序列(Tandem repeat,TRs),主要指1~200 bp的核心重复单位的重复序列,其广泛存在于真核生物和一些原核生物的基因组中,并表现出种属、碱基组成等的特异性[3]。在同一物种基因组中,串联重复序列在编码区和非编码区都有分布,并且在非编码区大量存在[4]。随着计算技术的进步及高通量数据分析的出现,重复序列研究已不仅仅局限于微卫星等短重复序列(通常指1~10 bp),中等重复序列(>10 bp)也已经被广泛研究,并且研究显示,这些重复序列在植物及绿藻的基因转录及翻译调控中扮演着重要的作用[5]。
本研究利用Phytozome数据库,下载高粱全基因组及基因组注解数据,然后使用Phobos软件分析1~50 bp重复单元在基因间和基因内的密度和分布特征,以便阐明重复序列在高粱基因组中的特征及可能的生物学功能。
1 材料和方法
1.1 高粱基因组数据的获得
从Phytozome数据库(http//www.Phytozome.net/)下载高粱(Sorghumbicolorv2.1)的全基因组及基因注解数据。其全基因组为738.54 Mb,有效基因组为697.58 Mb,这里的有效基因组指测序中可测的A、T、G、C 4种核苷酸。
依据真核基因结构图(图1),本研究中每个基因分为基因内区域[包括5′UTR、CDS(基因编码区)、Intron(内含子)和 3′UTR]和基因间区域(包括上游基因间隔区和下游基因间隔区),主要研究串联重复序列密度及串联重复序列分布[5]特征。
1.2 串联重复序列的检测和分析
为了搜索完美匹配和不完美匹配的串联重复序列,使用全基因组串联重复序列的搜索工具(Phobos version 3.3.12)[6]。考虑所需处理高粱基因组的计算资源和执行时间,采用1~50 bp作为重复单位的大小,检测重复序列的最小长度被设定为12 bp。对于循环的串联重复序列,按照字母顺序只有一个序列模体被选择为代表[5],例如AAG、AGA和GAA都是(AAG)n的重复单元。此外,分别检测串联重复序列以及它的反向互补序列(例如,AAG和CTT),这是因为正链和负链上的基因涉及到正义和反义转录[7],因此需要强调基因定位(正链或负链)的重要性,类似的策略已经被他人采用[8]。
串联重复序列的密度被定义为每兆碱基对(Mb)含有的串联重复序列的碱基对数(bp/Mb),表示串联重复序列长度在总检测序列长度中所占的比例。为了研究串联重复序列在不同区域的分布数量,首先将所研究的基因内和基因间区域长度规定为0~99(即百分化),然后切分为0~9、10~19、……、90~99共10个子区域,分别计算串联重复序列在每一个子区域出现的频数,这样所有基因间和基因内区域的串联重复序列数目就具有可比性[5]。
2 结果与分析
2.1 高粱基因组中1~50 bp串联重复序列的密度分析
在整个高粱基因组中(697.58 Mb),5′UTR中的串联重复序列密度最高,为25 655 bp/Mb,其次为UI200(16 761 bp/Mb)和UI500(10 718 bp/Mb),3′UTR中的串联重复序列密度最低,为5 710 bp/Mb,其余则为6 000 bp/Mb左右。5′UTR和UI200中的串联重复序列密度最高,这可能与启动子的保护和RNA聚合酶对启动子的识别有关(图2)。
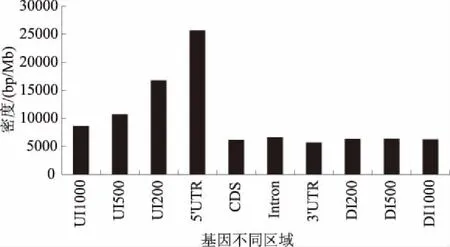
图2 基因不同区域串联重复序列密度
全基因组中1~50 bp串联重复序列密度排在前7位的依次是二碱基(1 123 bp/Mb)、三碱基(996 bp/Mb)、六碱基(650 bp/Mb)、二十一碱基(510 bp/Mb)、四碱基(438 bp/Mb)、五碱基(278 bp/Mb)和单碱基(253 bp/Mb),其中除了二十一碱基,其余全是微卫星DNA(图3)。二碱基和三碱基为主要的重复单元,其密度分别占总密度的17.04%和15.12%。二碱基重复类别中,密度最大为AT (746 bp/Mb),CG (9 bp/Mb)密度最小。三碱基重复类别中,重复密度较大的为ATT (108 bp/Mb)和AAT (107 bp/Mb),密度较小的为GAT (21 bp/Mb)和GGT (20 bp/Mb)。
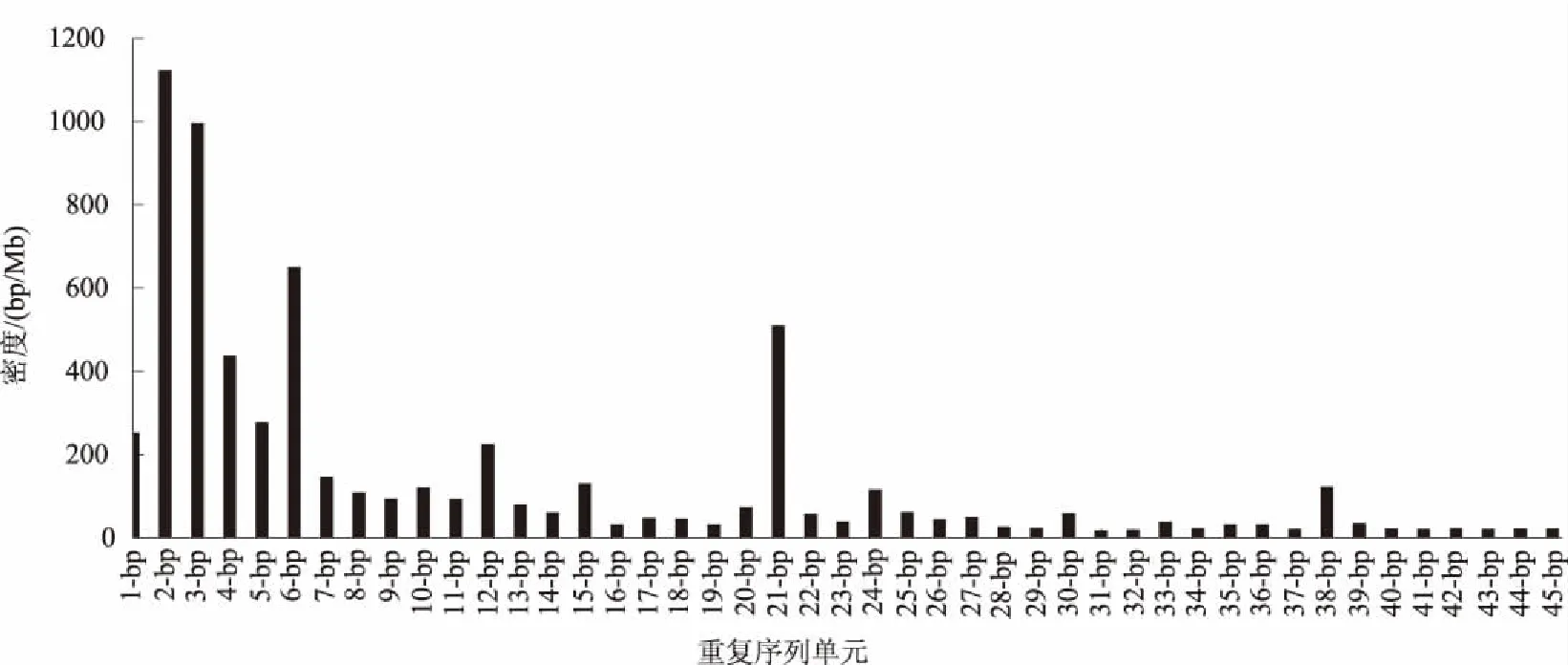
图3 高粱基因组中1~50 bp串联重复序列密度
2.2 基因内不同区域1~50 bp串联重复序列的密度分析
真核基因结构主要包括5′UTR、CDS、Intron和3′UTR等区域,这些区域与DNA的转录(如开放阅读框,ORF)、翻译密切相关。
2.2.1 串联重复序列1~50 bp的重复单元在5′UTR中的密度 如图4所示,5′UTR中重复密度从高到低主要为三碱基(8 949 bp/Mb)、二碱基(4 064 bp/Mb)、五碱基(3 897 bp/Mb)、六碱基(3 773 bp/Mb)和四碱基(2 821 bp/Mb),它们占总密度的91.62%。其中三碱基重复密度最大,占总密度的34.88%,且CCG(3 127 bp/Mb)为最高的重复模体。在二碱基重复单元中,AG(1 702 bp/Mb)、CT(1 585 bp/Mb)重复模体最高,GT(72 bp/Mb)最小。可见,在5′UTR中,重复序列主要为短的2~6 bp的微卫星重复,且主要为富含CG的重复模体。

图4 5′UTR中不同重复单元的密度
2.2.2 串联重复序列1~50 bp的重复单元在CDS中的密度 由图5知,CDS中重复序列最高的为三碱基重复(3 334 bp/Mb),占总重复序列的53.96%,其次为六碱基重复(1 599 bp/Mb),占总的25.87%,二者合计高达79.83%。在三碱基重复模体中,CGG(800 bp/Mb)和CCG(673 bp/Mb)最高,而AAT和GTT最低,均为1 bp/Mb。六碱基重复模体与三碱基重复类似,最高的为富含CG的CCGGCG (113 bp/Mb)。其余的重复单元主要为十二碱基(127 bp/Mb)、九碱基(96 bp/Mb)、十八碱基(92 bp/Mb)等,它们均为3 bp的倍数。由于CDS是蛋白质的编码序列,这可能与三联体密码子的翻译有关。
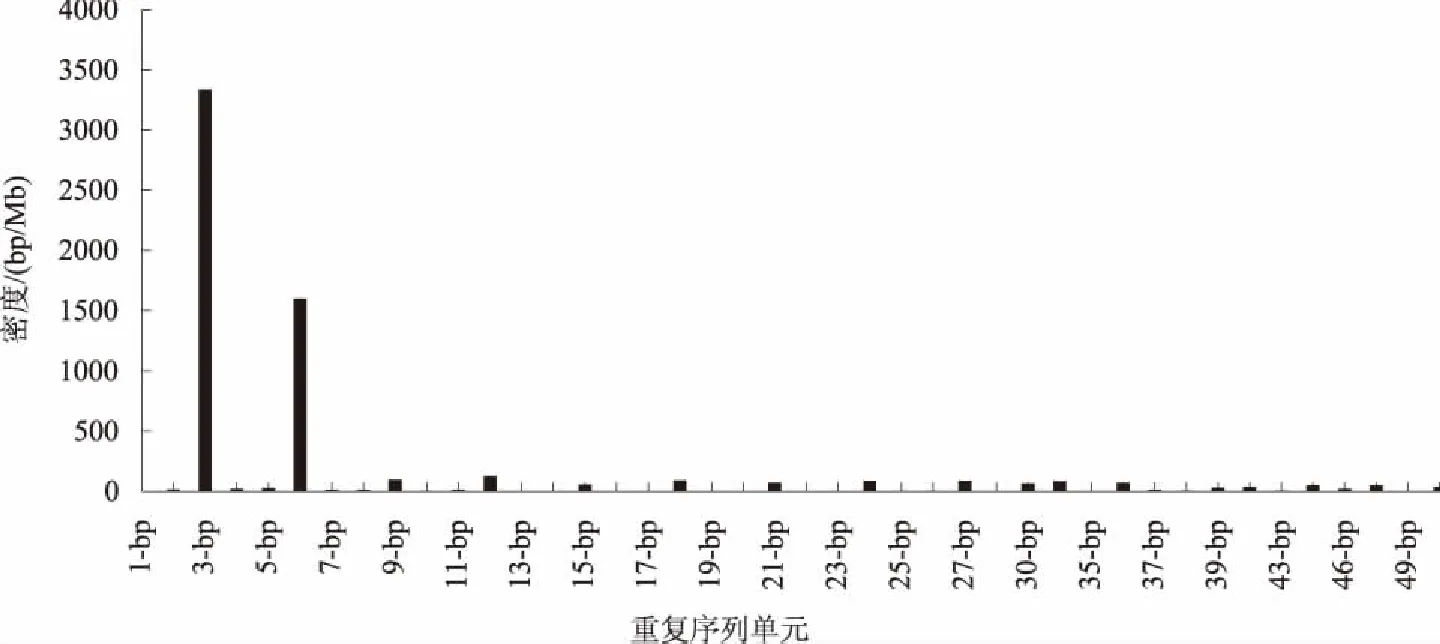
图5 CDS中不同重复单元的密度
2.2.3 串联重复序列1~50 bp的重复单元在Intron中的密度 高粱基因组Intron中,1~50 bp的重复单元主要是二碱基(1 613 bp/Mb)、单碱基(759 bp/Mb)、三碱基(654 bp/Mb)、四碱基(603 bp/Mb)、五碱基(440 bp/Mb)和六碱基(433 bp/Mb)等微卫星DNA(图6)。其中二碱基占总密度的24.40%,单碱基为11.48%,二者合计占总密度的35.88%。在二碱基重复单元中,AT重复模体密度最高(821 bp/Mb),AG、GT、AC重复密度相近(200 bp/Mb),CG重复密度最小(14 bp/Mb)。故可知,Intron中主要为富含AT的微卫星序列重复(1~6 bp),其他长碱基的重复单元较少。

图6 Intron中不同重复单元的密度
2.2.4 串联重复序列1~50 bp的重复单元在3′UTR中的密度 3′UTR区为非翻译区,与基因序列中5′UTR相对,它含有编码一段蛋白质的终止信号和Poly(A)信号,这一区域主要负责基因转录的终止。由图7知,在3′UTR中,重复碱基的主要类别是三碱基(1 148 bp/Mb)、二碱基(1 104 bp/Mb)、四碱基(863 bp/Mb)和五碱基(582 bp/Mb),合计占总密度的64.75%。在三碱基重复单元中,重复密度最大为GCT(163 bp/Mb)和GTT(141 bp/Mb),最小为ATC(11 bp/Mb)和ACC(8 bp/Mb);在二碱基重复单元中,AT(404 bp/Mb)和GT(349 bp/Mb)为优势重复碱基,AG(38 bp/Mb)和CG(26 bp/Mb)为弱势碱基。可见,3′UTR 类似于5′UTR,重复序列主要为富含GT的2~5 bp的微卫星重复。
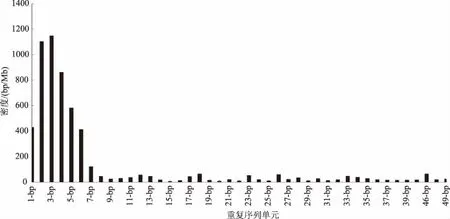
图7 3′UTR中不同重复单元的密度
2.3 基因间不同区域1~50 bp串联重复序列的密度分析
基因间序列指基因编码区的上游序列和下游序列,它们含有转录调节的元件,如增强子、启动子、弱化子、沉默子等属于非编码区,目前对它们的研究不多,但是在非编码区中存在大量重复序列。
2.3.1 串联重复序列1~50 bp的重复单元在上游序列中的密度 基因序列的上游区分为3个区,UI200(5′UTR之前的200 nt,指1~200 nt)、UI500(5′UTR之前500 nt,指201~700 nt)、UI1000(5′UTR之前1 000 nt,指701~1 700 nt)。该区域一般含有调控转录的元件,如启动子等。
在UI200中,二碱基(3 612 bp/Mb)和三碱基(3 437 bp/Mb)重复密度占总重复密度的42.05%。在二碱基重复单元中,优势模体为AT (1 170 bp/Mb)和CT (1 075 bp/Mb),弱势模体为GC (73 bp/Mb);在三碱基重复单元中,优势模体是CCG (627 bp/Mb)和CCT (565 bp/Mb) ,弱势模体为AAC (6 bp/Mb)(图8A)。可知,二碱基重复主要为富含AT的模体,而三碱基则为富含CG的模体。
在UI500中,二碱基(2 757 bp/Mb)和三碱基(1 395 bp/Mb)重复密度占总重复密度的38.74%。在二碱基重复单元中,AT (1 504 bp/Mb)为优势模体,而CG(35 bp/Mb)属于弱势模体;在三碱基重复单元中,ATT (173 bp/Mb )为优势模体,弱势模体是GTT (10 bp/Mb)(图8B)。可知在UI500中,二碱基和三碱基重复均为富含AT的模体。
类似于UI500,在UI1000中,二碱基(2 184 bp/Mb)和三碱基(1 153 bp/Mb)重复密度占总重复密度的38.59%。在二碱基重复单元中,优势模体为AT (1 336 bp/Mb),弱势模体为CG (14 bp/Mb);在三碱基重复单元中,优势模体为ATT (188 bp/Mb),弱势模体为GGT (22 bp/Mb)(图8C)。可知在UI1000中,二碱基和三碱基重复均为富含AT的模体。
在基因上游3个不同区域中,高重复密度的单元主要为1~7 bp,属于微卫星序列,其他重复单元较低。与UI500和UI1000相比, UI200重复单元的种类较少(只有30种),但密度较大,这或许与UI200和5′UTR位置较近有关系,此位置主要为启动子的调控区域,可能与转录起始及调控有关。
2.3.2 串联重复序列1~50 bp的重复单元在下游序列中的密度 基因序列的下游区分为3个区,DI200(3′UTR之后200 nt,指1~200 nt)、DI500(3′UTR之后500 nt,指201~700 nt)、DI1000(3′UTR之后1 000 nt,指701~1 700 nt)。该区域一般含有调控转录的终止元件,如Poly(A)位点等。
在DI200中,二碱基(856 bp/Mb)和三碱基(684 bp/Mb)重复密度占总重复密度的24.41%。在二碱基重复单元中,优势模体为AG (293 bp/Mb)和AT (215 bp/Mb),弱势模体为AC (25 bp/Mb);在三碱基重复单元中,优势模体是ATT (106 bp/Mb)和CGG (100 bp/Mb),弱势模体为ATC (5 bp/Mb)和GAT (5 bp/Mb)(图9A)。可知,二碱基和三碱基重复没有明显的偏向性。
在DI500中,二碱基(848 bp/Mb)重复为主要的重复单元,占总重复密度的13.29%。在二碱基重复单元中,AT (295 bp/Mb)为优势模体,而CG(13 bp/Mb)属于弱势模体(图9B)。可知在UI500中,二碱基重复主要为富含AT的模体。
在DI1000中,三碱基(717 bp/Mb)和二碱基(701 bp/Mb)重复密度占总重复密度的22.67%。在三碱基重复单元中,优势模体为CGG (90 bp/Mb)和CCG (90 bp/Mb),弱势模体为GAT (3 bp/Mb);在二碱基重复单元中,AT (241 bp/Mb)为优势模体,而CG(25 bp/Mb)属于弱势模体(图9C)。可知在DI1000中,三碱基为富含CG的模体,而二碱基重复主要为富含AT的模体。
不同于基因上游区域,在高粱基因组下游区域中,虽然1~7 bp的重复单元密度较高,但其密度的绝对值(150~850 bp/Mb)远小于基因上游区域(250~3 600 bp/Mb)。与上游区域相比,下游区域大于10 bp重复单元的重复密度明显较高。

图8 基因上游序列不同重复单元的密度
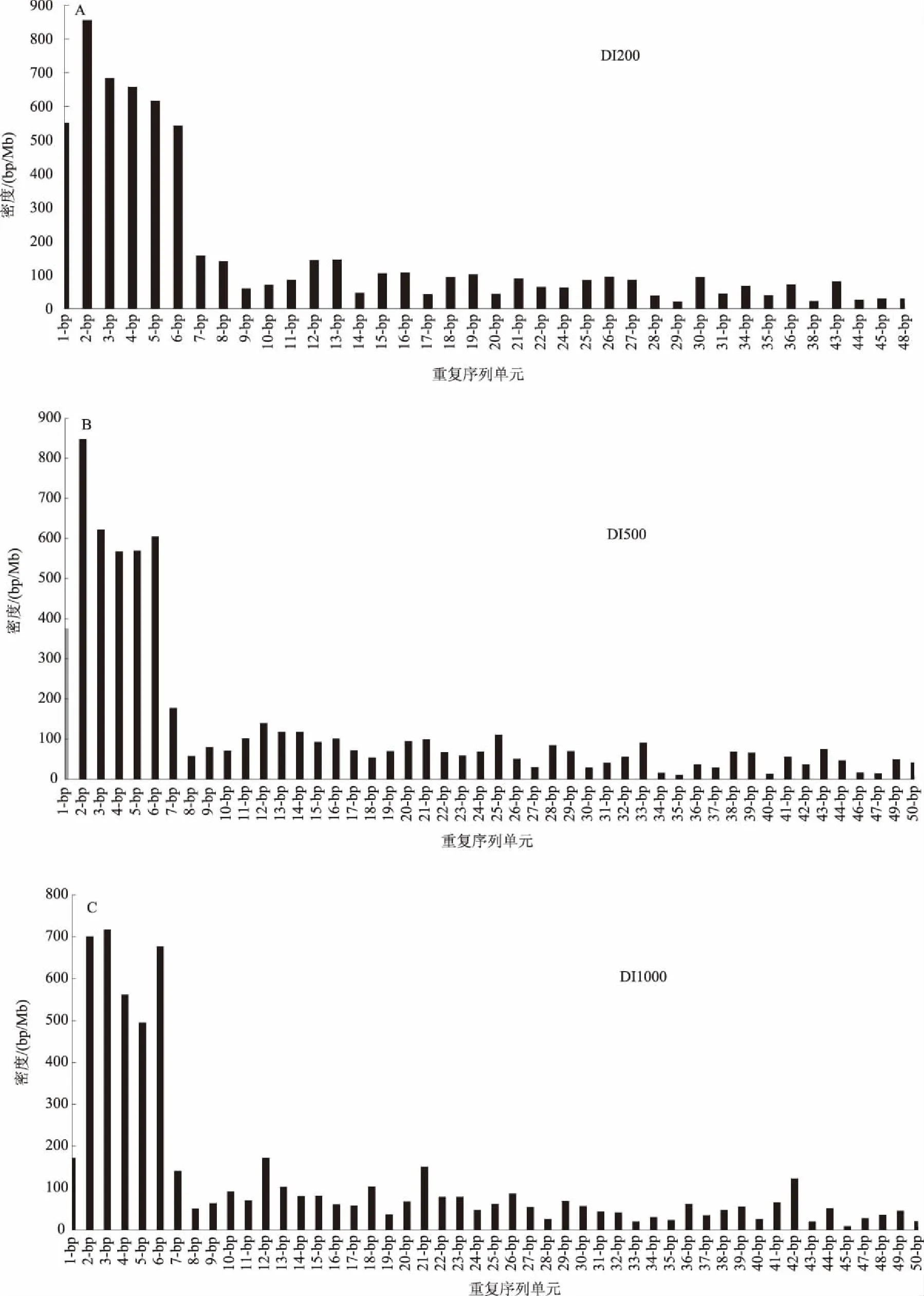
图9 基因下游序列不同重复单元的密度
2.4 高粱基因组串联重复序列在各个特征序列的分布
2.4.1 串联重复序列在基因内的分布 图10显示1~50 bp串联重复序列在基因内不同区域的分布情况。在5′UTR和CDS中,串联重复序列较均匀地分布于除两端以外的区域(>9.83%),两端分布较低(<9.14%),特别是在5′UTR中。在Intron中,两端的串联重复序列分布较多(10%左右),而中间60~69部位则最低(9.17%),由于Intron的两端紧靠着CDS,这可能与Intron的识别、剪接有关。与Intron相反,在3′UTR中,重复序列主要分布于中间,特别是在60~69部位最高(12.84%),两端则较低(接近9%)。


图10 串联重复序列频率在基因内的分布
2.4.2 串联重复序列在基因间的分布 在上游基因间隔区中,如图11,在UI200、UI500、UI1000中,距离基因5′UTR端越远,串联重复序列数量越少。在UI200和UI500中表现尤为明显,其最大与最小值分别相差2.55倍(UI200, 12.32/4.83)和1.72倍(UI500, 12.62/7.32)。

图11 串联重复序列频率在上游基因间隔区的分布
类似于上游基因间隔区,在下游基因间隔区内,距离基因3′UTR端越远,串联重复序列数量也越少(图12)。特别是在3′UTR下游1~700 bp内(DI200和DI500),靠近3′UTR端串联重复序列分布较高,远离3′UTR端其分布逐渐降低,其最大与最小值相差1.99倍(DI200,13.02/6.54)和1.57倍(DI500,11.63/7.39)。而在DI1000区域内,串联重复序列分布逐渐降低的趋势则不明显,可能揭示此区域与3′UTR转录终止相关性已较弱。

图12 串联重复序列频率在下游基因间隔区的分布
3 结论与讨论
3.1 串联重复序列在高粱基因组中的密度
在不同区域微卫星重复单元类别中(1~6 bp),主要是二碱基和三碱基等微卫星,占总密度的30%以上,而单碱基重复单元密度不高,同时部分长碱基的重复单元密度也不低,说明高粱基因组中重复单元的出现不是随机的,而是有一定的自然选择性,这与他人的研究结果相一致[3,5-6]。
已知重复序列在5′UTR、UI200和UI500中的密度较高,其余区域密度差别不大。就区域基因组大小而言,UI200基因组为2.73 Mb、5′UTR基因组为2.06 Mb,但其微卫星(1~6 bp)的重复密度高达80%以上,而Intron基因组为43.56 Mb,其微卫星重复密度只有35.88%。显然微卫星重复密度与基因组大小没有明显关系,可能与它所在位置的功能有关[3]。
本研究显示,高粱基因组中最高和次高的串联重复序列密度在5′UTR和它的直接上游区域,即UI200区,而这个区域通常为启动子区域。5′UTR被认为是串联重复序列的热点区域,之前的研究表明,5′UTR中的串联重复序列可参与转录或翻译的调控[5-6,9]。CDS中串联重复序列的密度较低,低密度的重复序列会降低蛋白质的复杂性从而增强其保守度,已经证实CDS的突变会导致蛋白质功能改变,功能丧失和蛋白质截短[10]。3′UTR和内含子中的串联重复序列密度也较低,可能暗示重复序列在这些区域保守度高,参与的生物学功能也可能较少[5]。
3.2 串联重复序列在高粱基因组中的位置分布
从重复序列在高粱基因内及基因间的分布可以看出,重复序列在整个基因组中的位置也并非随机存在,这与此前研究相一致[3]。基因间隔区的串联重复序列的分布明显偏向于靠近基因两端(5′UTR和3′UTR),串联重复序列已经被定位到基因和基因调节区,并参与转录和翻译的调控[11],本研究显示,串联重复序列偏向基因调节区,也支持了这一观点。另外,Intron两端的串联重复序列分布较高,考虑到可能与内含子剪接有关[9],也可对与其相邻的CDS起到保护的作用。
本研究显示,串联重复序列在基因不同区域具有显著的特征差异,并且重复序列的区域分布与基因调控具有紧密的联系,同时本研究将有助于对串联重复序列进化及在基因表达中调控作用的理解。但由于计算资源的局限性(如CPU、内存和运算时间),本研究只探究了1~50 bp重复序列,而对于更长的重复序列(>50 bp)进行研究或许能揭示更多潜在的重复序列功能。

