柑橘品种鉴定的SSR标记开发和指纹图谱库构建
李益,马先锋,唐浩,李娜,江东,龙桂友,李大志,牛英,韩瑞玺,邓子牛
柑橘品种鉴定的SSR标记开发和指纹图谱库构建
李益1,马先锋1,唐浩2,李娜1,江东3,龙桂友1,李大志1,牛英4,韩瑞玺2,邓子牛1
(1湖南农业大学园艺园林学院,长沙 410128;2农业部科技发展中心,北京 100122;3中国农业科学院柑桔研究所,重庆 400712;4广西特色作物研究院,广西桂林 541004)
【目的】筛选出一套SSR核心引物,用于构建柑橘品种的DNA指纹图谱库,为柑橘种苗认证、品种登记和植物新品种保护提供技术支撑。【方法】以柑橘属8个主要栽培种类为试材进行PCR扩增,扩增产物经琼脂糖凝胶电泳检测,筛选出多态性丰富、扩增条带稳定的引物对部分参试品种进行PCR扩增,扩增产物通过变性聚丙烯酰胺凝胶电泳进行进一步的引物筛选;筛选得到的引物进行荧光标记,并选用部分柑橘品种进行PCR扩增,扩增产物利用基因分析仪检测,分别计算各引物的值、等位基因数目,选取一套多态性丰富的适合进行荧光毛细管电泳检测的SSR引物组合,进而对所有参试品种进行分析,构建柑橘品种的DNA指纹图谱库,同时利用筛选的SSR标记对参试品种进行鉴定。【结果】初期以柑橘属8个种的代表品种(太田椪柑、沙田柚、沅江酸橙、大红甜橙、邓肯葡萄柚、枸橼、费米耐劳柠檬和墨西哥来檬)为材料,用362对SSR引物进行PCR扩增,扩增产物经4%琼脂糖凝胶电泳检测,初步筛选出80对种间多态性高、扩增稳定的SSR引物;进一步从8个种类里选择不同类型的64个柑橘品种,用上述筛选出的80对SSR引物进行PCR扩增,扩增产物经6%变性聚丙烯酰胺凝胶电泳检测,再筛选出60对品种类型间多态性高、扩增稳定、条带清晰的SSR引物,并分别进行荧光标记;另以168份柑橘品种为材料对上述60对SSR引物进行PCR扩增,经荧光毛细管电泳检测,依据各引物的值、等位基因数目、峰图读取难易程度、扩增稳定性及染色体位置等,最终筛选出21对SSR引物作为指纹图谱库构建的核心引物。为消除不同仪器、不同试验批次等引起的误差,为21对SSR引物各主要等位变异选择相应的参照品种。根据各引物标记的荧光颜色及扩增片段大小进行合理组合,21对SSR引物可分成5组进行多重电泳,共采集500份柑橘品种的DNA指纹。利用SSR标记,有111份品种仅用1对引物即可鉴别,86份品种用2对引物组合即可鉴别,24份品种用3对引物组合即可鉴别,另外279份品种虽无法单独一一鉴别,但可分为87个小组,组内品种无法区分,组间品种易鉴别。【结论】利用筛选出的21对SSR核心引物,构建了包含500份柑橘品种的DNA指纹图谱库,筛选出部分品种的特异标记,可以鉴别221份柑橘品种和87个品种组合,构建了基于毛细管电泳平台的柑橘SSR分子标记品种鉴定体系。
柑橘;SSR;毛细管电泳;品种鉴定;DNA指纹图谱库
0 引言
【研究意义】柑橘是多年生木本植物,可跨种属杂交,种属间杂种多,遗传背景复杂。柑橘的主栽品种多起源于芽变,如甜橙、温州蜜柑等,品种间遗传背景高度一致,增加了品种鉴定工作的难度。中国作为柑橘的原生国之一,柑橘种质资源丰富,近年通过引种和育种,中国柑橘品种的类型及结构更加多样化,国内各柑橘产区也培育了不少优良品种[1]。《中华人民共和国种子法》最新修改版规定品种需进行DUS(Distinctness, Uniformity, Stability)测试才能登记、审定及保护,因此,柑橘品种的鉴定工作不断推进。尽管柑橘可根据枝叶形态等鉴别其属种,但柑橘的形态学标记较少,种内品种间的差异不明显。随着分子生物学的发展,DNA分子标记鉴定因快速准确、不受环境影响等优点逐渐发挥重要作用,满足品种快速准确鉴定和标准指纹图谱构建的要求[2]。【前人研究进展】DNA分子标记技术广泛应用于植物的遗传多样性研究、遗传图谱构建和种质鉴定等,其中SSR标记和SNP标记均为共显性遗传,染色体定位清楚且具有高通量自动化的潜力,因此,国际新品种保护联盟(UPOV)的生物化学和分子生物学技术工作组(BMT)将其作为构建DNA指纹数据库的首选标记;而SSR标记在单个位点上的多态性高于SNP标记,技术研究更加成熟,且SNP的仪器设备成本高,不利于推广应用,因此,SSR标记因其各种优势在品种鉴定和数据库构建领域发挥越来越重要的作用[3]。近年来,主要农作物的DNA指纹检测技术已经成熟,基于SSR分子标记的玉米、水稻、大豆、小麦等主要农作物的指纹图谱库已构建完成[3-10]。SSR分子标记已广泛应用于柑橘的研究[11-12],韩国辉[13]建立了基于EST-SSR、Genomic-SSR、SCoT的柑橘连锁图谱及杂种和多倍体遗传分析;郭雁君等[14]利用SSR标记评价了广东省肇庆地区地方柑橘品种与其他部分柑橘属和近缘属植物的亲缘关系,为其分类和育种提供理论依据;曾涛[15]利用SSR分子标记构建了南丰蜜桔的指纹图谱并分析了各南丰蜜桔的遗传多样性;雷天刚等[16]以聚丙烯酰胺凝胶电泳为平台,用12对SSR引物和2个ISSR引物构建柑橘栽培品种(系)的DNA指纹图谱库,并赋予70个柑橘品种特征指纹代码。【本研究切入点】目前,国内柑橘品种DNA指纹库的构建大多基于琼脂糖凝胶电泳或聚丙烯酰胺凝胶电泳,但是琼脂糖凝胶电泳分辨率低,聚丙烯酰胺凝胶电泳制胶难度较大、步骤多且操作繁琐,都不适合大量样品的指纹采集。随着优良品种的不断选育、品种权保护的推广及育种家对品种保护意识的提高等,这两个平台已无法满足大量品种的DNA指纹鉴定。柑橘的指纹图谱虽有报道,但不够完善。【拟解决的关键问题】本研究从一系列基因组SSR和EST-SSR引物中筛选出一套多态性高、PCR扩增稳定、荧光峰型易识别的SSR引物组合,建立更加丰富的柑橘品种指纹图谱库,为柑橘种苗认证和DUS测试过程中近似品种的鉴定提供参考。
1 材料与方法
1.1 供试材料
用于引物筛选和DNA指纹图谱库构建的种质共503份,其中456份来自中国农业科学院柑桔研究所国家资源圃,47份来自国家柑橘改良中心长沙分中心,包括9份大翼橙亚属种质、16份枳、10份金柑、9份属间杂种、20份枸橼种质、162份宽皮柑橘、2份来檬、51份柠檬、12份葡萄柚、12份酸橙、19份甜橙、8份香橙、121份柚、52份柑橘属内杂种。
1.2 DNA提取
每份种质取5片同龄幼嫩叶片等量混合,液氮研磨,采用改良的CTAB法[17]提取基因组总DNA。
1.3 SSR引物的选择
依据参考文献[17-25]报道的序列共合成362对SSR引物。在经琼脂糖凝胶电泳和聚丙烯酰胺凝胶电泳筛选后的SSR引物上游5’端随机标记6-FAM、HEX、ROX和TAMRA 4种荧光染料。普通引物由上海生工生物工程技术服务有限公司合成,荧光引物由英潍捷基(上海)贸易有限公司合成。
1.4 PCR扩增
PCR反应体系为dNTP 0.20 mmol·L-1、上下游引物各0.25 μmol·L-1、DNA聚合酶0.05 U·μL-1、1×buffer(含Mg2+2.5 mmol·L-1)和样品DNA 2.5 ng·μL-1。PCR程序为94℃ 5 min;94℃ 30 s,55℃—60℃ 30 s,72℃ 40 s,共35个循环;72℃ 10 min。
PCR扩增产物经4%琼脂糖凝胶电泳40—50 min(恒压120 V),凝胶成像系统下拍照记录。变性后的PCR扩增产物经6%变性聚丙烯酰胺凝胶电泳分离1—1.5 h(80 W恒定功率),银染检测[26]。将6-FAM、HEX荧光标记的PCR产物稀释30倍,TAMRA和ROX荧光标记的PCR产物稀释15倍,取等量稀释后的产物混合并变性,上样至3130XL基因分析仪进行片段分析,使用SSR指纹分析器读取数据[7]。
1.5 引物多态性评价
利用(Polymorphism Information Content)评价引物多态性,计算公式:PIC=1-∑P2,其中P表示标记的第个等位基因在群体中的频率[27]。
2 结果
2.1 引物筛选和标记开发
2.1.1 引物初选 以柑橘属8个种的代表品种(太田椪柑、沙田柚、沅江酸橙、大红甜橙、邓肯葡萄柚、枸橼、费米耐劳柠檬、墨西哥来檬)为材料,对362对SSR引物进行PCR扩增及4%琼脂糖凝胶电泳检测,其中340对SSR引物扩增出目的片段,评价每对引物的多态性、扩增稳定性,初步筛选出80对种间多态性高、扩增稳定的SSR引物。
2.1.2 引物复选 从柑橘属8个种中选择不同类型的64份品种作为材料,对上述80对SSR引物进行PCR扩增及6%变性聚丙烯酰胺凝胶电泳检测,评价每对引物的多态性、扩增稳定性和条带清晰度等,筛选出60对多态性高、扩增稳定、带型较清晰的SSR引物(图1)。
2.1.3 引物精选 对上述筛出的60对SSR引物标记荧光染料,从柑橘属8个种中的选择不同类型的168份柑橘品种为材料进行PCR扩增,扩增产物经毛细管电泳检测,根据各引物的值、等位基因数目、峰图读取的难易程度及染色体位置等,最终筛选出21对多态性高、扩增稳定、毛细管电泳峰型易读取且在染色体上分布均匀的SSR引物作为指纹图谱库构建的核心引物[28]。21个位点的多态信息量()变幅为0.550—0.871,平均为0.733(图2、表1和表2)。
2.1.4 标记的校正统一与参照品种的选择 统计各柑橘(品)种在21个SSR位点上的扩增片段大小并取整,用取整后的片段大小命名各等位变异。为消除不同仪器型号、不同试验批次等造成的系统误差并保证不同电泳平台数据的一致性,为每对SSR引物的各等位变异选择相应的参照品种(代表核心位点主要等位变异的一组样品,辅助确定待测样品在某个位点上的等位变异大小)(表2)。
2.2 柑橘(品)种DNA指纹图谱库的构建
根据引物标记的荧光颜色及等位变异的大小范围进行组合,21对SSR引物可分成5组(表3),每组3—6对引物,同一组引物的PCR扩增产物混合后进行多重电泳,可提高试验效率、降低成本。提取各柑橘样品的DNA并进行PCR扩增,扩增产物依据引物分组进行毛细管多重电泳,采集各柑橘种质在21个SSR位点上的指纹数据,建立了包含500份柑橘种质的DNA指纹图谱库。在21个位点中共检测到253个等位变异,每个位点的等位变异数为4—19个,平均约12个(表3)。

M:25 bp DNA Step ladder,1:福橘;2:兴义大红袍;3:南橘;4:黄果柑;5:早熟;6:阳山橘;7:本地早;8:瓯柑;9:早熟克里曼丁;10:贡柑;11:永春椪柑;12:黄岩椪柑;13:太田椪柑;14:日南1号;15:大分4号;16:伊予柑;17:温岭高橙;18:秋辉;19:诺瓦;20:清见;21:维诺拉;22:世界蜜柚;23:默科特;24:凯旋柑;25:早香;26:阳香;27:桂平朱砂橘;28:满头红;29:印度酸橘;30:枸头橙;31:代代;32:朱栾;33:无核雪柑;34:莫洛;35:大红甜橙;36:夏沙蜜柚;37:北碚柚;38:金兰柚;39:五布红心柚;40:强德勒柚;41:勐伦早柚;42:橘湘红;43:沙田柚;44:马家柚;45:玉环柚;46:四季抛;47:曼赛龙柚;48:奥罗布兰克;49:邓肯葡萄柚;50:马叙葡萄柚;51:鸡尾葡萄柚;52:木里香橼;53:小香橼;54:长果香橼;55:美国枸橼;56:枸橼;57:野生香橼;58:大香橼;59:墨西哥来檬;60:印特多纳多;61:尤力克柠檬;62:橙花;63:白黎檬;64:北京柠檬
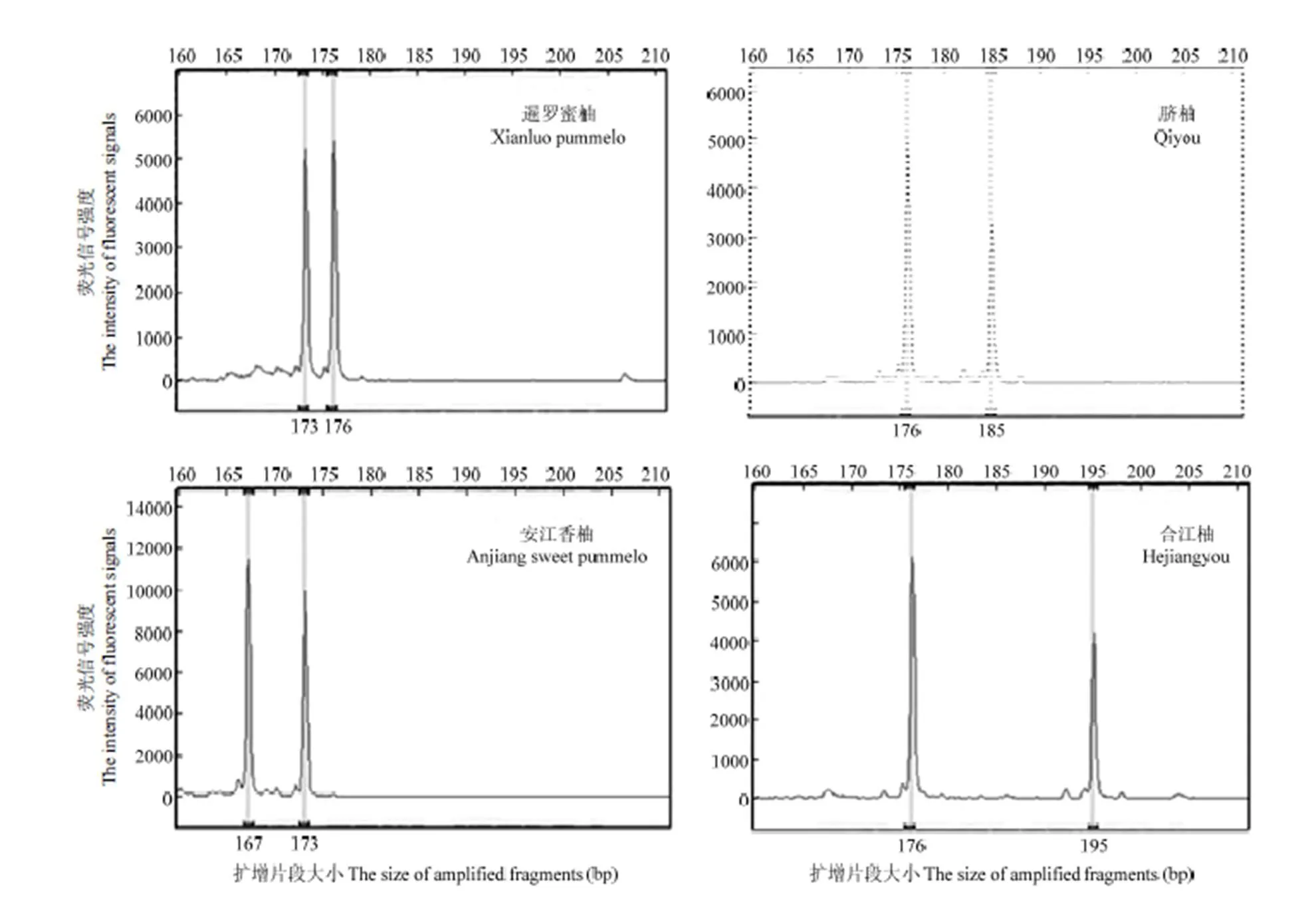
图2 引物P8在4个柑橘品种中检测到的等位变异
2.3 特异标记的筛选和种质鉴定
筛选了部分柑橘属种的特异标记(属、种内品种间条带一致,同时区别于其他属、种的品种),为跨属种杂交后代的鉴定及柑橘品种的属种鉴别提供参考(表4)。
分析500份柑橘种质在21个位点上的指纹数据,221份柑橘种质可准确鉴别:独立标记可鉴别111份种质,2对引物组合可鉴别86份种质,3对引物组合可鉴别24份种质,大翼橙亚属、枸橼、杂柑、柚类等易鉴别;279份种质暂时无法鉴别,但可分成87组,每组包含2—17份种质,同组的种质暂时无法区分,但组间种质易鉴别;不能鉴别的种质主要为芽变品种或同物异名,如温州蜜柑、椪柑、葡萄柚、柠檬、甜橙等种质难以鉴别。
3 讨论
良种是现代农业发展的基础,优良品种的选育工作受到越来越多的重视,本研究的植物材料“柑橘”为木本果树,开花结果需要经历“童期”这一特殊植物生理阶段,因此果树新品种的选育需投入大量的时间、精力和财力,如果选育的新品种不能得到有效保护,会打击育种者的积极性,不利于品种的持续创新[29]。植物新品种保护是国家知识产权战略的重要组成部分,中国越来越重视植物品种权的保护,育种者的保护意识不断提高,柑橘新品种保护的申请量和DUS测试量逐年增加;但是由于柑橘具有:(1)不同属、种或品种间可相互授粉杂交,杂种类型繁多;(2)柑橘栽培品种主要通过嫁接等无性繁殖方式繁育,易通过自我繁殖窃取新品种权益;(3)同一品种在不同种植区域往往存在不同名称的现象,导致柑橘品种的监管难度大。近年来,柑橘产业受黄龙病、溃疡病等病害的影响,产量和质量都大大降低,优良苗木需求旺盛[30-31],各柑橘主产省份已建成或正在新建无病毒苗圃,且部分研究机构已开展柑橘的种苗认证工作,明确柑橘苗木的真实身份。UPOV将DUS测试作为新品种登记和保护的基本条件[32],本研究筛选的柑橘SSR引物及建立的DNA指纹图谱库可用于柑橘新品种DUS测试中近似品种的筛选以及种苗市场监管的身份快速鉴定,有利于柑橘育种和品种资源保护。
表1 引物编号及序列
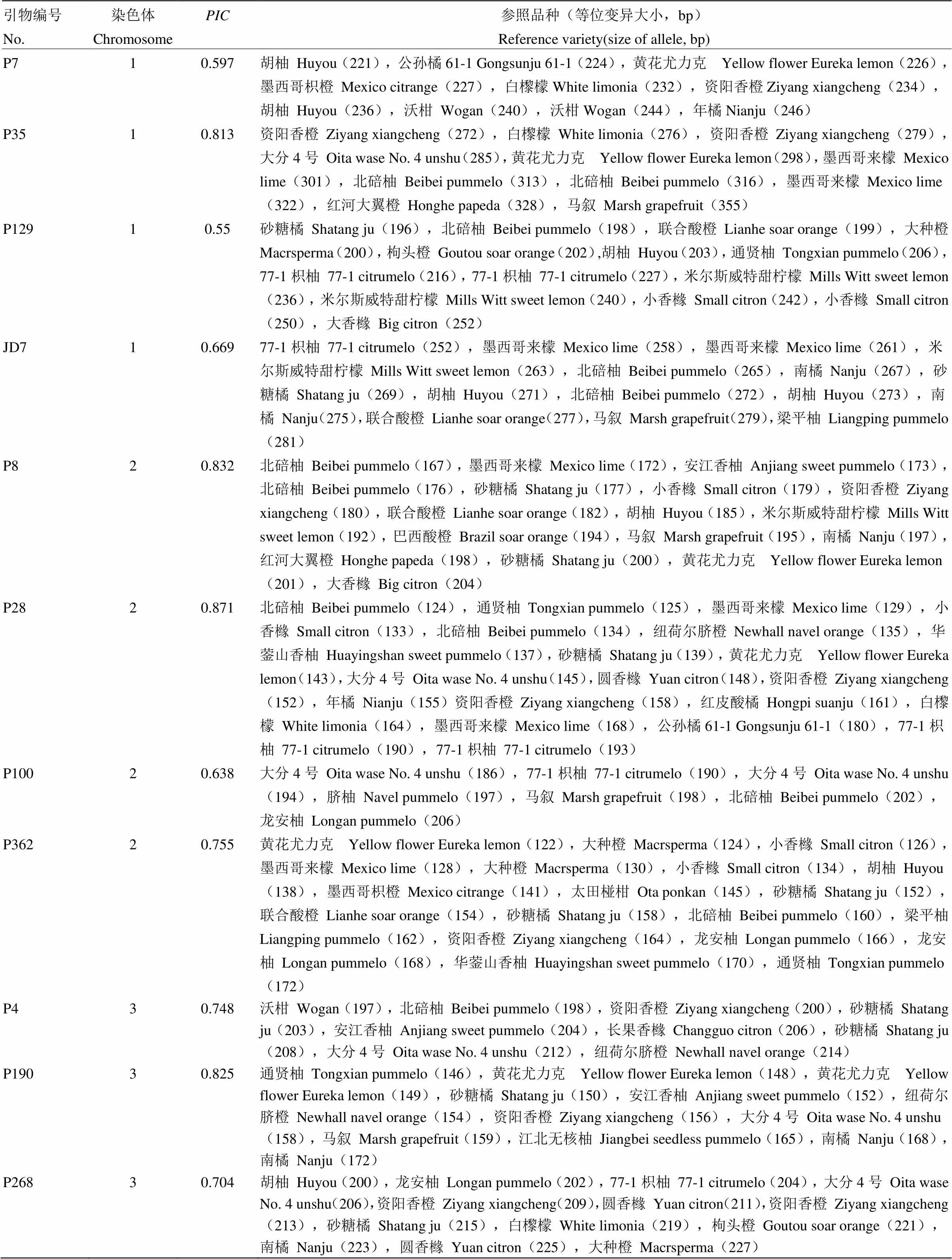
表2 用于柑橘属(品)种DNA指纹鉴定的21个标记
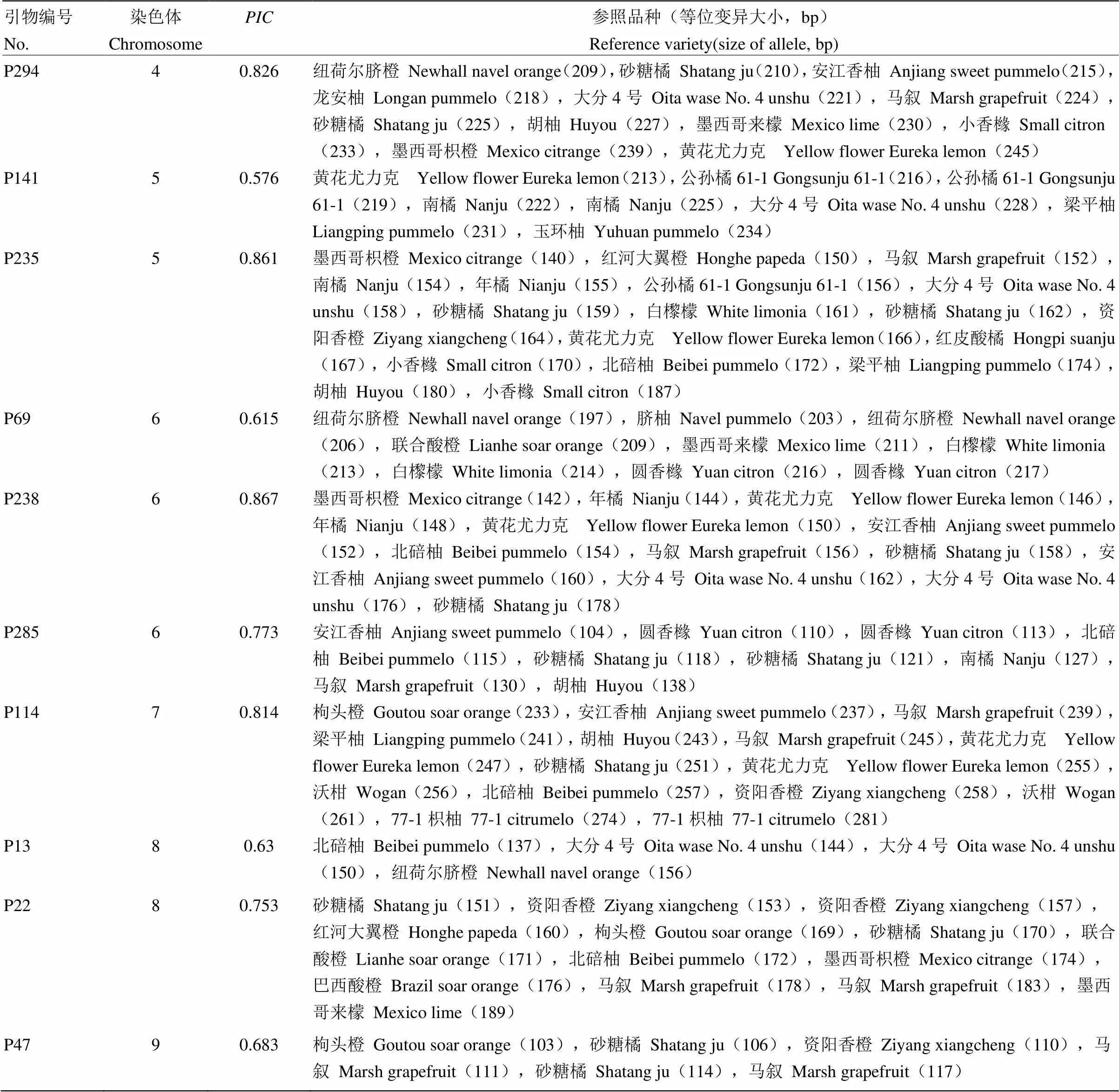
续表2 Continued table 2
参照品种是代表核心位点主要等位变异的一组样品。辅助确定待测样品在某个位点上等位变异的大小,校正仪器设备的系统误差
The reference variety is a set of samples representing the main alleles of the core site. Being used to assist in determining the size of an extended segment of the size of alleles at a certain site, correcting of system error of instrument and equipment

表3 21个荧光标记引物的分组
括号内为等位变异的大小范围 The number in the brackets indicate the range of allele sizes

表4 柑橘属种的特异标记
252/265代表引物在样品中扩增出2条片段,一条为252 bp,一条为265 bp。“/”表示暂未找到该属种的特异标记
252/265 represent that primer is amplified in two fragments in the sample, with a size of 252 bp and a size of 265 bp. “/” means that this specific molecular of genus-species currently have not be found
目前,柑橘品种鉴定和指纹图谱库构建取得了较好的进展。吴鑫[33]筛选出34对SSR引物和3个ISSR引物对126份柑橘材料进行鉴定,SSR标记可鉴定66份品种,ISSR标记可鉴定27份品种;雷天刚等[16]筛选出12对SSR引物和2个ISSR引物对102份柑橘材料进行鉴定,SSR标记可鉴定42份品种,ISSR标记可鉴定28份品种。本研究用21对SSR引物鉴定500份柑橘种质,其中221份材料可准确鉴别,SSR标记在宽皮柑橘、杂柑、柚类等品种间多态性高,与前人的结果一致;将暂时区分不开的材料归为一类,剩余的279份材料分为87类,不同类的品种易鉴别。由于SSR标记在甜橙、温州蜜柑、红橘等芽变品种中的多态性较低,前人利用ISSR标记能够鉴别部分芽变品种,考虑到ISSR标记重复性差、非特异性片段多、数据间难以整合,不适于毛细管电泳平台,因此,本研究未选用ISSR标记。本研究选用的品种覆盖了柑橘属各主栽品种、枸橼、酸橙及枳属和金柑属的部分(品)种,具有较好的代表性。筛选的21对SSR引物在染色体上分布较均匀、多态性高,通过特异标记及其组合可以有效鉴别目前大多数广泛栽培的品种。另外,本研究利用毛细管电泳平台构建DNA指纹图谱库,克服了聚丙烯酰胺凝胶电泳操作繁琐、费时费力、自动化程度低的缺点,能够高通量地采集数据,提高品种鉴定的效率[34]。柑橘是多年生木本植物,具有童期长、遗传背景复杂、部分栽培品种为芽变品种等特点,而芽变属体细胞变异,与芽变母本的遗传背景一致,SSR引物难以覆盖到变异位点,因此,SSR标记用于柑橘品种的鉴定存在一定的局限性[35]。随着基因组测序技术的发展,可结合SNP或简化基因组测序用于进一步鉴定芽变品种[36-38]。
4 结论
筛选出21对SSR核心引物,建立了基于毛细管电泳平台的柑橘品种SSR分子标记鉴定体系,构建了包含500份柑橘种质的DNA指纹图谱库,可用于柑橘品种的鉴别。
[1] 沈兆敏. 我国柑橘产销现状、发展趋势及对策建议. 果农之友. 2012(03): 3-4.
SHEN Z M. Citrus’ production, trend of development and countermeasures in China., 2012(03): 3-4. (in Chinese)
[2] 朱岩芳. 作物品种分子标记鉴定及指纹图谱构建研究[D]. 杭州: 浙江大学, 2013.
ZHU Y F. Research on cultivar identification and DNA fingerprinting of crops based on molecular markers[D].Hangzhou: Zhejiang University, 2013. (in Chinese)
[3] 王凤格, 杨扬, 易红梅, 赵久然, 任洁, 王璐, 葛建镕, 江彬, 张宪晨, 田红丽, 侯振华. 中国玉米审定品种标准SSR指纹库的构建. 中国农业科学, 2017, 50(1): 1-14.
WANG F G, YANG Y, YI H M, ZHAO J R, REN J, WANG L, GE J R, JIANG B, ZHANG X C, TIAN H L, HOU Z H. Construction of an SSR-based standard fingerprint database for corn variety authorized in China., 2017, 50(1): 1-14. (in Chinese)
[4] 刘文彬. 适于玉米品种鉴定的高通量SSR标记开发及复合体系建立[D]. 长春: 吉林农业大学, 2017.
LIU W B. Development high throughput SSR markers about suitable maize cultivar identification and buildig compound system[D]. Changchun: Jilin Agricultural University, 2017. (in Chinese)
[5] 邓姗, 褚云霞, 黄志城, 杨华, 顾可飞, 陈海荣. 上海地区水稻已知品种SSR指纹图谱库构建. 中国农学通报, 2015, 31(3): 7-15.
DENG S, CHU Y X, HUANG Z C, YANG H, GU K F, CHEN H R. Establishment of DNA fingerprint database of rice varieties in Shanghai., 2015, 31(3): 7-15. (in Chinese)
[6] 徐海风, 程保山, 杨加银, 沈业松. 黄淮海地区夏大豆品种(系)指纹图谱的构建及其遗传多样性分析. 西南农业报, 2014, 27(5): 1814-1819.
XU H F, CHENG B S, YANG J Y, SHEN Y S. Establishment of SSR fingerprint map and analysis of genetic diversity among soybean variety in Huang-Huai-Hai region., 2014, 27(5): 1814-1819. (in Chinese)
[7] 郑永胜, 张晗, 王东建, 孙加梅, 王雪梅, 段丽丽, 李华, 王玮, 李汝玉. 基于荧光检测技术的小麦品种SSR鉴定体系的建立. 中国农业科学, 2014, 47(19): 3725-3735.
ZHENG Y S, ZHANG H, WANG D J, SUN J M, WANG X M, DUAN L L, LI H, WANG W, LI R Y. Development of a wheat variety identification system based on fluorescently labeled SSR markers., 2014, 47(19): 3725-3735. (in Chinese)
[8] 宋伟林. 基于SSR荧光标记毛细管电泳的油菜品种DNA指纹鉴定技术平台的建立与应用[D]. 北京: 中国农业科学院, 2013.
SONG W L. Establishment and application of a technology platform for DNA fingerprint identification of rapeseed cultivars based on capillary electrophoresis with SSR fluorescence markers[D]. Beijing: Chinese Academy of Agricultural Sciences,2013. (in Chinese)
[9] 王瑞, 张福耀, 程庆军, 田承华, 凌亮. 利用SSR荧光标记构建20个高粱品种指纹图谱. 作物学报, 2015, 41(4): 658-665.
WANG R, ZHANG F Y, CHENG Q J, TIAN C H, LING L. Establishment of 20 sorghum hybrids fingerprints using SSR fluorescent marker., 2015, 41(4): 658-665. (in Chinese)
[10] 张晗, 王东建, 孙加梅, 郑永胜, 姚凤霞, 许金芳, 宋国安, 段丽丽, 李华, 王雪梅, 李汝玉. 大白菜高通量SSR标记鉴定体系的建立和应用. 植物遗传资源学报, 2014, 15(4): 815-823.
ZHANG H, WANG D J, SUN J M, ZHEN Y S, YAO F X, XU J F, SONG G A, DUAN L L, LI H, WANG X M, LI R Y. Development and application of a high-throughput chinese cabbage dna profiling system based on SSR markers., 2014, 15(4): 815-823. (in Chinese)
[11] 梁武军, 解凯东, 郭大勇, 谢宗周, 徐强, 伊华林, 郭文武. 柑橘10个品种实生后代多倍体的发掘及SSR鉴定. 园艺学报, 2014, 41(3): 409-416.
LIANG W J, XIE K D, GUO D Y, XIE Z Z, XU Q, YI H L, GUO W W. Spontaneous generation and SSR characterization of polyploids from ten citrus cultivars., 2014, 41(3): 409-416.(in Chinese)
[12] 刘通, 邓崇岭, 程玉芳, 李秋景, 陈传武,刘冰浩, 伊华林. 利用SSR和SRAP技术分析广西柑橘种质遗传多样性. 华中农业大学学报, 2016, 35(2):23-29.
LIU T, DENG C L, CHENG Y F, LI Q J, CHEN C W, LIU B H, YI H L. Analysis of genetic diversity of citrus in Guangxi by SSR and SRAP., 2016, 35(2): 23-29.(in Chinese)
[13] 韩国辉. 基于EST-SSR、Genomic-SSR和SCoT标记的柑橘连锁图谱构建及杂种和多倍体遗传分析[D]. 重庆: 西南大学, 2012.
HAN G H. Construction of molecular linkage map and genetic analysis of hybrids and polyploidy of citrus based on EST-SSR, genomic-SSR and Scot markers[D]. Chongqing: Southwest University, 2012(in Chinese)
[14] 郭雁君, 曾继武, 胡亚平,郭丽英, 蒋惠, 周希琴, 吉前华. 基于SSR标记的肇庆地区柑橘品种分类地位研究. 中国农学通报, 2014, 30(4):137-143.
GUO Y J, ZENG J W, HU Y P, GUO L Y, JIANG H, ZHOU X Q, JI Q H. Classification of Zhaoqing local citrus germplasm resources based on simple sequence repeat molecular marker analysis., 2014, 30(4):137-143. (in Chinese)
[15] 曾涛. 南丰蜜橘SSR指纹图谱构建及遗传多样性分析[D]. 南昌: 江西农业大学, 2012.
ZENG T. Construction of fingerprinting and analysis of genetic diversity with SSR markers for Nangfeng tangerine[D]. Nanchang: Jiangxi Agricultural University, 2012. (in Chinese)
[16] 雷天刚, 何永睿, 吴鑫, 姚利晓, 彭爱红, 许兰珍, 刘小丰, 陈善春. 柑橘栽培品种(系)DNA指纹图谱库的构建. 中国农业科学, 2009, 42(8): 2852-2861.
LEI T G, HE Y R, WU X, YAO L X, PENG A H, XU L Z, LIU X F, CHEN S C. Construction of DNA fingerprinting database of citrus cultivars(line)., 2009, 42(8): 2852-2861. (in Chinese)
[17] 张文. 抗溃疡病枸橼C05杂交后代的获得及分子标记鉴定[D]. 长沙: 湖南农业大学, 2013.
ZHANG W. The hybrids obtainment of citron C05 resistant to canker disease and the identification by molecular[D]. Changsha: Hunan Agricultural University, 2013. (in Chinese)
[18] Kijas J M, Thomas M R. An evaluation of sequence tagged microsatellite site markers for genetic analysis within citrus and related species., 1995, 38: 349-355.
[19] 鲁玉洋. 利用SSR和RAPD技术构建柑桔分子遗传图谱[D]. 重庆: 西南大学, 2006.
LU Y Y. Construction of genetic linkage maps of citrus based on SSR and RAPD marker master candidate[D]. Chongqing: Southwest University, 2006. (in Chinese)
[20] 江东, 钟广炎, 洪棋斌. 柑橘EST-SSR分子标记分析. 遗传学报, 2006, 33(4): 345-353.
JIANG D, ZHONG G Y, HONG Q B. Analysis of microsatellites in citrus unigenes., 2006, 33(4): 345-353. (in Chinese)
[21] 曾柏全. 宽皮柑橘资源遗传多样性的分子评价[D]. 长沙: 湖南农业大学, 2009.
ZENG B Q. Genetic diversity of mandarin investigated by molecular markers[D]. Changsha: Hunan Agricultural University, 2009. (in Chinese)
[22] 范达. 基于DNA指纹图谱的柑橘种苗纯度与真实性检测技术研究[D]. 重庆: 西南大学, 2011.
FAN D. DNA fingerprinting inspection technique for citrus nursery tree purity and genuineness[D]. Chongqing: Southwest University, 2011. (in Chinese)
[23] 谭洪泉. 柑橘杂种胚组织培养挽救及分子标记早期鉴定技术研究[D]. 重庆: 西南大学, 2011.
TAN H Q. Early rescue and identification of citrus hybrid embryo in vitro via SSR technology[D]. Chongqing: Southwest University, 2011. (in Chinese)
[24] 韩会君. 红橘优系遗传鉴定及起源研究[D]. 武汉: 华中农业大学, 2013.
HAN H J. The genetic identification and origin study of red tangerine elite accession[D]. Wuhan: Huazhong Agricultural University, 2013. (in Chinese)
[25] 龙荡. 基于转录组测序大规模开发早实枳SSR标记[D]. 武汉: 华中农业大学, 2014.
LONG D. Large-scale development of SSR markers based on transcriptome sequence of precocious trifoliate orange[D]. Wuhan: Huazhong Agricultural University, 2014. (in Chinese)
[26] 鲍思元. DNA变性聚丙烯酰胺凝胶电泳的实验教学改进. 实验科学与技术, 2015, 13(2): 122-124.
BAO S Y. Improvement of DNA denaturing polyacrylamide gel electrophoresis in experiment teaching., 2015, 13(2): 122-124. (in Chinese)
[27] Anderson J A, Churchill G A, Autrique J E, Tanksley S D, Scorells M E. Optimizing parental selection for genetic linkage maps., 1993, 36(1): 181-186.
[28] NY/T 2594-2016, 植物品种鉴定DNA分子标记法总则.
NY/T 2594-2016, General guideline for identification of plant varieties using DNA markers. (in Chinese)
[29] 胡凯. UPOV公约下我国植物新品种保护制度分析. 中国科技论坛, 2013(9): 91-96+145.
HU K. The system of new plant varieties of china under the framework of UPOV convention., 2013 (9):91-96+145. (in Chinese)
[30] 张雪英. 浅谈柑橘无病毒苗木繁育技术. 农业与技术, 2015, 35(3): 104-105.
ZHANG X Y. A brief discussion on citrus seedless seedling breeding technology., 2015, 35(3): 104-105. (in Chinese)
[31] 张建梅, 刘永忠, 李凤英, 张欣, 郑吉祥. 桂林市柑橘无病毒苗木生产现状调查及发展建议. 南方园艺,2014,25(5):36-38+40.
ZHANG J M, LIU Y Z, LI F Y, ZHANG X, ZHENG J X. Investigation and development Suggestions on the production of citrus seedless seedlings in Guilin., 2014, 25(5): 36-38+40. (in Chinese)
[32] Pimenta S, Rodrigues R, Sudré C P, Moraes J G T, Bento C S, Medeiros A M. Protecting vegetable cultivars in Brazil: a chili pepper case-study research., 2016, 34(2): 161-167.
[33] 吴鑫. 柑桔DNA指纹图谱库的构建与分析[D]. 重庆: 西南大学, 2008.
WU X. Construction and analysis of DNA fingerprinting database for citrus cultivars[D]. Chongqing: Southwest University, 2008. (in Chinese)
[34] 任小平, 郑艳丽, 黄莉, 陈玉宁, 周小静, 陈伟刚, 雷永, 晏立英, 万丽云, 廖伯寿, 姜慧芳. 花生SSR核心引物筛选及育成品种DNA指纹图谱构建. 中国油料作物学报, 2016, 38(5): 563-571.
REN X P, ZHENG Y L, HUANG L, CHEN Y N, ZHOU X J, CHEN W G, LEI Y, YAN L Y, WAN L Y, LIAO B S, JIANG H F. Selection of core SSR markers and identification of fingerprint on peanut cultivars., 2016, 38(5): 563-571. (in Chinese)
[35] 李小孟, 雷天刚, 陈伟, 吴安辉, 黎秋刚, 周天平, 王燕, 孙世秀, 陈善春, 何永睿. 沙田柚芽变品种“真龙柚”的分子标记鉴定. 中国南方果树, 2016, 45(5): 52-54.
LI X M, LEI T G, CHEN W, WU A H, LI Q G, ZHOU T P, WANG Y, SUN S X, CHEN S C, HE Y R. Identification of Shatinyu mutant cultivar “zhenlong pumelo” molecular markers., 2016, 45(5): 52-54. (in Chinese)
[36] Sun X W, Liu D Y, Zhang X X F, Li W B, Liu H, Hong W G, Jiang C B, Guan N, Ma C X, Zeng H P, Xu C H, Song J, Huang L, Wang C M, Shi J J, Wang R, Zheng X H, Lu C Y, Wang X W, Zheng H K. SLAF-seq: An efficient method of large-scale de novo SNP Discovery and genotyping using high- throughput sequencing., 2013, 8(3): e58700.
[37] Ma J Q, Huang L, Ma C L, JIN J Q, LI C F, WANG R K, ZHENG H K, YAO M Z, CHEN L.Large-scale SNP discovery and genotyping for constructing a high-density genetic map of tea plant using specific-locus amplified fragment sequencing (SLAF-seq)., 2015, 10(6): e0128798.
[38] ZHANG Y X, WANG L H, XIN H G, LI D H, MA C X, DING X, HONG W G, ZHANG X R. Construction of a high-density genetic map for sesame based on large scale marker development by specific length amplified fragment (SLAF) sequencing., 2013, 13(1): 141.
(责任编辑 李莉)
SSR markers screening for identification of citruscultivar and construction of DNA fingerprinting library
LI Yi1, MA XianFeng1, TANG Hao2, LI Na1, JIANG Dong3, LONG GuiYou1, LI DaZhi1, NIU Ying4, HAN RuiXi2, DENG ZiNiu1
(1College of Horticulture and Landscape, Hunan Agricultural University, Changsha 410128;2Development Center for Science and Technology, Ministry of Agriculture, Beijing 100122;3Institute of Citrus Research, Chinese Academy of Agricultural Sciences, Chongqing 400712;4Guangxi Academy of Specialty Crops, Guilin 541004, Guangxi)
【Objective】The purpose of the present work aims at the construction of DNA fingerprint library of citrus cultivar by using a series of SSR core primers in order to provide references for citrus nursery plant identification, cultivars registration and plant variety protection. 【Method】SSR primers were primarily screened by analyzing 8 mainly cultivated species belonging towhile the PCR products were separated by agarose gel electrophoresisthe primers that generated high polymorphic and stable PCR products were chosen for further PCR amplification with more citrus cultivars, and the PCR products were separated by polyacrylamide gel electrophoresis. the further screened primers were labeled with fluorescent and used for amplification of the all tested cultivars. The PCR products were detected by fluorescence capillary electrophoresis and the primers suitable for fluorescent capillary electrophoresis were screened according to their, number of alleles identified, and employed to construct the citrus cultivar DNA fingerprinting library, and the selected primers were used to identify the tested cultivars.【Result】Firstly, 80 pairs of polymorphic and stable primers were chosen from 362 SSR primers by 4% agarose gel electrophoresis with representative cultivars of 8 species ofgenus(including Ota ponkan, Shatian pummelo, Yuanjiang soar orange, Dahong sweet orange, Duncan grapefruit, citron, Femminello lemon, Mexico lime); Secondly, 60 pairs of SSR primers with high polymorphism, stability and clear amplification products were further selected from the above 80 pairs of primers by 6% polyacrylamide gel electrophoresis with 64 citrus cultivars and labeled with fluorescent dyes; Thirdly, 21 pairs of SSR core primers were verified for the construction of fingerprint library from above 60 pairs of primers by capillary electrophoresis with 168 citrus cultivars on the basis of, the allele number, peak reading difficulty, amplification stability and chromosome location. The corresponding reference cultivars were selected for 21 pairs of primers to eliminate the errors caused by different instruments and experimental batches. Through the multiple electrophoresis with the 5 combination groups of 21 primers according to the color and size of amplified fragment, we established 500 citrus cultivars fingerprinting library. Among the tested cultivars, 111 were identified by using only one pair of primers, 86 with the combination of two pairs of primers; and 24 with the combination of 3 pairs of primers, the remaining 279 cultivars were not able to be identified individually, they could, however, be divided into 87 groups, between which it was clearly distinguished but within which the cultivars were indistinguishable.【Conclusion】The DNA fingerprint library containing 500 citrus cultivars was constructed by using 21 pairs of screened SSR core primers, the specific markers for some cultivars were selected; 221 cultivars and 87 cultivar groups were identified; and a citrus cultivar identification system based on SSR fluorescence labeling was constructed.
; SSR; capillary electrophoresis; variety identification; DNA fingerprints
2018-01-19;
2018-03-28
国家自然科学基金(31720103915)、2018年农产品质量安全监管专项(111721301092361007)、广西柑橘新品种选育及无病毒苗木繁殖研究特聘专家岗位
李益,E-mail:1946494060@qq.com。通信作者邓子牛,E-mail:deng7009@163.com。通信作者韩瑞玺,E-mail:wudifeixue007@163.com
10.3864/j.issn.0578-1752.2018.15.0012

