大豆GmbZIP16的抗旱功能验证及分析
赵婉莹,于太飞,杨军峰,刘沛,陈隽,陈明,周永斌,马有志,徐兆师,闵东红
大豆的抗旱功能验证及分析
赵婉莹1,2,于太飞2,杨军峰3,刘沛2,陈隽2,陈明2,周永斌2,马有志2,徐兆师2,闵东红1
(1西北农林科技大学/旱区作物逆境生物学国家重点实验室,陕西杨凌 712100;2中国农业科学院作物科学研究所/农作物基因资源与 基因改良国家重大科学工程/农业部麦类生物学与遗传育种重点实验室,北京 100081;3河北旺丰种业有限公司,河北邢台 054900)
【目的】通过分析干旱条件下大豆的转录组数据,筛选获得大豆锌指蛋白GmbZIP16,对其进行功能验证,确定参与大豆抵抗干旱的分子机理。【方法】大豆干旱转录组数据分析得到上调倍数较高的锌指蛋白GmbZIP16,以大豆cDNA为模板克隆获得。并通过In-Fusion连接酶技术,构建pCAMBIA1302- GmbZIP16和pCAMBIA3301-GmbZIP16表达载体。通过液氮冷冻法将重组载体pCAMBIA1302- GmbZIP16和pCAMBIA3301-GmbZIP16分别转入农杆菌GV3101和大豆发根农杆菌K599的感受态细胞中,通过农杆菌侵染拟南芥花序以及大豆子叶节技术,产生过表达拟南芥植株以及过表达大豆毛状根复合体植株。通过半定量RT-PCR和qRT-PCR分析,确定在转基因拟南芥和大豆毛状根中能够超表达。分别将正常条件下生长2周龄的转基因和野生型拟南芥植株转移至含有不同PEG浓度(6% PEG和8% PEG)的MS0培养基上继续培养7 d,观察转基因拟南芥和对照野生型拟南芥之间的生物量差异;利用qRT-PCR分析转基因拟南芥和野生型拟南芥植物体中胁迫相关的基因表达情况。将生长良好的转大豆毛状根复合体施加25% PEG处理1周后,分别采取转大豆毛状根复合体和转空载体大豆毛状根复合体的叶片,用酶标仪测定植株的脯氨酸、丙二醛和叶绿素的含量。【结果】通过PCR技术扩增得到正确的序列,通过农杆菌转化技术得到2个稳定过表达的转拟南芥株系。通过对转基因拟南芥的表型鉴定发现转基因拟南芥在干旱处理下的生物量(鲜重和根长)及存活率比野生型显著提高。在过表达拟南芥植株中,一些与胁迫相关的基因的表达要高于在野生型,如、和。转大豆毛状根复合体植株在25% PEG处理1周后,大豆毛状根复合体叶片中叶绿素和脯氨酸的含量要显著高于转空载体大豆毛状根复合体叶片中叶绿素和脯氨酸的含量,而转大豆毛状根复合体叶片中丙二醛的含量显著低于转空载体大豆毛状根复合体叶片中丙二醛的含量。【结论】在拟南芥中过表达大豆提高了转基因拟南芥的抗旱性。过表达可以提高转基因大豆毛状根复合体对干旱的抗性。提高植物的抗旱性主要是通过影响与抗逆相关基因的表达来实现的。
大豆;GmbZIP16;抗旱性;实时荧光定量PCR
0 引言
【研究意义】细菌和真菌等病原微生物引起的生物胁迫以及干旱、高盐和低温等引起的非生物胁迫给作物的产量和品质造成的损失越来越引起人们的关注,这些环境因素严重影响了作物的生长和发育。植物在长期适应环境变化的过程中进化出自己的防御系统,主要是通过改变其基因表达和代谢途径来抵抗外界环境的变化。因此,研究植物的抗逆机制、培育抗逆新品种对提高作物产量有着重要的意义[1]。【前人研究进展】转录因子在植物抵抗生物及非生物胁迫的过程中发挥着至关重要的作用。锌指蛋白转录因子在其蛋白内部含有一个具有手指状的结构域,该结构域由若干个半胱氨酸残基组成,并通过与Zn2+结合折叠成稳定的手指状结构,锌指转录因子蛋白通过该区域实现与DNA的结合[2]。bZIP类转录因子识别DNA顺式作用元件的核心序列为ACGT,一些受ABA及光诱导的基因的启动子区域都含有这些元件[3],Choi等[4]和Fujita等[5]报道bZIP类转录因子能够激活启动子中含有ABRE元件的基因的表达。锌指蛋白也广泛存在于植物体中,且在植物体中拥有庞大的家族。1996年,Berg等[6]依据锌指蛋白的序列以及功能的不同将其分为9类,分别为C4、C6、C8、CCCH、C2H2、C2HC、C2HC5、C3HC4和C4HC3(C和H分别代表半胱氨酸和组氨酸),其中关于C2H2类锌指蛋白的研究最多。锌指蛋白在植物体中参与许多重要的调控过程,如花粉发育[7]、花形态建成以及胚发育等。近年来,越来越多的文章报道锌指蛋白在植物抵抗非生物胁迫过程中发挥着重要的作用。例如,将马绊草()锌指蛋白基因在烟草中过表达可增强转基因烟草的耐旱性,并且在干旱条件下转基因烟草能够存活并完成整个生活史,产生可育的种子,然而野生型烟草却不能在干旱条件下存活[8-9];在水稻中过表达水稻锌指蛋白基因,能增强转基因水稻对干旱胁迫的抵抗能力[10-11]。郭书巧等[12]和Huang等[13]从水稻中得到2个锌指蛋白基因和,二者属于C2H2型锌指蛋白,研究中通过组织特异性分析显示和在根、茎、叶以及幼穗中均有表达,能够被高盐、干旱、低温和ABA诱导上调表达,而的表达只被高盐和干旱诱导,不被低温和ABA诱导。将转入烟草或水稻都能增强转基因植株的耐盐性。【本研究切入点】大豆(L. Merr)是中国重要的油料作物之一,近年来,干旱等一些非生物胁迫严重影响中国大豆的产量和品质。目前,ZIP家族基因功能挖掘的研究已成为热点。ZIP家族基因在水稻、玉米等作物受到胁迫时的研究机理比较深入,对于大豆ZIP基因在干旱胁迫下如何发挥功能还未形成完整的体系。通过对大豆抗逆机制研究,挖掘大豆抗逆相关基因,应用基因工程培育大豆新品种对解决中国粮食安全具有重要的意义。【拟解决的关键问题】本研究从大豆干旱转录组数据中发现一个被干旱诱导上调表达的锌指蛋白转录因子GmbZIP16,并从大豆cDNA中克隆出,通过对过表达拟南芥和过表达大豆毛状根复合体植株的表型鉴定,确定该基因具有增强植物体抵抗非生物胁迫的功能,为进一步研究验证在植物抵抗干旱过程中的作用奠定基础。
1 材料与方法
1.1 植物及菌株材料
植物材料:普通大豆(L. Merr)William 82栽培品种及野生型拟南芥(Col-0)是由中国农业科学院作物科学研究所提供。文中WT(Wild Type)代表野生型拟南芥(Col-0)。菌株材料:农杆菌感受态GV3101由北京博迈德生物科技有限公司提供,K599发根农杆菌由中国农业科学院作物科学研究所张辉课题组提供。
1.2 大豆干旱转录组测序
剪取生长2周龄的William82大豆幼苗叶片1—2 g,将叶片放在滤纸上模拟干旱处理4 h。同时剪取正常生长下的大豆叶片作为对照,样品利用液氮速冻保存。样品由华大基因公司进行测序。后续对转录组测序结果进行差异表达基因聚类分析和代谢通路分析。
1.3 大豆GmbZIP16的扩增
利用Trizol方法,提取大豆叶片的总RNA,按照反转录试剂盒(Takara,北京)说明书的具体操作步骤,合成大豆cDNA。通过Primer Premier 5软件设计的特异性引物(bZIP16-F:5′-ATCCAATC TTCACTCACTCTCTAAT-3′;bZIP16-R:5′-CAATAA TAAGAAAAGAAGCCAAGGA-3′)。以大豆cDNA为模板,PCR扩增得到
1.4 过表达载体的构建
酶切载体:选择限制性内切酶Ⅰ对载体pCAMBIA1302进行酶切,用限制性内切酶Ⅰ和EⅡ对载体pCAMBIA3301进行酶切,并对酶切后的产物进行胶回收,合成含有载体酶切位点上端15 bp和下端15 b的载体序列的引物序列bZIP16-1302F:5′-GGGACTCTTGACCATGATGAGTAGTGTTTTTTCA-3′;bZIP16-1302R:5′-TCAGATCTACCCATGGC TACATCAGTAATTCATT-3′;bZIP16-3301F:5′-GGA CTCTTGACCATGATGAGTAGTGTTTTTTCA-3′;bZIP16-3301R:5′-ATTCGAGCTGGTCACCCTACAT CAGTAATTCATT-3′,通过DNA聚合酶的作用将载体酶切位点两端的序列插入到的两端,并对其PCR产物进行胶回收,将回收的目的基因片段及酶切的载体片段进行连接、转化后挑取单克隆进行测序,筛选阳性克隆,并通过DNAMAN软件比对验证重组质粒pCAMBIA1302-和pCAMBIA3301-连接正确。
1.5 转GmbZIP16拟南芥的获得
将重组的质粒pCAMBIA1302-转入农杆菌GV3101感受中,并通过拟南芥花序侵染法侵染野生型拟南芥。收取T0代种子后利用具有潮霉素抗性的MS培养基筛选阳性株系。阳性株生长4周后,提取其叶片的RNA反转录为cDNA,以WT的模板为对照,设计引物(:5′-GCACAAGTACCT TCCCCACA-3′::5′-AGTAATGAAAC CGGCGAGGG-3′)进行PCR检测,确定能够在转基因拟南芥中超表达。继续繁殖到T3代。
1.6 转GmbZIP16拟南芥的表型鉴定
将T3代转基因拟南芥的种子和野生型经过清洗和春化后,移入MS培养基上,在正常条件下生长9 d左右,将拟南芥幼苗移入含有不同浓度PEG(6% PEG和8% PEG)的MS固体培养基上继续生长1周,观察转基因拟南芥和野生型拟南芥在不同PEG浓度处理下的表型,并对其生物量进行测定分析。
将T3代转基因拟南芥的种子和野生型经过清洗和春化后,移入MS培养基上,在正常条件下生长2周左右,将拟南芥幼苗移入土中继续生长1周后。控水处理1周,观察转基因拟南芥和野生型拟南芥的表型,记录控水1周后的存活率。并剪取转基因拟南芥和野生型拟南芥叶片测量其相对电导率。
1.7 叶片相对电导率的测定
剪取上述试验中控水1周和在正常条件下生长的转基因拟南芥和野生型拟南芥的叶片0.2 g,将其冲洗干净后,剪成0.5 cm左右的小长条,放入到含有6 mL去离子水的试管中,待室温静置25 min后,用电导仪分别测量6 mL去离子水的电势即为V0及含有叶片的去离子水试管中的电势即为Vm,再用沸水将含有叶片的去离子水煮沸30 min,待冷却至室温后在测量含有叶片的离心管中的电势即为Vn。相对电导率的计算公式为:(Vm-V0)×100/(Vn-V0)。
1.8 转GmbZIP16拟南芥中胁迫响应基因的表达
选取正常生长4周龄的转拟南芥和野生型,用清水冲洗根部后,放到滤纸上分别在速旱0 h和4 h时进行取样,利用Trizol方法,提取大豆叶片的总RNA,将其反转录为cDNA。以上述不同时间段不同样品cDNA为模板,对胁迫响应基因进行qRT-PCR分析。
1.9 转GmbZIP16大豆复合体植株的产生
将大豆种子种入土中大约生长1周龄左右对大豆幼苗进行农杆菌侵染。在大豆种子萌发生长期间准备农杆菌材料。将方法1.4中构建的重组载体pCAMBIA3301-及空载体pCAMBIA3301通过液氮冷冻的方法转入发根农杆菌K599的感受态细胞中,并将其涂于含有卡那霉素和链霉素的二抗固体培养基中让其生长2—3 d。用注射器针头挑取在上述培养基中生长的农杆菌菌落,并将其扎入大豆子叶节中,将已转入农杆菌的大豆幼苗在黑暗中培养12 h,黑暗培养过后,将已侵染的大豆幼苗放置在高温高湿的环境中生长,大约生长1周左右,覆土至盖过大豆的侵染部位,充分浇水,至大豆毛状根生长至5—10 cm,采取少许的大豆毛状根,提取总RNA并反转录cDNA,对其进行qRT-PCR检测分析。确定能够在大豆毛状根中过表达。将大豆幼苗取出,剪去侵染部位以下的大豆真根,并将剩余地含有大豆毛状根复合体植株继续移入土中,生长1周,待大豆植株能够恢复正常生长时,进行后续试验。
1.10 转GmbZIP16大豆复合体植株的表型鉴定及生理指标的测定
使用25% PEG处理上述方法中得到的大豆复合体(转大豆复合体植株和转空载体大豆复合体植株),处理2周,观察其在25% PEG处理下的表型。在25% PEG处理1周时,采取大豆复合体的叶片,并通过试剂盒对大豆叶片的脯氨酸、丙二醛及叶绿素进行提取,并通过酶标仪测量其在不同波长下的吸光值。通过计算得出在不同处理下的叶片中脯氨酸、丙二醛及叶绿素的含量。
2 结果
2.1 干旱胁迫处理条件下大豆的转录组数据分析
将干旱处理的大豆与在正常生长条件生长的大豆进行转录组测序分析,聚类分析结果显示,当大豆植株遇到干旱胁迫时,其体内许多转录因子家族出现差异表达(图1-A),在这些差异表达的转录因子数据库中,进行了数据统计分析结果显示有60种类型的转录因子在干旱胁迫下出现了差异表达(图1-B),数据统计显示,大量MYB和AP2-EREBP类型的转录因子的在干旱处理前后表达量发生了改变,在转录组数据中发现了与激素ABA合成相关的一些转录因子的表达量同样发生了变化,如ABI3。在这些转录因子中,报道最多的与干旱胁迫相关的转录因子家族有WRKY类型转录因子、MYB类型转录因子、AP2/EREBP类型的转录因子及bZIP家族类型的转录因子。以上多种转录因子在植物抵抗非生物胁迫过程中扮演着重要角色。
将这些差异表达转录因子及其所在的调控网络途径进行统计发现,这些转录因子主要富集于30种植物调控网络途径中(图2),在这30种途径中,涉及到植物激素信号传导途径的转录因子最多,其次是涉及甘油脂代谢和生物周期节律途径的转录因子。在这些转录因子家族中,锌指蛋白类转录因子在干旱胁迫下表达量在干旱处理前后的log2(FoldChange)值达到10左右。说明,在大豆干旱处理下诱导表达倍数较高(电子附表1,其中加粗为,表中提供部分转录组数据内容,且按照差异表达量由高到低的顺序依次排列)。因此,将锌指转录因子作为进一步研究的对象。
2.2 转GmbZIP16拟南芥的获得
通过对转基因拟南芥T1代苗期取样进行RT-PCR检测(图3-A)。并通过荧光定量PCR进行阳性株系相对表达量进行定量(图3-B)。说明外源的成功转化到野生型拟南芥中,相对表达量最高达到10倍左右。继续种子繁殖,得到T3代纯合植株,进行后续试验。
2.3 转GmbZIP16拟南芥的表型鉴定
对T3代转基因拟南芥和野生型使用不同浓度PEG处理进行表型鉴定(图4)。结果显示,在6% PEG和8% PEG处理条件下,转基因拟南芥的长势要优于野生型拟南芥。PEG处理条件下转基因拟南芥的总根长要长于野生型拟南芥,分别增长了39.6%(6% PEG,2个株系的平均值)和74.2%(8% PEG,2个株系的平均值);鲜重分别增加了35.4%(6% PEG,2个株系的平均值)和57.9%(8% PEG,2个株系的平均值)。
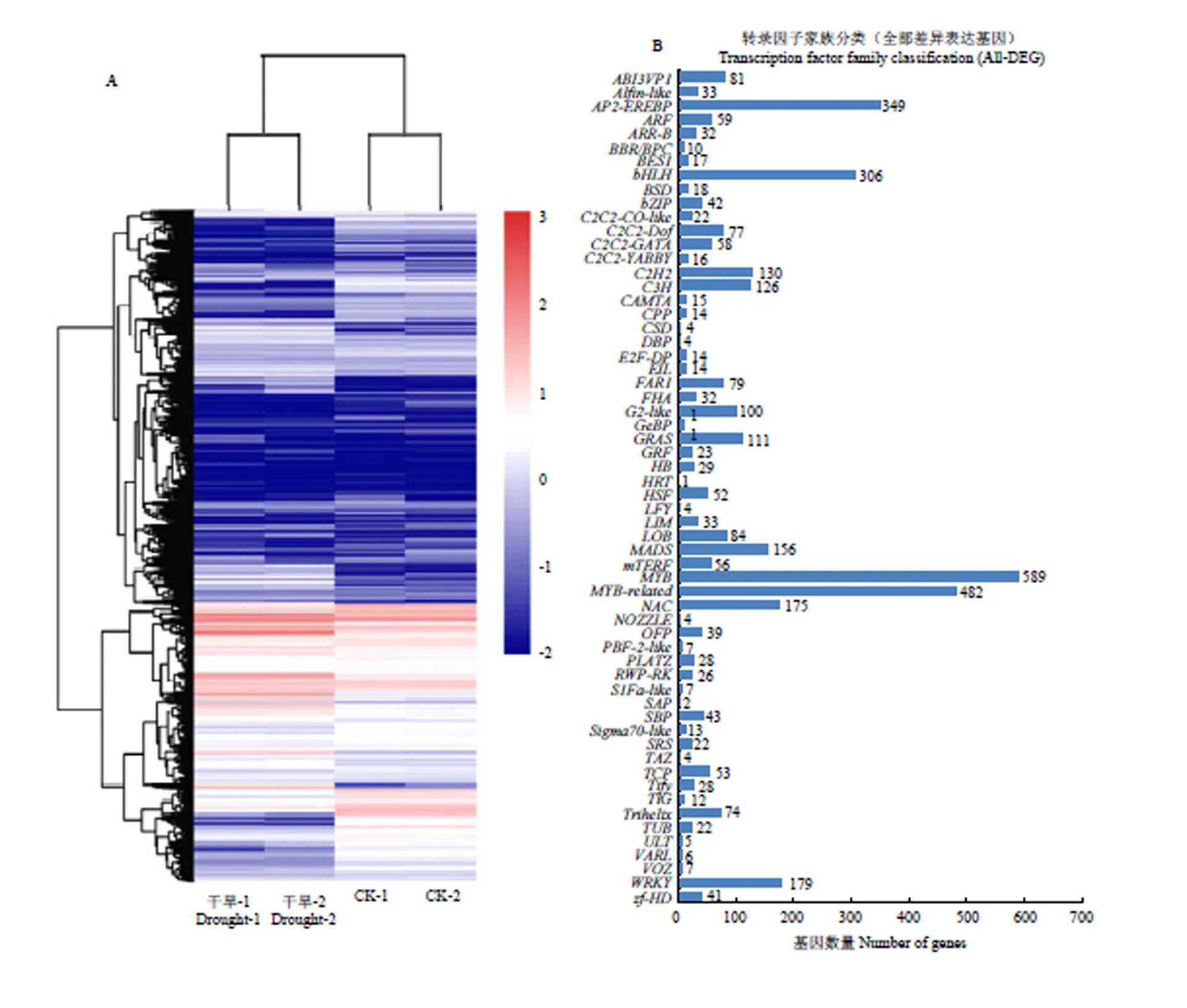
A:转录组数据中差异表达的转录因子家族的聚类分析;B:转录组数据中差异表达的转录因子家族的统计分析
将T3代纯合的转基因拟南芥和野生型拟南芥的种子经过清洗和春化后转移至MS0培养基上生长2周,将其转移至土中继续培养1周后对拟南芥的进行控水处理,结果显示,在控水前转基因拟南芥和野生型拟南芥的生长并无差异(图5-A),但是控水1周后80%以上的转基因拟南芥能够存活,然而只有20%左右的野生型拟南芥能够存活(图5-C)。采取控水1周后的转基因拟南芥和野生型拟南芥的叶片,对叶片的相对电导率进行了测定,结果显示在干旱处理1周后,转基因拟南芥的相对电导率要低于野生型的相对电导率(图5-D)。
2.4 大豆GmbZIP16增强了拟南芥胁迫响应基因的表达
通过提取干旱处理0和4 h的4周龄的T3代纯合转基因拟南芥和野生型拟南芥幼苗的总RNA,并将其反转录cDNA。通过qRT-PCR检测胁迫响应基因的表达。结果显示,在正常生长条件下,转基因拟南芥植株中的胁迫响应基因、和的表达量与在野生型拟南芥中的表达量相比有所提高,特别是在干旱处理4 h后,这些胁迫响应基因在转基因拟南芥植株中的表达量显著高于在野生型拟南芥中的表达量(图6)。
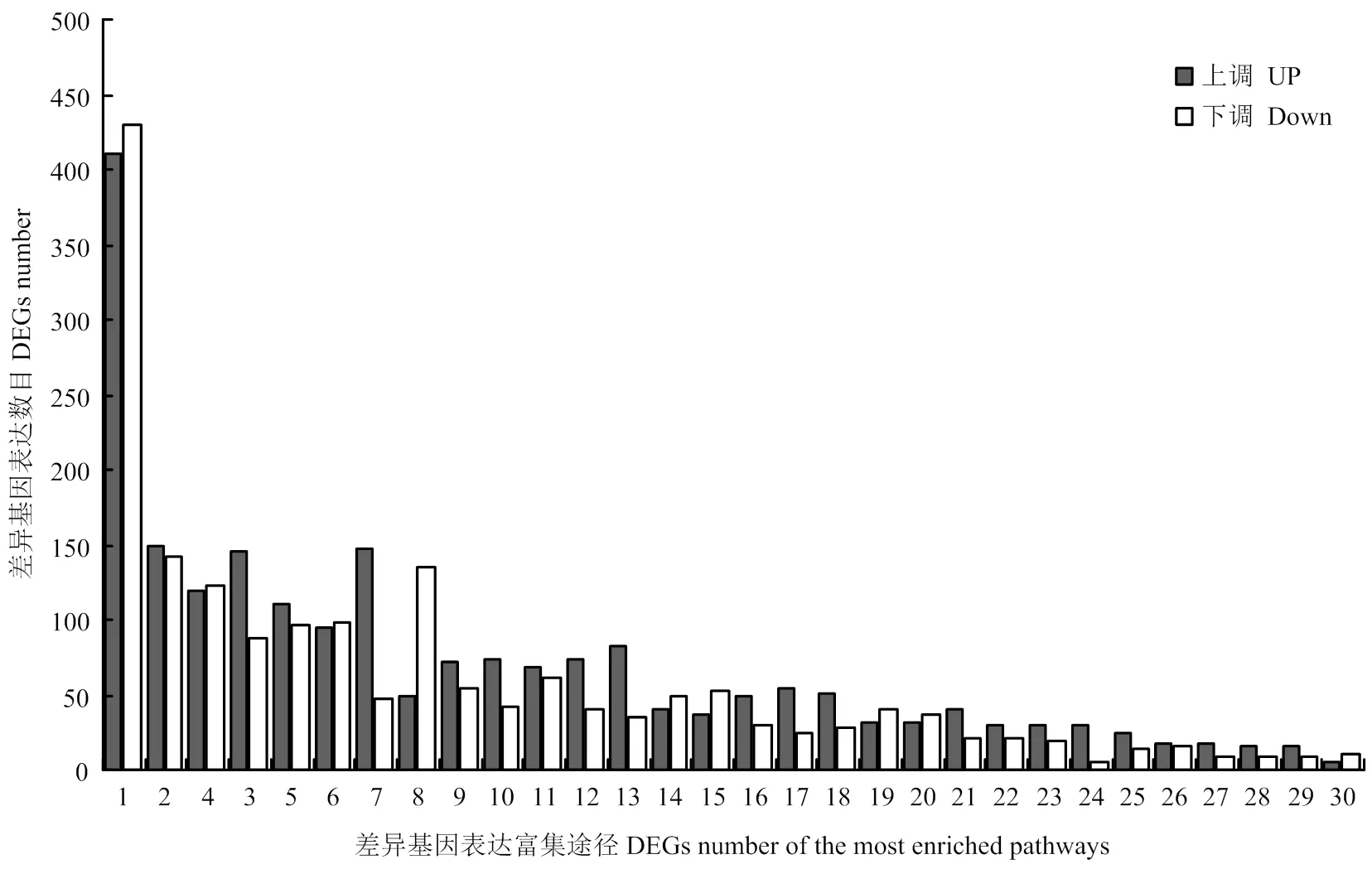
1:植物激素信号转导Plant hormone signal transduction;2:甘油酯代谢Glycerolpidmetabolism;3:昼夜节律Circadian rhythm-plant;4:糖酵解/糖质新生Glycolysis/Gluconeogenesis;5:过氧物酶体Peroxisome;6:甘油磷脂代谢Glycerophospholipid metabolism;7:卟啉和叶绿素代谢Porphyrin and ChlorophyII metabolism;8:调节自噬Regulation of autophagy;9:鞘脂类代谢Sphingolipid metabolism;10:果糖和甘露糖Fructose and mannose metabolism;11:类黄酮生物合成Flavonoid biosynthesis;12:磷酸戊糖途径Pentose phosphate pathway;13:脂肪酸代谢Fatty acid metabolism;14:亚麻酸代谢Alpha-Linolenic acid metabolism;15:醚脂类代谢Ether lipid metabolism;16:角质、软木脂和蜡合成Cutin, Suberine and wax biosynthesis;17:鞘糖脂生物合成-神经节Glycosphingolipid biosynthesis-ganglio series;18:脂肪酸生物合成Fatty acid biosynthesis;19:异黄酮生物合成Isoflavonoid biosynthesis;20:黄酮和黄酮醇生物合成Flavone and flavonol biosynthesis;21:脂肪酸延伸Fatty acid elongation;22:叶酸生物合成Folate biosynthesis;23:柠檬烯和松萜降解Limonene and pinene degradation;24:光合作用-天线蛋白Photosynthesis-antenna proteins;25:生物素代谢Biotin metabolism;26:牛磺酸和亚牛磺酸代谢Taurine and hypotaurine metabolism;27:花青素生物合成Anthocyanin biosynthesis;28:葡萄糖鞘脂类Glycosphingolipid biosynthesis-globo series;29:单萜类合成Monoterpenoid biosynthesis;30:酮体的合成和降解Synthesis and degradation of ketone bodies
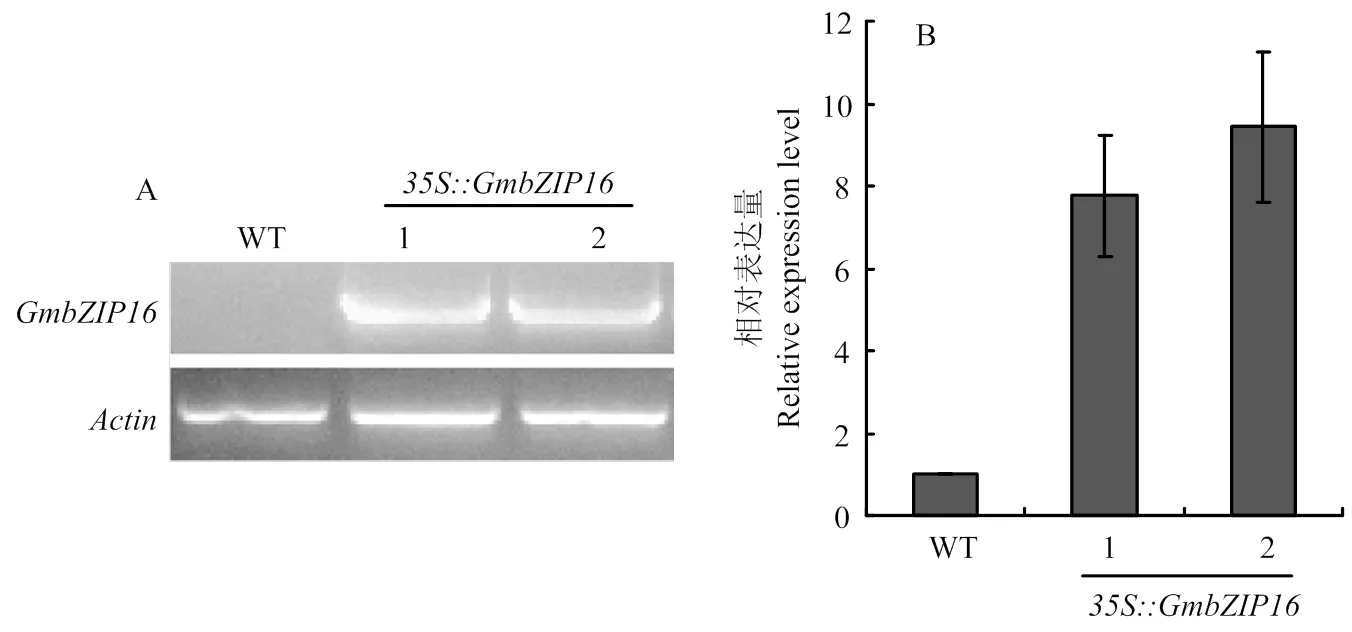
A:转GmbZIP16基因的半定量PCR鉴定;B:转GmbZIP16基因拟南芥株系相对表达量检测
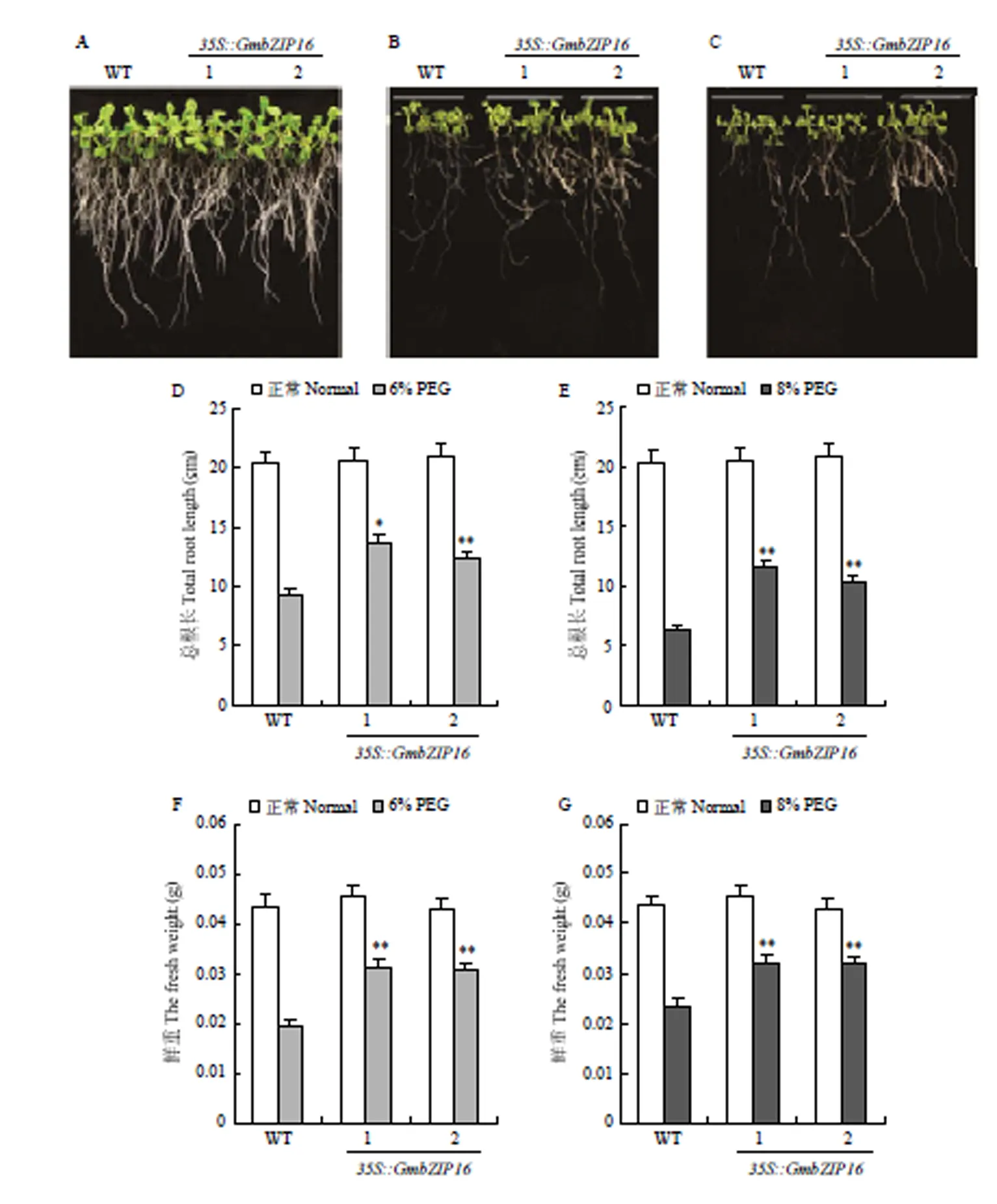
A:转GmbZIP16拟南芥和野生型拟南芥在正常生长条件下的表型;B:转GmbZIP16拟南芥和野生型拟南芥在6% PEG处理下的表型;C:转GmbZIP16拟南芥和野生型拟南芥在8% PEG处理下的表型;D和E:转GmbZIP16拟南芥和野生型拟南芥在PEG处理下的总根长;F和G:转GmbZIP16拟南芥和野生型拟南芥在PEG处理下的鲜重。*:差异显著(P<0.05);**:差异极显著(P<0.05)。下同
2.5 转GmbZIP16大豆毛状根复合体的表型鉴定及生理指标的测定
为了进一步分析大豆能够增强植物的抗旱性,对过表达大豆复合体中毛状根进行qRT-PCR检测(图7-A),结果表明,在过表达复合体毛状根中,表达水平是空载对照复合体毛状根的4倍以上。表明已在大豆毛状根中超表达。通过对大豆毛状根复合体植株的表型鉴定,在25% PEG处理2周后,转空载体大豆毛状根复合体植株叶片的黄化程度要大于转基因大豆毛状根复合体植株(图7-C),通过对处理1周的大豆毛状根复合体植株的叶片脯氨酸、丙二醛及叶绿素的提取分析,得出在25% PEG处理1周后,转大豆毛状根复合体的丙二醛含量显著低于对照(图7-D),且脯氨酸和叶绿素含量均高于对照组(图7-E和图7-F)。
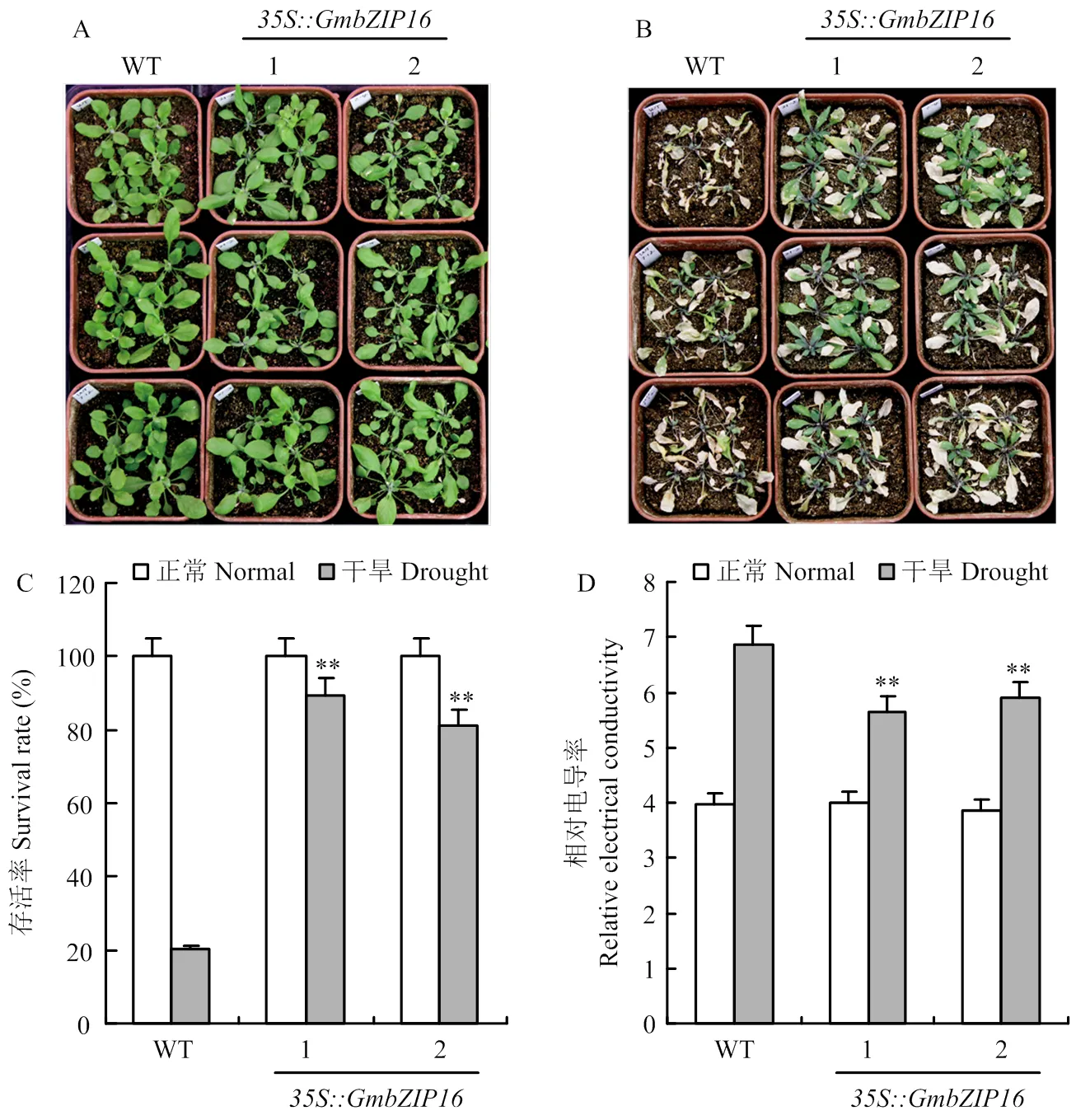
A:转GmbZIP16拟南芥和野生型拟南芥在处理前的表型;B:转GmbZIP16拟南芥和野生型拟南芥在干旱处理7 d后的表型;C:转GmbZIP16拟南芥和野生型拟南芥在干旱处理下的存活率;D:转GmbZIP16拟南芥和野生型拟南芥在干旱处理下的相对电导率
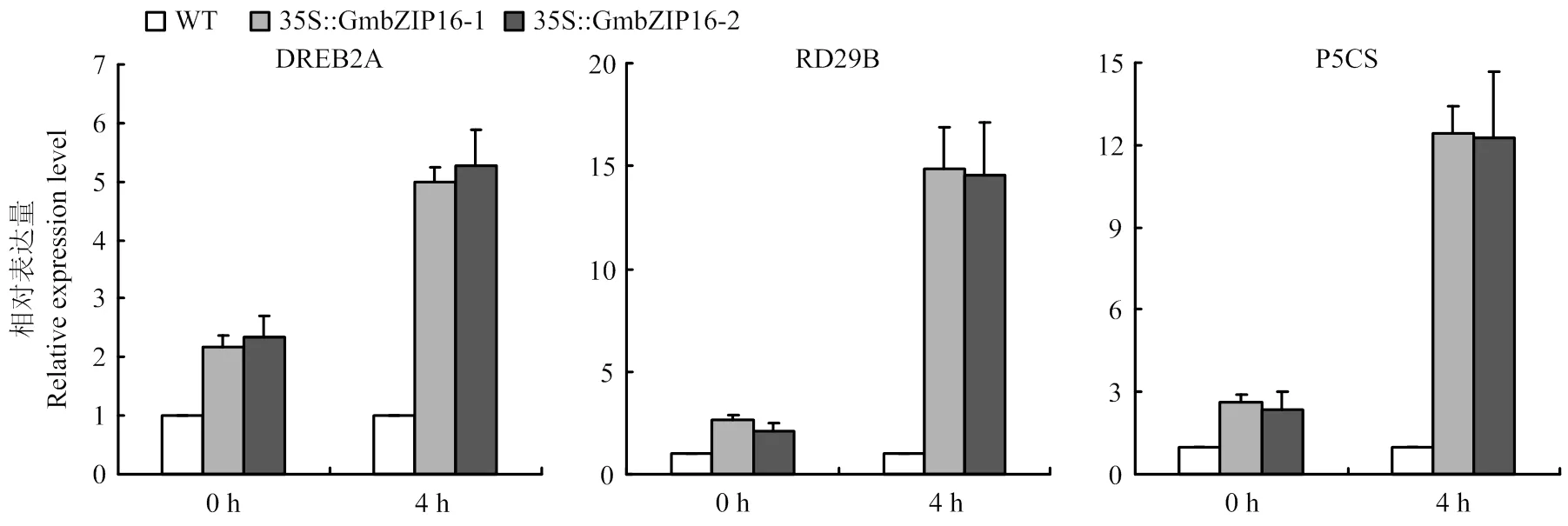
图6 胁迫响应基因在转基因拟南芥和野生型拟南芥植株中的qRT-PCR分析
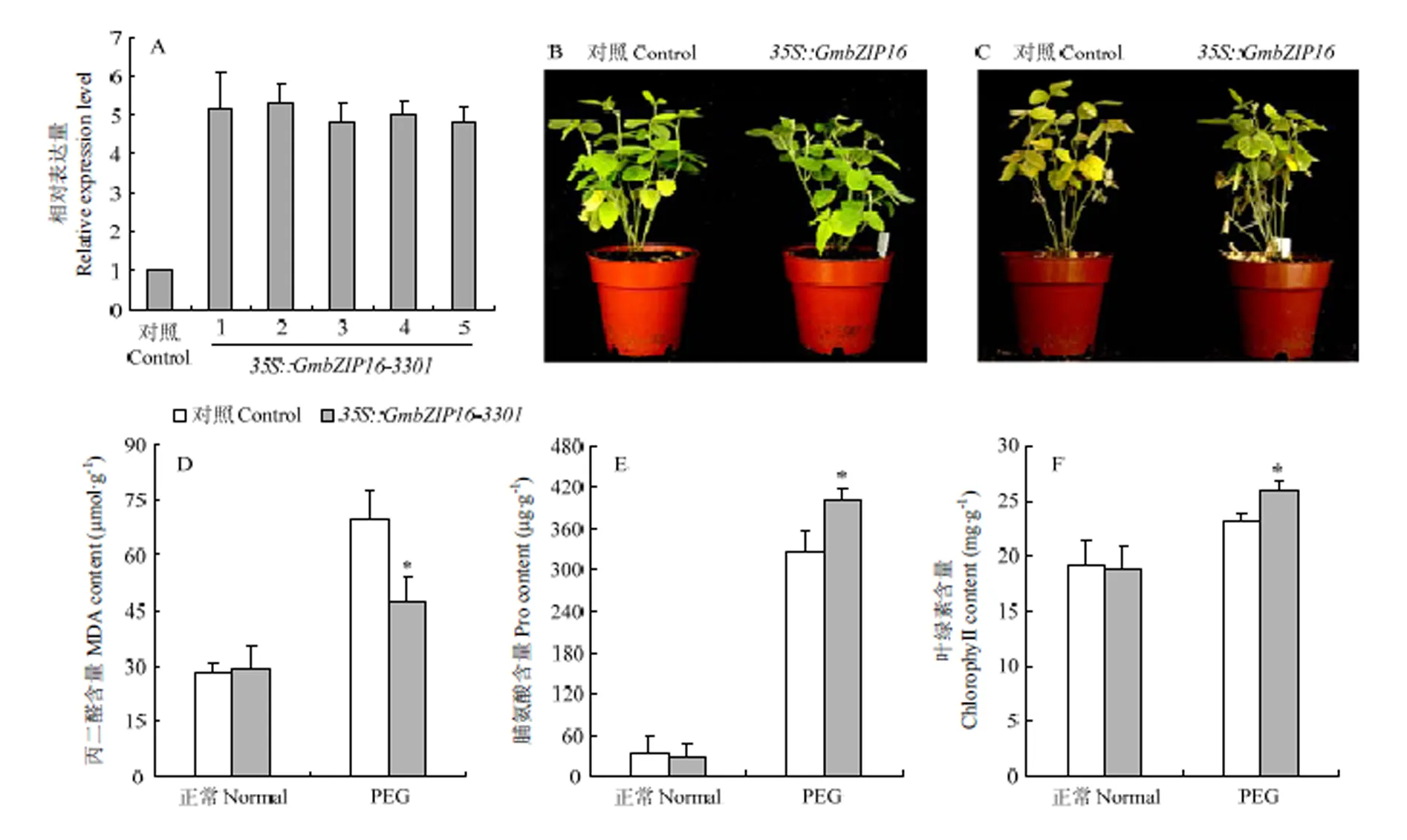
A:转GmbZIP16大豆毛状根复合体不同株系表达量检测;B:转GmbZIP16大豆毛状根复合体及对照在25% PEG处理前的表型;C:转GmbZIP16大豆毛状根复合体及对照在25% PEG处理2周后的表型;D:转GmbZIP16大豆毛状根复合体及对照在25% PEG处理1周后的叶片丙二醛含量;E:转GmbZIP16大豆毛状根复合体及对照在25% PEG处理1周后的叶片脯氨酸含量;F:转GmbZIP16大豆毛状根复合体及对照在25% PEG处理1周后的叶片叶绿素含量
3 讨论
全球气候的不断变化,使植物的生长受到了严峻考验。为了适应环境的变化,植物体通过对自身信号传导及能量代谢等途径的调控来增强对外界不良环境的抵抗[14]。在这一系列复杂的过程中,转录因子发挥着重要的调控作用。植物体通过信号的逐级传导使与抗逆相关的转录因子的活性增加,从而调控胁迫相关基因的表达来增强植物体对外界环境的抵抗[15]。因此,研究转录因子的功能,对提高植物的抗逆性具有重要的意义。锌指蛋白转录因子广泛存在真核生物体中[16],且具有广泛的生物学功能,在生物体中参与很多重要的代谢途径,如影响植物细胞的生长发育、调控植物的光周期以及参与植物抵抗非生物胁迫等等[17]。本研究通过对大豆干旱转录组数据分析,发现一个受到干旱诱导高表达的锌指转录因子,并将其命名为GmbZIP16。并从大豆的转录本中成功的将其克隆。
近年来越来越多文章报道大豆锌指转录因子参与植物抵抗非生物胁迫的过程。Liao等[18]报道在拟南芥中过表达大豆增强了转基因拟南芥对盐的抗性,而这种耐盐性仅仅体现在种子的萌发阶段;然而在拟南芥中过表达、和,发现这3个大豆锌指转录因子负调控ABA信号途径,减弱了转基因拟南芥对ABA的敏感性,增强了转基因拟南芥对低温和高盐的抗性[19]。Gao等[20]发现一个大豆锌指转录因子,并将其过表达与拟南芥和小麦中,结果显示,大豆在拟南芥和小麦中过表达提高了转基因植株对多种非生物胁迫的抵抗。本文将大豆在拟南芥及大豆毛状根中过表达,并通过对转基因拟南芥及转基因大豆毛状根复合体植株的表型鉴定,发现在植物中过表达大豆提高了转基因植株的抗旱性(图4、图5和图7)。这些结果表明大豆在大豆的抗旱性方面发挥着重要的作用。
当非生物胁迫来临时,植物体中的锌指转录因子蛋白以二聚体的形式与DNA顺式作用元件相结合,从而调控下游与胁迫相关的靶基因的表达[20]。利用qRT-PCR技术,对转拟南芥植株与野生型拟南芥中的、和表达量进行了实时检测。拟南芥被报道与拟南芥抵抗非生物胁迫过程中扮演这重要的角色,将在拟南芥中过表达能够增强拟南芥植株中、及等胁迫响应基因的表达[21]。在拟南芥中,是一个与脯氨酸合成相关的关键基因[22],而脯氨酸是植物抵抗非生物胁迫过程中一个重要的渗透保护物质[23]。结果显示,在过表达大豆拟南芥的植物体中,、和的表达量会有显著提高,特别是在干旱处理4 h后,它们的表达量会有明显的提高(图6),这些结果表明大豆在拟南芥中过表达,增强了胁迫基因的表达从而增强了转基因植株对干旱的抵抗能力。
4 结论
获得大豆,并确定能够增强转基因植株对干旱的抵抗能力,在拟南芥中过表达可以增强拟南芥中与胁迫响应相关的基因的表达,从而增强转基因植株的抗逆性。
附表1 差异表达转录因子基因的统计分析
Table S1 Statistical analyses of differential expressions of the transcription factors
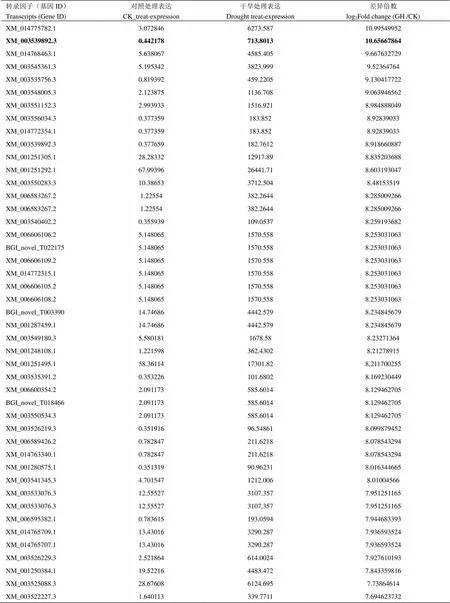
转录因子(基因ID)Transcripts (Gene ID)对照处理表达CK_treat-expression干旱处理表达Drought treat-expression差异倍数log2Fold change (GH /CK) XM_014775782.13.0728466273.58710.99549952 XM_003539892.30.442178713.801310.65667864 XM_014768463.15.6380674585.4059.667632729 XM_003545361.35.1953423823.9999.52364764 XM_003535756.30.819392459.22059.130417722 XM_003548005.32.1238751136.7089.063946562 XM_003551152.32.9939331516.9218.984888049 XM_003556034.30.377359183.8528.92839033 XM_014772354.10.377359183.8528.92839033 XM_003539892.30.377659182.76128.918660887 NM_001251305.128.2833212917.898.835203688 NM_001251292.167.9939626441.718.603193047 XM_003550283.310.386533712.5048.48153519 XM_006583267.21.22554382.26448.285009266 XM_006583267.21.22554382.26448.285009266 XM_003540402.20.355939109.05378.259193682 XM_006606106.25.1480651570.5588.253031063 BGI_novel_T0221755.1480651570.5588.253031063 XM_006606109.25.1480651570.5588.253031063 XM_014772315.15.1480651570.5588.253031063 XM_006606105.25.1480651570.5588.253031063 XM_006606108.25.1480651570.5588.253031063 BGI_novel_T00339014.746864442.5798.234845679 NM_001287459.114.746864442.5798.234845679 XM_003549180.35.5801811678.588.23271364 NM_001248108.11.221598362.43028.21278915 NM_001251495.158.3611417301.828.211700255 XM_003535391.20.353226101.68028.169230449 XM_006600354.22.091173585.60148.129462705 BGI_novel_T0184662.091173585.60148.129462705 XM_003550534.32.091173585.60148.129462705 XM_003526219.30.35191696.548618.099879452 XM_006589426.20.782847211.62188.078543294 XM_014763340.10.782847211.62188.078543294 NM_001280575.10.35131990.962318.016344665 XM_003541345.34.7015471212.0068.01004566 XM_003533076.312.555273107.3577.951251165 XM_003533076.312.555273107.3577.951251165 XM_006595382.10.783615193.05947.944683393 XM_014765709.113.430163290.2877.936593524 XM_014765707.113.430163290.2877.936593524 XM_003526229.32.521864614.00247.927610193 NM_001250384.119.522164483.4727.843359816 XM_003525088.328.676086124.6957.73864614 XM_003522227.31.640113339.77117.694623732 XM_006582175.242.613688600.9677.657038471 XM_006582174.242.613688600.9677.657038471 XM_003526128.342.613688600.9677.657038471 BGI_novel_T0034421.639543327.21937.640820568 XM_003536435.32.072706406.01177.613862265 XM_003540037.3112.010120959.487.547830256 XM_003555003.30.769511140.79347.51542311 XM_003524356.30.33667560.285317.484307845 NM_001248578.11.634442290.49667.473578999 XM_006601151.20.765817135.67597.468947932 XM_006601149.20.765817135.67597.468947932 XM_003553745.31.632353279.70697.420818746 NM_001248972.126.046944443.0077.414278518 XM_003520654.34.246435721.35447.408312447 XM_006573726.28.1686671385.5927.406186143 NM_001255872.20.76422128.34927.39187046 XM_006576060.20.33283155.778347.388770909 NM_001250398.11.636804273.56077.384835537 NM_001248860.123.438173904.3617.380082399 NM_001248860.123.438173904.3617.380082399 XM_003542945.310.35191635.8567.304006045 XM_003526787.368.4063410458.037.25626551 XM_014762182.10.32787647.287717.172171038 XM_003533977.30.32787647.287717.172171038 XM_006587303.20.32787647.287717.172171038 XM_014762181.10.32787647.287717.172171038 XM_006587302.20.32787647.287717.172171038 BGI_novel_T01845129.099594100.3917.138619044 BGI_novel_T01845129.099594100.3917.138619044 XM_006600337.229.099594100.3917.138619044 XM_006600337.229.099594100.3917.138619044 XM_003550526.329.099594100.3917.138619044 XM_003550526.329.099594100.3917.138619044 XM_003549364.32.929455372.2036.989313287 XM_003549364.32.929455372.2036.989313287 XM_003538059.36.84471865.79516.982892432 XM_003543209.34.227683512.6446.92194612 BGI_novel_T00508224.723332863.5646.855794636 NM_001248384.124.723332863.5646.855794636 XM_014766943.16.838204766.41496.808364244
表中加粗部分代表在转录组测序中的差异表达数据
The bold part of the table is represented that the differential expression data ofin transcriptome sequencing
[1] Zhu J K. Plant salt tolerance., 2001, 6: 66-71.
[2] Frankel A D, Pabo C O. Fingering too many proteins., 1988, 53: 675.
[3] Nakagawa H, Ohmiya K, Hattori T, A rice bZIP protein, designated, is rapidly induced by abscisic acid., 1966, 9: 217-227.
[4] Choi H, Hong J, Kang J. ABFs, a family of ABA-responsive element binding factors., 2000, 21: 1723-1730.
[5] Fujita Y, Fujita M, Satoh R, KIM S Y. AREB1 is a transcription activator of novel ABRE-De-pendent ABA signaling that enhances drought stress tolerance in., 2005, 17: 3470-3488.
[6] Berg J M, Shi Y. The galvanization of biology: a growing appreciation for the roles of zinc., 1996, 271: 1081-1085.
[7] 韩莹琰, 张爱红, 范双喜, 曹家树. 十字花科植物C2H2型锌指蛋白新基因同源序列克隆与进化分析. 核农学报, 2011, 25(5): 916-921.
Han Y Y, Zhang A H, Fan S X, Cao J S. Cloning and evolutionary analysis of homologous sequences of a novel gene encoding C2H2 zinc finger protein in cruciferae., 2011, 25(5): 916-921. (in Chinese)
[8] Saad R B, Zouari N, Ramdhan W B, Azaza J, Meynard D, Guiderdoni E, Hassairi A. Improved drought and salt stress tolerance in transgenic tobacco overexpressing a novel A20/AN1 zinc-fingergene isolated from the halophyte grasss., 2010, 72: 171-190.
[9] Saad R B, Romdhan W B, Zouari N, Azaza J, Meynard D, VerdeilJ L, Guiderdoni E, Hassairi A. Promoter of the AlSAP gene from the halophyte grassdirects developmental-regulated, stress-inducible, and organ-specific gene expression in transgenic tobacco., 2010, 20(5): 1003-10018.
[10] Huang J, Sun S J, Xu D Q, Yang X, Bao Y M, Wang Z F, Tang H J, Zhang H. Increased tolerance of rice to cold, drought and oxidative stresses mediated by the overexpression of a gene that encodes the zinc finger protein ZFP245.2009, 389: 556-561.
[11] Jain M, Tyagi A K, Khurana J P. Constitutive expression of a meiotic recombination protein gene homolog, OsTOP6A1, from rice confers abiotic stress tolerance in transgenicplants., 2008, 27: 767-778.
[12] 郭书巧, 黄骥, 江燕, 张红生.水稻C2H2型锌指蛋白基因RZF71的克隆与表达分析. 遗传, 2007, 29(5): 607- 613.
Guo S Q, Huang J, Jiang Y, Zhang H S. Cloning and characterization of RZF71 encoding a C2H2-type zinc finger protein from rice., 2007, 29(5): 607-613. (in Chinese)
[13] Huang J, Yang X, Wang M M, Tang H J, Ding L Y, Shen Y, Zhang H S. A novel rice C2H2-type zinc finger protein lacking DLN-box/EAR-motif plays a role in salt tolerance., 2007, 1769: 220-227.
[14] 李芳兰, 包维楷.植物叶片形态解剖结构对环境变化的响应与适应.植物学通报, 2005, 22: 118-127.
Li F L, Bao W K. Responses of the morphological and anatomical structure of the plant leaf to environmental change, 2005, 22:118-127. (in Chinese)
[15] 田超, 王冉, 彭艳, 张志昌, 曹林. 植物抗逆胁迫相关蛋白激酶的研究进展. 安徽农业科学, 2015, 43(20): 4-6.
Tian C, Wang R, Peng Y, Zhang Z C, Cao L. Research advance of protein kinase in plant resistant to adversity stress.2015, 43(20): 4-6. (in Chinese)
[16] 杨颖, 高世庆, 唐益苗, 冶晓芳, 王永波, 刘美英, 赵昌平.植物bZIP转录因子的研究进展. 麦类作物学报, 2009, 29(4): 730-737.
Yang Y, Gao S Q, Tang Y M, Ye X F, Wang Y B, Liu M Y, Zhao C P. Advance of bZIP transcription factors in plants., 2009, 29(4): 730-737. (in Chinese)
[17] 王伟英, 李海明, 戴艺民, 林江波.植物锌指蛋白的功能研究进展. 中国园艺文摘, 2016, 32(7):3-5.
Wang W Y, Li H M, Dai Y M, Lin J B. Advances on the function of plant zinc finger protein., 2016, 32(7): 3-5. (in Chinese)
[18] Liao Y, Zhang J S, Chen S Y, Zhang W K. Role of soybeanunder abscisic acid and salt stresses.y, 2008, 50: 221-230.
[19] Liao Y, Zou H F, Wei W, Hao Y J, Tian A G, Huang J, Liu Y F, Zhang J S, Chen S Y. Soybean,andgenes function as negative regulator of ABA signaling and confer salt and freezing., 2008, 228: 225-240.
[20] Gao S Q, Chen M, Xu Z S, Zhao C P, Li L C, Xu H J, Tang Y M, Zhao X , Ma Y Z. The soybean GmbZIP1 transcription factor enhances multiple abiotic stress tolerances in transgenic plants., 2011, 75: 537-553.
[21] Sakuma Y, Maruyama K, Osakabe Y, Qin F, Seki M, Shinozaki K, YAMAGUCHI S K. Functional analysis of antranscription factor, DREB2A, involved in drought- responsive gene expression., 2006, 18: 1292-1309.
[22] Strizhov N, Abraham E, Okresz L, BLICKLING S, ZILBERSTEIN A, SCHELL J, KONCZ C, SZABADOS L. Differential expression of twogenes controlling proline accumulation during salt stress requires ABA and is regulated by ABA1, ABI1 and AXR2 in., 1997, 12: 557-569.
[23] 李玲, 余光辉, 曾富华.水分胁迫下植物脯氨酸累积的分子机理. 华南师范大学学报, 2003, 1: 126-134.
Li L, Yu G H, Zeng F H. The plant molecular mechanism of proline accumulation under water stress., 2003, 1: 126-134. (in Chinese)
(责任编辑 李莉)
Verification and analyses of soybeangene resistance to drought
ZHAO WanYing1,2, YU TaiFei2, YANG JunFeng3, LIU Pei2, CHEN Jun2, CHEN Ming2, ZHOU YongBin2, MA YouZhi2, XU ZhaoShi2, MIN DongHong1
(1Northwest A & F University/State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi;2Institute of Crop Science, Chinese Academy of Agricultural Sciences/National Key Facility for Crop Gene Resources and Genetic Improvement/ Key Laboratory of Biology and Genetic Improvement of Triticeae Crop, Ministry of Agriculture, Beijing 100081;3Hebei Wangfeng Seed Industry Co., Ltd. Xingtai 054900, Hebei)
【Objective】Soybean GmbZIP16 protein was screened by analyzing soybean drought transcriptome. Functions of soybeanwere verified by analyzing the phenotypic characterization of transgenicand soybean hairy root complexes. On the basis of experimental result analyses above-mentioned, we could determine thatwas involved in the drought resistance process.【Method】soybeanwas found out by analyzing soybean drought transcriptome and cloned by PCR, which soybean cDNA as a template, and then ligated the fragment to pCAMBIA1302 and pCAMBIA3301 expression vectors by in-fusion ligase. The recombinant pCAMBIA1302-GmbZIP16 and pCAMBIA3301-GmbZIP16 vectors were transferred intoGV3101 and K599 competent cells by liquid nitrogen freeze-melt method, respectively. The transgenicplants and transgenic soybean hairy root complexes were generated by-mediated transformation method. The transgenicplants were identified by semi quantitative PCR and quantitative real time PCR. which demonstratedcould over express in transgenicand transgenic soybean hairy root complexplants. 2 weeks old transgenicand WT plants grew under normal conditions were transferred into MS0 solid medium supplement with 6% and 8% PEG for 7d. Biomass differences between transgenicand WT plants were investigated and analyzed. The different expression of stress- related genes betweenand WT plants were analyzed by qRT-PCR. The transgenicsoybean hairy root complexes and control group plants were treated with 25% PEG for 7 days, and then the leaf samples of transgenicsoybean hairy root complex and control group plants were taken. The proline content, MDA content and chlorophyll content of leaf samples were measured by Multiskan Spectrum Microplate Spectrophotometer.【Result】Thegene was isolated by PCR technology. The two transgeniclines were obtained by-mediated transformation method. Compared with the WT plants, the transgeniclines had higher biomass (the fresh weight and the root length) and survival rate under drought stress by phenotypic characterization experiment. The expression levels of some relative genes such as,andwere improved in transgenic, compared with the WT plants. The leaves of transgenicsoybean hairy root complex plants had the higher proline and chlorophyll content and a lower MDA content than that of the control group plants under the deal with 25% PEG. 【Conclusion】The drought resistance of transgenicwas improved by expressing soybean. Over expression ofcould enhance the drought resistance of transgenic soybean hairy root complex.can improve the drought resistance of plants mainly by affecting the expression of genes related to stress tolerance.
soybean; GmbZIP16; drought resistance; qRT-PCR
2018-04-01;
2018-05-14
国家转基因生物新品种培育科技重大专项(2018ZX08009100,2016ZX08002-002)、西北农林科技大学2017年唐仲英育种基金
赵婉莹,E-mail:zhaowanying199406@126.com。通信作者闵东红,E-mail:mdh2493@126.com。通信作者徐兆师,E-mail:xuzhaoshi@caas.cn
10.3864/j.issn.0578-1752.2018.15.001

