基因组预测中先验分布的两种超参数设定策略比较
董林松,方 铭,王志勇
(集美大学农业部东海海水健康养殖重点实验室,福建 厦门 361021)
基因组选择(GS)是一种通过全基因组范围内的标记进行基因组估计育种值(GEBV)并加以选择的方法[1].传统的最佳线性无偏预测(BLUP)方法可以结合系谱和表型信息来预测个体的育种值[2];相比简单的表型选择方法,BLUP方法可以提高育种值估计的准确性.然而,系谱记录无法准确捕获等位基因在个体间传递的具体信息.换言之,传统方法无法很好地估计出个体育种值中所包含的孟德尔抽样误差项[3].与传统BLUP方法相比,基因组选择方法捕获了个体的标记基因型信息,可以更加准确地了解到个体之间的亲缘关系,从而提高GEBV的准确性[4-6].基于其较高的GEBV准确性,并且可以进行早期选种[7],目前基因组选择已经成为奶牛育种的标准方法,并在其他动植物育种中被广泛应用[8].但由于基因组的标记数量往往多于参考群个体数量,所以基因组预测在算法上存在挑战,即所谓的“大P小N”的问题[9].
为解决该问题,近年来出现大量的关于基因组预测的方法,其中较为典型的是Meuwissen等[1]在2001年提出的岭回归BLUP(RRBLUP)、BayesA和BayesB方法,在此基础上又有学者提出基因组BLUP(GBLUP)[10]、GBLUP-最小绝对值收缩与选择操作(LASSO)[11]、性状特异的关系矩阵BLUP(TABLUP)[12]、BayesCπ[13]、BayesLASSO[14-15]、Bayes-随机搜索变量选择(SSVS)[16]、快速BayesB(FBayesB)[17]、期望最大化BayesB(emBayesB)[18]和基于帕雷托法则的混合正态分布(MixP)[19-20]等方法.这些方法都是在一些先验假设下进行推导的,RRBLUP和GBLUP假定基因组内所有标记效应值的方差相等,而Bayes方法则假定标记的方差(或效应)服从某一分布,如逆卡方分布[1,13]或拉普拉斯(双指数)分布[15,17]等.但不论何种分布,都需要在计算之前对超参数进行设置.朱波等[21]已经证明当BayesA的先验分布超参数取不同数值时,GEBV的预测准确性会产生变化,说明设定合理的超参数值是非常重要的.同时,由于部分研究者对先验超参数的设定产生一些误解,因此有必要对基因组选择算法中先验分布的超参数设定策略加以探讨.
本研究提出基因组选择算法的2种设定先验超参数的策略,并利用QTLMAS2012的模拟数据,采用7种基因组选择算法来探讨和验证超参数设定策略的可行性.
1 计算与实验方法
1.1 计算模型
基因组预测模型分为两个水平,第一个水平是对观测值y的剖分,基因组预测模型的第二个水平是对单核苷酸多态性(SNP)效应或其方差进行解释.对于表型值的剖分可以用如下混合模型表示:
y=Xb+Za+e.

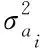
其中,π是SNP效应值方差为0的概率,当π取值为0时即是BayesA方法[1].BayesCπ的先验分布与BayesB相似,区别在于所有非零效应SNP的方差被认为是相等的,并且π值可以通过程序进行更新,而不是一个固定值[13].FBayesB是2009年由Meuwissen等[17]提出的一种基于迭代条件期望(ICE)算法的快速Bayes方法,该方法同样认为只有一部分SNP存在效应,并且这部分SNP效应值的方差相等,但SNP效应服从拉普拉斯分布:
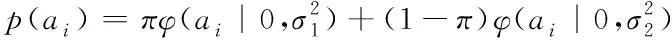
1.2 加性遗传方差与σ2的关系
这里先对单个SNP位点进行研究,然后将问题扩展到所有SNP位点.假设基因组中的某一SNP位点存在2种等位基因M和m,就有可能出现3种基因型,即MM、Mm和mm.为便于计算,这里将3种基因型转化为0,1,2,其中Mm为1,MM和mm分别为0和2.假定该位点基因型符合哈代-温伯格平衡(HWE);等位基因M的频率为pi,m的频率为qi=1-pi,则该位点各种基因型期望值E(w)和方差V(w)分别为[23]:
E(w)=2qi,
(1)
V(w)=E(w2)-E2(w)=2piqi.
(2)
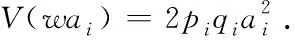
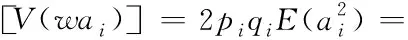
(3)
根据式(3)可以看出一个位点的期望加性遗传方差与该位点的等位基因频率和SNP效应值的方差有关.
现将该问题扩展到所有SNP位点,设所有位点总的期望加性遗传方差为VA,且每个位点效应值方差都等于σ2,则[24]
故:
(4)
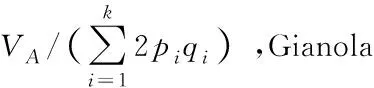
1.3 先验分布超参数的设定
RRBLUP可通过求解下列混合模型方程组来获得SNP的效应值[1]:
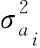
(5)
式(5)中有两个未知数,即v和s2.为获得这两个参数值,通常需对v任意设定一个参数值,例如设定v等于4.2[13]或5.0[32]等,这样s2就可以通过式(5)求出.
在BayesB[1]和BayesCπ[13]中,先验分布假设只有一部分的标记存在效应,而其余标记效应的方差为0.此时s2可以通过如下式获得[13]:
其中π为一个位点效应值为0的概率.
1.4 先验超参数的另一种设定方法
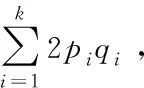
鉴于不同研究者对SNP基因型的编码习惯不同,为统一超参数设定公式,可以在计算参数前对标记基因型进行优化.优化的方法就是将基因型进行如下转换[17]:

1.5 模拟验证
本研究采用QTLMAS2012的模拟数据[34]对比2种先验分布参数设定方案的结果.该群体参考群个体数为3 000,估计群为1 020.共模拟了5条染色体,每条染色体的长度均为100 Mb.基因组共模拟产生10 000个SNP位点,剔除最小等位基因频率(MAF)小于0.05的位点之后,共保留9 042个有效的SNP标记.基因组中共模拟50个数量性状基因座(QTL),每个QTL的效应从gamma(0.42,5.4)抽样而来,其中0.42为分布的形状参数,5.4为尺度参数.本研究选取其模拟的第一个性状(产奶量)进行分析,该模拟性状的遗传力为0.35.
利用上述提到的7个预测方法(RRBLUP、BayesA、BayesB、BayesCπ、FBayesB、FMixP、MMixP)估计SNP的效应值.在BayesB,FBayesB和FMixP模型中,π值设定为0.99(假定一个QTL大概与周围两个SNP发生紧密连锁).
本研究对SNP基因型采用进行标准化和不进行标准化2种策略.在基于MCMC算法的Bayes方法中,逆卡方分布的自由度v取值为5.0,相应的尺度参数s2的计算和其他Bayes方法的超参数计算公式如表1所示.RRBLUP采用高斯-赛德尔(Gauss-Seidel)迭代法求解,FBayesB和FMixP采用ICE算法获得SNP的效应值,判断迭代收敛的标准为:(at-at-1)T(at-at-1)/((at)Tat)<10-8,其中t表示第t次迭代.BayesA、BayesB、BayesCπ和MMixP中,运行Gibbs抽样20 000次循环,并舍弃前5 000次循环,取后面15 000次的平均值作为SNP的效应值.在BayesB的每个Gibbs抽样循环中,对SNP效应值的方差通过Metropolis-Hastings算法抽样100次.估计群体的GEBV通过各个位点的SNP效应值的累加获得,估计准确性定义为GEBV与真实育种值的相关系数,并作为2种先验超参数设定结果的比较依据.所有的计算程序均使用Fortran90代码来运行.
表1 2种SNP基因型编码策略下的各种预测模型的先验超参数的计算方法
Tab. 1 Calculations of prior hyper-parameters in various prediction models in the two strategies of SNP genotypes coding

预测模型先验分布类型策略1策略2BayesAσ2ai~χ-2(v,s2)s2=(v-2)VAvks2=(v-2)VAv∑ki=12piqiBayesBσ2ai~χ-2(v,s2)s2=(v-2)VAv(1-π)ks2=(v-2)VAv(1-π)∑ki=12piqiBayesCπσ2ai~χ-2(v,s2)s2=(v-2)VAv(1-π)ks2=(v-2)VAv(1-π)∑ki=12piqiFBayesBai~Laplace(0,λ)λ=2(1-π)kVAλ=2(1-π)∑ki=12piqiVAFMixPai~N(0,σ21)或~N(0,σ22)σ21=(1-π)VAπkσ22=πVA(1-π)kσ21=(1-π)VAπ∑ki=12piqiσ22=πVA(1-π)∑ki=12piqiMMixPσ2ai~χ-2(v,s21)或~χ-2(v,s22)s21=(v-2)(1-π)VAvπks22=(v-2)πVAv(1-π)ks21=(v-2)(1-π)VAvπ∑ki=12piqis22=(v-2)πVAv(1-π)∑ki=12piqi
注:策略1为SNP基因型进行标准化,策略2为基因型不进行标准化.
2 结 果
通过各种预测模型获得的2种基因型编码策略下的预测结果如表2所示,从结果中可以看出,不同方法间的GEBV预测准确性相差较大,其中预测准确性较高的为4种基于MCMC算法的Bayes方法(其中MMixP和BayesCπ预测的准确性相对较高,而BayesB相对较低),而2种快速Bayes方法的预测结果与RRBLUP相似.在BayesCπ结果中,π值(无效应的SNP比例)在SNP基因型进行标准化和不进行标准化下的预测值非常接近,分别为98.22%和98.24%.基于该结果,本试验中重新将BayesB中的π值设定为98.2%,再次运行BayesB程序,在SNP基因型标准化和非标准化下获得的GEBV准确性结果分别为0.783和0.780,较之前有所提高.Usai等[34]在使用相同的模拟数据下,通过RRBLUP、BayesA、BayesB和BayesC方法获得的GEBV预测准确性分别为0.74,0.79,0.79和0.79,这个结果与本试验的结果比较吻合,出现稍许差异的原因可能是因为本研究剔除了MAF小于0.05的SNP位点,导致参考群中可用于分析的SNP数量减少.但是在任意一种计算方法下,2种参数设定策略获得的GEBV预测准确性差异都非常小,几乎可以忽略,说明这2种参数设定策略都是可行的.
表2 两种SNP基因型编码策略下的GEBV预测准确性
Tab. 2 GEBV predictive accuracies in the two strategies of SNP genotypes coding
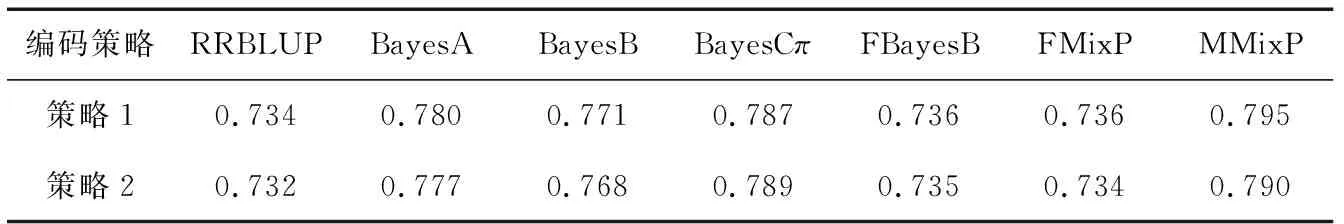
编码策略RRBLUPBayesABayesBBayesCπFBayesBFMixPMMixP策略10.7340.7800.7710.7870.7360.7360.795策略20.7320.7770.7680.7890.7350.7340.790
注:策略1为SNP基因型进行标准化,策略2为基因型不进行标准化.
3 讨 论
本研究使用了7种基因组预测方法来观察GEBV的预测准确性,总体上,基于MCMC算法的Bayes方法的预测结果要优于基于ICE算法的Bayes方法和RRBLUP方法.在4种基于MCMC算法的Bayes方法中,BayesCπ和MMixP的结果较为相似并且最好,原因可能是因为这两种方法在计算过程中可以更新π值,从而获得一个最优的解.从BayesCπ方法预测的π值也可以看出,无效应的SNP比例接近98.2%,而本研究在BayesB中将该比例设定为99%,这可能是导致BayesB准确性略低于BayesCπ和MMixP的原因.重新设定π值之后,再次运行BayesB就提高了GEBV预测准确性,验证了上述猜测.快速Bayes方法的预测结果低于基于MCMC方法的预测结果,这与Meuwissen等[17]的研究结果吻合,原因可能是因为ICE本身是一种近似算法[17],此外π在计算时取值不够合理,因此更加降低了这些方法的预测效果.虽然BayesA和RRBLUP都认为所有位点存在效应,但BayesA认为不同SNP位点效应值的方差是有差异的,因此能够将无效SNP位点的效应值压缩至接近0,而RRBLUP受限于其先验分布的问题,压缩能力明显不如BayesA.换言之,BayesA的先验假设更接近模拟的QTL的分布情况,因此预测准确性高于RRBLUP.
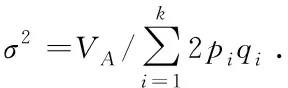
4 结 论
本研究对比了基因组选择算法中先验分布的两种超参数设定策略的结果,阐述了标记效应方差与位点加性遗传方差的区别,并给出了相应的计算公式.从模拟结果来看,在计算SNP效应之前,基因型是否进行标准化对最终的结果影响很小.但需要注意的是,如果SNP基因型不进行标准化,在计算标记效应的期望方差时,分母是所有SNP位点的基因型方差之和;如果进行标准化,该项变为k,即总的标记位点数.由于基因型标准化可以突出效应较大的SNP位点,故建议先将SNP基因型标准化,然后再设定先验分布的超参数值.

