棘孢木霉葡聚糖酶基因Glu1的克隆及原核表达
刁桂萍,杨 帅,遇文婧
(1.东北林业大学林学院,黑龙江 哈尔滨150040; 2.黑龙江省林科院森林保护研究所,黑龙江 哈尔滨 150040)
植物致病真菌的细胞壁主要由几丁质、β-1,3-葡聚糖、α-1,3-葡聚糖和α-1,4-葡聚糖组成[1-3]。葡聚糖酶具有分解植物病原真菌细胞壁的能力,因此能够起到抑制植物真菌的作用[4-6]。Cuddihy等[7]从番茄(Lycopersiconesculentum)的代谢产物中分离纯化到一种葡聚糖酶,体外抑菌试验表明,其能抑制番茄病原真菌茄链格孢菌(Altemariasolani)的菌丝生长。而在豌豆(Pisumsativum)种子中发现分子量为26 kDa的葡聚糖酶,能抑制菜豆炭疽菌(Colletotrichumlindemuthianum)和香蕉炭疽菌(Gloeosporiummusarum)菌丝的生长[8]。此外,大豆疫霉菌(Phytophthorasojae)能诱导大豆葡聚糖酶活性的增强已被证实,而且大豆(Glycinemax)对大豆疫霉根腐病(P.sojae)的抗性与葡聚糖酶的活性呈正相关关系[9]。
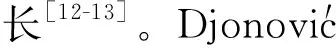
1 材料与方法
1.1 试验材料
供试菌株棘孢木霉(T.asperellum)ACCC30536,由中国农业微生物保藏管理中心(ACCC)提供。
1.2 葡聚糖酶基因Glu1的克隆及序列分析
利用Trizol法提取棘孢木霉ACCC30536的总RNA,并反转录获得cDNA。根据引物g1:5′-ATGACGATCCGCATCCTCGTC-3′和g2:3′-GGTGGAGGATGTTTCTATATC-5′进行cDNA的PCR扩增,扩增产物转化到大肠杆菌Top10中,经过PCR检测后进行测序[15]。对获得的cDNA序列进行生物信息学分析:NCBI Conserved Domains search(http:∥www.ncbi.nlm.nih.gov/)进行保守区和蛋白家族预测,并寻找相似序列;MEGA5.1软件进行多序列比对和进化树的建立;ExPASy ProtParam(http:∥www.expasy.org/)分析分子量、等电点和疏水性;SignalP 4.0(http:∥www.cbs.dtu.dk/services/SignalP/)预测信号肽;ProtComp 9.0进行亚细胞定位分析;Swissmodel(https:∥www.swissmodel.expasy.org/)预测蛋白的三维结构,模式选自动建模[16-17]。
1.3 葡聚糖酶基因Glu1在不同培养条件下表达分析
将棘孢木霉ACCC30536菌丝体分别培养在MM[0.5%葡萄糖,15 g·L-1NaH2PO4,5 g·L-1(NH4)2SO4,600 mg·L-1CaCl22H2O,600 mg·L-1MgSO47H2O,5 mg·L-1FeSO4,2 mg·L-1CoCl2,1.6 mg·L-1MnSO4,1.4 mg·L-1ZnSO4]、C饥饿(MM-葡萄糖),N饥饿(MM-(NH4)2SO4),羧甲基纤维素钠(M-葡萄糖+羧甲基纤维素钠),山新杨(Populusdavidiana×P.albavar.pyramidlis)根粉(MM+1%山新杨根磨粉),山新杨茎粉(MM+1%山新杨茎磨粉),山新杨叶粉(MM+1%山新杨叶磨粉),病原菌细胞壁(MM+1%杨树烂皮病细胞壁)和病原菌发酵液(MM+5%杨树烂皮病发酵液)[17]共9种诱导培养基中,分别在诱导0、12、24、48和72 h收取菌丝,提取总RNA,反转录后进行qRT-PCR,每组重复3次。qRT-PCR反应体系:10 μL的2×SYBR Green Real-time PCR Master mix,0.5 μL引物,2 μL的cDNA,ddH2O定量至总体积20 μL。qRT-PCR反应条件为:95 ℃预变性30 s;95 ℃变性5 s,59 ℃退火15 s,72 ℃延伸10 s;82 ℃,读板1 s;45个循环;55~95 ℃,每间隔0.5 ℃读板1 s。并采用2-ΔΔCT方法计算基因表达量[17]。Glu1基因及内参基因(Actin,α-tubulin和β-tubulin)引物如表1所列。
1.4 葡聚糖酶基因Glu1原核表达载体构建
根据Glu1基因序列和原核表达载体序列设计带有酶切位点的引物,G1e:5′-ATCGGGATCCATGACGATCCGCATCCTCGTC-3′(下划线为BamHI酶切位点)G2e:5′-CGATGCGGCCGCGGTGGAG-GATGTTTCTATATC-3′(下划线为NotI酶切位点)。以棘孢木霉ACCC30536的cDNA为模板,G1e和G2e为引物进行PCR扩增。用BamHI和NotI限制性内切酶对PCR的回收产物及pGEX-4T-2质粒分别进行双酶切,用T4 DNA Ligase进行连接后,转入大肠杆菌TOP10感受态中,在含终浓度为50 mg·L-1羧苄青霉素的LB固体培养基中筛选,并对获得的转化子进行PCR和双酶切检测[18]。

表1 qRT-PCR引物Table 1 qRT-PCR primers
1.5 葡聚糖酶基因Glu1的原核表达
将重组质粒转入大肠杆菌表达菌株BL21中。将获得的阳性转化子接种于3 mL含50 mg·L-1羧苄青霉素的LB液体培养基中,37 ℃过夜培养12 h。次日用新鲜培养基稀释过夜的菌液,待菌液OD600为0.4~0.6时,加入终浓度为1.0 mmol·L-1IPTG,于30 ℃、200 r·min-1培养6 h,进行重组蛋白诱导表达,以IPTG诱导的含pGEX-4T-2空载的大肠肝菌BL21及未诱导的含重组质粒的大肠杆菌BL21作为对照,并通过电转化进行蛋白纯化[15]。
1.6 重组葡聚糖酶的酶活特性分析
采用还原糖测定法[19],测定不同pH(3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5和7.0)及不同反应温度(30、35、40、45、50、55、60、65和70 ℃)对重组葡聚糖酶酶活的影响。然后在最适温度和pH条件下测定不同诱导时间(1、2、3、4、5和6 h)对重组葡聚糖酶活性的影响。以沸水浴中失活的粗酶液作对照,每组重复3次。
2 结果
2.1 棘孢木霉葡聚糖酶基因Glu1的克隆与序列分析
棘孢木霉Glu1基因的cDNA序列全长为984 bp,编码327个氨基酸(图1)。ProtParam软件分析表明,该葡聚糖酶的分子由4 956个原子构成,分子式为C1592H2456N406Q494S8,相对分子量为35 443.92,等电点为4.76,含有20种氨基酸(表2)。通过BlastP预测该蛋白属于Glyco-hydro-12家族(图2A)。葡聚糖酶Glu1具有多个磷酸化位点,其中有18个苏氨酸磷酸化作用位点(Thr:18)、13个丝氨酸磷酸化作用位点(Ser:13)和5个酪氨酸磷酸化作用位点(Tyr:5),说明该葡聚糖酶磷酸化以苏氨酸和丝氨酸磷酸化为主,兼有酪氨酸磷酸化(图2B)。在葡聚糖酶Glu1第22和23个氨基酸中间有一个信号肽酶剪切位点LGI∥LF存在,第35和36个氨基酸中间有一个信号肽酶剪切位点TGO∥PP存在(图2C)。棘孢木霉ACCC30536葡聚糖酶的三级结构与模板β-1,4-葡聚糖酶(4h7m.1)的结构有36%的相似性(大于30%可以期望得到较好的预测结果),由此推测,棘孢木霉ACCC30536葡聚糖酶属于β-1,4-葡聚糖酶(图2D)。
2.2 棘孢木霉葡聚糖酶Glu1多序列比对及进化树分析
通过Blastp获得Glu1相似的氨基酸序列,从中选取相似性较高的13个序列进行分析(图3),其中深绿木霉IMI 206040(XP_013940397.1,相似度91%)、贵州木霉(OPB37643.1,相似度82%)、绿色木霉Gv29-8(XP_013954335.1,相似度81%)和里氏木霉QM6a(XP_006964596.1,相似度79%)的Glyco-hydro-12家族葡聚糖酶相似度最高。将Glu1和13个相似序列进行进化树分析(图4),结果表明Glu1和13个相似序列被分成5组,其中Glu1和所有木霉属的Glyco-hydro-12家族葡聚糖酶分成一组(Group 1),其中Glu1和深绿木霉IMI 206040(XP_013940397.1)的Glyco-hydro-12家族葡聚糖酶同源性最高,亲缘关系最近,而其他属的Glyco-hydro-12家族葡聚糖酶被分为4组(Group 2、Group 3、Group 4和Group 5)。
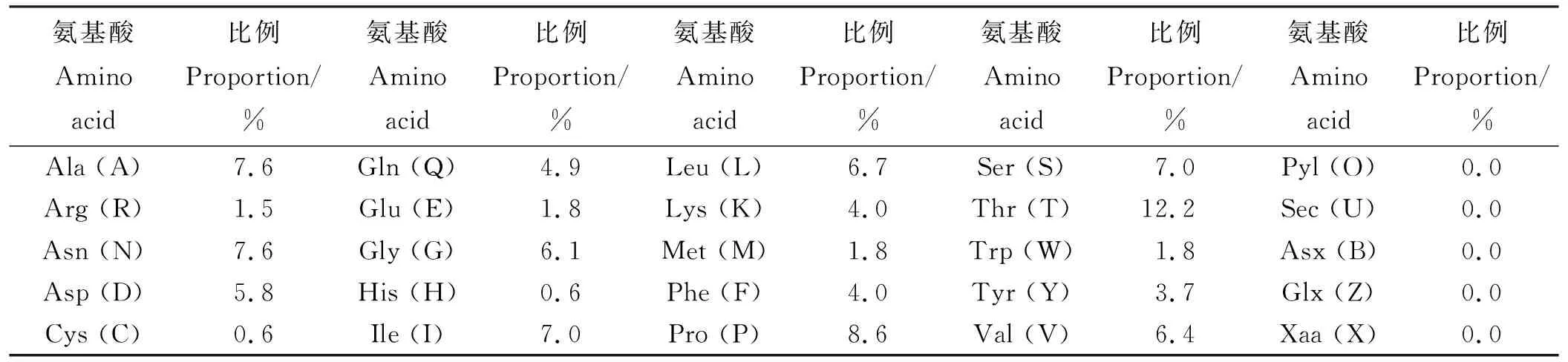
表2 Glu1基因的氨基酸组成Table 2 Amino acid composition of Glu1

1ATGACGATCCGCATCCTCGTCAACGCTGCGCTGCTGGCCATCCCCATCGCCGTGACGCTGMTIRILVNAALLAIPIAVTL61GGCATCTTGTTCGGCCTCCAATCGCACCGAGATGCCACGGGCCAGCCGCCTTTGTTTGCGGILFGLQSHRDATGQPPLFA121CCTCAACCGCCGCCGATTCCAACTCCTGCACCGCCGAAACCCCCAAAGAATGGCGTCACCPQPPPIPTPAPPKPPKNGVT181AAGACGAGATATTGCCAAAAGTCGTATGGAATTACACCAGATACCACTGGGCAGCAGTATKTRYCQKSYGITPDTTGQQY241ATATTAAATCCTAATCAATGGAACTGGAACGTCGGCGATCCTGGACGCCTGTGTATGAATILNPNQWNWNVGDPGRLCMN301GTCACCACGTTTAACAACGGCACATATGCCACAAGCACTACCGCTCCTCAGTTTATGGTTVTTFNNGTYATSTTAPQFMV361TCATGGCAATATCCCCGTGGCCCAGAGGACAACCCTGTACACGCTTTCCCCAACATCCAGSWQYPRGPEDNPVHAFPNIQ421ATTGATACCGACGTCATTCCCGCCACTGTGAACAGCATTTCCAAAGTCGACATCGACATCIDTDVIPATVNSISKVDIDI481GAATGGTCCTACGGCGTCGGCAATGAGACCACAACGTCTTCTACCCTCGCGGATCTTACTEWSYGVGNETTTSSTLADLT541GCCGACAATCTCAACACCAACGTTGCTGTCGATATGTTTATTGACAGCGATAAGACGGCCADNLNTNVAVDMFIDSDKTA601GCTCAGAACCCTGCCAAGGCCAAGTTCGAACTCATGGTTTGGTTTGCGGCCTATGGTAGTAQNPAKAKFELMVWFAAYGS661TCCACTCAGCCCATCGGTTTTAGCAACGGTGCTGTGTCCACCCAAACGCTTAATGGCAACSTQPIGFSNGAVSTQTLNGN721ACATTCAATCTTTATGCTGGCATTAACACAGCGACCAAGCAAAACGTCATGACCTGGTACTFNLYAGINTATKQNVMTWY781ACCGACACGCCTATACAAAAGTTCAATGGAGACATATCTCCACTCATCAACAGCGTGTTCTDTPIQKFNGDISPLINSVF841AAGCTCACCACTGTTACCGATATTCCCAAGTCTAGCGACTACTTGGGATACCTGGCTCTGKLTTVTDIPKSSDYLGYLAL901GGCTCCGAGGCCTTTTCCGTTGACAAGACCGTCACGCTTTACGTACCCCAACTCTCGATAGSEAFSVDKTVTLYVPQLSI961GATATAGAAACATCCTCCACCTAG*DIETSST
图1Glu1基因的cDNA序列及推导的氨基酸序列
Fig.1cDNAsequenceoftheGlu1geneandpredictedaminoacidsequenceoftheGlu1protein
粗体表示cDNA序列;斜体表示cDNA编码的氨基酸序列。
Bold text indicates the cDNA sequence; italic text indicates the amino acid sequence encoded by the cDNA.
图2 葡聚糖酶Glu1的保守区、磷酸化位点、信号肽及三级结构预测图Fig. 2 Image of the conserved region, phosphorylation site, signal peptide site, and tertiary structure of the predicted Glu1 sequence
A:Glu1的保守区;B:Glu1的磷酸化位点;C:Glu1的信号肽;D:Glu1的三级结构。
A: conserved region of Glu1; B: phosphorylation site of Glu1; C: signal peptide site of Glu1; D: tertiary structure of Glu1.
2.3 棘孢木霉葡聚糖酶基因Glu1在9种诱导条件下的表达分析
运用qRT-PCR技术检测9种诱导条件下棘孢木霉Glu1基因的调控表达水平(图5)。在MM(图5A)、N饥饿(图5C)、1%山新杨根粉(图5E)、1%山新杨茎粉(图5F)、1%山新杨叶粉(图5G)、1%杨树烂皮病菌细胞壁(图5H)和5%杨树烂皮病菌发酵液(图5I)培养条件下,Glu1基因均呈上调表达,分别在12、24、24、12、12、24和48 h达到峰值,分别为未诱导时的12.13(23.6)、13.93(23.8)、45.25(25.5)、10.56(23.4)、8.57(23.1)、6.06(22.6)和8.57(23.1)倍。但是,在C饥饿(图5B)和羧甲基纤维素钠(图5D)培养条件下,Glu1基因呈下调表达,分别在24和12 h下调至最低点,分别低于未诱导时的6.96(22.8)和21.11(24.4)倍。说明Glu1基因在棘孢木霉响应环境压力中起到重要作用,并且能够参与棘孢木霉对杨树(Populus)或杨树病原菌的识别。
2.4 棘孢木霉葡聚糖酶Glu1的SDS-PAGE分析
SDS-PAGE检测结果(图6)显示,重组转化子经IPTG诱导后,泳道1-5清晰可见蛋白条带,大小与61.44 kDa的预测蛋白一致,而对照泳道6在相应位置没有条带出现,证明Glu1基因成功整合到大肠杆菌BL21中;而且诱导6 h的重组蛋白明显多于诱导2和4 h的重组蛋白,说明IPTG诱导时间越长,转化子分泌的蛋白越多。对变性复性后的蛋白进行电纯化后,获得纯化后的目的蛋白rGlu1。

图3 葡聚糖酶Glu1及其相似序列的多序列比对Fig. 3 Multiple sequence alignment of Glu1 and the similar sequences
*表示同源identity;XP_013940397.1: 深绿木霉T.atrovirideIMI 206040; OPB37643.1:贵州木霉T.guizhouense; XP_013954335.1:绿色木霉T.virensGv29-8; XP_006964596.1:里氏木霉T.reeseiQM6a; XP_006964596.1:里氏木霉T.reeseiQM6a; EQL01618.1:冬虫夏草OphiocordycepssinensisCO18; KIM99495.1:大孢树粉孢菌OidiodendronmaiusZn; KJK64291.1:寄生曲霉AspergillusparasiticusSU-1; XP_007817188.1:罗伯茨绿僵菌MetarhiziumrobertsiiARSEF 23; XP_014543006.1:罗伯茨绿僵菌MetarhiziumbrunneumARSEF; XP_014572972.1:大孢绿僵菌MetarhiziummajusARSEF 297; KDB11778.1:稻曲病菌Ustilaginoideavirens; KXG45790.1:灰黄霉Penicilliumgriseofulvum。
2.5 重组葡聚糖酶rGlu1酶活特性
首先测定不同浓度葡萄糖的吸光度绘制标准曲线,获得线性回归方程y=0.000 8x-0.003 9,R2=0.973 2。根据回归方程,分析在不同pH、不同反应温度和不同反应时间下rGlu1的酶活特性。在不同pH条件下,rGlu1活性呈先增加后降低的趋势,当pH为4.5时,rGlu1活性最大,为1.81 U·mL-1(图7A);在不同反应温度条件下,rGlu1活性也呈先增加后降低的趋势,当温度达到45 ℃时,rGlu1活性最大,为1.86 U·mL-1(图7B);在不同反应时间条件下,rGlu1活性呈递增的趋势,在5 h达到峰值,为1.80 U·mL-1,之后趋于平缓(图7C)。这些结果表明,重组葡聚糖酶最适pH为4.5,偏酸性,最适温度为45 ℃,且随着时间的增加,重组酶活性逐渐增加,并于5 h后趋于平稳。
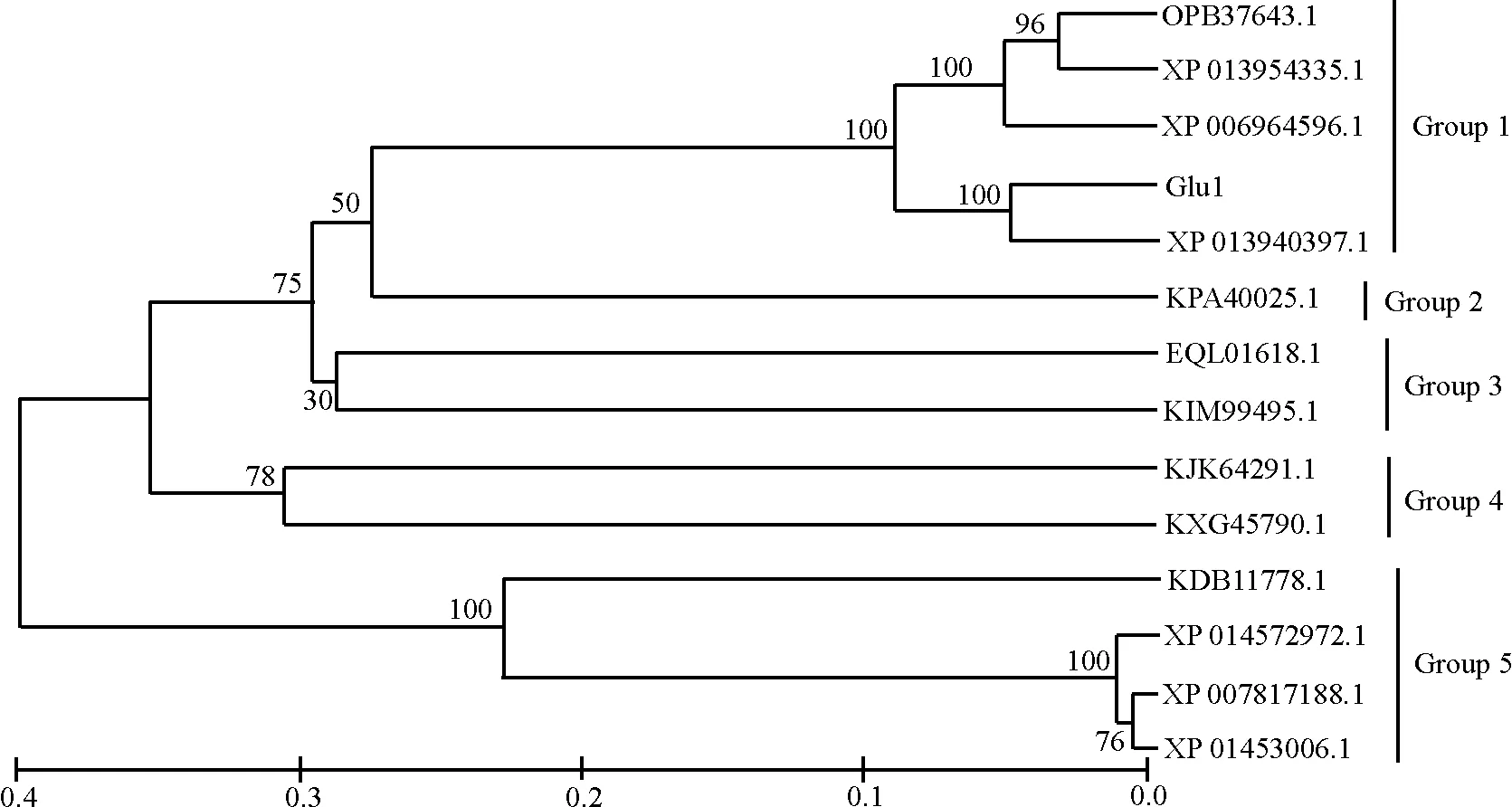
图4 葡聚糖酶Glu1及其相似序列的进化树Fig. 4 Phylogenetic tree of Glu1 and the similar sequences
所有分支显示了可靠的置信度(超过50%的引导值);线段上的数字表示碱基替换率。
All branches show significant support (>50% bootstrap values); the numbers on the line segments indicate the base replacement rates.
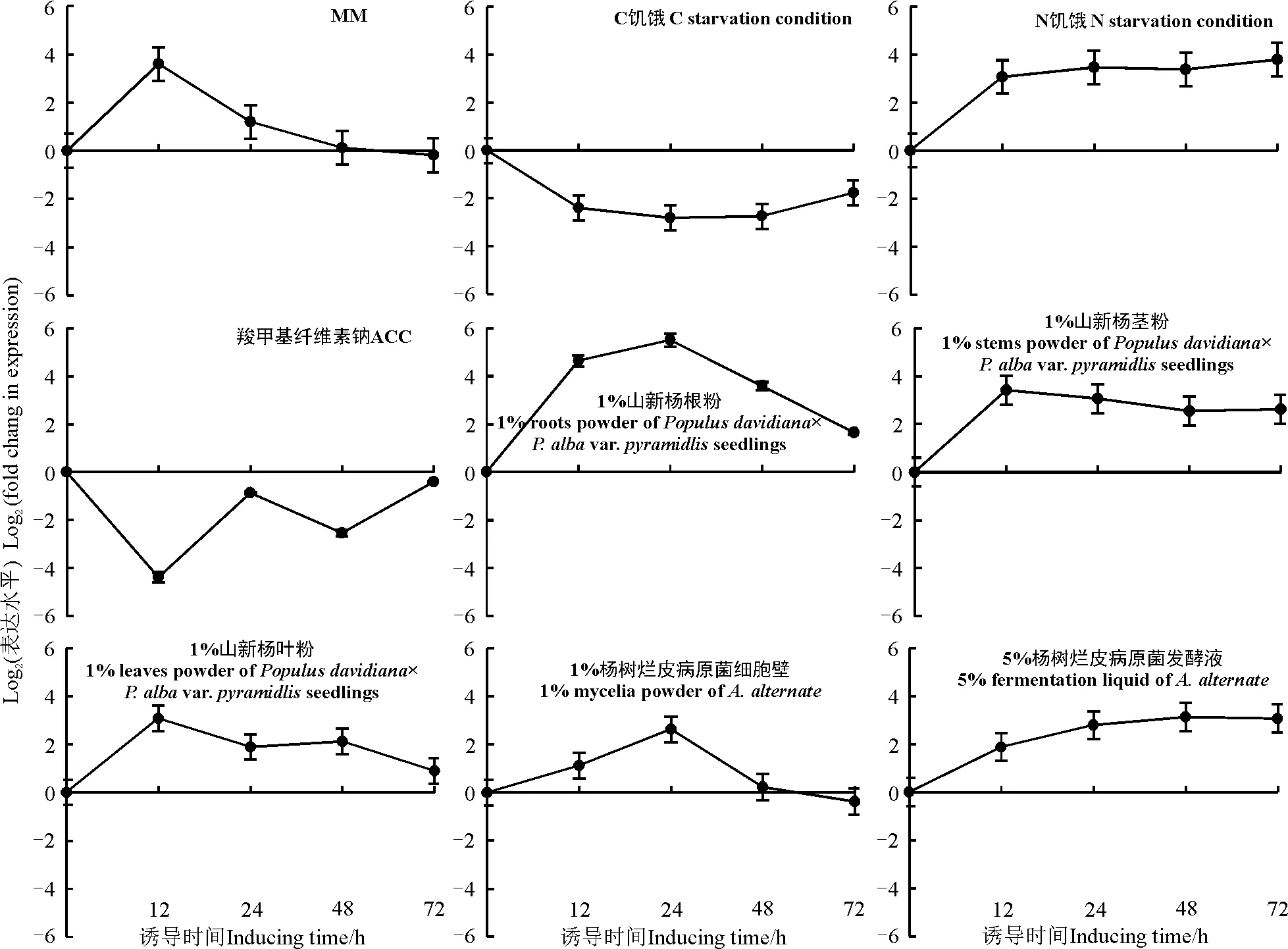
图5 9种诱导条件下Glu1基因差异表达Fig. 5 Expression level of the gene Glu1 under nine different induction conditions
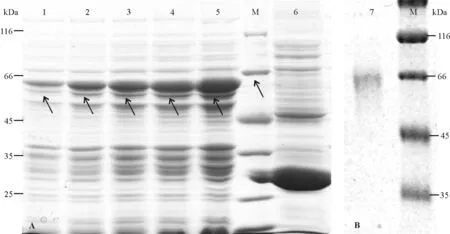
图6 重组蛋白的SDS-PAGE检测Fig. 6 SDS-PAGE detection of recombinant protein
M:Protein marker;1-5:IPTG分别诱导2、3、4、5和6 h,重组转化子BL21-Glu1蛋白SDS-PAGE电泳图;6:IPTG诱导6 h,对照转化子BL21-BL21-pGEXSDS-PAGE电泳图;7:纯化后的重组蛋白rGlu1 SDS-PAGE电泳图。
M: Protein Marker; 1-5: SDS-PAGE analysis of the transformant BL21-Glu1 induced for 2, 3, 4, 5, and 6 h, respectively; 6: SDS-PAGE analysis of the control transformant BL21-pGEX induced for 6 h; 7: SDS-PAGE analysis of purified recombinant rGlu1.
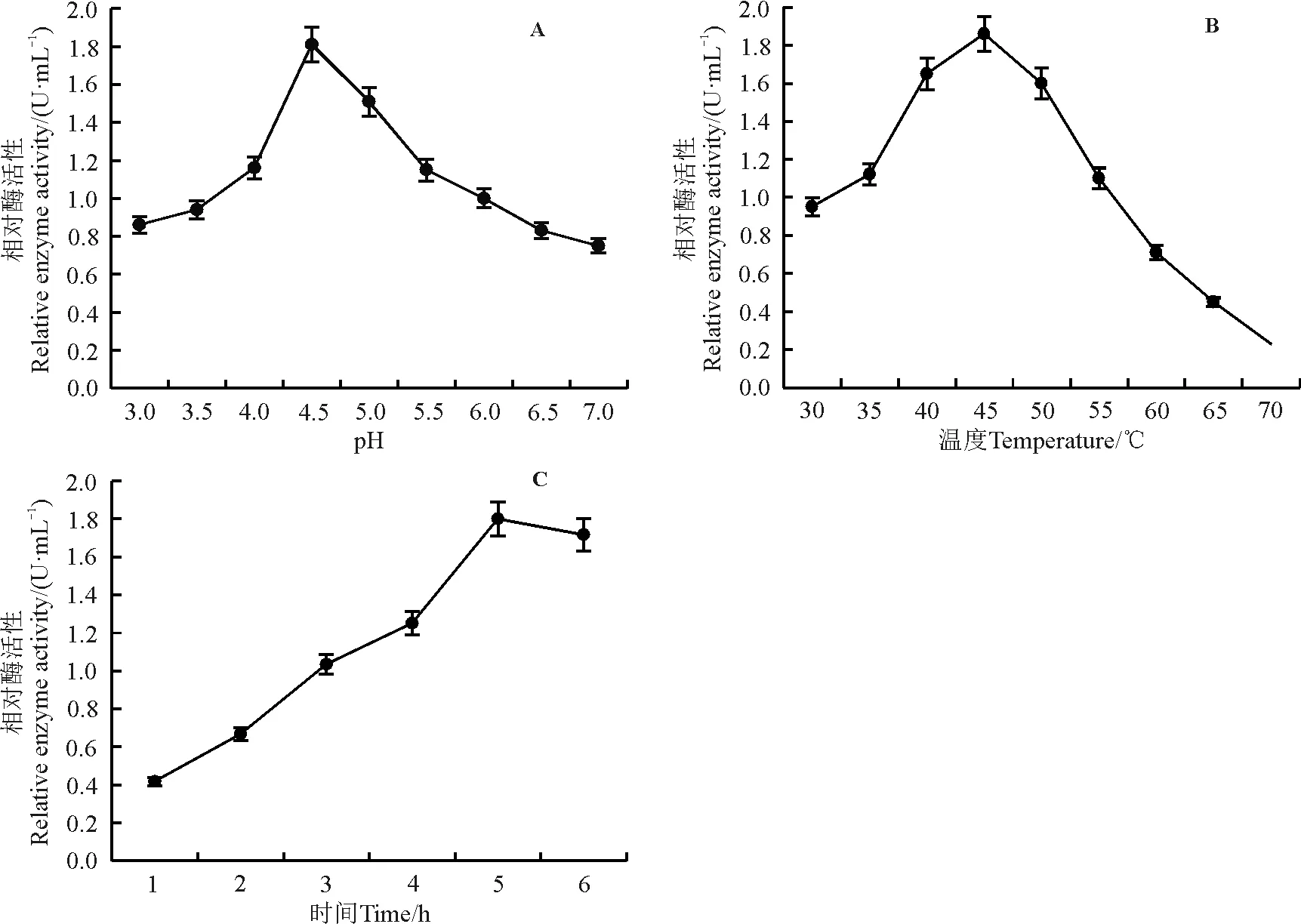
图7 重组葡聚糖酶rGlu1酶活特性Fig. 7 Enzyme activity properties of recombinant rGlu1
3 讨论与结论
木霉菌种类繁多,其分泌的代谢产物也相当丰富,其中抗真菌次生代谢物和细胞壁水解酶协同作用可导致植物病原真菌死亡[20]。细胞壁水解酶中的葡聚糖能够水解植物病原真菌细胞壁最外层覆盖的葡聚糖层,使细胞壁骨架和细胞受损,而且抑菌试验也表明葡聚糖酶对多种真菌菌丝的生长有抑制作用[21]。本研究从棘孢木霉ACCCC30536菌株基因组中克隆到一个葡聚糖酶基因Glu1,cDNA序列全长984 bp,由327个氨基酸组成,通过生物信息学分析推测此酶属于β-1,4-葡聚糖酶。一些研究表明,β-1,4-葡聚糖酶在纤维素的水解过程中起着非常关键的作用,它能够作用于纤维素链内的β-1,4糖苷键,将长链纤维素分子水解成更小分子量的葡萄糖聚合物[20,22]。插入β-1,4-葡聚糖酶的绿色木霉对小麦纹枯病菌(Rhizoctoniacerealis)的抑制效果较原始木霉菌株明显提高,抑制率提高了27%[21]。因此,本研究中的Glu1蛋白可能具有与β-1,4-葡聚糖酶一样的生防特性。
在正常环境下葡聚糖酶在生物体中的含量较少,活性很低。当生物体受到外界刺激时,葡聚糖酶可以被刺激物所诱导和积累[9]。研究表明,霍尔斯单轴霉(Plasmoparahalstedii)侵染后的向日葵(Helianthusannuus),其葡聚糖酶基因表达量明显升高[23]。在本研究中,当棘孢木霉ACCC30536与山新杨及杨树病原菌互作后,Glu1基因的表达也明显上升,表明Glu1蛋白参与了生防菌棘孢木霉与杨树及杨树病原菌的识别,从而使杨树(Populus)可能通过调节自身防御基因表达和生理变化来适应环境或抵御病原真菌。在此基础上构建了Glu1基因的原核表达载体并获得重组葡聚糖酶rGlu1。酶活特性分析表明,该重组rGlu1活性的最适pH为4.5,偏酸性,最适温度为45 °C,与玉蜀黍赤霉菌(Gibberellazeae)的β-1,4-葡聚糖酶重组蛋白酶酶活特性报道结果一致[23]。此外,重组酶酶活受时间影响较大,随着时间的增加,酶活也逐渐增加,并于5 h后趋于平稳。 以上这些研究结果将为进一步探究葡聚糖酶的生防功能提供理论基础,也为进一步开发和应用葡聚糖酶类生物农药奠定良好的物质基础。

