基于全基因组De novo测序的异常汉逊酵母菌株792基因组重复序列的分布特征研究
王 婷, 张寒玉, 蔡长龙, 唐 朝, 毛培宏,,钱卫东*, 李永东
(1.陕西科技大学 食品与生物工程学院, 陕西 西安 710021; 2.新疆大学 离子束生物技术中心, 新疆 乌鲁木齐 830046; 3.西安工业大学 离子束生物工程与生物多样性研究中心, 陕西 西安 710032; 4.宁波市疾病预防控制中心, 浙江 宁波 315010)
0 引言
酵母菌根据其合成乙醇能力的差异分为酿酒酵母和非酿酒酵母.大量研究表明一些非酿酒酵母对酒的品质发挥积极作用,特别是产香气型酵母,其赋予果酒浓郁的发酵香味,对果酒中醇类、酯类物质的形成扮演着重要的角色.因此,近年来非酿酒酵母的酿酒作用、生物多样性、分离鉴定及其潜在应用价值研究已成为国内外的研究热点[1].前期本课题组从陕西洛川苹果表面分离一株产香气能力强的异常汉逊酵母(Hansenulaanomala),命名为Hansenulaanomala792.
异常汉逊酵母作为一种重要的非酿酒酵母,具有高产乙酸乙酯的能力,且在较高温度的下具有较强的发酵力和酯化力.同时还具有一定的产酒精能力,并可以降低乙酸含量,提高丙三醇含量,提高有益香气成分含量[1-5].目前,一些研究主要关注非酿酒酵母异常汉逊酵母的产香气代谢能力研究,对其基因组的遗传背景的研究报道相对较少.
重复序列是真核生物基因组中重要的组成部分,按其在基因组中的分布方式,分为串联重复序列(Tandem Repeat Sequences)和散在重复序列(Interspersed Repeat Sequences)[6].串联重复序列又可根据其重复单元长度划分为卫星DNA (Satellite DNA)[7]、小卫星DNA (Minisatellite DNA)[8]和微卫星DNA (Microsatellite DNA)[9].其中微卫星DNA又称为短串联重复序列(Short Tandom Repeat,STR)或简单重复序列(Simple Sequence Repeats,SSRs),随机分布于生物体整个基因组中.微卫星标记作为理想的分子遗传标记,被广泛地用于目的基因筛选、基因诊断多样性分析及遗传连锁图谱构建等工作中.而散在重复序列(又称转座子元件,Transposable Element,TE)分为RNA介导的转座元件(又称 RNA转座子)和DNA介导的转座元件(又称DNA 转座子),不仅可以影响基因组的大小,还能直接或间接促成基因组重排,并可影响基因表达水平、改写基因调控网络[10].
本研究在异常汉逊酵母菌株792(Hansenulaanomala792)全基因组denovo测序的基础上,利用生物信息学方法分析其基因组中各种重复序列的类型及分布特点,以期深入了解汉逊酵母菌株基因组结构中重复序列的特征,为基于重复序列定向进化的分子育种及开发SSR分子标记提供理论依据.
1 实验部分
1.1 菌株及其全基因组De nove测序
菌株分离自陕西洛川苹果表面,经常规培养,分离纯化,收集菌体,利用18S rDNA、28S rDNA和ITS(Internal Transcribed Spacer)的分子生物学方法鉴定为异常汉逊酵母,命名为异常汉逊酵母792,现保存于陕西科技大学食品与生物工程学院微生物制造研究室.异常汉逊酵母792菌株由北京诺禾致源生物信息科技有限公司微生物部制备基因组DNA,并应用PacBio单分子测序技术对其进行全基因组Denove测序,所获得的全基因组DNA序列,作为本研究的基本数据.
1.2 基因组中重复序列的获取
应用TRF(Tandem Repeat Finder)方法 (http://tandem.bu.edu/trf/trf404.linux64.download.html)获取异常汉逊酵母菌株792全基因组DNA序列中的串联重复序列,最大的重复单元bp数设置为2 000 bp.
对TRF获取的结果进行细分,设置微卫星DNA序列重复单位为2~6 bp,小卫星DNA序列重复单位为10~60 bp.
使用RepeatMasker 方法(http://www.repeatmasker.org/RMDownload.html)获取异常汉逊酵母菌株792全基因组DNA序列中的散在重复序列.
2 结果与讨论
2.1 微卫星DNA序列在基因组中的分布特征
应用TRF方法在异常汉逊酵母菌株792基因组中发现了175个SSR,总长分别为8 163 bp,占基因组DNA序列总长度的0.059%,平均每78.63 Kb就能检测到一个SSR.
SSR在三核苷酸(Tri-)模体中的数目最多,为94条,占重复序列总数的53.71%;其次是六核苷酸(Hexa-)模体,为47条,占26.86%;五核苷酸(Penta-)和四核苷酸(Tetra-)模体数目相对较少,分别13~16条之间,占比约为7.43%~9.14%;二核苷酸(Di-)模体的重复序列最少,仅有3条,占1.71%.具体如图1所示.

图1 异常汉逊酵母菌株792基因组中不同模体类型的SSR分布
碱基类型的重复基序分析结果如表1所示.由表1可知,在4种两碱基类型重复中,仅有AT重复基序.
三碱基类型重复基序有10种,其中数量较多的碱基类型依次是AAC(49条,52.13%)、ACT(15条,15.96%)、AAG(11条,11.70%).累积长度最长的依然是上述三个类型的重复序列:AAC(2011 bp)、ACT(744 bp)、AAG(545 bp).
四碱基类型重复序列中含有AAAT、AACT、ATTA、GAAT和GTTG重复类型,且前两种类型数量较多,占四碱基重复序列数目的73.33%,长度较长,约占四核苷酸重复序列累积长度的74.05%.五碱基类型重复序列共有16条,其中AAAAC、TATAC和TGAAT重复单元的序列各有2条,共占五核苷酸重复的37.5%,其余各类型基序重复序列均只有1条.六碱基类型重复序列共46条,其每种重复单元基序数目均为1~2个.

表1 异常汉逊酵母菌株792基因组微卫星DNA的重复基序分布
续表1

重复类型重复序列数目占SSR总数的百分比/%累积长度/bp占SSR总长度百分比/%拷贝数范围平 均拷贝数GATGAC10.57410.506.86.8GATGGT10.57510.628.58.5GGATCA10.57380.476.36.3GGTTCA10.57330.405.55.5GTGAAA10.571121.371919TATTAC10.57520.649.39.3TCATAA10.57330.405.55.5TCATCC10.57350.435.85.8TCATTT10.57290.364.84.8TCTTCA21.141201.478.3~11.29.75TCTTCC21.141301.5910~11.710.85TGAAGA21.141121.378.7~109.35TGAGGT21.14841.035.5~8.57TGATGG10.57350.435.85.8TGATGT10.57300.3755TGCTGT10.57320.395.35.3TTATGT10.57330.405.55.5TTCGTC10.57330.405.55.5TTCTGA21.14750.925.3~7.26.25TTCTTC10.571932.3632.232.2TTGCTG10.57270.334.54.5TTGTTT10.57340.425.75.7Subtotal4726.86239029.284.2~32.28.51
各种重复类型的拷贝数分析结果如表2所示.由表2可以看出,微卫星序列均在低拷贝区出现频率较高,拷贝数低于15次的微卫星序列占比75.43%;拷贝数在15~27之间的微卫星序列,占比17.14%;拷贝数在27~39之间的占比6.86%;拷贝数大于39次的微卫星序列最少,仅占0.57%.五种重复单位的平均拷贝数分别为33、15.04、12.76、7.33、8.51.从图2可以看出,拷贝数越大,微卫星序列数目越少,微卫星平均拷贝数随着重复单位长度的增加而减少.

表2 异常汉逊酵母菌株792基因组微卫星DNA不同模体的拷贝数分布
上述研究数据表明,重复序列是基因组的重要组成部分,对生物的进化、遗传和基因的表达与调控有重要作用.重复序列是考察遗传物质在进化中无数次的重组及整合的活化石,其出现说明基因组中的遗传物质在不断地进行自我复制,并进行水平交换和垂直交换,对丰富生物的遗传信息具有重要作用[11].生物体中许多关键基因是单拷贝的,重复序列的存在能保护这些重要的基因结构不受破坏,同时也是新基因产生的物质基础,是驱动生物进化的重要因素之一[12].

图2 异常汉逊酵母菌株792基因组微卫星DNA的重复单元长度与拷贝数关系
2.2 小卫星DNA序列在基因组中的分布特征
利用TRF方法在异常汉逊酵母菌株792基因组的串联重复序列中发现了1 384条小卫星DNA序列,总长分别为79 849 bp,占串联重复序列长度的33.80%,占基因组序列总长的0.58%,平均每10 Kb出现一个小卫星序列.
小卫星DNA的长度介于25 bp至958 bp之间,根据其序列长度可分为142种类型,长度为25~78 bp的序列数目占76.81%,30 bp的小卫星序列最多,有80条,长度大于82 bp的序列各含一条.长度为15 bp的重复单位序列数目最多,有211条,占小卫星序列总数的15.25%.重复单元为15 bp的序列累积长度最长,高达8 328 bp,占小卫星序列总长的10.43%.各重复单元的拷贝数范围为1.9~42.5,平均拷贝数为2.6的重复序列数目最多,有211条.小卫星DNA序列的重复类型数目、序列长度及拷贝数见图3所示.

图3 异常汉逊酵母菌株792基因组小卫星DNA序列的重复单元长度与其数量关系
小卫星DNA序列数目与重复单位长度有一定关系,随着重复单位长度的增加呈下降趋势,这一特征在重复单元大于15 bp的小卫星序列中尤为显著;与微卫星DNA类似,小卫星DNA序列重复单位拷贝数较低,主要分布在1~3次;重复单元拷贝数与小卫星DNA序列之间无显著相关关系,见图4所示.
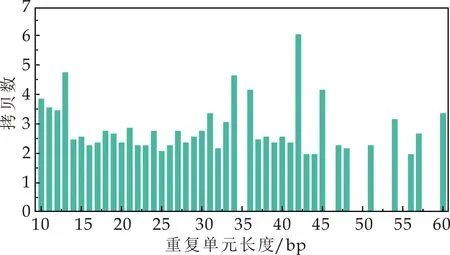
图4 异常汉逊酵母菌株792基因组小卫星DNA序列的重复单元长度与其拷贝数关系
2.3 散在重复序列在基因组中的分布特征
运用RepeatMasker方法,获得了异常汉逊酵母菌株792基因组中的多种散在重复序列(表3所示),其在基因组中占比很小,仅为0.79%左右.其中长末端重复序列(LTR)数目最多,为695条,占总数的46.30%;其次是DNA转座子,为459个;长散在重复序列(LINE)共303条;而短散在重复序列(SINE)只有25条;滚环(RC)14个.各类型散在重复的总长度分布与数目分布保持一致,其长度大小关系为LTR>DNA>LINE>SINE>RC,各占散在重复序列总长的48.54%、30.62%、23.42%、1.38%和1.27%.值得注意的是,虽然RC的重复序列数目较少,但其平均长度约为SINE的两倍.
本研究所采用的RepeatMasker方法具有较高的效率和搜索速度,可以发现低拷贝数量的家族,但只能搜索同源序列,不能产生新的元素.这类方法被认为是黄金准则,通常作为查找重复序列的第一步.

表3 异常汉逊酵母菌株792基因组中散在重复序列的分布
3 结论
本研究利用生物信息学RepeatMasker方法分析了异常汉逊酵母菌株792全基因组中的重复序列在其基因组中的分布及特征.结果表明,重复序列在基因组中含量较少,为全基因组的2.52%;微卫星DNA序列在其基因组中的占比不到千分之一,重复单元的拷贝数大多低于15个,重复单位长度与其拷贝数间存在着负相关;优势重复类型为三核苷酸重复,AAC为所有微卫星DNA类型中数目最多的基序;两核苷酸重复序列数目最少,且仅有AT重复.
在微卫星DNA和小卫星DNA中,AT含量均大于50%.这与Edwards等人的研究结果一致,AT类型重复的在植物、酵母和真菌类串联重复序列中的频率最高[13].串联重复串联序列中富含AT,与其全基因组DNA序列中AT含量较高有关,异常汉逊酵母菌株792全基因组的AT含量高达65.47%,为串联重复序列中富含AT提供了基础.对重复序列中的转座元件的分析发现,RNA转座子的数目与长度均高于DNA转座子,这与其他酵母菌的研究结果一致[14].三核苷酸类型重复和六核苷酸类型重复是异常汉逊酵母菌株792基因组微卫星DNA序列的优势核苷酸类型.
异常汉逊酵母菌作为汉逊酵母属中常见的一个种,具有一些酿酒酵母缺乏的酿造特性,是酿造产品香气成分的主要贡献者之一[15,16],有助于最终产品感官特性的提高[17],本研究结果为其分子育种和SSR分子标记的开发提供了理论基础,也为其遗传多样性研究提供了基础数据.

