葡萄无核性状的SSR新分子标记开发及应用
马亚茹,冯建灿,刘崇怀,樊秀彩,孙海生,姜建福,张颖
葡萄无核性状的SSR新分子标记开发及应用
马亚茹1,冯建灿2,刘崇怀1,樊秀彩1,孙海生1,姜建福1,张颖1
(1中国农业科学院郑州果树研究所,郑州 450009;2河南农业大学园艺学院,郑州 450002)
【目的】开发葡萄无核性状的SSR新分子标记,对葡萄杂种后代进行早期无核性状鉴定,为分子标记辅助育种奠定基础。【方法】对母本‘红地球’与父本‘森田尼无核’及F1代杂交群体的133份葡萄材料进行RAD-seq,将测序结果比对到参考基因组上;运用SAMtools软件生成CaSFS软件所需的pileup和glf文件,过滤获得亲本之间的有效SNP集;采用窗口滑动的方法,确定每个窗口的基因型,选择以每15个SNP为一个窗口,每次滑动一个SNP,得到每个个体的基因型并生成bin图;对生成的bin图采用拟测交的方式利用Joinmap软件进行连锁分析,构建‘红地球’与‘森田尼无核’遗传连锁图谱;用WindowQTLCartographer2.0软件进行QTL定位,在定位区间通过Perl程序语言找到符合SSR特征序列的35个SSR分子标记,用Primer premier5.0软件设计35个SSR分子标记引物对,通过HRM技术筛选亲本之间在无核性状上存在差异的SSR分子标记,并在131株F1代杂交群体及65个葡萄品种的自然群体中检测无核分型正确率、无核检测率、无核保持率。【结果】在‘红地球’与‘森田尼无核’构建的遗传连锁图谱上,将葡萄无核性状定位在chr18号染色体上,定位区间26 835 846—26 960 426,对无核的贡献率为77.9%,LOD阈值为26.3。亲本之间在无核性状上存在差异的SSR分子标记为VvSD10,其在111 bp等位点能对葡萄无核性状进行鉴定,利用分子标记VvSD10上的111 bp等位点通过HRM技术在葡萄F1代杂交群体及自然群体中进行葡萄无核性状的鉴定。结果表明,分子标记VvSD10在F1代杂交群体及自然群体中的无核分型正确率分别为97%、94%,其无核检测率均为56%,无核保持率分别为77%、85%。【结论】分子标记VvSD10在遗传群体及自然群体中的无核分型均能提供较高的准确信息,为无核葡萄的分子标记辅助育种奠定了基础。
葡萄;无核;QTL定位;SSR分子标记;辅助育种
0 引言
【研究意义】葡萄(L.)属于葡萄科葡萄属浆果,是中国第二大栽培果树,可以广泛应用于酿酒、鲜食、制汁、制干[1]等工艺。在鲜食葡萄中,无核化已经成为消费者最喜爱的特性之一。无核葡萄果汁丰富,含糖量高,食用方便,品质优良,深受广大消费者喜爱。在发达国家,消费的鲜食和制干葡萄超过50%为无核葡萄[2-3]。在中国,新疆是葡萄主产区,且大部分是无核葡萄品种[4],无核葡萄育种成为葡萄育种的主要目标之一。在无核葡萄育种过程中,使用最多的是常规杂交育种,通常使用有核品种做母本,无核品种做父本的杂交组合[5]。但是常规杂交育种周期长、后代选育率低,极大阻碍了无核葡萄的育种进程。近几年,分子标记的不断发展,给无核葡萄选育提供了便利[6-7]。利用分子标记对杂种后代进行早期的无核性状鉴定,极大地加速了无核葡萄的育种进程[8]。【前人研究进展】目前,有关分子辅助育种在葡萄上的应用已有部分研究。EMANUELLI等[9]利用SSR标记找到了与葡萄中的玫瑰香味相关联的一个候选基因,并将其命名为。CONSTANTINI[10]、MEJIA[11]等在两个鲜食葡萄品种的163株杂交个体中筛选到了与果粒大小、成熟期、种子数分别相关的基因。FISCHER等[12]在‘Regent’בLemberger’的153株杂交个体中定位到与葡萄真菌抗性连锁的QTL位点。HANANIA等[13]在有核与无核白系的葡萄花朵基因不同表达的转录分析中识别到一个叶绿素伴侣蛋白(ch-Cpn21),其沉默表达导致了烟草与西红柿种子的败育,泛激素延伸蛋白(S27a)在控制葡萄组织发育过程中可能存在某种角色[14],然而这些基因均不是控制葡萄无核性状的基因。Cabezas等[15]在‘赤霞珠’ב秋无核’的118株杂交子代中定位到了与葡萄无核性状相关联的QTL位点SDI(seed development inhibitor),该位点位于葡萄chr18染色体,与该位点紧密连锁的在198 bp等位点上能对后代的无核性状进行有效的分离,表型变异解释率为50%。MEJIA等[16]研究发现在SDI位点存在一个([17]、[18]),该基因位于下游463 bp处。葡萄与拟南芥的序列同源性较高,拟南芥MADS-box家族中的MADS5参与调控种子的生长发育[19-20]。【本研究切入点】目前,在葡萄无核育种中被广泛应用。然而,在本研究的F1代杂交群体中的分型正确率只有45%,不能作为辅助育种的有效工具。基于RAD-seq测序利用高密度连锁图谱,较为准确地定位到了无核性状的QTL,并结合高分辨率熔解曲线(HRM)开发了一个新的用于葡萄无核性状的SSR分子标记VvSD10,该标记位于葡萄内部,能够更好地连锁基因的分离。【拟解决的关键问题】本研究在前人研究结果基础上,在母本‘红地球’与父本‘森田尼无核’两亲本及F1代杂交群体中又重新筛选到了与葡萄无核性状相关联的SSR分子标记VvSD10。用分子标记VvSD10在F1代杂交群体以及从中国农业科学院郑州果树研究所的国家葡萄品种资源圃随机选择的葡萄自然群体中进行鉴定,检测该分子标记在杂交子代及自然群体无核性状中的分型率及保持率,进一步提高和补充前人研究结果。
1 材料与方法
田间试验于2015—2016年在中国农业科学院郑州果树研究所国家葡萄品种资源圃进行,其他试验于中国农业科学院郑州果树研究所果树基因资源实验室进行。
1.1 试验材料
以母本‘红地球’与父本‘森田尼无核’两亲本和131株F1代杂交群体,以及从中国农业科学院郑州果树研究所国家葡萄品种资源圃随机选取的65个葡萄品种的自然群体为试验材料(表1)。
1.2 试验方法
1.2.1 无核性状的调查 葡萄成熟期,调查F1代杂交群体每个单株的有无核性状,以种子的有或无作为评价标准,有种子且发育良好的单株属于有核,无核、残核与软核的单株均属于无核。
1.2.2 指标的测定方法
无核分型正确率(%)=存在111 bp等位点且表型为无核的葡萄植株个数/表型为无核的葡萄植株总数×100;
无核保持率(%)=存在111 bp等位点的植株个数/葡萄植株总个数×100;
无核检测率(%)=存在111 bp等位点且表型为无核的葡萄植株个数/存在111 bp等位点的葡萄植株个数×100。
1.2.3 葡萄叶片DNA的提取 采摘F1代杂交群体以及自然群体的无病害的鲜嫩叶片,采用植物基因组DNA提取试剂盒(由洛阳爱森生物科技有限公司提供)提取叶片基因组DNA,浓度用NanoDrop1000检测(Thermo Scientific),DNA样本浓度用无菌水统一稀释至10 ng∙µL-1。
1.2.4 分子标记遗传连锁图谱的构建 对两亲本及已排除假杂交情况的131株F1代杂交群体进行RAD-seq,运用SOAP比对软件将测序序列比对到参考基因组序列上(NCBI-GCF-000003745.3-12X);根据比对结果,运用SAMtools软件生成CaSFS软件所需的pileup和glf文件,鉴定群体中每个位点的情况,过滤获得亲本之间的有效SNP集;由于SNPs数量多,采用窗口滑动的方法,选择以每15个SNP为一个窗口,每次滑动一个SNP,确定每个窗口的基因型,得到每个个体的基因型并生成bin图;对生成的bin采用拟测交的方式,利用Joinmap软件进行连锁分析,构建‘红地球’与‘森田尼无核’遗传连锁图谱。
1.2.5 QTL定位 结合遗传连锁图谱与无核性状的调查数据,用WindowQTLCartographer2.0软件进行QTL定位。
1.2.6 SSR分子标记引物对的设计 在QTL定位区间,用Perl程序语言找到了符合SSR序列特征的35个SSR分子标记,利用Primer premier5.0软件设计35个SSR分子标记的引物对(引物由上海生工生物有限公司合成)。
1.2.7 SSR分子标记的分型与检测 以两亲本和131株F1代杂交群体以及自然群体的65个葡萄品种的基因组DNA为模板,利用筛选到的分子标记引物通过HRM技术进行基因分型分析,以两亲本作为对照样本,与母本分为一组的样本为有核,与父本分为一组的样本为无核。
HRM分析仪器为罗氏Light Cycler®480Ⅱ,试剂为Light Cycler®480 High Resolution Melting Master。反应体系为15 µL:1.6 µL浓度为3 µmol∙L-1的Mgcl2,7.5 µL 2×HRM Master Mix,SSR上、下游引物各0.8 µL(浓度为10 µmol∙L-1),1 µL模板DNA,无菌蒸馏水3.3 µL。
HRM技术包括PCR和HRM分析过程。PCR反应条件为:95℃预变性3 min;95℃变性10 s,62℃复性15 s,72℃延伸10 s,共48个循环;升温和降温过程温度变化率分别为4.4和2.2℃∙s-1。HRM分析过程为:95℃变性1 min,40℃杂交1 min;65—95℃读取熔解曲线,温度变化率为1℃∙s-1
2 结果
2.1 葡萄无核性状的QTL定位
通过形成的bin图构建‘红地球’与‘森田尼无核’遗传连锁图谱,并结合无核性状调查数据,将葡萄无核性状定位在chr18号染色体上,定位区间26 835 846—26 960 426,对无核表型的贡献率为77.9%,LOD阈值为26.3(表2,图1)。
2.2 SSR分子标记引物对的筛选
根据已设计的35对SSR分子标记引物对,在母本‘红地球’与父本‘森田尼无核’两个亲本中通过HRM技术筛选亲本之间在有无核性状上存在差异的分子标记引物对,命名为VvSD10,其正向引物序列为5′-agagctcatttggattaagagcg agtaattatattgt-3′,反向引物序列为3′-ggaa aaatccatcgctaacaaagtattaattctcttca-5′。同时,用含有6-FAM荧光标记的PCR产物进行毛细管电泳(图2)。结果显示,分子标记VvSD10在父本‘森田尼无核’中出现了111 bp等位点,在母本‘红地球’中出现了99 bp与120 bp两个等位点。
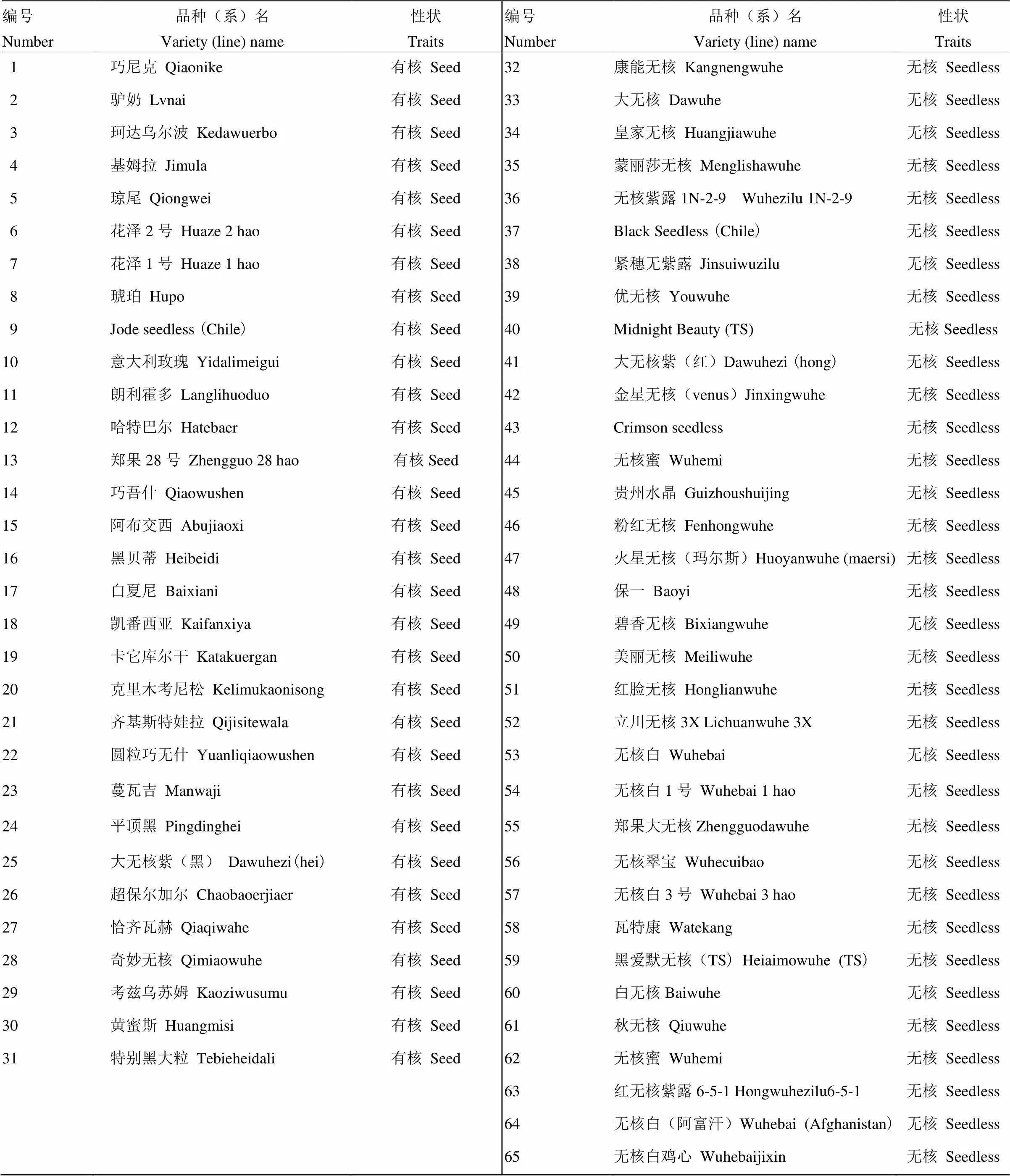
表1 自然群体葡萄品种信息

表2 葡萄无核性状的QTL定位分析
2.3 分子标记VvSD10在不同葡萄群体无核性状中的分析
基于分子标记VvSD10中存在的111 bp等位点(即与父本分为一组)对葡萄F1代杂交群体及自然群体的无核性状进行鉴定,结果显示,在被检测的131株F1代杂交群体中,无核性状的植株有59个,其中存在111 bp等位点且表现无核性状的植株有57个。由此可知,分子标记VvSD10在F1代杂交群体中的无核分型正确率为97%,无核保持率为77%,无核检测率为56%。在65个葡萄品种的自然群体中,有33株为无核植株,其中存在111 bp且表现无核性状的有31株。由此可知,分子标记VvSD10在自然群体中的无核分型正确率、无核保持率、无核检测率分别为94%、85%、56%(表3,图3)。
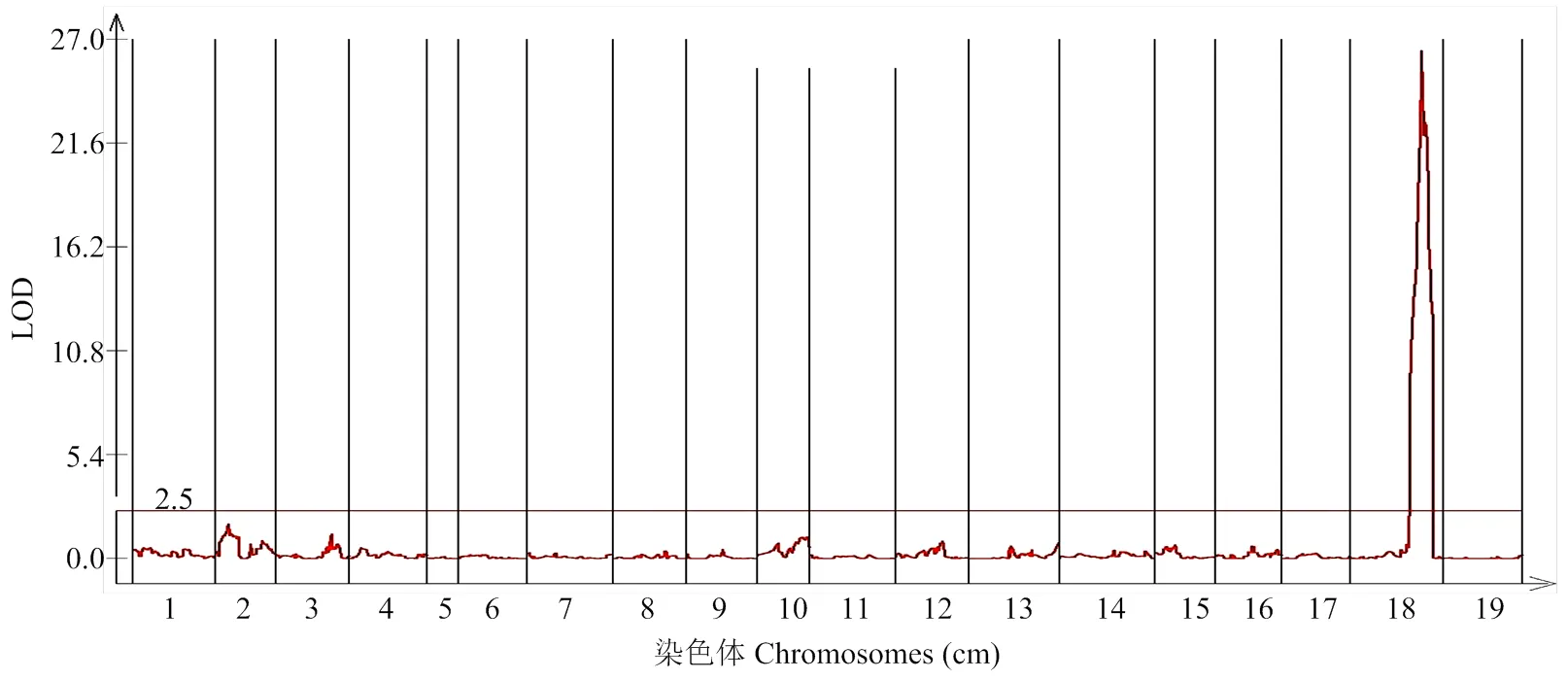
图1 葡萄无核性状定位图
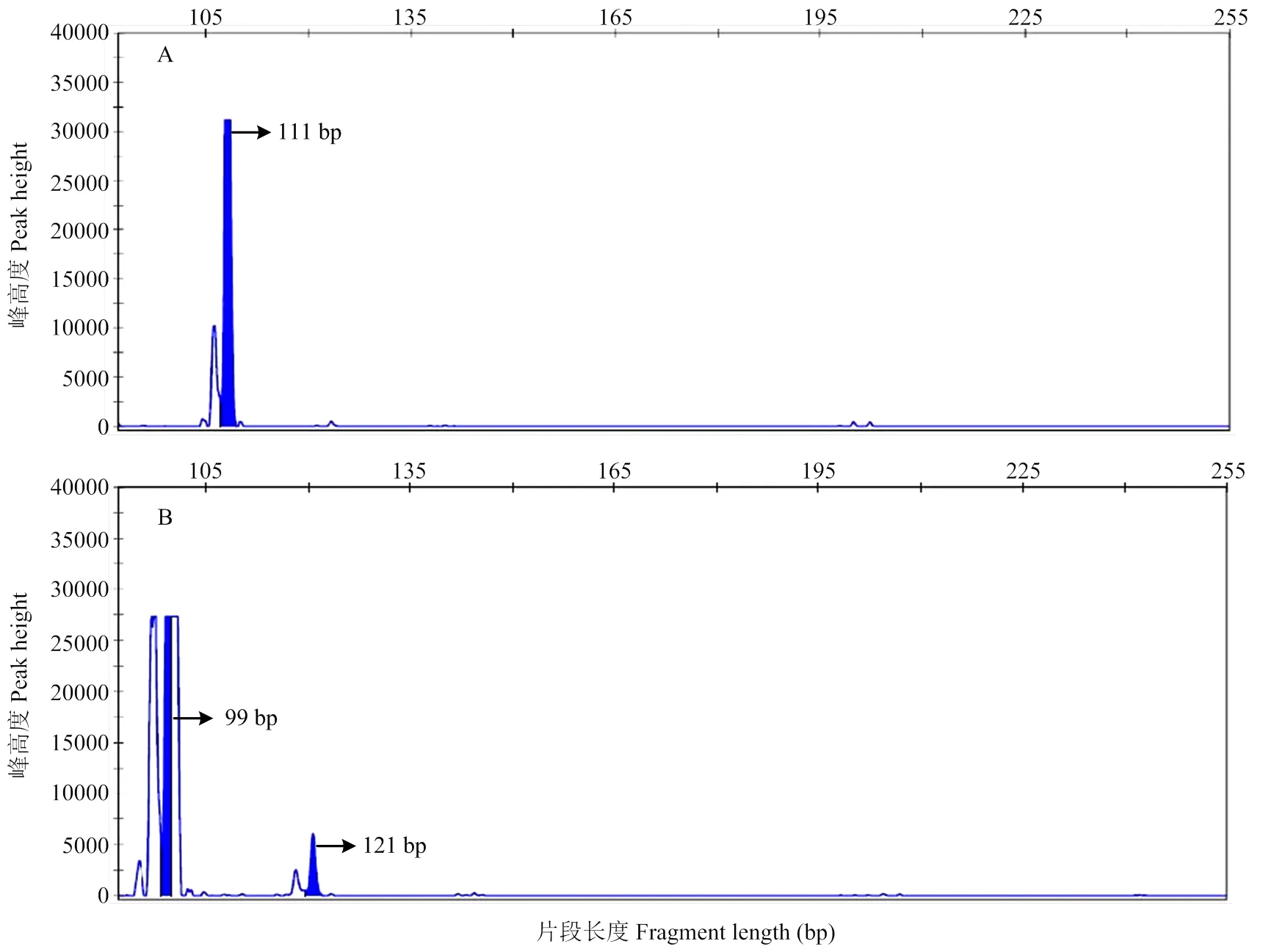
图2 分子标记VvSD10在母本‘红地球’(A)与父本‘森田尼无核’(B)中的毛细管电泳
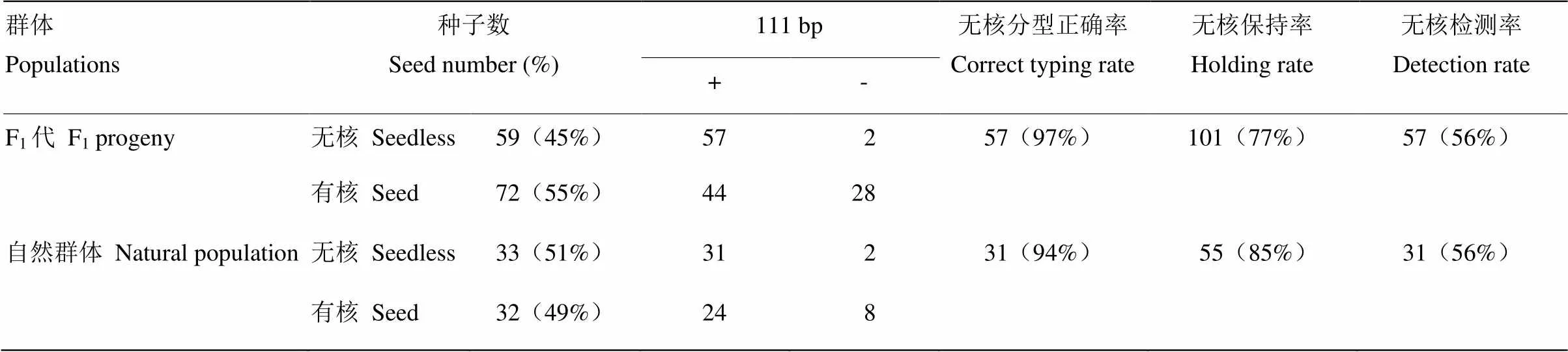
表3 分子标记VvSD10在不同葡萄群体无核性状中的分析
“+”代表存在111 bp等位点;“-”代表不存在111 bp等位点
“+” represents the existence of 111 bp loci; “-” represents the absence of 111 bp loci
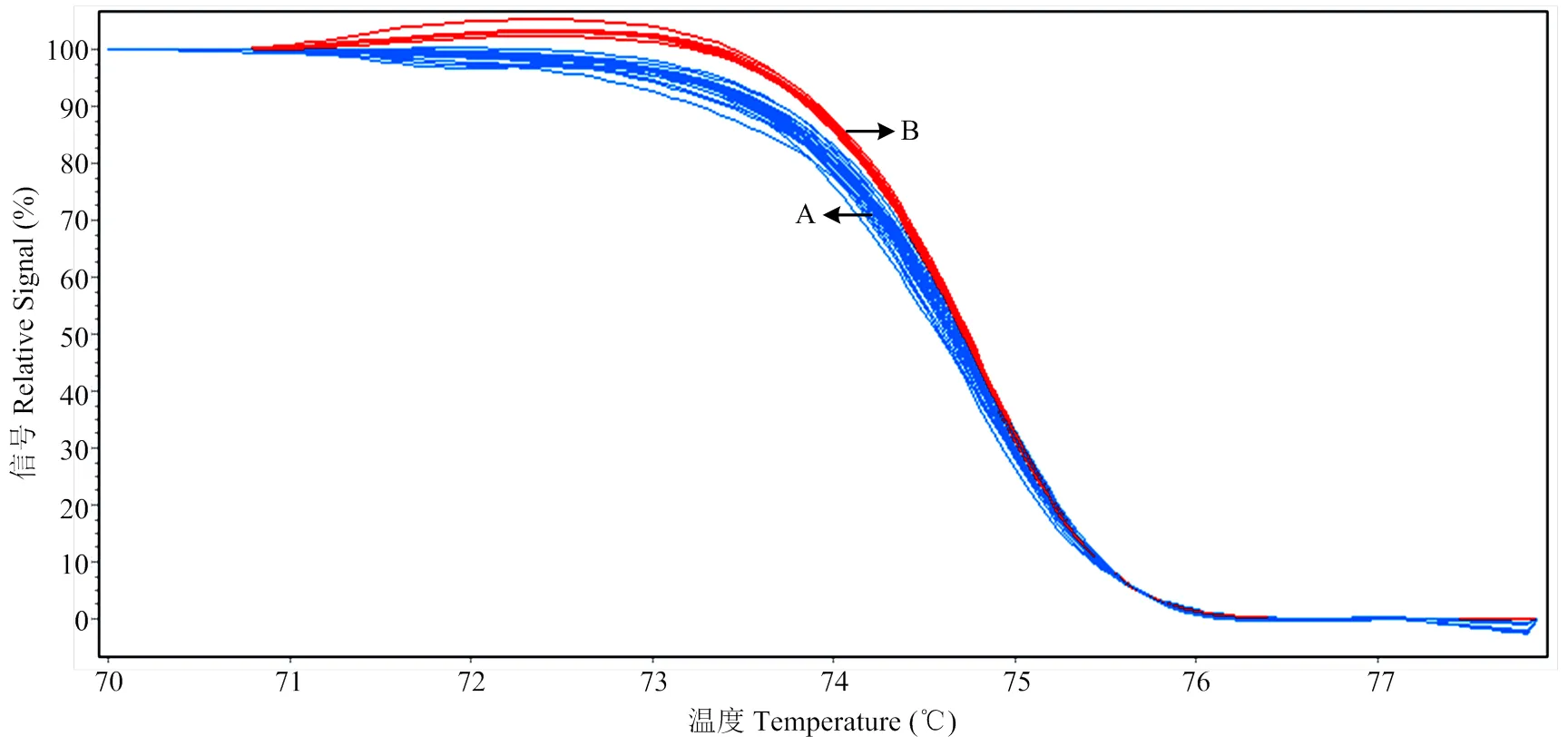
A:重叠区域代表样本分型结果为有核(与母本归为一组);B:重叠区域代表样本分型结果为无核(与父本归为一组)
3 讨论
3.1 HRM技术在科学研究中的应用
高分辨率熔解曲线(High-Resolution Melting,HRM)技术,是国际上发展起来的一种基于单核苷酸熔解温度不同而形成不同熔解曲线的突变扫描和基因分型新技术。该技术的原理是利用饱和的荧光染料对PCR产物进行熔解曲线分析,根据熔解曲线的形状进行基因分型[21-24]。本研究使用了HRM技术,它是一种有效的、高分辨率的DNA差异检测技术,因操作简便迅速,使用成本低,结果准确,实现了真正的闭关操作,而受到普遍关注。目前可以应用于基因突变扫描[25]、基因分型[26]、甲基化分析[27]、法医学鉴定[28]等方面。曹春鸽等[29]应用HRM技术对CYP2C19*2和CYP2C19*3进行了双重SNP分型,结果显示,64个随机DNA样本的HRM分型结果与测序结果一致性达到100%,与邓建强等[28]应用HRM技术进行法医昆虫种属鉴定研究中的结果相似。嘎利兵嘎[30]利用HRM技术在动物病毒基因分型中的研究及应用中,建立了6种常见动物病毒的PCR-HRM检测方法,成功对6种动物病毒进行了基因分型,为动物传染病分子诊断提供了一种快速、简单、成本低廉的PCR-HRM检测方法。在本研究中,应用HRM技术对葡萄无核性状进行基因分型鉴定,筛选出了能检测葡萄无核性状的标记VvSD10,同时该标记在F1代杂交群体及自然群体中的无核分型正确率分别为97%、94%,提供了较高的准确信息。
3.2 SSR标记在葡萄分子辅助育种中的应用
SSR(simple sequence repeats)标记是近些年发展起来的一种以特异引物PCR为基础的分子标记技术,是一类由几个核苷酸为重复单位组成的串联重复序列,因其具有高多态性、多等位基因、共显性遗传等优势而被广泛应用[31-32]。CABEZAS等[15]利用SSR标记方法定位到了与葡萄无核性状紧密连锁的,与MEJIA等[11]的研究结果相似。在CABEZAS等[15]的研究中,在无核葡萄品种‘森田尼无核’中检测到198 bp与200 bp两个等位点,在有核葡萄品种‘红地球’中检测到200 bp与202 bp两个等位点。并且,在‘Dona Maria’与‘Muscat of Alexandria’2个有核葡萄品种中也检测到198 bp等位点。类似报道中,在‘红地球’中仅检测到200 bp一个等位点[33]。本研究中,用在母本‘红地球’中检测到200 bp与202 bp两个等位点,在父本‘森田尼无核’中检测到200 bp与205 bp两个等位点,但并未检测到中的典型等位点198 bp。
KARAAGAC等[33]利用的198 bp等位点对‘Red Globe’בCrimson Seedless’224株杂交个体的有无核性状进行检测,存在198 bp等位点的有112株,无核保持率为50%;在‘96-71-7’בCrimson Seedless’的126株杂交个体中,存在198 bp等位点的有96株,无核保持率为76%。本研究中,在‘红地球’ב森田尼无核’F1代杂交群体中的分型正确率仅有45%,远不能达到检测该群体无核性状的目的。本研究基于SSR分子标记筛选到了与葡萄无核性状紧密连锁的SSR分子标记VvSD10,其在无核性状上的典型等位点为111 bp。并且在131株F1代群体中有101株可以检测到111 bp等位点,保持了77%的无核率,在分子标记辅助育种中可以提前舍去23%个体。由于不同遗传背景的差异,本研究中的分子标记VvSD10在不同杂交群体的无核分型率、无核保持率等会有不同。
4 结论
本研究基于RAD-seq测序方法对葡萄无核性状进行QTL定位,将葡萄无核性状定位在葡萄Chr18染色体上,定位区间位于26 835 846—26 960 426,约124 kb。在定位区间设计35对SSR分子标记引物,通过高分辨率熔解曲线(HRM)与毛细管电泳结合的方法,筛选到了能检测葡萄无核性状的SSR分子标记VvSD10,该标记具有分型明确,表现稳定的特点,在遗传群体和自然群体中的无核分型均能提供较高的准确信息,可以在育种中提供辅助信息。
[1] MENCARELLI F, ANDREA B, GIANCARLO D. Grape: Post- harvest Operations[D]Italy: University of Viterbo, 2005.
[2] 崔梦杰, 王晨, 张文颖, 汤崴, 朱旭东, 李晓鹏, 房经贵. 无核葡萄研究进展. 植物生理学报, 2017, 53(3): 317-330.
CUI M J, WANG C, ZHANG W Y, TANG W, ZHU X D, LI X P, FANG J G. Research advance of seedlessness in grape., 2017, 53(3): 317-330. (in Chinese)
[3] 孙红, 李晓萍. 美国新注册的葡萄品种. 落叶果树, 2001, 33(5): 57-58.
SUN H, LI X P. Newly registered grape varieties in the United States., 2001, 33(5): 57-58. (in Chinese)
[4] 蒲胜海, 张计峰, 丁峰, 马雪琴. 新疆葡萄产业发展现状及研究动态. 北方园艺, 2013(13): 200-203.
PU S H, ZHANG J F, DING F, MA X Q. Development status and research trends of grape industry in Xinjiang., 2013(13): 200-203. (in Chinese)
[5] 陶建敏. 葡萄无核育种技术研究及种质创新[D]. 南京: 南京农业大学, 2007.
TAO J M. Study on the technology in breeding of seedless grape and innovation of germplasm [D]. Nanjing: Nanjing Agricultural University, 2007. (in Chinese)
[6] BOUQUET A, DANGLOT Y. Inheritance of seedlessness in grapevine (L.)., 1996, 35(1): 35-42.
[7] WEI X, SYKES S R, CLINGELEFFER P R. An investigation to estimate genetic parameters in CSIRO's table grape breeding program., 2002, 128(3): 343-351.
[8] DOLIGEZ A, BOUQUET A, DANGLOT Y, LAHOGUE F, RIAZ S, MEREDITH C P, EDWARDS K J, THIS P. Genetic mapping of grapevine (L.) applied to the detection of QTLs for seedlessness and berry weight., 2002, 105(5): 780-795.
[9] EMANUELLI F, BATTILANA J, COSTANTINI L, LE CUNFF L, BOURSIQUOT J M, THIS P, GRANDO M S. A candidate gene association study on muscat flavor in grapevine (L.)., 2010, 10(1): 241.
[10] COSTANTINI L, BATTILANA J, LAMAJ F, FANIZZA G, GRANDO M S. Berry and phenology-related traits in grapevine (L.): From quantitative trait loci to underlying genes., 2008, 8(1): 38.
[11] MEJIA N, GEBAUER M, MUNOZ L, HEWSTONE N, MUNOZ C, HINRICHSEN P. Identification of QTLs for seedlessness, berry size, and ripening date in a seedless x seedless table grape progeny., 2007, 58(4): 499-507.
[12] FISCHER B M, SALAKHUTDINOV I, AKKURT M, EIBACH R, EDWARDS K J, TOPFER R, ZYPRIAN E M. Quantitative trait locus analysis of fungal disease resistance factors on a molecular map of grapevine., 2004, 108(3): 501-515.
[13] HANANIA U, VELCHEVA M, OR E, FLAISHMAN M, SAHAR N, Perl A. Silencing of chaperonin 21, that was differentially expressed in inflorescence of seedless and seeded grapes, promoted seed abortion in tobacco and tomato fruits.,2007, 16: 515-525.
[14] HANANIA U, VELCHEVA M, SAHAR N, FLAISHMAN M, OR E, DEGANI O, PERL A. The ubiquitin extension protein S27a is differentially expressed in developing flower organs of Thompson seedless versus Thompson seeded grape isogenic clones., 2009, 28(7): 1033-1042.
[15] CABEZAS J A, CERVERA M T, RUIZGARCIA L, CARRENO J, MARTINEZ-ZAPATER J M. A genetic analysis of seed and berry weight in grapevine., 2006, 49(12): 1572-1585.
[16] MEJIA N, SOTO B, GUERRERO M,CASANUEVAL X, HOUEL C, MICCONO M A, RAMOS R, CUNFF L L, BOURSIQUOT J M, HINRICHSEN P, ADAM-BLONDON A F. Molecular, genetic and transcriptional evidence for a role of VvAGL11 in stenospermocarpic seedlessness in grapevine.y, 2011, 11(1): 57.
[17] BOSS P K, SENSI E, HUA C, DAVISE C, THOMAS M R. Cloning and characterisation of grapevine (L.) MADS-box genes expressed during inflorescence and berry development., 2002, 162(6): 887-895.
[18] DIAZ-RIQUELME J, LIJAVETZKY D, MARTINEZ-ZAPATER J M, CARMONA M J. Genome-wide analysis of MIKCC-type MADS box genes in grapevine., 2009, 149(1): 354-369.
[19] PARENICOVA L, FOLTER S D, KIEFFER M, HORNER D S, FAVALLI C, BUSSCHER J, COOK H E, INGRAM R M, KATER M M, DAVIES B, ANGENENT G C, COLOMBO L. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in: New openings to the MADS world., 2003, 15: 1538-1551.
[20] IMMINK R G, TONACO I A, FOLTER S D, SHCHSNNIKOVA A, DIJK A D V, BUSSCHER-LANGE B, BORST J W, ANGENENT G C. SEPALLATA3: The ‘glue’ for MADS box transcription factor complex formation., 2009, 10(2): R24.
[21] 谭昌渊, 罗九甫, 任兆瑞. 高分辨率熔解曲线分析—分子诊断的新技术. 生命科学研究, 2009, 13(3): 268-271.
TAN C Y, LUO J F, REN Z R. High Resolution Melting-New method for molecular diagnosis., 2009, 13(3): 268-271. (in Chinese)
[22] SOUZA R A, FALAAO J P. A novel high-resolution melting analysis- based method for, genotyping., 2014, 106(3): 129-134.
[23] 舒庆尧, 张华丽, 黄建中. 一种用于水稻光敏雄性核不育基因lncR分型的引物及其应用. CN 103146696 A[P]. 2013.
SHU Q Y, ZHANG H L, HUANG J Z. Primer and application for lncR typing of photoperiod sensitive genic male sterile gene in rice. CN 103146696 A[P]. 2013. (in Chinese)
[24] 尤崇革, 李玉民, 马克君, 施鑫鹤, 郜莉娜, 李菲菲. 基于高分辨熔解技术进行单核苷酸多态性基因分型的方法学评价. 中华临床医师杂志(电子版), 2012(7): 1786-1790.
YOU C G, LI Y M, MA K J, SHI X H, GAO L N, LI F F. Evaluation on the methodology of SNP genotyping based on high resolution melting., 2012(7): 1786-1790. (in Chinese)
[25] NOMOTO K, TSUTA K, TAKANO T, FUKUI T, YOKOZAWA K, SAKAMOTO H, YOSHIDA T, MAESHIMA A M, SHIBATA T, FURUTA K, OHE Y, MATSUNO Y. Detection of EGFR mutations in archivedcytologic specimens of non-small cell lung cancer using high-resolution melting analysis., 2006, 126(4): 608-615.
[26] LOCHLAINN S Ó, AMOAH S, GRAHAM N, ALAMER K, RIOS J J, KURUP S, STOUTE A, HAMMOND J P, OSTERGAARD L, KING G J, WHITE P J, BROADLEY M R. High resolution melt (HRM) analysis is an efficient tool to genotype EMS mutants in complex crop genomes.s, 2011, 7(1): 1-9.
[27] MAAT W, VELDEN P A V D, OUTLUITING C, PLUG M, DIRKS A, JAGER M, GRUIS N A. Epigenetic inactivation of RASSF1a in uveal melanoma., 2007, 48(2): 486-490.
[28] 邓建强, 李文慧, 李亮, 张文彬, 龙仁, 蔡继峰. 应用HRM技术进行法医昆虫种属鉴定的初步研究. 中国热带医学, 2014, 14(1): 9-11.
DENG J Q, LI W H, LI L, ZHANG W B, LONG R, CAI J F. Preliminary study of species identification by HRM in forensic entomology., 2014, 14(1): 9-11. (in Chinese)
[29] 曹春鸽, 孙海燕, 周芳芳, 王诗铭, 陈红岩, 卢大儒. 应用HRM技术对CYP2C19*2和CYP2C19*3进行双重SNP分型. 遗传, 2013, 35(7): 923-930.
CAO C G, SUN H Y, ZHOU F F, WANG S M, CHEN H Y, LU D R. Duplex genotyping ofandby high-resolution melting curve analysis., 2013, 35(7): 923-930. (in Chinese)
[30] 嘎利兵嘎. 高分辨率熔解曲线(HRM)技术在动物病毒基因分型中的研究及应用[D]. 北京: 中国农业大学, 2015.
GA L B G. Research and application of high resolution melting curve analysis in the genotyping and detection of animal viruses [D]. Beijing: China Agricultural University, 2015. (in Chinese)
[31] SONG Q J, JIA G F, ZHU Y L, GRANT D, NELSON R T, HWANG E Y, HYTEN D, CREGAN P B. Abundance of SSR motifs and development of candidate polymorphic SSR markers (BARCSOYSSR_1.0) in soybean., 2010, 50(5): 1950-1960.
[32] KUBOTA S, LIU Q, KESSUWAN K, OKAMOTO N, SAKAMOTO T, NAKAMURA Y, SHIGENOBU Y, SUGAYA T, SANO M, UJI S, NOMURA K, OZAKI A. High-throughput simple sequence repeat (SSR) markers development for the kelp grouper (), and cross-species amplifications forspecies., 2014, 5(2): 117-130.
[33] KARAAGAC E, VARGAS A M, ANDRES M T D, CARRENO I, IBANEZ J, CARRENO J, MARTINE-ZAPATER J M, CABEZAS J A. Marker assisted selection for seedlessness in table grape breeding., 2012, 8(5): 1003-1015.
(责任编辑 赵伶俐)
Development and application of SSR new molecular marker for seedless traits in grape
MA YaRu1, FENG JianCan2, LIU ChongHuai1, FAN XiuCai1, SUN HaiSheng1, JIANG JianFu1, ZHANG Ying1
(1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009;2College of Horticulture, Henan Agricultural University, Zhengzhou 450002)
【Objective】The objective of this study is to develop a new SSR molecular marker for seedless traits in grape, and to identify the early seedless traits of grape hybrids, and to lay the foundation for molecular marker assisted selection breeding. 【Method】RAD-seq was carried out in 133 grape materials including the female parent ‘Red Globe’, the male parent ‘Centennial Seedless’ and their F1hybrids, and the sequencing results were compared to the reference genome. SAMtools software was used to generate CaSFS software required for pileup and GLF files, and the effective SNP sets between parents were obtained by filtration. A window sliding method was used to determine the genotype of each window, and selected one window per 15 SNP and slided one SNP at a time. The genotype of each individual was obtained and the bin generated. Linkage analysis was carried out based on Joinmap software for generated bin, and genetic linkage map was constructed between ‘Red Globe’ and ‘Centennial Seedless’. The QTL loci was mapped using WindowQTLCartographer2.0 software. 35 SSR molecular markers consistent with the SSR characteristic sequence were identified by Perl programming language in the QTL mapping interval, and then 35 pairs of SSR molecular marker primers were designed. The molecular marker was screened in the differences with the seedless traits between parents by HRM technology, and the accuracy of seedless typing, the rate of seedless detection and the rate of seedless holding were detected in 131 F1hybrid population and natural population including 65 grape varieties.【Result】On the genetic linkage map between ‘Red Globe’ and ‘Centennial Seedless’, the seedless traits of grape were mapped on chromosome 18, and the mapping interval was 26 835 846-26 960 426, and the contribution rate to seedless was 77.9%, and the LOD threshold was 26.3. The molecular marker VvSD10 was screened in the differences with the seedless traits between parents, and there was a 111 bp loci in seedless traits of grape. Identification of seedless traits in grape F1hybrid population and natural population by HRM technology using 111 bp loci on VvSD10 marker, the results indicated that in F1hybrid population and natural population, the correct typing rate of molecular marker VvSD10 was 97% and 94%, respectively, and the rate of seedless detection of both populations was 56%, the rate of seedless holding was 77% and 85%, respectively. 【Conclusion】Seedless typing in grape by molecular marker VvSD10 could provide high accurate information in both genetic and natural populations and laid the foundation for molecular marker assisted breeding program in seedless grape.
L.; seedless; QTL mapping; SSR molecular marker; assisted breeding
2017-11-20;
2018-01-04
国家葡萄产业技术体系(CARS-29-yc-1)、中国农业科学院科技创新工程(CAAS-ASTIP-2017-ZFRI-02)、果树(桃、枣、苹果、葡萄等)优异种质资源创新和高效育种体系构建(151100110900)
马亚茹,Tel:13223070473;E-mail:1256381782@qq.com。
张颖,Tel:13525530042;E-mail:zhangying05@caas.cn
10.3864/j.issn.0578-1752.2018.13.017

