Loxp序列位置对基因表达的影响
许盈盈,靳伟,代敏敏,樊宝良
Loxp序列位置对基因表达的影响
许盈盈,靳伟,代敏敏,樊宝良
(河北农业大学动物科技学院 河北保定 071001)
【目的】先选用绿色荧光蛋白基因()作为试验基因构建loxp位点位于egfp基因上游或下游不同位置的表达结构,对loxp位点的位置对基因表达的影响进行初步分析。然后以植酸酶()基因为另一个试验基因对通过egfp基因得出的结果进行进一步验证。研究结果为开展通过基因打靶技术将外源基因与内源基因通过2A序列连接共表达的转基因动物制作中,打靶位点的选择提供可靠依据。【方法】先选择增强绿色荧光蛋白基因()为试验基因,红色荧光蛋白基因(为内参基因。以pEGFP-N2、pGEM-5zf-loxp质粒为基础,构建了ploxp-EGFP(loxp序列位于egfp基因开放阅读框的Kozak序列上游的5′非翻译区)、pEGFP-loxp(loxp序列位于egfp基因开放阅读框的终止密码子下游的3′非翻译区)、ploxp-EGFP-loxp(egfp基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列)。以pEGFP-N2质粒为对照质粒,以pDsRed2-N1为内参质粒,与所构建的egfp基因各表达质粒共转染PK15细胞,转染24h后在荧光显微镜下观察荧光表达情况,用荧光分析软件Image J进行荧光强度分析并记录荧光强度,进而应用SPSS19.0软件对各转染荧光表达情况进行分析,确定loxp序列的位置对基因表达的影响。为了对以egfp基因为试验基因所得到的结果进行进一步验证,本试验选择植酸酶(phytase基因)基因为试验基因,egfp基因为转染内参基因萤火虫荧光素酶基因(luciferase基因)为表达内参基因。以pIREsNEo、pGEM-5zf-loxp和pT-phytase、pGL4.13[luc2/SV40]质粒为基础。构建了p-SV40-luciferase-CMV-loxp-phytase(loxp序列位于phytase基因开放阅读框的Kozak序列上游的5′非翻译区)、p-SV40-luciferase-CMV-loxp-phytase-loxp(phytase基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列)、p-SV40-luciferase-CMV-phytase-loxp(loxp序列位于phytase基因开放阅读框的终止密码子下游的3′非翻译区)和p-SV40-luciferase-CMV-phytase(phytase基因开放阅读框的上下游均无loxp序列)等试验质粒,以egfp基因(pEGFP-N2)为转染内参基因与所构建的各种phytase基因表达结构共转染PK15细胞,同时设置一个转pEGFP-N2+ pDsRed2-N1的空白对照。转染48h后在荧光显微镜下观察绿色荧光蛋白基因表达情况,用荧光分析软件Image J进行荧光强度分析并记录荧光强度,应用SPSS19.0软件对各转染绿色荧光表达情况进行分析。同时应用钼蓝法测定各转染的植酸酶活性。应用萤火虫萤光素酶报告基因检测试剂盒检测各转染萤火虫荧光素酶活性。用SPSS19.0软件对各转染的植酸酶和萤火虫荧光素酶表达水平(活性)进行统计分析。【结果】试验中以为内参基因,对转染后每个转染的不同区域红色荧光强度平均值(代表各转染红色荧光蛋白的表达水平)进行统计分析表明egfp基因各结构每个转染的间红色荧光蛋白表达水平差异不显著,表明不同转染间排除了转染操作、质粒纯度、细胞活性等的差异对分析结果的影响,转染前质粒的电泳结果表明,各质粒间不同构象的质粒比例基本一致,这也基本排除了转染用质粒质量的差异对转染效果的影响,对各转染不同区域绿色荧光强度的平均值(代表各转染的egfp基因表达水平)进行的统计分析表明,同一结构不同转染间egfp基因表达水平差异不显著,不同结构的转染间存在差异,其中ploxp-EGFP和ploxp-EGFP-loxp结构转染中绿色荧光蛋白的表达水平显著低于pEGFP-loxp和pEGFP-N2,而pEGFP-loxp和pEGFP-N2转染间基因间的表达差异不显著。这一结果初步说明loxp序列在外源基因开放阅读框下游时对基因表达水平没有影响,在外源基因开放阅读框上游时对基因表达呈负影响,为了进一步验证上述结果的可靠性,本研究以植酸酶基因作为另一试验基因,以萤火虫荧光素酶基因为表达内参基因,以egfp基因为转染内参基因再次进行了验证,验证试验得到了相似的结果。【结论】开展通过基因打靶技术将外源基因与内源基因通过2A序列连接,实现外源基因和内源基因共表达的转基因动物制作研究中,以Cre/loxp系统作为报告基因删除工具时,则内源基因的下游为最佳的打靶位点。
loxp序列; 基因表达; 2A序列; 基因打靶
0 引言
【研究意义】转基因动物技术是一种行之有效的快速动物育种新技术,可用于高附加值[1-2]、环境友好[3]、生产性能[4]或产品品质改良[5]以及抗病[6]等的动物新品种培育。目前转基因动物制作所获得的转基因动物,外源基因多以多拷贝串联方式随机整合到受体动物基因组中。这就使得目前转基因动物制备中存在导致内源基因表达异常[7]、外源基因异位表达[8-9]、外源基因表达及遗传稳定性差[10]且外源基因的表达水平缺乏可预见性和可控性[11]等问题。这些因素一直制约着转基因动物的研究与应用,探讨解决上述问题的有效方法是推动转基因动物技术进步并有效服务于人类的重要前提。基因打靶技术[12]能够将外源基因精确的插入到受体基因组中的特定位点。探讨通过基因打靶技术将外源基因定点插入到内源基因的开放阅读框的上游或者下游,通过融合肽2A序列[13]连接,使外源基因与内源基因融合表达,是解决上述问题的思路之一。早期的基因打靶技术因为效率低下[14],无法实现上述目标。对基因打靶机理的研究表明,在特定位点高效实现DNA双链断裂能大大提高了基因打靶的效率[15]。近年来一些人工核酸酶技术如:锌指核酸酶[16-17]、类转录激活因子效应物核酸酶[18-19],和CRISPR/Cas 系统[20-21]等的出现。使得人们可以在基因组的任意位点高效产生DNA双链断裂而使得基因打靶效率得到了显著提高[22],这使得上述思路的实现成为可能。然而这些新的技术的出现与应用虽然使得基因打靶效率获得了显著提高但仍然较低。在大多数情况下利用这些技术进行基因打靶仍然需要使用报告基因来有效富集阳性克隆以提高筛选效率[23-24]。而从生物安全角度考虑在获得了同源重组的转基因细胞后需要将报告基因表达结构删除。Cre/Loxp系统是目前应用较为广泛的报告基因删除系统[25-27]。【前人研究进展】Cre/loxp系统是1981年人们从P1噬菌体及其宿主细胞中发现的重组酶系统[28]。loxp序列是由34个碱基对构成的一个反向重复序列。Cre酶能够对该序列进行识别和加工。如果在一段序列的两侧各有一个loxp序列且方向相同,则在Cre酶作用下位于两个loxp序列之间的DNA就会被从原来的DNA分子中切下,也就是删除。人们应用这一原理建立了基于Cre/loxp的删除系统,并应用于报告基因的删除以及在基因功能研究中用于条件敲除内源基因,来阐明该基因在特定组织细胞类型或特定发育时期的功能[29-30]。Cre/loxp系统用于报告基因或作为条件敲除内源基因时loxp序列都是位于报告基因或欲敲除基因完整表达结构的两侧,已有的研究表明这种结构对报告基因或欲敲除的内源基因的表达没有影响。【本研究切入点】而上述希望通过基因打靶技术将外源基因与内源基因通过2A序列链接实现共表达的设想中会将loxp序列留置于基因开放阅读框的上游或下游的非翻译区中。而loxp序列已如前述是一种反向重复序列,其出现在基因开放阅读框的上游或下游的非翻译区中时,将在转录产生的mRNA上游或下游非翻译区中形成发卡型二级结构,而这种二级结构的形成有可能对mRNA的翻译效率产生影响,而且loxp位点的位置不同可能影响程度有所不同,阐明这一效应对开展上述思路的研究中打靶位点的选择具有重要意义,而目前还缺乏这方面的相关研究报道。【拟解决的关键问题】以增强的绿色荧光蛋白(egfp)基因为试验基因,探究loxp序列在egfp基因开放阅读框的上游或下游的非翻译区序列中对基因表达的影响,并进一步以植酸酶基因为试验基因对以egfp基因为试验基因得到的结果进行进一步验证,为上述思路的研究中打靶位点的选择提供参考依据。
1 材料与方法
1.1 材料
1.1.1 细胞系、菌株、载体 猪肾细胞系PK15(ATCC,美国);大肠杆菌()DH5α(TaKaRa, 中国大连);质粒pEGFP-N2(Clonth,美国)、pIREsNeo(Clonth,美国)、pGL4.13[luc2/SV40](Promaga, 美国)、pGEM-5zf-loxp(河北农业大学动物科技学院实验室构建)和pDsRed2-N1(TaKaRa, 中国大连)等由河北农业大学动物科技学院实验室保存。
1.1.2 试剂 无内毒质粒DNA提取试剂盒QIAGEN Plasmid Mini Kit购自QIAGEN(德国);限制性内切酶、DNA 分子量标准1kb ladder marker、DNA连接酶等购自Fermentas(美国);LipofectamineTM2000脂质体转染试剂购自Invitrogen(美国);胎牛血清、DMEM、0.25%胰酶购自GIBCO(美国);无水乙酸钠、植酸钠购自Sigma公司(德国),其他试剂均为国产,萤火虫萤光素酶报告基因检测试剂盒购自碧云天生物技术公司(中国);DNA测序工作由上海生工生物工程有限公司完成。
1.2 试验方法
1.2.1 ploxp-EGFP、pEGFP-loxp、ploxp-EGFP--loxp质粒的构建 试验于2017年3月于河北农业大学动物遗传育种与繁殖实验室进行。以pEGFP-N2、pGEM-5zf-loxp质粒为骨架质粒,通过一系列酶切连接反应构建所需质粒。这些质粒的结构见图1。
1.2.2 pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-lox、p-SV40- luciferase-CMV-phytase-loxp和pSV40-luciferase- CMV-phytase质粒的构建 试验于2018年1月于河北农业大学动物遗传育种与繁殖实验室进行。以pIREsNeo、pGEM-5zf-loxp、pT-phytase、pGL4.13[luc2/ SV40]为基础通过一系列酶切连接反应构建了这些质粒。这些质粒的结构见图2。
1.2.3 转染用质粒DNA提取 各质粒DNA提取按QIAGEN Plasmid Mini Kit 质粒提取试剂盒说明书进行,所提取质粒电泳结果见图3.
1.2.4 细胞培养与转染 猪肾细胞系PK15在37℃,5% CO2条件下和含10%新生牛血清的DMEM培养基中常规方法进行培养和传代。
试验基因1 (egfp基因)相关结构转染试验:试验于2017年4月于河北农业大学动物遗传育种与繁殖实验室进行。在转染前24 h接种1×105个/ml指数生长期的细胞于24孔板中。培养24 h后细胞汇合度达90%时进行脂质体转染。每孔转染质粒800 ng,其中包括对照质粒pEGFP-N2或每种构建的试验质粒(plxop-EGFP、pEGFP-loxp、ploxp-EGFP-loxp)400 ng和pDsRed2-N1内参质粒400 ng,每组转染设置3个重复,转染24 h后将各转染组置于倒置显微镜下观察各孔内参基因和试验基因1表达情况。
试验基因2 phytase基因相关转染试验:试验于2018年3月于河北农业大学动物遗传育种与繁殖试验室进行。在转染前24 h接种1×105个/mL指数生长期的细胞于24孔板中。培养24 h后细胞汇合度达90%时进行脂质体转染。每孔转染质粒800 ng,其中每种构建的试验质粒(pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-lox、p-SV40-luciferase-CMV-phytase-loxp和pSV40-luciferase-CMV- phytase)各400 ng和pEGFP-N2内参质粒400 ng,另以400 ng pDsRed2- N1+400 ng pEGFP-N2的共转染细胞为酶活性空白对照,每组转染设置3个重复,转染48 h后将各转染组置于倒置显微镜下观察各孔转染内参egfp基因表达情况,并制备酶粗体液做酶活性测定。
1.2.5 试验基因1egfp基因转染结果检测与分析 试验于2017年4月于河北农业大学动物遗传育种与繁殖实验室进行。

A为ploxp-EGFP(loxp序列位于egfp基因开放阅读框中kozak上游的5′非翻译区);B为pEGFP-loxp(loxp序列位于egfp基因开放阅读框终止密码子下游3′非翻译区);C为ploxp-EGFP—loxp(egfp基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列)

A为pSV40-luciferase-CMV-loxp-phytase(loxp序列位于phytase基因开放阅读框中kozak上游的5′非翻译区);B为p-SV40-luciferase-CMV-phytase-loxp(loxp序列位于phytase基因开放阅读框终止密码子下游3′非翻译区);C为pSV40-luciferase-CMV-loxp-phytase-lox(phytase基因开放阅读框的Kozak序列上游5′非翻译区和终止密码子下游3′非翻译区各有一个loxp序列);D为pSV40-luciferase-CMV-phytase(phytase基因上下游非翻译区序列中均添加loxp序列)
1.2.5.1 荧光显微镜观察荧光蛋白基因表达情况 转染细胞前在细胞培养板上用记号笔对每个孔进行划线均分为6个区域,转染24 h后在荧光显微镜下观察并记录所分区域,对各区域egfp与dsred2基因表达情况以及明视野中细胞照相并用Image J软件分析光密度值。整理数据,进行统计学分析。
1.2.5.2 结果的统计与分析 提取每孔中不同区域的红色荧光和绿色荧光的光密度值后,依据所得数据应用SPSS 19.0 统计软件进行一维方差分析,分析方法采用Tukey法,对每转染组的三个重复间以及不同转染组间的两种荧光强度的差异显著性进行分析,来确认loxp序列的位置对外源基因表达的影响。
1.2.6 试验基因2(phytase基因)转染结果检测与分析本试验于2018年3月于河北农业大学动物遗传育种与繁殖实验室进行。
1.2.6.1 荧光显微镜观察绿色荧光蛋白基因表达情况 转染细胞前在细胞培养板上用记号笔对每个孔进行划线均分为6个区域,转染48 h后在荧光显微镜下观察并记录所分区域,对各区域egfp基因表达情况以及明视野中细胞照相并用Image J软件分析光密度值。整理数据,进行统计学分析。
提取每孔中不同区域的绿色荧光光密度值后,依据所得数据应用SPSS 19.0 统计软件进行一维方差分析,分析方法采用Tukey法,对每转染组的三个重复间以及不同转染组间的绿色荧光强度的差异显著性进行分析,来确定各转染间是否存在因转染质粒纯度、细胞活性以及转染操作带来的误差。
1.2.6.2 表达内参基因和试验基因2(phytase基因)表达情况水平分析 转染48 h后,将细胞版中的待测细胞用PBS(pH7.3)洗一次,去除残余液体,加入细胞裂解液200 µL,室温裂解20 min,将细胞裂解物移至1.5 mL离心管中,12 000 r/min 4℃离心5 min去除细胞碎片,称为酶粗提液,然后用酶粗提液测定荧光素酶和植酸酶的活性。
荧光素酶活性测定:参考试剂盒说明书进行,大致过程为:取20 µL酶粗提液,加入底物100 µL,使用LKB公司的化学发光仪Wallac 1250 Luminoneter,输出值为毫伏(mV),测定10秒的发光强度(代表试样的荧光素酶活性)。
植酸酶基因活性测定:采用钼酸显色法进行测定,主要参照程海娜等[31-32]方法进行。颜色/终点混合液:250 mL钼酸铵(100 g·L-1)+ 250 mL钒酸铵(2.35 g·L-1)+ 165 mL 65%硝酸+335 mL水。取100 µL酶粗提液加入100 µL 1. 25 mol·L-1乙酸缓冲液(pH 5. 5)进行稀释,同时设置一个空白对照,即200 µL 1. 25 mol·L-1乙酸缓冲液(pH 5.5)替代酶粗提液稀释液,与其他试样同时进入后续步骤。向稀释的酶粗提液和对照样中加入0.6 mL 1.25 mol·L-1乙酸缓冲液(pH 5.5),混匀,在37℃保温5 min,然后加入1.6 mL的8.4 g·L-1植酸钠,继续保温30 min,加入颜色/终点混合液1.6 mL,以空白对照校正,采用分光光度计在415 nm波长下测定吸光值,记录每试样的吸光值(代表试样植酸酶活性)。
2 结果
2.1 转染用质粒DNA提取
图3显示所提取的转染用质粒质量一致性很好,质粒质量引起的各转染间产生的差异基本可以忽略。
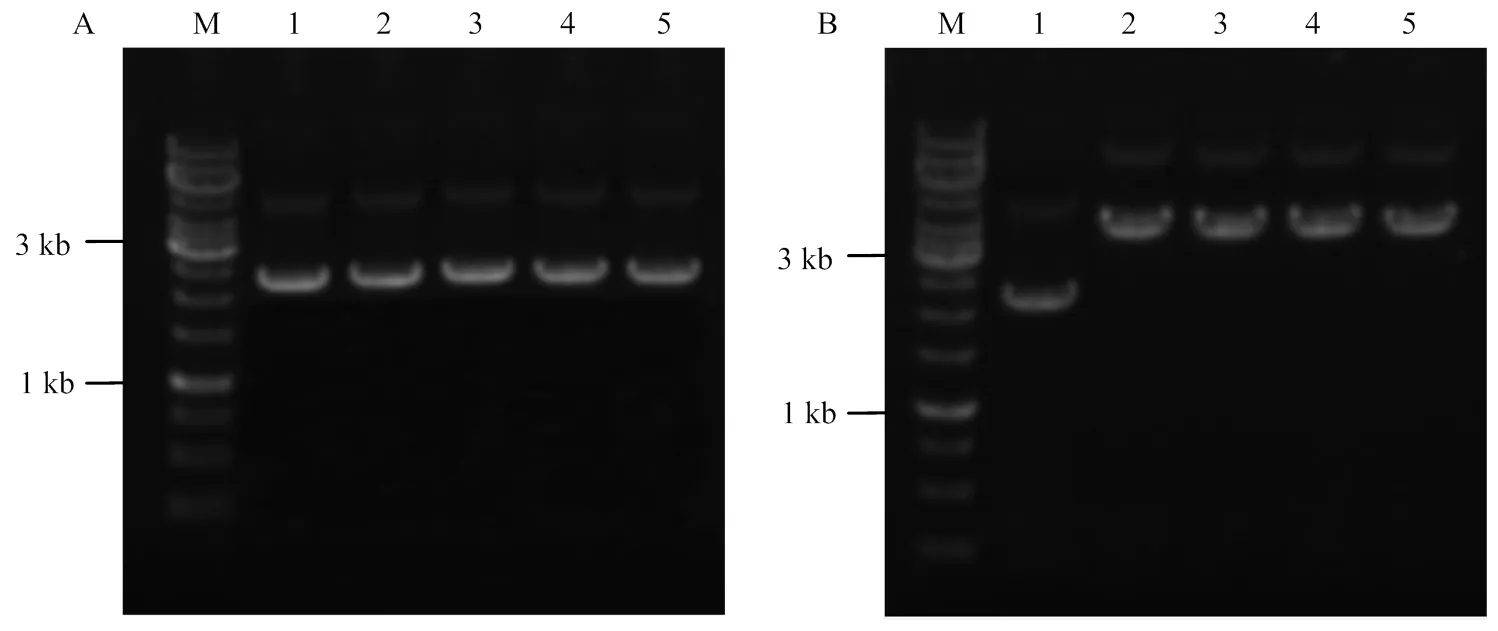
A 为试验基因1(egfp基因)系列质粒电泳结果M为1kb ladder 分子量标准;1为pEGFP-N2质粒;2为ploxp-EGFP质粒, 3为ploxp-EGFP-loxp质粒,4为pEGFP-loxp质粒, 5为pDsRed2-N1质粒;B为试验基因2(phytase基因)系列质粒电泳结果,其中M为1kb ladder 分子量标准,1为pEGFP-N2质粒, 2为pSV40-luciferase-CMV-loxp-phytase质粒, 3为pSV40-luciferase-CMV-loxp-phytase-loxp质粒,4为pSV40-luciferase-CMV- phytase-loxp质粒, 5为pSV40-luciferase-CMV-phytase质粒
2.2 试验基因1(egfp基因)试验
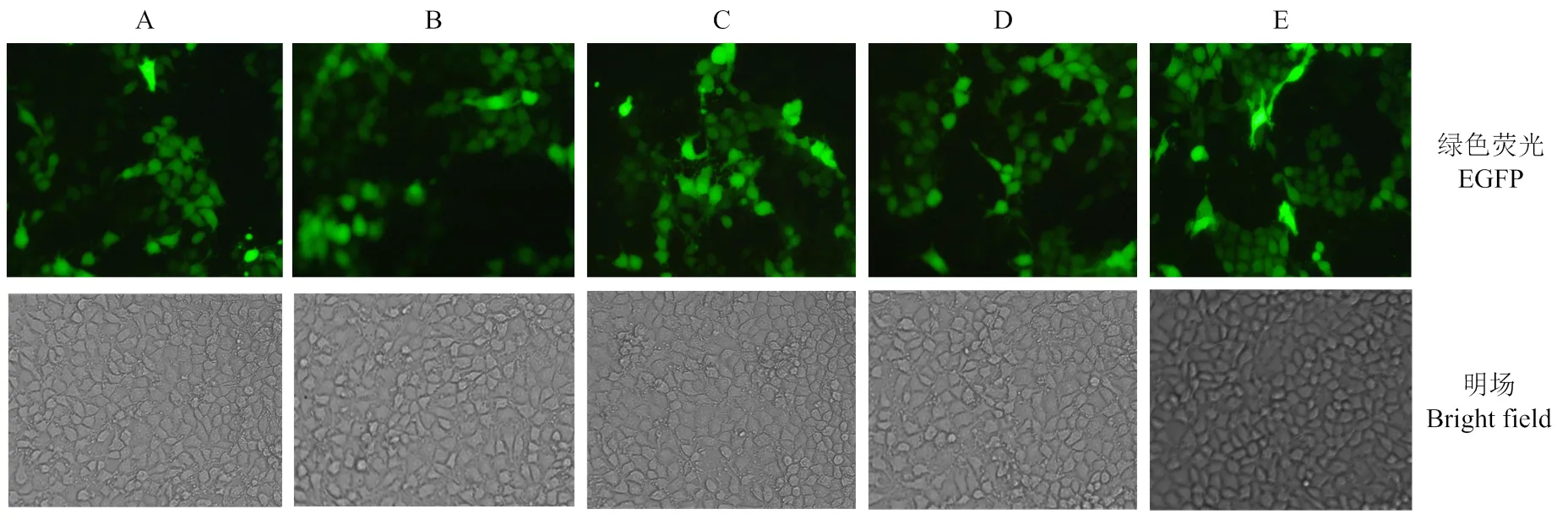
2.2.1 定性分析 图4为对照质粒及不同结构的试验基因1质粒与内参质粒共转染后绿色荧光蛋白和红色荧光蛋白基因表达情况。结果显示,不同转染组中红色荧光蛋白表达基本一致,表明各不同转染组间因细胞活性、转染质粒纯度及转染操作产生的误差不显著,在此情况下,不同loxp位置的质粒的绿色荧光蛋白表达情况不完全一致,pEGFP-loxp与pEGFP-N2转染组的绿色荧光蛋白表达强度基本一致,ploxp- EGFP、ploxp-EGFP-loxp转染组绿色荧光蛋白表达要比pEGFP-N2转染组绿色荧光蛋白表达强度明显低,这一结果表明,loxp序列在基因开放阅读框的下游对基因表达基本无影响,而loxp序列在基因开放阅读框上游时对基因表达呈显著负影响。
2.2.2 egfp基因转染结果分析 图4结果只是对转染结果的定性分析,为了得到更为确切的结论,本研究应用Image J软件对所拍照片的荧光强度进行了提取,得到了每个区域的两种荧光的光密度值(表1)。
依据表1中的数据应用SPSS 19.0 统计软件进一步对每组转染三个重复间及不同转染组间绿色荧光蛋白和红色荧光蛋白表达的差异显著性进行分析(表2)。
由表2可看出,转染孔中红色荧光蛋白平均光密度值组内与组间均差异不显著,排除了转染过程中试验操作误差或不同质粒纯度以及细胞活性差异等对试验结果造成的影响。而转染构建质粒表达的绿色荧光蛋白平均光密度值在4个转染组中不完全相同,转染pEGFP- loxp与pEGFP-N2质粒中绿色荧光蛋白平均光密度值差异不显著(<0.05,<0.01),转染ploxp-EGFP、ploxp-EGFP-loxp质粒中绿色荧光蛋白平均光密度值与转染pEGFP-N2质粒中绿色荧光蛋白平均光密度值有极显著差异(>0.01),表明在转染条件一致的情况下,loxp序列在基因下游位置对基因表达影响不显著,loxp序列在基因上游对基因表达影响极显著,且为负影响。

A.为对照质粒pEGFP-N2与内参质粒pDsRed2-N1共转染;B为试验质粒ploxp-EGFP与内参质粒pDsRed2-N1共转染;C为试验质粒pEGFP-loxp与内参质粒pDsRed2-N1共转染;D为试验质粒ploxp-EGFP-loxp与内参质粒pDsRed2-N1质粒共转染
2.3 试验基因2(phytase基因)转染分析
2.3.1 转染内参基因表达分析 从图5的定性分析结果显示各转染间转染内参egfp基因表达水平差异并不明显,初步显示质粒纯度、细胞活性以及转染操作等误差对试验结果没有显著影响。
依据表3中的数据应用SPSS 19.0 统计软件进一步对每组转染三个重复间及不同转染组间绿色荧光蛋白表达的差异显著性进行分析(表4)。表3和表4的分析表明不同转染间绿色荧光蛋白表达水平差异不显著,从这一分析结果可以认为各转染间由于质粒的纯度、细胞活性差异以及转染操作带来的误差很小,不影响最终得出结论的准确性。
2.3.2 表达内参基因和试验基因表达分析
2.3.2.1 表达内参基因表达测定结果从表5的分析可以看出,除了空白对照组(pDsRed2-N1+ pEGFP-N2)外其他各转染的不同转染间荧光素酶的活性差异不显著(>0.05),而空白对照的三个重复间差异也不显著(>0.05),这一结果进一步说明各转染间因质粒质量、细胞活性以及和转染误差以及酶活测定中的操作误差造成的差异很小可以忽略。
A.为表达对照质粒pSV40-luciferase-CMV -phytase与转染内参质粒pEGFP-N2共转染结果;B为试验质粒pSV40-luciferase-CMV-loxp-phytase与内参质粒pEGFP-N2共转染结果;C为试验质粒pSV40-luciferase-CMV -phytase-loxp与内参质粒pEGFP-N2共转染结果;D为试验质粒pSV40-luciferase-CMV-loxp-phytase-loxp与内参质粒pEGFP-N2质粒共转染结果;E为pDsRed2-N1与pEGFP-N2共转染作为试验基因phytase和表达内参基因luciferase的阴性对照

表1 不同转染三个重复试验的不同区域的两种荧光光密度值
Re:内参质粒pDsRed2-N1;RP:红色荧光平均光密度值;GP:绿色荧光平均光密度值;1.1-1.6:每组转染的第一个平行的6个区域,2.1-2.6:每组转染的第二个平行的6个区域,3.1-3.6:每组转染的第三个平行的6个区域
Re: The internal reference plasmid pDsRed2-N1: RP: the average density of red fluorescent; GP: the average density of green fluorescent; 1.1-1.6: the 6 regions of the first parallel; 2.1-2.6: the 6 regions of the second parallel; 3.1-3.6 : the 6 regions of the third parallel
表2 荧光蛋白平均光密度值差异显著性分析
GP:绿色荧光平均光密度值;RP:红色荧光平均光密度值。同列数字标不同小写字母表示差异显著(<0.05),不同大写字母表示差异极显著(<0.01);Re:内参质粒pDsRed2-N1
GP: The average density of green fluorescent;RP: The average density of red fluorescent. Different superscripts lowercase letters within column indicate significant difference (<0.05), different capital letters within column indicate extremely significant difference(<0.01); Re: Reference plasmid pDsRed2-N1

表3 不同转染三个重复试验的不同区域的绿色荧光光密度值
A:pSV40-luciferase-CMV –phytase+pEGFP;B:pSV40-luciferase-CMV- loxp-phytase+pEGFP;C:pSV40-luciferase-CMV -phytase-loxp+pEGFP- N2;D:pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP;E:pDsRed2- N1+pEGFP-N2;1.1-1.6:每组转染的第一个平行的6个区域,2.1-2.6:每组转染的第二个平行的6个区域,3.1-3.6:每组转染的第三个平行的6个区域
A: pSV40-luciferase-CMV–phytase+pEGFP:B:pSV40-luciferase-CMV- loxp-phytase+pEGFP; C: pSV40-luciferase-CMV-phytase-loxp+pEGFP- N2; D: pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP; E: pDsRed2-N1+pEGFP-N2; 1.1-1.6: the 6 regions of the first parallel; 2.1-2.6: the 6 regions of the second parallel; 3.1-3.6: the 6 regions of the third parallel
2.3.2.2 试验基因1表达测定结果 表6的结果显示试验质粒的转染间植酸酶活性存在差异,试验质粒pSV40-luciferase-CMV-phytase和试验质粒pSV40-luciferase-CMV-phytase-loxp之间植酸酶活性差异不显著,试验质粒pSV40-luciferase-CMV-loxp -phytase和试验质粒pSV40-luciferase-CMV-loxp- phytase-loxp之间差异不显著,而试验质粒pSV40- luciferase-CMV-phytase、pSV40-luciferase-CMV-phytase- loxp与试验质粒pSV40-luciferase-CMV-loxp-phytase、pSV40- luciferase-CMV-loxp-phytase-loxp两两间差极异显著(<0.01),而所有试验质粒植酸酶活性与空白对照间差异都极显著(<0.01)综合表5和表6的结果进一步证实了试验基因1(egfp基因)试验所得出的结论,即loxp序列位于外源基因Kozak序列上游5′非翻译区时对外源基因表达具有负影响,而只在外源基因的3′端终止密码子下游时对外源基因的表达无显著影响。
表4 绿色荧光蛋白平均光密度值差异显著性分析
表中所列数值为绿色荧光平均光密度值;同列数字标不同小写字母表示差异显著(<0.05),不同大写字母表示差异极显著(<0.01)
The value in the table is the average density of green fluorescent;Different lowercase letters within column indicate significant difference (<0.05), different capital letters within column indicate extremely significant difference (<0.01)
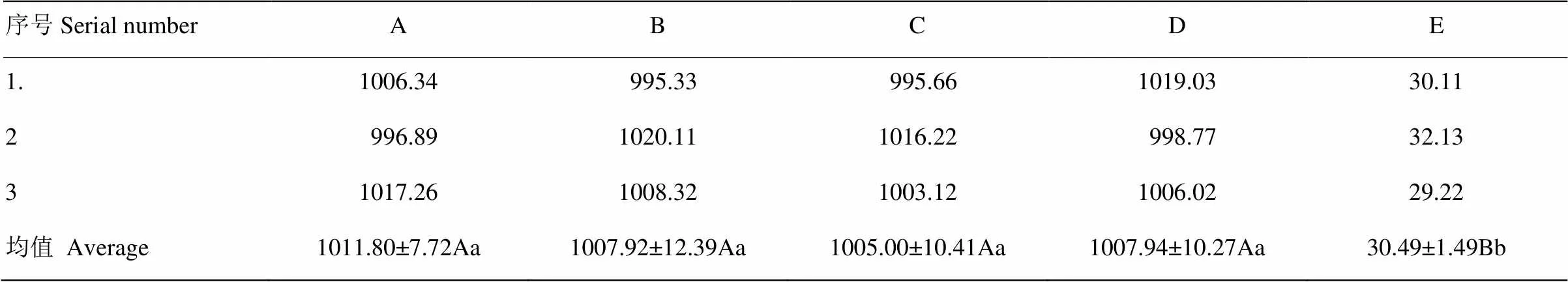
表5 不同转染三个重复试验的荧光素酶活性(mV)测定及统计分析
A: pSV40-luciferase-CMV –phytase+pEGFP:B:pSV40-luciferase-CMV-loxp-phytase+pEGFP;C: pSV40-luciferase-CMV -phytase-loxp+pEGFP-N2; D: pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP; E: pDsRed2-N1+pEGFP-N2; 1是每组转染的第一个重复;2为每个转染的第二个重复;3为每个转染的第三个重复。表中所列数值为每一转染的荧光素酶活性测定值(mV)及统计分析结果;同列数字上标不同小写字母表示差异显著(<0.05),不同大写字母表示差异极显著(<0.01)
A: pSV40-luciferase-CMV –phytase+pEGFP:B:pSV40-luciferase-CMV-loxp-phytase+pEGFP;C: pSV40-luciferase-CMV -phytase-loxp+pEGFP-N2; D: pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP; E: pDsRed2-N1+ pEGFP-N2;1. The first transfection repeat for every experimental plasmid;2. The second transfection repeat for every experimental plasmid;3. The third transfection repeat for every experimental plasmid. The value in the table is the estimated value of luciferase activity(mV) for every transfection and the statistic analysis result; Different superscripts lowercase letters within column indicate significant difference (<0.05),different capital letters within column indicate extremely significant difference(<0.01)

表6 不同转染三个重复试验的植酸酶活性(吸光值)测定及统计分析
A: pSV40-luciferase-CMV –phytase +pEGFP:B:pSV40-luciferase-CMV-loxp-phytase+pEGFP;C: pSV40-luciferase-CMV -phytase-loxp+pEGFP-N2; D: pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP; E: pDsRed2-N1+pEGFP-N2; 1是每组转染的第一个重复;2为每个转染的第二个重复;3为每个转染的第三个重复;表中所列数值为每一转染的植酸酶活性测定值(吸光值)及统计分析结果;同列数字标不同小写字母表示差异显著(<0.05),不同大写字母表示差异极显著(<0.01)
A: pSV40-luciferase-CMV –phytase+pEGFP:B:pSV40-luciferase-CMV-loxp-phytase+pEGFP;C: pSV40-luciferase-CMV -phytase-loxp+pEGFP-N2; D: pSV40-luciferase-CMV-loxp-phytase-loxp+pEGFP; E: pDsRed2-N1+pEGFP-N2;1. The first transfection repeat for every experimental plasmid;2. The second transfection repeat for every experimental plasmid;3. the third transfection repeat for every experimental plasmid. The value in the table is the estimated value of luciferase activity (mV) for every transfection and the statistic analysis result;Different lowercase letters within column indicate significant difference (<0.05), different capital letters within columnindicate extremely significant difference (<0.01)
3 讨论
mRNA作为蛋白质合成的模板指导蛋白质的合成。虽然mRNA为单链分子,但由于其局部互补配对在生理条件下可互补配对形成二级结构,这种二级结构的形成对mRNA 指导蛋白质的合成效率的影响是多方面的。首先,在mRNA指导蛋白质合成过程中需要打开这些二级结构才能阅读密码子进行正常翻译,因此大量的二级结构的存在势必会减慢蛋白质合成的速度。尤其是在mRNA的5′非翻译区大量的RNA二级结构的存在会影响蛋白质合成的起始,5′非翻译区的二级结构对蛋白质合成效率的影响可能更为强烈[33]。对酵母全基因组水平的研究表明,起始密码子周围-40—+40 nt范围内高含量的二级结构对基因翻译的确存在一定的抑制作用,同时mRNA 5′非翻译区及起始密码子下游40nt的序列中二级结构的存在不利于mRNA出核[34],而成熟的mRNA出核是其进行蛋白质合成前提条件。这一研究似乎表明mRNA 5′非翻译区中RNA二级结构的存在对蛋白质具有更多不利的影响,人们对小鼠[35]、斑马鱼[36]和拟南芥[37]的研究也发现了同样的规律,即多数基因5′非翻译区二级结构相对含量比较低,这表明这一现象在进化上具有一定的保守性,而mRNA3′非翻译区在酵母和拟南芥中二级结构含量相对较低,而在小鼠、斑马鱼中却相对较高这似乎说明mRNA 3′非翻译区的二级结构含量保守性较低,因此对基因表达效率的影响并不重要。然而对小鼠胚胎干细胞mRNA二级结构的研究[38],却发现这种细胞类型中mRNA的5′非翻译区和3′非翻译区的二级结构含量都相对较高,这可能与小鼠胚胎干细胞中表达的有些基因5′非翻译区中某些二级结构有特殊的生物学功能如屏蔽micoRNA作用位点等有关。因此从上述分析中可知在一个基因的5′或(和)3′非翻译区添加一个反向重复序列其效应需要进行评价。
Cre/loxp删除系统中loxp序列基本结构为长34 bp,中间是8 bp的核心序列,两边是13 bp的反向重复序列。当Cre 重组酶识别loxp 位点后,可以在该位置切断DNA,并在断端形成中间体,在Cre 重组酶催化下可重组连接。因此,同一条DNA 分子上若有两个loxp位点,且方向相同,重组后则loxp位点间的核酸序列被特异性删除,并在原位点留置一个loxp序列,如果以Cre/loxp系统作为报告基因删除系统通过基因打靶将外源基因定点整合到内源基因的上游或下游并通过2A序列链接形成共表达结构,会在融合表达结构的上游或(和)下游非翻译区留置一个loxp序列。当基因表达时转录产生的mRNA分子中上游或(和)下游会存在一个loxp序列,该序列会形成一个13 bp长的发卡型二级结构,该二级结构对外源基因的影响需要进行评估以便为上述设想的研究中打靶位点的选择提供依据。
本研究先选择egfp基因作为试验基因1,开展了研究结果初步显示loxp序列位于外源基因上游时对基因表达形成了强烈的抑制作用,而在外源基因下游时对外源基因的表达没有显著的影响,为了进一步证实试验基因1(egfp基因)的结果,笔者选用phytase基因作为试验基因2进一步验证试验基因1的结果,试验基因2(phytase基因)的结果和试验基因1(egfp基因)的结果基本一致。据此可以认为loxp序列位于基因上游时对基因表达有强烈的抑制作用,而在外源基因终止密码子下游时对外源基因的表达没有显著的影响。
分析产生这一现象的原因在于Cre/loxp结构是感染大肠杆菌的P1噬菌体的基因和序列,loxp序列在真核生物细胞基因组中并不存在,因此真核细胞中不会有针对loxp序列的相应的调控因子,因此loxp经转录出现在成熟mRNA的5′非翻译区时,不会有类似屏蔽micoRNA结合位点或被其他反式调控因子识别增强蛋白质翻译的作用,其自身形成的发卡结构会使核糖体的翻译效率下降甚至提前脱落,而因此其作用只有抑制了核糖体的翻译起始过程,从而导致外源基因表达下降;而loxp序列在基因开放阅读框下游的3′非翻译区内时其序列形成的发卡结构在外源基因终止密码子之后,即核糖体在翻译完成后才遇到相应的二级结构,因此该二级结构对蛋白翻译效率没有明显影响。这一结果也和前人对真核生物mRNA二级结构对基因表达的影响的研究结果基本一致。
4 结论
在开展通过基因打靶技术将外源基因与内源基因通过2A序列连接,实现外源基因和内源基因共表达的转基因动物制作研究中,以Cre/loxp系统作为报告基因删除工具时,则内源基因的下游为最佳的打靶位点。
[1] BERTOLINI L R, MEADE H, LAZZAROTTO C R, MARTINS L T, TAVARES K C, BERTOLINIM, MURRAY J D. The transgenic animal platform for biopharmaceutical production., 2016 25(3): 329-343.
[2] ShIMATSU Y, YAMADA K, HORII W, HIRAATAK A, SAKAMOTO Y, WAKI S, SANO J, SAITOH T, SAHARA H, ShIMIZU A, YAZAWA H, SACHS D H, NUNOYA TProduction of cloned NIBS (Nippon Institute for Biological Science) and α-1, 3-galactosyltransferase knockout MGH miniature pigs by somatic cell nuclear transfer using the NIBS breed as surrogates., 2013, 2(3): 157-164.
[3] GOLOVAN S P, MEIDINGER R G, AAKAIYE A, COTTILL M, WIEDERKEHR M Z, BARNEY D J, PLANTE C, POLLARD J W, FAN M Z, HAYES M A, LAURSEN J, HJORTH J P, HACKER R R, PHILLIPS J P, FORSBERG C W. Pigs expressing salivary phytase produce low-phosphorus manure., 2001, 19(8): 741-745.
[4] DAMAK S, SU H, JAY N P, BULLOCK D W. Improved wool production in transgenic sheep expressing insulin-like growth factor 1., 1996, 14(2): 185-188.
[5] SAEKY K, MATSUMOTO K, KINOSHITA M, SUZUKI I, TASAKA Y, KANO K, TAGUCHI Y, MIKAMI K, HIRABAYASHI M, KASHIWAZAKI N, HOSOI Y, MURATA N, IRITANI A. Functional expression of a Delta12 fatty acid desaturase gene from spinach in transgenic pigs., 2004, 101(17): 6361-6366.
[6] JUNE BYUN S, YUK S S, JANG Y J, CHOI H, JENO M H, ERDENE-OCHIR T O, KWON J H, NOH J Y, SUN KIM J, GYU YOO J, SONG C S. Transgenic chickens expressing the 3D8 single chain variable fragment protein suppress avian influenza transmission., 2017, 7(1): 5938.
[7] ROCHA-MARTINS M, CAVALHEIRO G R, MATOS-RODRIGUES G E, MARTINS R A. From gene targeting to genome editing: Transgenic animals applications and beyond.2015, 87(2 Suppl): 1323-1348.
[8] ROBERTS B, DITULLIO P, VITALE J, HEHIR K, GORDON K.. Cloning of the goat beta-casein-encoding gene and expression in transgenic mice.1992. 121(2), 255-262.
[9] ADAMS V, KRANKEL N, KAUFFOLD H, HAMBRECHT R, SCHULTHEISS H, SCHULER G, WALTHER T. Is the MLC1/3 promoter / enhancer the right system to generate skeletal muscle specific transgenic animals?, 2004(8): 157-161.
[10] 孔庆然, 刘忠华.外源基因在转基因动物中遗传和表达的稳定性遗传, 2011, 33(5): 504-511.
KONG Q R, LIU Z H. Inheritance and expression stability of transgene in transgenic animals., 2011, 33(5): 504-511. (in Chinese)
[11] 于政权樊宝良戴蕴平郑敏牛慧玲王美丽王丽丽费菁李宁. 转基小鼠乳腺表达重组人溶菌酶. 科学通报, 2003, 48(20): 2149-2153.
YU Z Q, FAN B L, DAI Y P, ZhENG M, NIU H L, WANG M L, WANG L L, FEI J, LI N. 2003. Expression of recombinant human lysozyme in the milk of transgenic mice., 2003, 48(20): 2149-2153. (in Chinese)
[12] TAN W, PROUDFOOT C, LILLICO S G, WHITELAW C B A. GENE targeting, genome editing: from Dolly to editors., 2016, 25(3): 273-287.
[13] 祁伟彦, 孙鹤, 周妮, 朱莉, 郎志宏, 黄大昉. 抗虫/耐草甘膦双价融合基因表达载体的构建及烟草转化. 生物技术通报, 2011, 10: 114-119.
QI W Y, SUN H, ZHOU N, ZhU L, LANG Z H, HUANG D F. Construction of Co-ordinated expression vectors with insect resistance /glyphosate tolerance and transformation of tobacco.2011, 10: 114-119. (in Chinese)
[14] SEDIVY J M, SHARP P A. Positive genetic selection for gene disruption in mammalian cells by homologous recombination., 1989, 86(1): 227-231.
[15] DONOHO G, JASIN M, BERG P. Analysis of gene targeting and intrachromosomal homologous recombination stimulated by genomic double-strand breaks in mouse embryonic stem cells., 1998, 18(7): 4070-4078.
[16] PORTEUS M H, BALTIMORE D. Chimeric nucleases stimulate gene targeting in human cells., 2003, 300(5620): 763.
[17] MILLER J C, HOLMES M C, WANG J, GUSCHIN D Y, LEE Y L, RUPNIEWSKI I, BEAUSEJOUR C M, WAITE A J, WANG N S, KIM K A, GREGORY P D, PABO C O, REBAR E J. An improved zinc-finger nuclease architecture for highly specific genome editing., 2007, 25(7): 778-785.
[18] HOCKEMEYER D, WANG H, KIANI S, LAI C S, GAO Q, CASSADY J P, COST G J, ZhANG L, SANTIAGO Y, MILLER J C, ZEITLER B, ChERONE J M, MENG X, HINKLEY S J, REBAR E J, GREGORY P D, URNOV F D, JAENISCH R. Genetic engineering of human pluripotent cells using TALE nucleases., 2011, 29(8): 731-734.
[19] TESSON L, USAL C, MENORET t S, LEUNG E, NILES BJ, REMY S, SANTIAGO Y, VINCENT A I, MENG X, ZhANG L, GREGORY P D, ANEGON I, COST G J. Knockout rats generated by embryo microinjection of TALENs.2011, 29(8): 695-696.
[20] CONG L, RAN FA, COX D, LIN S, BARRETTO R, HABIB N, HSU P D, WU X, JIANG W, MARRAFFINI LA, ZhANG F. Multiplex genome engineering using CRISPR/Cas systems., 2013, 339(6121): 819-823.
[21] MALI P, YANG L, ESVELT K M, AACH J, GUELL M, DICARLO J E, NORVILLE J E, ChURCH G M. RNA-guided human genome engineering via Cas9., 2013, 339(6121): 823-826.
[22] PORTEUS M H, CARROLL D. Gene targeting using zinc finger nucleases.. 2005 23(8): 967-73.
[23] WU H, WANG Y, ZHANG Y, YANG M, LV J, LIU J, ZHANG Y. TALE nickase-mediated SP110 knockin endows cattle with increased resistance to tuberculosis., 2015, 112(13): 1530-1539.
[24] 王敏, 石翾, 黄翔, 刘小凤, 覃玉凤, 刘小红, 陈瑶生, 何祖勇.应用RGS 双荧光替代性报告载体提高CRISPR/Cas9对猪BMP15基因的打靶效率. 遗传, 2017, 39(1): 48-55.
WANG M, SHI H, HUANG X, LIU X F, TAN Y F, LIU X H, CHEN Y S, HE Z Y. 2017 Improving gene targeting efficiency on the porcine BMP15 gene mediated by CRISPR/Cas9 by using the RGS surrogate reporter system.2017, 39(1): 48-55. (in Chinese)
[25] BELTEKI G, HAIGH J, KABACS N, HAIGH K, SISON K, COSTANTINI F, WHITSETT J, Quaggin S E, NAGY A. Conditional and inducible transgene expression in mice through the combinatorial use of Cre mediated recombination and tetracycline induction.2005, 33(5): e51.
[26] 兰翀, 任丽娜, 吴敏, 刘思国, 刘国辉, 徐旭俊, 陈建泉, 马恒东, 成国祥.利用Cre/LoxP系统删除转基因山羊体内的选择标记基因.生物工程学报, 2013, 29(12): 1847-1854.
Lan C, REN L N, WU M, LIU S G, LIU G H, XU X J, CHEN J Q, MA H D, CHENG G X. Deletion of marker gene in transgenic goat by Cre/LoxP system.2013, 29(12): 1847-1854. (in Chinese)
[27] ZOU X, PENG A, XU L, LIU X, LIE T, YAO L, HE Y, CHEN S. Efficient auto-excision of a selectable marker gene from transgenic citrus by combining the Cre/loxP system and ipt selection., 2013, 32(10): 1601-1613.
[28] SEGEV N, COHEN G. Control of circularization of bacteriophage P1 DNA in., 1981, 30;114(2): 333-342.
[29] WU Y1, H E Y, ZHANG H, DAI X, ZHOU X, GU J, WANG G, ZHU JJ. A stringent dual control system overseeing transcription and activity of the Cre recombinase for the liver-specific conditional gene knock-out mouse model., 2008, 35(7): 431-439.
[30] GAVERIAUX-RUFF CL, KIEFFER B L. Conditional gene targeting in the mouse nervous system: Insights into brain function and diseases., 2007;113(3): 619-634.
[31] 程海娜, 莫湘涛, 陈宇, 谭海滨, 周雄, 夏立秋. 产植酸酶菌株的筛选及产酶条件的研究. 生命科学研究, 2002, 6(4): 343-346.
CHENG H N, MO X T, CHEN Y, TAN H B, ZHOU X, XIA L Q. Studies on screening of strains producing phytase and conditions of producing phytase., 2002, 6(4): 343-346. (in Chinese)
[32] 王金华, 穆跃林, 黄遵锡基因工程菌产植酸酶(phytA)的纯化及性质初步研究. 云南师范大学学报, 2003, 23(1): 43-47.
WANG J H, MU Y L, Huang Z X. Purification and characterization of phytase (PhytA) from genetic engineering barm.2003, 23(1): 43-47. (in Chinese)
[33] MAKRIDES S C. Components of vectors for gene transfer and expression in mammalian cells., 1999, 17(2): 183-202.
[34] KERTESZ M, WAN Y, MAZOR E, RINN J L, NUTTER R C, CHANG H Y, SEGAL E. Genome-wide measurement of RNA secondary structure in yeast., 2010, 467(7311): 103-107
[35] SHABALINA S A, OGURTSOV A Y, SPIRIDONOV N A. A periodic pattern of mRNA secondary structure created by the genetic code., 2006, 34(8): 2428-2437.
[36] KAUSHIK K, SIVADAS A, VELLARIKKAL S KVERMA A, JAYARAJAN R, PANDEY S, SETHI T, MAITI S, SCARIA VSIVASUBBU S. RNA secondary structure profiling in zebrafish reveals unique regulatory features., 2018, 19(1): 147.
[37] DING Y, TANG Y, KWOK C K, ZHANG Y, BEVILACQUA P C, ASSMANN S M.genome-wide profiling of RNA secondary structure reveals novel regulatory features., 2014, 505(7485): 696-700. doi: 10.1038/2013 Nov 24.
[38] INCARNATO D, NERI F, ANSELMI F, OLIVIERO S. Genome-wide profiling of mouse RNA secondary structures reveals key features of the mammalian transcriptome., 2014, 15(10): 491.
(责任编辑 林鉴非)
Researching on the Effect of the Location of Loxp Sequence on the Gene
XU YingYing, JIN Wei, DAI MinMin, FAN BaoLiang
(College of Animal Sciences, Hebei Agricultural University, Baoding 071001, Hebei)
【Objective】In order to get a solid foundation for the site selection of a gene targeting experiment which aimed to realize the co-expression of the foreign gene and the endogenous gene linked through 2A sequence, the effect on the gene expression of different location of the loxp sequence at the open reading frame of a gene need to be clarified when the Cre/loxp system was selected as a tool for reporter gene deletion. 【Method】The enhanced green fluorescent protein (egfp) gene was selected as experimental gene, and red fluorescent protein (dsred2) gene as internal reference gene. Based on pEGFP-N2, pGEM-5zf-loxp plasmid, three plasmids with different location of loxp sequence at the open reading frame of egfp gene named ploxp-EGFP (The loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region of egfp gene), pEGFP-loxp (The loxp sequence located at the downstream of the termination codon in the 3′un-translation region of egfp gene) and ploxp-EGFP-loxp (There is one loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region and another downstream of the termination codon in the 3′un-translation region of egfp gene) were constructed. As the internal reference, Plasmid pDsRed2-N1 was co-transfected with constructed plasmids into PK15 cells. Twenty-four hours later, the fluorescence intensity of every transfection was observed and analyzed using fluorescence microscope and software Image J. In order to verify the result obtained using egfp gene as experimental gene, phytase gene was selected as another experimental gene. Based on pIREsNeo, pGEM-5zf-loxp, pT-phytase, pGL4.13[luc2/SV40] plasmid and luciferase gene as expression internal reference gene four plasmids with different location of loxp sequence at the open reading frame of phytase gene named p-SV40-luciferase-CMV-loxp-phytase (The loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region of phytase gene, p-SV40-luciferase-CMV-loxp-phytase-loxp (There is one loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region and another downstream of the termination codon in the 3′un-translation region of phytase gene), p-SV40-luciferase-CMV-phytase-loxp (The loxp sequence located at the downstream of the termination codon in the 3′un-translation region of phytase gene), p-SV40-luciferase-CMV-phytase (There is no loxp sequence located at the upstream of the Kozak sequence in the 5′un-translation region and another downstream of the termination codon in the 3′un-translation region of phytase gene)were constructed. As transfection internal reference, pEGFP-N2 was co-transfection with the differenct plasmid constructed above into PK15 cells, pEGFP-N2 and pDsRed2-N1 were co-transfected into PK15 cells to work as blank control for enzyme activity of phytase and luciferase. Forty-eight hours later, the fluorescence intensity of every transfection was observed and analyzed using fluorescence microscope and software Image J. These data were analyzed using statistics software SPSS19.0. At the same time, the activity of phytase and luciferase of every transfection were measured by Molybdenum Blue Method or using Firefly Luciferase Assay Kit, and these data were analyzed using statistics software SPSS19.0.【Result】In the experiment, thegene worked as internal reference gene. Statistics analysis using the average value of red fluorescence of different area of every transfection as basic data shows there is no significant difference between every different transfection. This result shows there is no significant difference in purity of plasmid, handling and cell activity between every transfection. Plasmid electrophoresis result shows there is no significant difference in quality between every plasmid used in transfections, this means the difference in quality between every plasmid used in every transfection is not enough to influence the accuracy for analysis. Analysis on the green fluorescence in the same rule shows there is no significant difference between different transfection of the same plasmid and between pEGFP-N2 and pEGFP-loxp, ploxp-EGFP and ploxp-EGFP-loxp, but there is significant difference between pEGFP-N2, pEGFP-loxp and ploxp-EGFP ploxp-EGFP-loxp. These result shows loxp sequence had no effect on egfp gene expression when it is located downstream of the open reading frame of the gene, but had a negative effect on egfp gene expression when it located upstream of the open reading frame of the gene. Replacing the egfp gene bygene and using egfp gene as transfection internal reference gene, luciferase gene as expression internal reference gene, it shows a same result.【Conclusion】These results indicate that if we try to prepare a transgenic animals in which the foreign gene and the endogenous gene is linked by 2A sequence and co-express under the regulation of the full regulation element of the endogenous gene in one copy manner and the Cre/loxp system is selected as a tool for reporter gene deletion, the downstream of the open reading frame of the endogenous gene is the suitable targeting site.
loxp sequence; gene expression; 2A sequence; gene targeting
2017-10-09;
2018-05-04
转基因重大专项(2014ZX08006-005)、国家自然科学基金(30972081)、河北省现代农业产业技术体系蛋肉鸡产业创新团队遗传资源开发与利用岗位项目(HBCT2018150201)、河北省科技支撑计划项目(14236602D-4(2014))
许盈盈,E-mail:289637781@qq.com。
樊宝良,Tel:13513125768;E-mail:fanbl119@vip.sina.com
10.3864/j.issn.0578-1752.2018.13.013

