陆地棉CRK基因家族的鉴定及其表达分析
张中起,王娇,2,靳炜,葛冬冬,刘康,吕芬妮,孙敬
陆地棉CRK基因家族的鉴定及其表达分析
张中起1,王娇1,2,靳炜1,葛冬冬1,刘康1,吕芬妮1,孙敬1
(1南京农业大学作物遗传与种质创新国家重点实验室,南京 210095;2太原理工大学,太原 030024)
【目的】富含半胱氨酸类受体激酶(CRK)是植物中最大的类受体激酶家族之一,在植物生长发育、激素信号传导和抗逆境胁迫中发挥重要作用。从全基因组水平鉴定陆地棉CRK基因家族并进行生物信息学和表达模式分析,为研究和利用陆地棉CRK基因家族奠定基础。【方法】从Pfam数据库下载stress-antifung结构域氨基酸序列,应用BLASTp程序搜索棉花基因组数据库,鉴定棉花CRK基因家族;利用Compute/Mw tool、SignalP、TMHMM Server V2.0、WoLF POSRT等在线工具预测陆地棉CRK家族蛋白的分子量、信号肽、跨膜结构域和亚细胞定位等;用ClustalX1.8软件对棉花和拟南芥CRK蛋白质进行氨基酸序列比对,MEGA5.0分析棉花和拟南芥CRK蛋白的系统进化关系;使用TBtools制作陆地棉CRK基因家族的染色体定位、基因结构和蛋白质结构域示意图;应用植物顺式调控元件数据库PlantCARE分析棉花启动子序列;通过植物磷酸化位点数据库PlantPhos预测陆地棉CRK家族蛋白的磷酸化位点;从NCBI数据库下载RNA-Seq数据,利用转录组定量工具Kallisto计算TPM值,通过在线工具Morpheus绘制陆地棉CRK家族基因表达热图。【结果】陆地棉基因组中有70个CRK基因,分布于14条染色体,其中52个基因(74.3%)集中串联成簇分布于A6/D6、A9/D9、A10/D10染色体,且在A/D染色体组之间呈现高度共线性关系。编码302—901个氨基酸,58个蛋白质(82.9%)具有跨膜结构域,主要定位于叶绿体、质膜和胞外。磷酸化位点预测结果表明,陆地棉和拟南芥CRK有5个相同的磷酸化位点基序,包括3种丝氨酸磷酸化位点基序和2种苏氨酸磷酸化位点基序。65个陆地棉CRK基因的启动子区(92.9%)至少含有一种逆境激素响应元件,69个基因启动子区(98.6%)至少含有一种生物或非生物胁迫响应元件。根据RNA-Seq数据分析结果,陆地棉CRK基因可分为3种不同的组织表达特征类型;盐、干旱、冷、热胁迫以及接种大丽轮枝菌均可以导致部分陆地棉CRK基因表达水平的改变。在根、茎、叶和胚珠中优势表达,在纤维中几乎不表达,ABA、GA3、SA、PEG-6000、氯化钠和大丽轮枝菌Vd991处理均能刺激迅速上调表达。应用病毒诱导的基因沉默技术(VIGS)沉默GhCRK25可导致棉花对大丽轮枝菌Vd991更为敏感。【结论】陆地棉CRK基因家族有70个成员,具有保守的基因结构和功能结构域,多样化的组织表达特征,大多数基因受激素和逆境调控。
陆地棉;富含半胱氨酸蛋白激酶;基因家族;抗逆性;
0 引言
【研究意义】富含半胱氨酸类受体激酶(cysteine- rich receptor-like kinase,CRK)是植物中最大的类受体激酶(receptor-like kinase,RLK)家族之一,在植物生长发育、激素信号传导、非生物胁迫和病原体防御以及细胞过敏性死亡中发挥至关重要的作用。利用生物信息学的方法研究棉花CRK基因家族的成员数量、染色体分布、基因结构和进化关系、时空表达特征以及对非生物和生物胁迫的响应等,对于研究棉花CRK基因的功能并应用于棉花的遗传改良具有重要的意义。【前人研究进展】CRK是广泛存在于动物和植物中的一类膜受体蛋白,具有1个N端信号肽、1个跨膜结构域、1个C端的胞内丝氨酸/苏氨酸蛋白激酶结构域(Ser/Thr protein kinase domain)以及1个通常由1—4个拷贝的strss-antifung结构域构成的胞外结构域(extracellular domain)[1]。拟南芥受病原菌和病原/微生物相关分子模板(pathogen/microbe associated molecular pattern,PAMP/MAPMP)诱导强烈表达,在免疫反应中相互协同作用增强防御病原菌反应[2-4]。过表达、、或均可增强拟南芥防御病原菌早期和中期的PTI(PAMP triggered immunity)反应[5-7]。过表达拟南芥受丁香假单胞菌DC3000侵染后积累更多的胼胝质从而增强了晚期PTI反应[6-7]。N端糖基化的AtCRK4参与AtBAK1/AtSERK4调控的细胞过敏性死亡,触发特异性防御反应ETI(effector triggered immunity)[8-9]。过表达或拟南芥受丁香假单胞菌DC3000侵染后,迅速引起细胞过敏性死亡,诱导病原菌防御相关基因、和上调表达,同时加速内源水杨酸(SA)积累[10-12]。AtCRK28与AtBAK1相互作用激活免疫受体复合物FLS2/AtBAK1,加速胞外ROS爆发进而诱导细胞死亡,增强转基因拟南芥对丁香假单胞菌DC3000的抗性[13]。菜豆受病原菌诱导后显著上调表达[14];沉默降低小麦对小麦纹枯病菌的抗性[15];OsCRK6和OsCRK10与OsNPR1互作介导水稻对多种病原菌的广谱性免疫反应[16]。但是,瞬时沉默增强大麦表皮细胞对大麦白粉病菌f. sp的抗性而不影响R基因介导的抗病途径[17];拟南芥突变体受丁香假单胞菌DC3000侵染后,加速胞外ROS爆发,诱导细胞过敏性死亡,抑制病原菌的生长[18];这些研究表明CRKs在植物免疫抗病中起重要调控作用,但是不同CRK的功能和作用方式可能不同。拟南芥CRK还参与对干旱、盐、臭氧(O3)、紫外光(UV)等非生物胁迫的应答[2-3,5,10,12,19]:过表达增强转基因拟南芥对紫外线的适应性[20];拟南芥突变体对ABA不敏感,在盐胁迫下具有更高的发芽率和良好的生长状态,过表达增强拟南芥对干旱胁迫的耐受性[21]。此外,CRKs还参与生长和发育的形态建成过程:过表达影响拟南芥的开花和荚果发育[13];过表达拟南芥抽薹延迟了5 d[21];番茄是花粉中特异表达的基因[22]。【本研究切入点】富含半胱氨酸类受体激酶,作为具有众多家族成员、对于植物生长发育和抗逆性具有重要功能的植物类受体激酶中的一大类,其在棉花基因组中的分布、表达及其功能迄今尚未见报道。随着四倍体栽培棉种基因组测序的完成[23-25],为利用模式生物的CRK基因信息发掘棉花CRK基因提供了条件。【拟解决的关键问题】本研究采用生物信息学分析方法,分析棉花CRK基因家族的成员数量、染色体定位、进化及其分类;预测编码蛋白质的分子结构和生理生化特性;分析CRK基因的时空和胁迫表达模式,为进一步研究棉花CRK基因家族的功能,发掘CRK基因并应用于棉花品种的遗传改良提供基础信息。
1 材料与方法
1.1 材料
植物试验材料为陆地棉遗传标准系TM-1,由南京农业大学作物功能组学实验室连续自交保存,开花后-1、0、1、3、5、15和25 d花蕾(铃)采自南京农业大学棉花育种圃,剥取胚珠之后液氮速冻-80℃保存。试验用根、茎、叶采自温室生长三叶期棉花幼苗。一周龄的MS培养基培养的棉花无菌苗用于激素、氯化钠、PEG-6000和大丽轮枝菌Vd991处理:ABA、GA3、SA、NaCl、PEG-6000处理浓度分别为0.1 mmol×L-1、0.1 mmol×L-1、2.5 mmol×L-1、200 mmol×L-1、20%(w/v),大丽轮枝菌接种用5×106cfu/mL Vd991孢子悬浮液浸泡无菌苗根30 s后,移入液体MS培养基中,培养条件为25℃、光照16 h/黑暗8 h、光照强度2 000 lx,分别于处理0、1、3、6、12、24和48 h后取棉苗根部,3次生物学重复,液氮速冻后-80℃保存备用。
1.2 棉花CRK基因家族的鉴定
以Pfam(http://pfam.sanger.ac.uk/)数据库下载的stress-antifung(PF01657)和Pkinase_Tyr (PF07714)种子序列为探针,用BLASTp为引擎分别搜索陆地棉和海岛棉的基因组数据库(E-value=1e-5)获得同源序列。剔除冗余序列后,分别利用Pfam和SMART(http://smart.embl-heidelberg.de)对所有候选的棉花CRK蛋白质序列进行结构域鉴定,保留同时含有stress-antifung和STKc_IRAK结构域的序列,最终获得棉花CRK基因家族。
1.3 棉花CRK基因家族编码蛋白质的分子结构和生化特性分析
利用Compute/Mw tool(https://web.expasy. org/compute_pi/)预测陆地棉CRK蛋白质的分子质量和等电点。通过SignalP(http://www.cbs.dtu.dk/services/ SignalP/)预测棉花CRK蛋白序列中潜在的信号肽位点(D-cutoff value>0.5)。利用TMHMM Server V2.0 (http://www.cbs.dtu.dk/services/TMHMM/)预测棉花CRK蛋白的跨膜结构。采用WoLF POSRT tool(https://www.genscript.com/psort.html)预测棉花CRK蛋白的亚细胞定位。
1.4 棉花CRK基因家族的系统进化分析
使用ClustalX1.8软件对陆地棉、海岛棉和拟南芥CRK蛋白质进行氨基酸序列比对,通过MEGA 5.0软件构建系统发育树:采用邻接法(Neighbor-Joining algorithm),泊松校正(Poission correction),成对删除(pairwise deletion),Bootstrap重复值1 000次。参考拟南芥CRK基因家族的亚族分类结果对系统发育进化树中棉花CRK基因家族进行亚族分类。
1.5 陆地棉CRK基因家族的基因结构、蛋白质结构域和染色体定位分析
通过棉花基因组数据库(https://www.cottongen. org/)查询棉花CRK基因家族的染色体位置信息,利用系列工具软件TBtools(https://github.com/CJ-Chen/ TBtools)中基因结构绘制工具、蛋白功能结构域绘制工具和基因定位工具绘制陆地棉CRK基因结构图、CRK蛋白功能结构域图和CRK基因家族的染色体定位图。
1.6 陆地棉CRK基因家族的启动子分析
从陆地棉全基因组数据库中提取每个陆地棉CRK基因起始密码子上游1 500 bp基因组序列搜索植物启动子顺式作用元件,在线数据库PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测顺式作用元件。
1.7 陆地棉CRK家族蛋白磷酸化位点分析
利用在线网站motif-X(http://motif-x.med.harvard. edu/motif-x.html)并以拟南芥磷酸位点数据库(IPIProteome)为背景分析拟南芥CRK家族蛋白磷酸化位点motif。通过植物磷酸化位点数据库PlantPhos(http://csb.cse.yzu.edu.tw/PlantPhos/Predict. php)预测陆地棉CRK家族蛋白的磷酸化位点,利用在线网站motif-X并以本地上传的P3DB数据库为背景分析预测棉花CRK磷酸化位点motif。
1.8 基于RNA-seq数据库分析陆地棉CRK基因的表达模式
从NCBI Sequence Read Archive数据库(https: //www. ncbi.nlm.nih.gov/sra)下载陆地棉转录组测序项目(PRJNA248163和PRJNA408075),首先利用NCBI SRA Toolkit(https://www.ncbi.nlm.nih.gov/sra/docs/ toolkitsoft/)将测序数据sra文件转换为fastq文件,再利用软件Trimmomatic[26]去除测序数据中的接头序列和低质量序列,使用FastQC软件(http://www. bioinformatics.babraham.ac.uk/projects/fastqc/)对测序数据进行质量控制,最后利用有参转录组定量工具Kallisto[27-28]计算TPM(Transcripts Per Kilobase of exonmodel per Million mapped reads)值作为基因相对表达量。利用在线工具(https://software.broadinstitute. org/morpheus/)绘制陆地棉CRK家族基因表达热图。
1.9 RNA提取和实时荧光定量PCR分析
采用EASYspin Plus多糖多酚/复杂植物RNA快速提取试剂盒(北京艾德莱生物科技有限公司)提取棉花RNA,反转录试剂盒PrimeScriptTM1st Strand cDNA Synthesis Kit(TaKaRa大连)反转录合成cDNA。使用Primer Premier 5.0软件设计(Gene ID Gh_A06G1272)的qRT-PCR特异引物(表1),以陆地棉(AY305737)作为内参基因,SYBR Premix Ex TaqTM(TaKaRa大连)为PCR反应试剂,稀释3倍后的cDNA作为实时荧光定量PCR的模板。PCR反应体系为cDNA 2 µL、SYBR Premix Ex TaqTM10 µL、正、反向引物各0.8 µL和ddH2O 6.4 µL。扩增程序为95℃30 s;95℃ 5 s,60℃ 1 min,72℃ 30 s,40个循环;在罗氏lightCycler480Ⅱ定量PCR仪进行,以2-△△Ct方法计算的相对表达量。
1.10 病毒介导的基因沉默(virus-induced gene silencing,VIGS)
利用在线工具(http://vigs.solgenomics.net)设计的最佳VIGS干扰片段,针对VIGS干扰片段设计引物并在正向引物VIGS-GhCRK25-F的5′端添加Ⅰ酶切位点,反向引物VIGS-GhCRK25-R的5′端添加HⅠ酶切位点,扩增的目标片段经酶切与VIGS表达载体TRV2(pLY156)连接获得重组表达载体TRV2﹕GhCRK25,将重组表达载体TRV2﹕GhCRK25和空载体TRV2﹕00转化农杆菌GV3101,以TRV2空载体为阳性对照,通过子叶注射法,转化陆地棉品种TM-1。以VIGS沉默棉花表现出的白化性状作为表型对照,通过qRT-PCR和RT-PCR的方法检测沉默效果,以(AY305724. 1)作为RT-PCR的内参。利用微量注射器分别对TRV2﹕GhCRK25沉默植株和TRV2﹕00对照植株接种2 µL浓度为5×106cfu/mL的大丽轮枝菌Vd991,接种15 d后观察接种植株表型并统计病情指数[29]。

表1 PCR引物及其序列
2 结果
2.1 棉花CRK家族的鉴定与系统进化分析
以Pfam数据库下载的stress-antifung(PF01657)和Pkinase_Tyr(PF07714)为种子序列,BLASTp搜索本地陆地棉和海岛棉蛋白数据库,去冗余后获得候选CRK蛋白,分别搜索Pfam、SMART、NCBI-CDD蛋白质数据库鉴定候选蛋白结构域,保留同时含有stress-antifung和Pkinase_Tyr结构域的CRK蛋白。分别从陆地棉和海岛棉中鉴定出70和54个CRK蛋白。与拟南芥CRK家族44个成员的蛋白序列进行多序列比对并采用邻接法构建系统发育树(图1)。根据进化树聚类结果,可将陆地棉、海岛棉和拟南芥3个物种的CRK蛋白分为5个亚族;第Ⅰ亚族成员总数最多,包括38个陆地棉CRK蛋白、32个海岛棉CRK蛋白和38个拟南芥CRK蛋白,分别占其总数的54.29%、59.26%和86.36%;第Ⅱ亚族包括11个陆地棉CRK蛋白和6个海岛棉CRK蛋白;第Ⅲ亚族仅包括1个陆地棉CRK蛋白和1个拟南芥CRK蛋白,成员数目最少且它们与其他亚族间的遗传距离较远;第Ⅳ亚族包括2个陆地棉CRK蛋白和3个海岛棉CRK蛋白;第Ⅴ亚族包括17个陆地棉CRK蛋白、13个海岛棉CRK蛋白和5个拟南芥CRK蛋白,成员总数仅次于第Ⅰ亚族。同一亚族内陆地棉和海岛棉CRK蛋白聚为一组而拟南芥CRK蛋白聚为一组,表明2个棉种的CRK蛋白遗传距离较近与拟南芥CRK蛋白的遗传距离较远。文章主要对陆地棉CRK基因家族的70个成员进行生物信息学分析和表达模式分析。
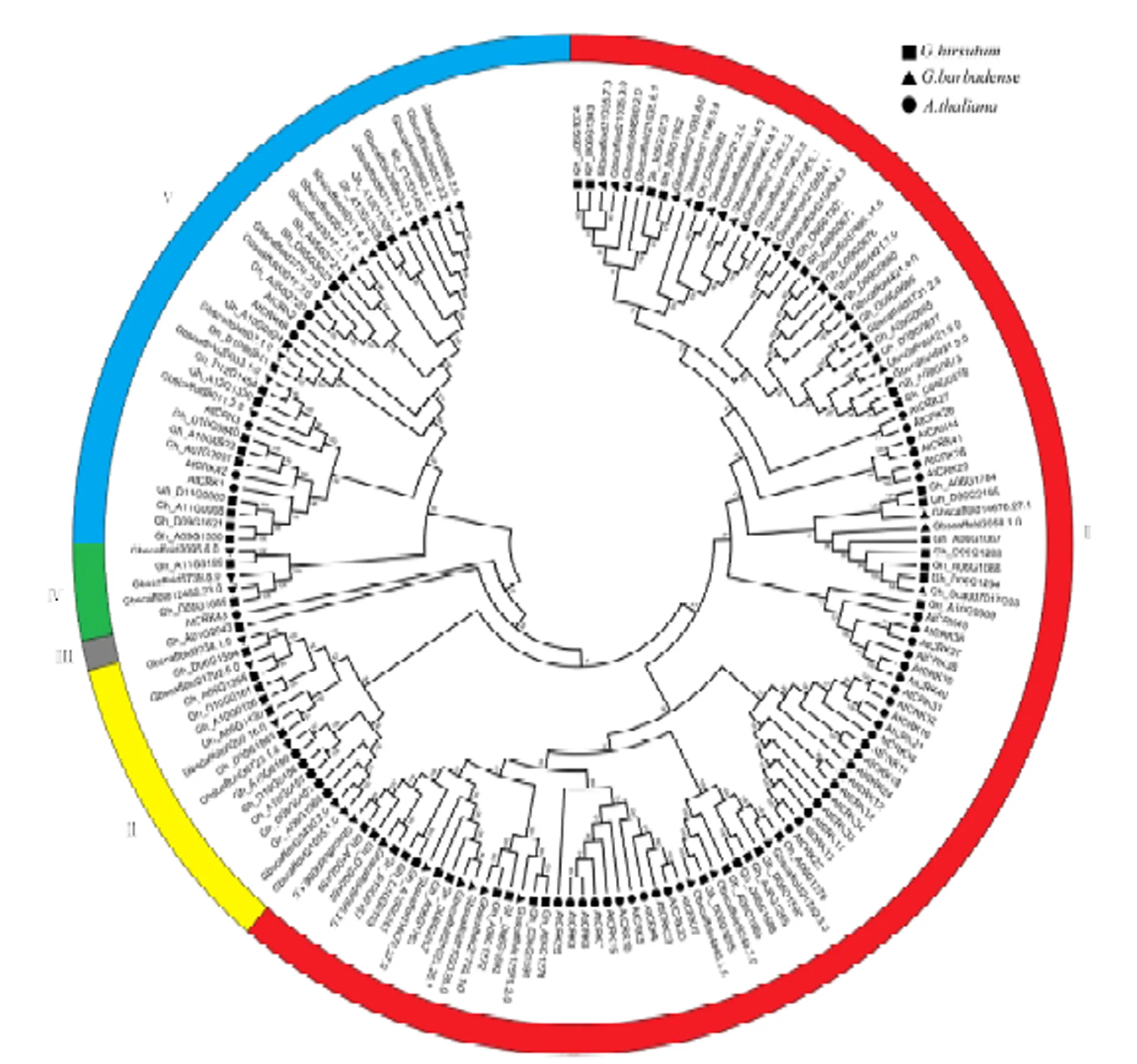
图1 陆地棉、海岛棉和拟南芥CRK基因家族的系统发育进化树
2.2 陆地棉CRK家族蛋白分子结构和生理生化特性
陆地棉CRK家族蛋白分子结构和生理生化特性见表2。陆地棉CRK家族编码302—901个氨基酸,平均623个,相对分子质量介于33.44—100.49 kD,理论等电点变化为4.97—9.06。44个CRK蛋白既含有信号肽又含有跨膜结构域,14个CRK有跨膜结构域而缺少信号肽,具有跨膜结构的CRK占陆地棉CRK总数的82.9%,与CRK受体激酶的功能和特性相符。9个CRK有信号肽而缺少跨膜结构域,3个CRK同时缺少信号肽和跨膜结构域。利用WOLF POSORT在线工具预测陆地棉CRK家族蛋白亚细胞定位,结果表明,25个CRK蛋白定位于叶绿体,21个CRK蛋白定位于质膜,12个CRK蛋白定位于胞外,其他少数CRK蛋白被定位于液泡、内质网、细胞核和胞质。表明棉花CRK蛋白分子结构和细胞定位的多样性,陆地棉CRK成员间具有不同的功能分化。
2.3 棉花CRK基因家族的染色体定位
70个陆地棉CRK基因定位于14条染色体上(表2、图2),其中33个CRK基因定位于A亚组,34个CRK基因定位于D亚组,、和等3个CRK基因分别定位于未能组装到染色体的scaffold32_ A01、scaffold2939_A11(+):118938-131953和scaffold7017片段。其中,A6(13个)、D6(13个)、A9(4个)、D9(7个)、A10(8个)和D10(7个)染色体上分布最多。呈现明显A和D亚组同源基因对平行进化特征,A6/D6染色体分布最多,共12对旁系同源基因;其次为A10/D10染色体,共7对旁系同源基因,而A9/D9染色体包含3对旁系同源基因,它们在A/D染色体对之间呈现高度共线性关系,且在染色体上串联成簇分布,串联基因间的物理距离较近。此外,A12/D12亚组、A5/D5亚组和A11/D11亚组也有少量平行进化的旁系同源基因对。还有部分CRK基因仅分布于A或D某一亚组,而在其对应的另一个亚组上未发现同源基因的存在,推测很可能是A/D亚组上的个别同源基因在进化过程中丢失,或者发生了易位。
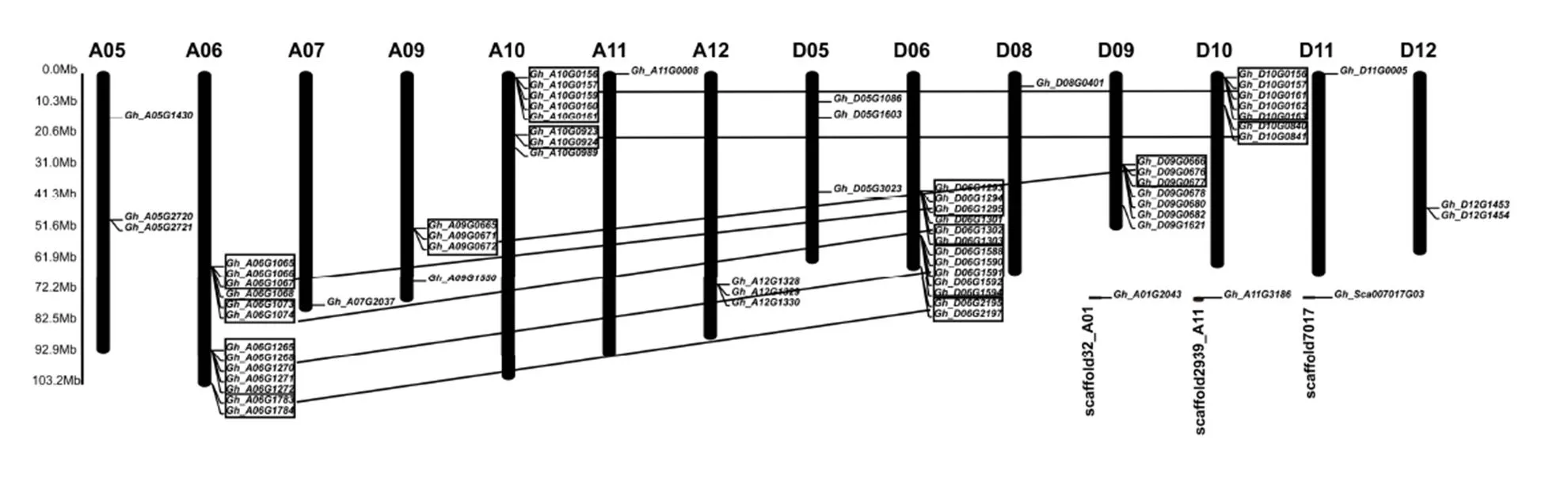
AD亚组染色体上由矩形标注的基因为同源基因并用直线连接
2.4 陆地棉CRK基因家族的结构和蛋白质功能结构域分析
陆地棉CRK基因具有相对保守的基因结构和蛋白功能结构域(图3)。陆地棉CRK基因长度介于2.1—19.8 kb,外显子数量4—10个,大部分CRK基因长度小于4 kb,大多数外显子数为7个。进化树中聚类在同一组的CRK基因通常具有类似的基因结构和外显子数量,保守性较高,当然,也有少数CRK基因长度和外显子数量缺乏规律性(图3-B)。保守结构域分析结果显示,陆地棉CRK家族中62个蛋白质具有2个stress-antifung结构域和1个Pkinase_Tyr结构域,7个陆地棉CRK蛋白只含有1个stress-antifung结构域和1个Pkinase_Tyr结构域,而Gh_D08G0401具有4个stress-antifung结构域和1个Pkinase_Tyr结构域。Gh_D08G0401、Gh_A10G0157、Gh_D10G0156和Gh_A10G0156除含有stress-antifung结构域和Pkinase_Tyr结构域外,还含有1功能未知的DUF3403(PF11883)结构域;而基因Gh_A05G1430除含有stress-antifung结构域和Pkinase_Tyr结构域外,还含有1个TauE(PF01925)结构域(图3-C)。结合陆地棉CRK蛋白家族系统发育树分析结果,位于进化树同一分支的同源基因具有相似的基因结构和功能结构域,推测它们具有相似的生物学功能。
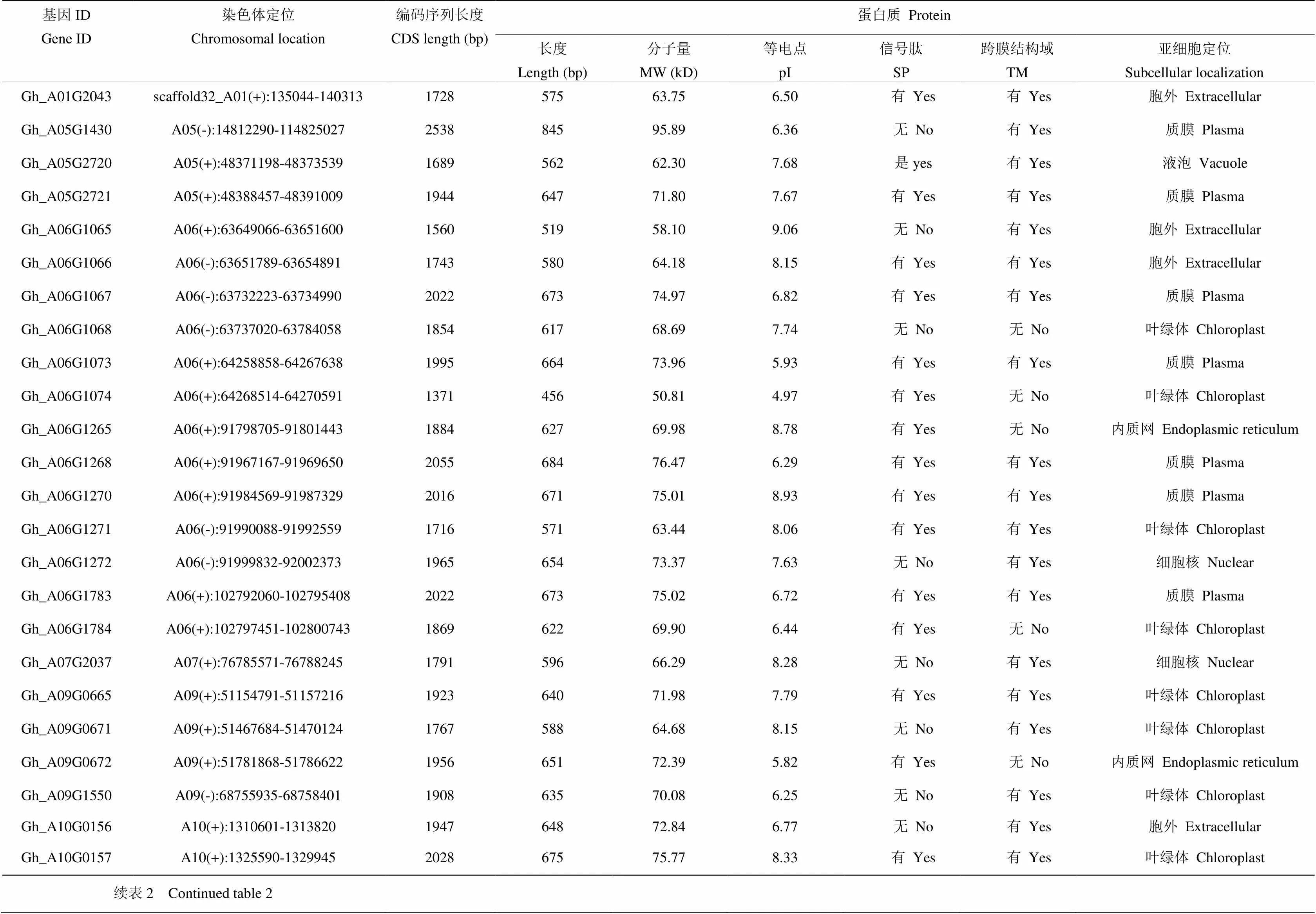
表2 陆地棉CRK家族蛋白分子结构和生理生化特性

A:陆地棉CRK家族进化树;B:陆地棉CRK家族基因结构;C:陆地棉CRK家族蛋白的功能结构域
2.5 陆地棉CRK基因家族的启动子分析
陆地棉CRK基因家族启动子分析结果表明陆地棉CRK家族基因的启动子区域富含响应植物激素和逆境胁迫的顺式作用元件(图4)。所有CRK基因启动子区至少含有一个植物激素响应元件,包括SA响应元件TCA-element、赤霉素响应元件GARE-motif、茉莉酸(JA)响应元件CGTCA-motif、乙烯响应元件ERE和脱落酸(ABA)响应元件ABRE等,其中65个(占总基因数92.9%)陆地棉CRK基因的启动子区至少含有一种逆境激素响应元件。69个(占总基因数98.6%)CRK基因至少含有一种生物或非生物胁迫响应元件,逆境胁迫响应元件包括热胁迫响应元件HSE、低温胁迫响应元件LTR、干旱胁迫响应元件MBS等非生物胁迫响应元件,38个基因启动子区含有真菌激发子响应元件Box-W1是棉花CRK基因的另一个突出特点。表明陆地棉CRK基因家族在调控植物生长发育、抵御逆境胁迫尤其是生物胁迫中发挥重要作用。
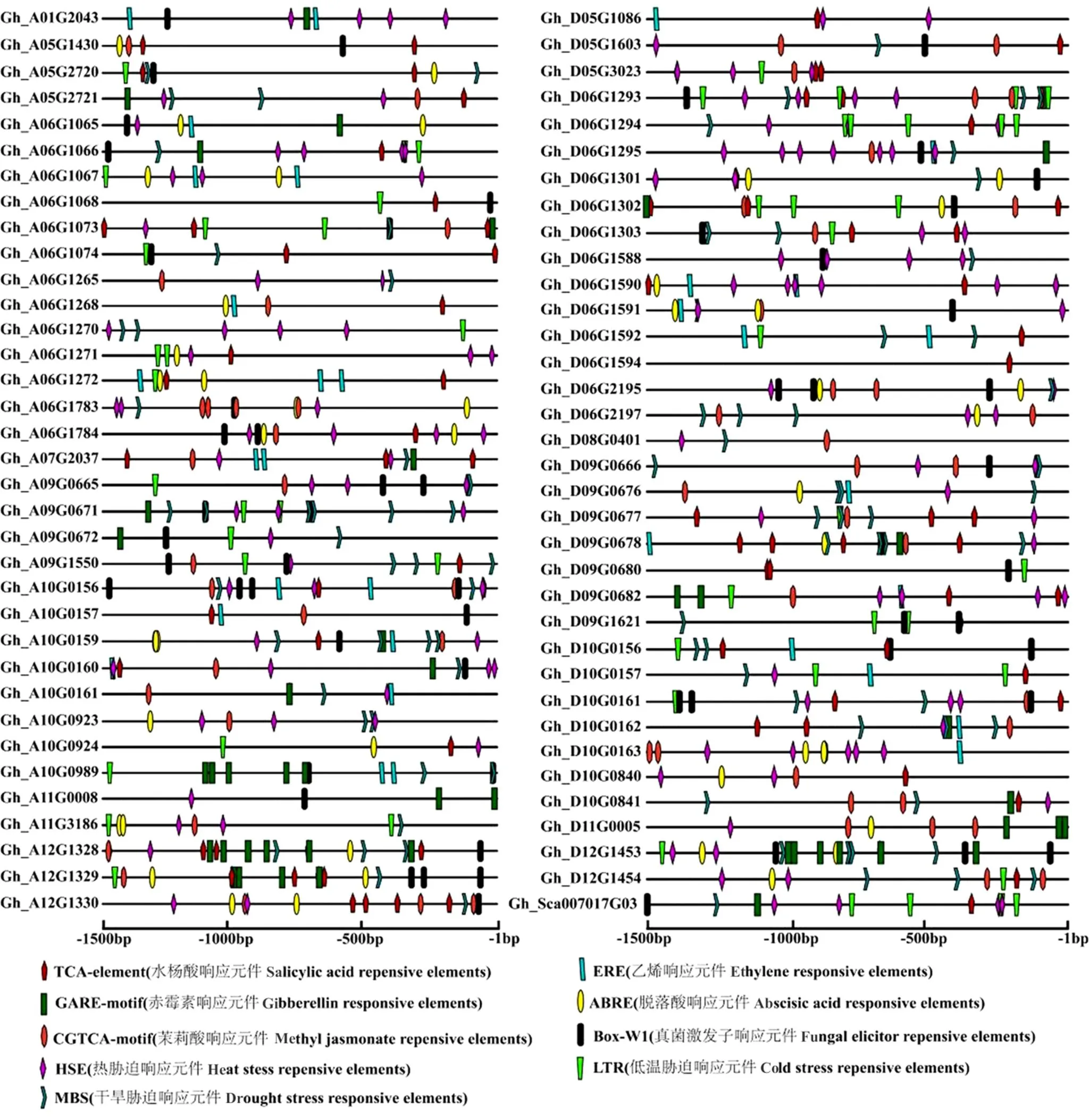
图4 预测陆地棉CRK家族基因启动子中顺式调控元件
2.6 陆地棉CRK家族蛋白质磷酸化位点分析
蛋白激酶往往通过自身及其互作蛋白的磷酸化发挥其生物学功能,通过预测陆地棉CRK家族蛋白质磷酸化位点,并与拟南芥CRK蛋白质磷酸化位点进行对比。陆地棉CRK家族共发现9个保守的丝氨酸磷酸化位点、6个苏氨酸磷酸化位点以及3个酪氨酸磷酸化位点;拟南芥CRK家族则包含8个保守的丝氨酸磷酸化位点、4个苏氨酸磷酸化位点以及2个酪氨酸磷酸化位点(图5)。其中,陆地棉和拟南芥CRK家族蛋白有3种丝氨酸磷酸化位点基序(···L···S*·······、·····N·S*·······和·······S*N······)和2种苏氨酸磷酸化位点基序(······N·T*·······和·······T*·····T·)相同,具有同样磷酸化基序的蛋白质可能具有相同或相似的生物学功能和分子机制,因此,对于预测棉花CRK基因的功能具有参考价值。
2.7 陆地棉CRK基因家族的表达分析
为初步了解陆地棉CRK基因的功能,利用陆地棉TM-1的转录组数据分析了CRK基因家族在根、茎、叶、花托、雌蕊、雄蕊、花瓣以及不同天数的胚珠和纤维中的表达特征(图6)。结果表明,陆地棉CRK基因具有多样化组织特异表达和时空表达特征,依据表达特征,陆地棉CRK家族基因可分为3组:第Ⅰ组基因几乎在所有组织和整个生育期中优势表达,主要包括、、和。第Ⅱ组基因在棉花胚珠和纤维中低水平表达,而在营养器官和生殖器官器官中优势表达,如、、、、、和在根中优势表达,、、、、、和在花托中优势表达,可能主要参与陆地棉营养生长和花器官发育。第Ⅲ组基因表达特征是:一部分基因在除纤维以外各个组织中优势表达,如、、、、和,另一部分基因在副萼中优势表达,如、和。结果表明,广泛参与陆地棉生长发育的生命过程。
植物CRK蛋白是一类膜受体蛋白激酶,参与调节植物对生物胁迫和非生物胁迫的应答。利用来源于陆地棉的RNA-Seq数据分析了陆地棉CRK基因在PEG-6000、盐、低温、高温和大丽轮枝菌Vd6处理下的表达特征(图7)。依据对生物胁迫和非生物胁迫的响应表现出不同的表达特征,陆地棉CRK基因家族可分为4组:第Ⅰ组基因主要在受到非生物胁迫后表现为上调表达,如、、、、等,这类CRK基因主要参与陆地棉对低温、高温、干旱和盐胁迫的应答和抵御。第Ⅱ组基因受到非生物胁迫和大丽轮枝菌处理后均表现为持续上调表达,如、和等。第Ⅲ组CRK基因主要在受大丽轮枝菌处理后表现为持续上调表达,这类基因可能主要与陆地棉抗病性相关,参与陆地棉对黄萎病菌的应答和信号传导等。第Ⅳ组陆地棉CRK基因受到非生物和生物胁迫处理后没有显著的变化,如、、、和等。以上结果表明,陆地棉CRK基因家族成员在进化过程中发生了功能分化,分工协作参与陆地棉不同刺激物质的响应。
2.8 陆地棉GhCRK25功能的分析
从陆地棉TM-1中克隆了1个CRK编码基因(),其结构与相似(图1),命名为。采用qRT-PCR技术分析了组织表达特征以及对ABA、赤霉素(GA3)、SA、PEG-6000、NaCl和大丽轮枝菌Vd991处理的响应(图8)。结果表明,在根、茎、叶和幼嫩胚珠中优势表达,在纤维中表达明显降低(图8-A)。
ABA、GA3、SA、PEG-6000、氯化钠和大丽轮枝菌Vd991处理均能刺激迅速上调表达(图8-B—图8-G)可能通过激素介导的信号途径参与棉花对非生物和生物胁迫抗性调控。

A:陆地棉磷酸化位点motif;B:拟南芥磷酸化位点motif
为初步验证在陆地棉抵御大丽轮枝菌Vd991侵染中的功能,利用VIGS技术沉默棉花,检测其对黄萎病抗性的影响。结果表明,接种大丽轮枝菌Vd991后15 d,TRV﹕GhCRK25沉默植株叶片黄化和枯萎等病征较对照更为明显,病情指数显著高于TRV﹕00植株(图9)。沉默降低了陆地棉对大丽轮枝菌Vd991的抗性。因此,是棉花黄萎病抗性所必需的基因之一。
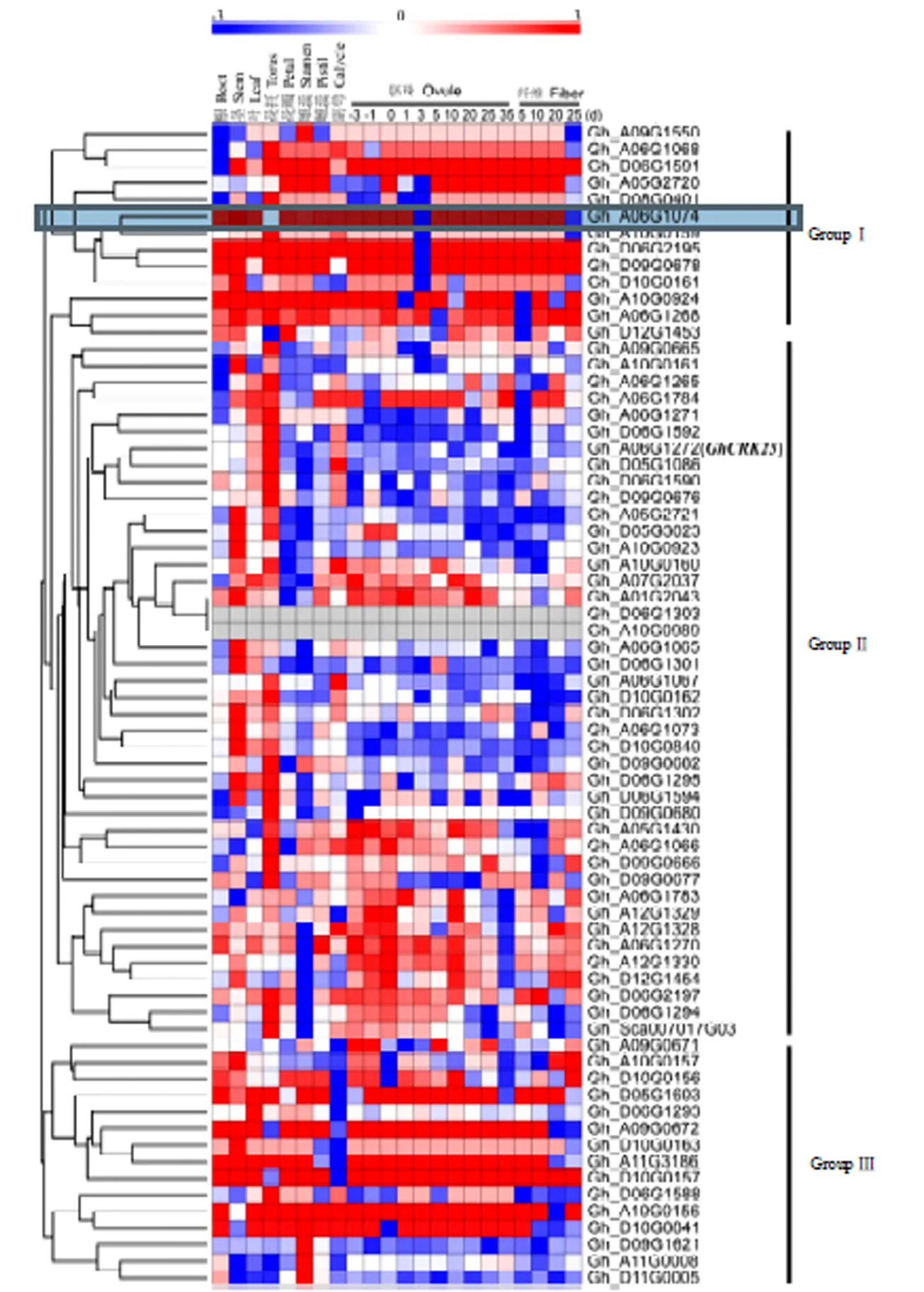
图6 陆地棉CRK基因在不同组织中的表达分析
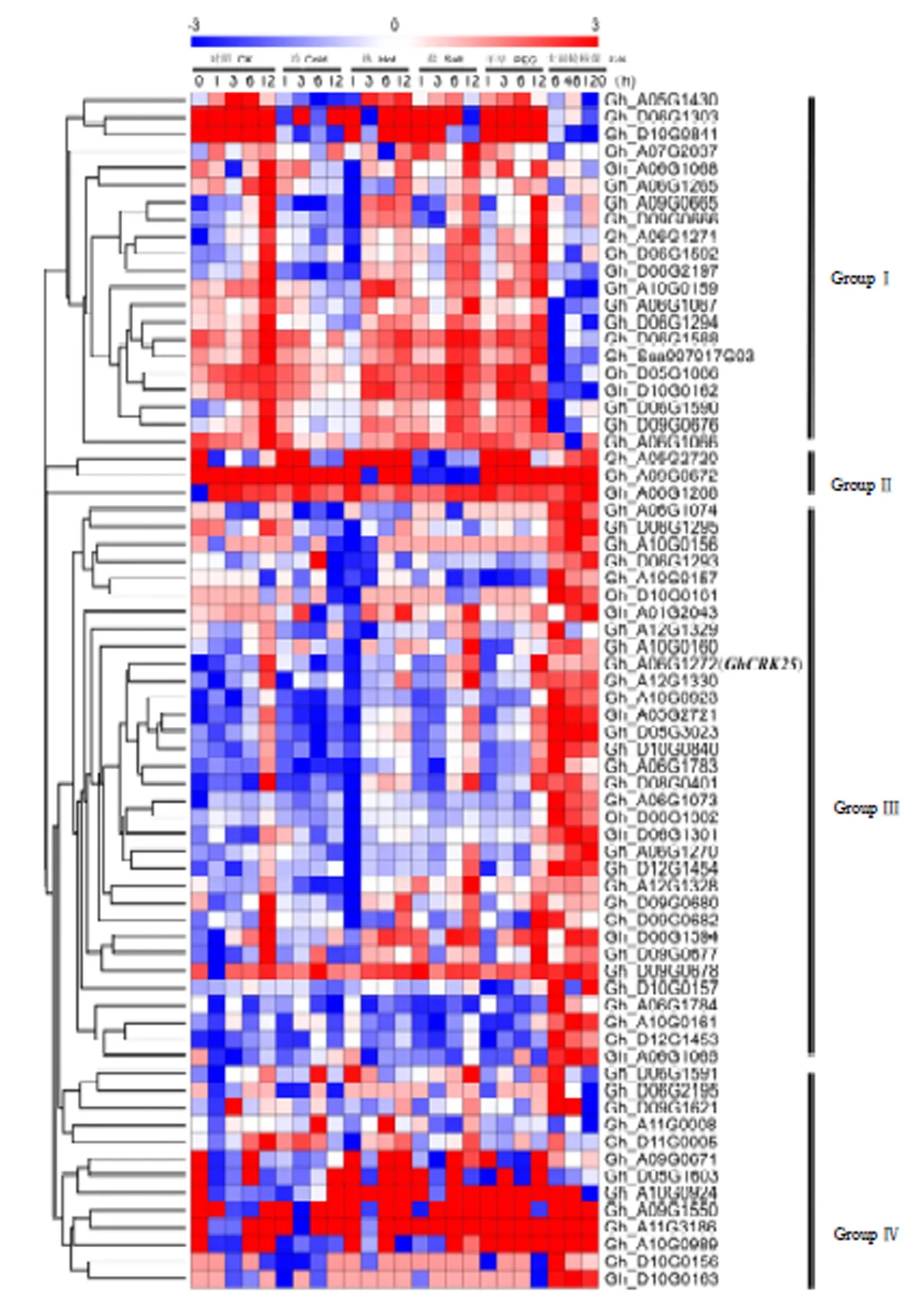
图7 陆地棉CRK基因在PEG-6000、盐、低温、高温和大丽轮枝菌处理条件下的表达模式
3 讨论
通过同源搜索,在陆地棉基因组中,鉴定出70个CRK基因。陆地棉CRK基因家族有26对平行进化的旁系同源CRK基因,主要以串联成簇的形式排列分布于A6/D6(12对)、A10/D10(7对)和A9/D9(3对),A和D亚组同源基因对平行进化特征明显,A和D亚组可能来源于同一个共同祖先。部分CRK基因在A、D亚组之间对应不平衡,则可能是A亚组和D亚组的非对称进化所形成的,A亚组蛋白质进化速率、染色体重排发生频率以及基因丢失和失活的频率均显著高于D亚组[23]。拟南芥CRK基因家族包含44个成员,其中19个成员以串联成族的形式排列于第4染色体且序列间具有高度相似性[31]。拟南芥CRK基因家族中串联排列的基因间具有相似的生物学功能,如,与参与响应并传导胞外O3信号保护质外体抵御过氧化胁迫[30],过表达或加速拟南芥胞外ROS的爆发进而诱导细胞死亡,提高了对丁香假单胞菌DC3000的抗性,免疫沉淀研究发现,AtCRK28不但可以自我相互作用而且还与AtCRK29相互作用[13]。但有研究表明,拟南芥41个CRK基因的单基因突变体株系受DC3000侵染后并未表现出强烈的免疫缺陷表型,说明拟南芥CRK成员间可能负责对不同病原菌的抗性或存在功能冗余[3]。陆地棉CRK基因家族中串联排列的CRK基因间是否具有类似的生物学功能以及如何相互作用,CRK基因家族成员间如何响应不同逆境,是否有不同的应答机制和功能冗余值得进一步研究。
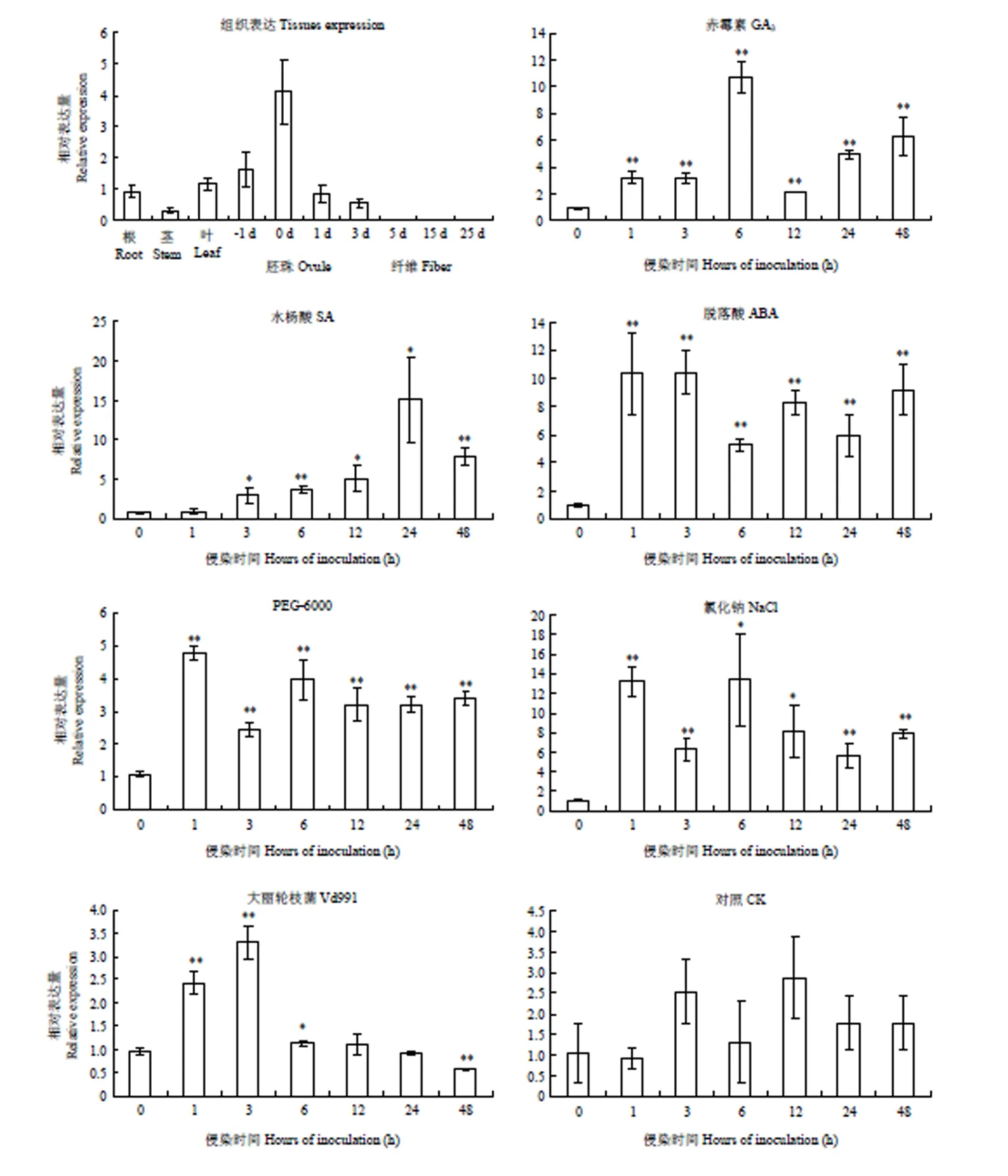
误差线代表3个生物学重复的标准偏差。星号代表显著性差异水平,采用Student t测验(*P <0.05;**P <0.01)。下同
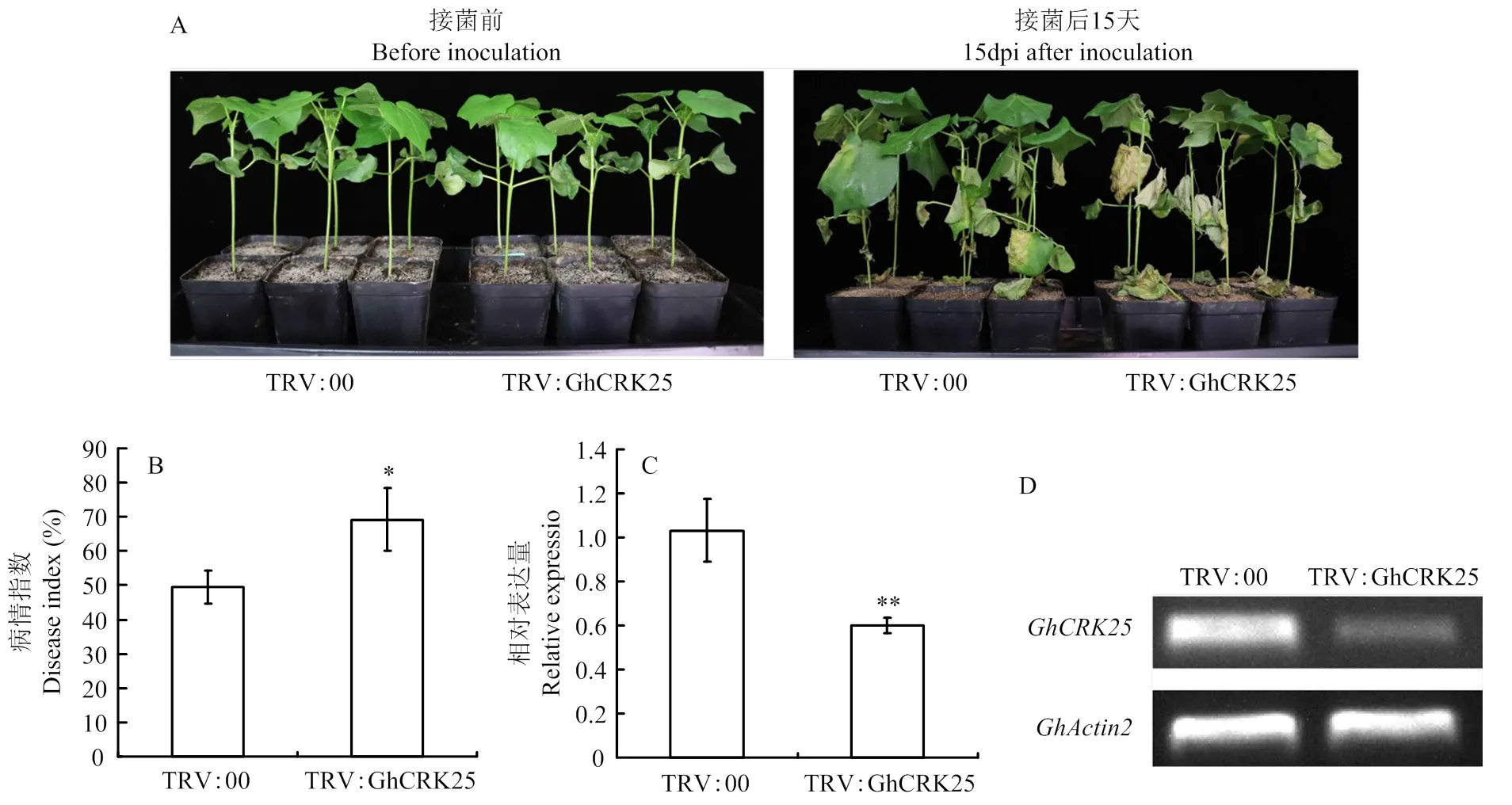
A:TRV﹕00对照植株和TRV﹕GhCRK25植株受大丽轮枝菌Vd991侵染后15 d的症状;B:TRV﹕00对照植株和TRV﹕GhCRK25植株受大丽轮枝菌Vd991侵染后15 d的病情指数;C:qRT-PCR检测基因GhCRK25在TRV﹕00对照植株和TRV﹕GhCRK25叶片中的转录水平;D:RT-PCR检测基因GhCRK25在TRV﹕00对照植株和TRV﹕GhCRK25叶片中的转录水平
陆地棉CRK基因家族的基因长度介于2.1—19.8 kb,外显子数量为4—10个,位于进化树同一亚族内CRK基因间具有类似的内含子/外显子排列模式和数目,同时它们的保守结构域也具有类似的数量和位置顺序,表明亚族内成员间在进化过程中较为保守。通过结晶技术和X射线技术发现,银杏种子中的抗真菌蛋白Gnk2与植物CRK蛋白的胞外stress-antifung结构域有同源性,它们都含有一个由3个半胱氨酸(Cys)残基构成的C-X8-C-X2-C基序,保守的3个半胱氨酸残基(Cys)可以形成分子内二硫键作为氧化还原修饰的靶位点[31-32],推测棉花CRK蛋白中的C-X8-C-X2-C基序与其抗病性相关。部分陆地棉CRK家族基因的基因结构和保守结构域特征显著不同于其他亚族,Gh_D08G0401、Gh_A10G0157、Gh_D10G0156和Gh_A10G0156除含有典型stress-antifung结构域和Pkinase_Tyr结构域外还含有1功能未知的DUF3403结构域(PF11883);而Gh_A05G1430除含有典型的stress-antifung结构域和Pkinase_Tyr结构域外还含有1个TauE结构域(PF01925),CRK家族基因在进化过程中基因结构和保守结构域的差异为其成员间功能分化提供了遗传变异的基础。
基因启动子区包含的顺式调控元件决定基因的表达水平和表达模式。前人研究表明,拟南芥、、、、、和以及马铃薯启动子的序列的启动子区均包含与病原菌防御相关的顺式调控元件Box-W1[10,19,33-34]。转录因子AtWRKY18识别启动子区的Box-W1并诱导表达[32],从而增强植物对病原菌的抗性。本研究发现,有38个棉花CRK基因的启动子区具有Box-W1,由基因表达热图(图7)可以看出,至少有35个棉花基因受大丽轮枝菌胁迫处理后显著上调表达,表明这些基因可能与WRKY转录因子相互作用参与陆地棉对黄萎病菌的抵御反应。上游启动子区具有对热胁迫响应元件的CRK基因达56个、具有干旱胁迫响应元件的CRK基因49个、具有低温胁迫响应元件的CRK基因33个,与RNA-Seq分析结果基本一致(图7)。表明棉花CRK还参与非生物胁迫的抗性反应。每个CRK基因启动子区至少含有一个植物激素响应元件,其中具有SA、JA、ABA、乙烯等所谓逆境激素的响应元件的基因分别为47、41、29和24个。分析本研究中克隆的(Gh_A06G1272)其上游1 500 bp以内的启动子区含有冷胁迫响应元件以及SA、ABA和乙烯的响应元件,qRT-PCR分析结果表明,不仅SA和ABA可以诱导棉花急剧上调表达,GA3、PEG-6000、氯化钠和大丽轮枝菌Vd991处理也均能刺激迅速上调表达。可见CRK是对细胞内外刺激都非常敏感的类受体激酶。具有赤霉素响应元件的CRK基因有24个,且GA3诱导上调表达,暗示某些CRK与植物生长调节有关。利用VIGS技术研究在陆地棉抵御大丽轮枝菌Vd991侵染中的功能发现TRV﹕GhCRK25沉默植株比TRV﹕00对照植株表现出更为严重的叶片黄化和枯萎的病症以及更高的病情指数。表明沉默陆地棉对大丽轮枝菌Vd991更为敏感,因此与黄萎病抗性有关。然而启动子区并没有Box-W1元件,因此推测CRK可以通过响应其他(如水杨酸等信号)通路参与对黄萎病病原菌的防御反应。
棉花CRK如何感受并传递内外环境刺激,从而调控植物体的生理机能,是解析其分子作用机制的关键。蛋白激酶往往通过自身及其互作蛋白的磷酸化发挥其生物学功能,因此,研究CRK家族蛋白质磷酸化功能具有十分重要的意义。本研究共发现陆地棉CRK家族的蛋白质有9个保守的丝氨酸磷酸化位点、6个苏氨酸磷酸化位点以及3个酪氨酸磷酸化位点,其中有3种丝氨酸磷酸化位点基序(···L···S*·······、·····N·S*·······和·······S*N······)和2种苏氨酸磷酸化位点基序(······N·T*·······和·······T*·····T·)与拟南芥相同,因此可以根据拟南芥中含有这些基序的CRK的研究结果,为研究棉花具有这类基序的CRK基因的功能及其作用机制提供参考。
4 结论
从陆地棉基因组中鉴定出70个CRK基因,陆地棉CRK基因具有保守的基因结构和功能结构域,多种响应激素和胁迫的启动子区调控元件,多样化的组织表达特征,大多数基因受激素和逆境调控。
[1] CHEN Z. A superfamily of proteins with novel cysteine-rich repeats, 2001, 126(2): 473-476.
[2] WRZACZEK M, BROSCHE M, SALOJARVI J, KANGASJARVI S, IDANHEIMO N, MERSMANN S, ROBATZEK S, KARPINSKI S, KARPINSKA B, KANGASJARVI J. Transcriptional regulation of the CRK/DUF26 group of receptor-like protein kinases by ozone and plant hormones in, 2010, 95(10): 1471-2229.
[3] BOURDAIS G, BURDIAK P, GAUTHIER A, NITSCH L, SALOJARVI J, RAYAPURAM C, IDANHEIMO N, HUNTER K, KIMURA S, MERILO E, VAATTOVAARA A, ORACZ K, KAUFHOLDT D, PALLON A, ANGGORO D T, GLOW D, LOWE J, ZHOU J, MOHAMMADI O, PUUKKO T, ALBERT A, LANG H, ERNST D, KOLLIST H, BROSCHE M, DURNER J, BORST J W, COLLINGE D B, KARPINSKI S, LYNGKJAER M F, ROBATZEK S, WRZACZEK M, KANGASJARVI J, CONSORTIUM C R K. Large-scale phenomics identifies primary and fine-tuning roles for CRKs in responses related to oxidative stress, 2015,11(7): e1005373.
[4] ZIPFEL C. Plant pattern-recognition receptors, 2014, 35(7): 345-351.
[5] YEH Y H, CHANG Y H, HUANG P Y, HUANG J B, ZIMMERLI L. Enhancedpattern-triggered immunity by overexpression of cysteine-rich receptor-like kinases, 2015, 6: 322.
[6] BOLLER T, FELIX G. A renaissance of elicitors: perception of microbe-associated molecular patterns and danger signals by pattern-recognition receptors, 2009, 60: 379-406.
[7] LEE D S, KIM Y C, KWON S J, RYU C M, PARK O K. Thecysteine-rich receptor-like kinase CRK36 regulates immunity through interaction with the cytoplasmic kinase BIK1, 2017, 8: 1856.
[8] CUI H, TSUDA K, PARKER J E. Effector-triggered immunity: from pathogen perception to robust defense, 2015, 66: 487-511.
[9] OLIVEIRA1 M, XU G, LI BO, VESPOLI L, MENG X, CHEN X, YU X, SUZANE A, INTORNE A, MANHÃES A, MUSINSKY A, KOIWA H, FILHO G, SHAN L, HE P. Specific control ofBAK1/SERK4-regulated cell death by protein glycosylation.2016, 2: 15218.
[10] CHEN K, FAN B, DU L, CHEN Z. Activation of hypersensitive cell death by pathogen-induced receptor-like protein kinases from, 2004, 56(2): 271-283.
[11] ACHARYA B R, RAINA S, MAQBOOL S B, JAGADEESWARAN G, MOSHER S L, APPEL H M, SCHULTZ J C, KLESSIG D F, RAINA R. Overexpression of, ancysteine-rich receptor-like kinase, results in enhanced resistance to, 2007, 50(3): 488-499.
[12] CHEN K G, DU L Q, CHEN Z X. Sensitization of defense responses and activation of programmed cell death by a pathogen-induced receptor-like protein kinase in, 2003, 53(1): 61-74.
[13] YADETA K A, ELMORE J M, CREER A Y, FENG B, FRANCO J Y, RUFIAN J S, HE P, PHINNEY B, COAKER G. A cysteine-rich protein kinase associates with a membrane immune complex and the cysteine residues rre required for cell death, 2017, 173(1): 771-787.
[14] JURG LANGE Z-P X, WILLIAM J. BROUGHTON, REGINA VOGELI-LANGE, THOMAS BOLLER. A gene encoding a receptor-like protein kinase in the roots of common bean is differentially regulated in response to pathogens, symbionts and nodulation factors, 1999, 144(2): 133-145.
[15] YANG K, RONG W, QI L, LI J, WEI X, ZHANG Z. Isolation and characterization of a novel wheat cysteine-rich receptor-like kinase gene induced by Rhizoctonia cerealis, 2013, 3: 3021.
[16] CHERN M, XU Q, BART R, BAI W, RUAN D, WING H, CANLAS P, JAIN R, CHEN X, RONALD P. A genetic screen identifies a requirement for cysteine-rich-receptor-like kinases in rice NH1 (OsNPR1)-mediated immunity, 2016, 12(5): e1006049.
[17] RAYAPURAM C, JENSEN M K, MAISER F, SHANIR J V, HORNSHOJ H, RUNG J H, GREGERSEN P L, SCHWEIZER P, COLLINGE D B, LYNGKJAER M F. Regulation of basal resistance by a powdery mildew-induced cysteine-rich receptor-like protein kinase in barley, 2012, 13(2): 135-147.
[18] EDERLI L, MADEO L, CALDERINI O, GEHRING C, MORETTI C, BUONAURIO R, PAOLOCCI F, PASQUALINI S. Thecysteine-rich receptor-like kinase CRK20 modulates host responses topv. tomato DC3000 infection, 2011, 168(15): 1784-1794.
[19] TOSTI N, PASQUALINI S, BORGOGNI A, EDERLI L, FALISTOCCO E, CRISPI S, PAOLOCCI F. Gene expression profiles of O3-treatedplants, 2006, 29(9): 1686-1702.
[20] BURDIAK P, RUSACZONEK A, WITON D, GLOW D, KARPINSKI S. Cysteine-rich receptor-like kinase CRK5 as a regulator of growth, development, and ultraviolet radiation responses in, 2015, 66(11): 3325-3337.
[21] ZHANG X, YANG G, SHI R, HAN X, QI L, WANG R, XIONG L, LI G.cysteine-rich receptor-like kinase 45 functions in the responses to abscisic acid and abiotic stresses, 2013, 67: 189-198.
[22] KIM W, YI S, OH S, LIM C, KIM H, JANG H, LEE H, PARK Y, KWON S. Identification of a pollen-specific gene, SlCRK1 (RFK2) in tomato, 2014, 36(3): 303-311.
[23] ZHANG T, HU Y, JIANG W, FANG L, GUAN X, CHEN J, ZHANG J, SASKI C A, SCHEFFLER B E, STELLY D M, HULSE-KEMP A M, WAN Q, LIU B, LIU C, WANG S, PAN M, WANG Y, WANG D, YE W, CHANG L, ZHANG W, SONG Q, KIRKBRIDE R C, CHEN X, DENNIS E, LLEWELLYN D J, PETERSON D G, THAXTON P, JONES D C, WANG Q, XU X, ZHANG H, WU H, ZHOU L, MEI G, CHEN S, TIAN Y, XIANG D, LI X, DING J, ZUO Q, TAO L, LIU Y, LI J, LIN Y, HUI Y, CAO Z, CAI C, ZHU X, JIANG Z, ZHOU B, GUO W, LI R, CHEN Z J. Sequencing of allotetraploid cotton (L. acc. TM-1) provides a resource for fiber improvement, 2015, 33(5): 531-540.
[24] LI F, FAN G, LU C, XIAO G, ZOU C, KOHEL R J, MA Z, SHANG H, MA X, WU J, LIANG X, HUANG G, PERCY R G, LIU K, YANG W, CHEN W, DU X, SHI C, YUAN Y, YE W, LIU X, ZHANG X, LIU W, WEI H, WEI S, HUANG G, ZHANG X, ZHU S, ZHANG H, SUN F, WANG X, LIANG J, WANG J, HE Q, HUANG L, WANG J, CUI J, SONG G, WANG K, XU X, YU J Z, ZHU Y, YU S. Genome sequence of cultivated upland cotton (TM-1) provides insights into genome evolution., 2015, 33(5): 524-530.
[25] YUAN D, TANG Z, WANG M, GAO W, TU L, JIN X, CHEN L, HE Y, ZHANG L, ZHU L, LI Y, LIANG Q, LIN Z, YANG X, LIU N, JIN S, LEI Y, DING Y, LI G, RUAN X, RUAN Y, ZHANG X. The genome sequence of Sea-Island cotton () provides insights into the allopolyploidization and development of superior spinnable fibres., 2015, 5: 17662.
[26] BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data, 2014, 30(15): 2114-2120.
[27] LI B, RUOTTI V, STEWART R M, THOMSON J A, DEWEY C N. RNA-Seq gene expression estimation with read mapping uncertainty, 2010, 26(4): 493-500.
[28] BRAY N L, PIMENTEL H, MELSTED P, PACHTER L. Near-optimal probabilistic RNA-seq quantification, 2016, 34(5): 525-527.
[29] ZHANG B, YANG Y, CHEN T, YU W, LIU T, LI H, FAN X, REN Y, SHEN D, LIU L, DOU D, CHANG Y. Island cotton Gbve1 gene encoding a receptor-like protein confers resistance to both defoliating and non-defoliating isolates of, 2012, 7(12): e51091.
[30] IDANHEIMO N, GAUTHIER A, SALOJARVI J, SILIGATO R, BROSCHE M, KOLLIST H, MAHONEN A P, KANGASJARVI J, WRZACZEK M. Thecysteine-rich receptor-like kinases CRK6 and CRK7 protect against apoplastic oxidative stress, 2014, 445(2): 457-462.
[31] MIYAKAWA T, MIYAZONO K, SAWANO Y, HATANO K, TANOKURA M. Crystal structure of ginkbilobin-2 with homology to the extracellular domain of plant cysteine-rich receptor-like kinases, 2009, 77(1): 247-251.
[32] MIYAKAWA T, SAWANO Y, MIYAZONO K, HATANO K, TANOKURA M. Crystallization and preliminary X-ray analysis of ginkbilobin-2 from Ginkgo biloba seeds: a novel antifungal protein with homology to the extracellular domain of plant cysteine-rich receptor-like kinases, 2007, 63(Pt 9): 737-739.
[33] DU L, CHEN Z. Identification of genes encoding receptor-like protein kinases as possible targets of pathogen- and salicylic acid-induced WRKY DNA-binding proteins in, 2000, 24(6): 837-847.
[34] 吴田, 谢从华. 马铃薯蛋白激酶基因启动子的克隆及活性分析中国农业科学, 2011, 44(5): 867-873.
WU T, XIE C H. Cloning and activity analysis of the promoter of potato protein kinase gene, 2011, 44(5): 867-873. (in chinese)
(责任编辑 李莉)
Identification and Expression Analysis ofGene family in Upland Cotton
ZHANG ZhongQi1, WANG Jiao1,2, JIN Wei1, GE DongDong1, LIU Kang1, LÜ FenNi1, SUN Jing1
(1National Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University, Nanjing 210095;2Taiyuan University of Technology, Taiyuan 030024)
【Objective】 The cysteine-rich receptor kinase (CRK) is one of the largest family of receptor-like kinases in plants, and plays important roles in plant growth and development, hormone signal transduction and stress tolerance. So far, few cottongenes have been reported.identification, bioinformatics and expression analysis offamily genes in upland cotton on whole genome level lays the foundation for in-depth study and utilization offamily genes in cotton. 【Method】 The conserved stress-antifung domain sequence downloaded from the Pfam database was used as query sequence to search the(cv. TM-1) genome database to identify cotton CRKs by using BLASTp program; the theoretical isoelectric point and molecular weight, signal peptide, transmembrane domain, subcellular localization of cotton CRK proteins was predicted applying Compute/Mw, SignalP, TMHMM Server V2.0, WoLF POSRT online program respectively; amino acid sequence alignment of CRK proteins in cotton andwas performed using ClustalX1.8 software. phylogenetic relationships of cotton andCRK proteins were analyzed with MEGA5.0; the chromosome location, gene structure and conserved domain were visualized with TBtools. The promoter sequences of cotton CRK genes wereanalyzed by searching PlantCARE database; phosphorylation sites were predicted with PlantPhos. RNA-Seq data were download the from the NCBI database, TPM values were calculated using transcriptome quantification tool Kallisto, the heatmap of CRK gene expression was drawn with online tools Morpheus. 【Result】There are 70genes in upland cotton genome distributed on 14 chromosomes, 52 genes (74.3% of the total) are intensively distributed in clusters on A6/D6, A9/D9 and A10/D10 chromosomes characterized by collinear relationships between A/D chromosomes. Thesegenes encode proteins containing 302-901 amino acids, 58 proteins (82.9%) have a transmembrane domain, mainly located in the chloroplasts, plasmalemma and extracellular. Phosphorylation site prediction results showed that cotton andCRK share 5 consensus phosphorylation sites, including three serine phosphorylation motifs and two threonine phosphorylation motifs. The promoter regions of 65 cotton CRK genes (account for 92.9%) contain at least one stress hormone response element, and 69 (98.6%) genes contain at least one biotic or abiotic stress response element. RNA-Seq data analyses showed that the tissue expression patterns ofcould be divided into three types, and that the expression of somegenes were altered in response to salt, drought, cold, heat stress and inoculation withKleb.was predominantly expressed in roots, stems, leaves, and ovules, but barely accumulated in fibers. ABA, GA3, SA, PEG-6000, NaCl, andVd991 can stimulate rapid up-regulation ofexpression.-silenced cottonby using virus induced gene silencing technology (VIGS) showed increased susceptibility toVd991. 【Conclusion】 There are 70 members offamily gene in the upland cotton genome. They have conserved gene structure and functional domain, diverse tissue expression characteristics; most of cottongenes are responsive to hormone and stress stimulus.
upland cotton; cysteine-rich protein kinase; gene family; stress tolerance;
2018-02-14;
2018-03-28
国家自然科学基金(31371672)、中国博士后科学基金(2016M600423)
张中起,E-mail:2015101104@njau.edu.cn。
刘康,E-mail:kangliu@njau.edu.cn
10.3864/j.issn.0578-1752.2018.13.002

