冠心病患者9p21染色体位点CDKN2A、CDKN2B、MTAP及ANRIL的表达变化
王文杰,申社林
(邢台医学高等专科学校,河北邢台054000)
据我国流行病学资料显示,我国冠心病患病人数已达2.3亿,且年新发病人数高达60万,每天死亡9 590人,是我国首要死亡原因[2]。当前冠心病病因研究虽然并未完全明确,但遗传因素和环境因素是公认的主要病机。环境因素包括性别、年龄、肥胖、高血脂、高血压、不良生活习惯等[3~5],但仅可解释50%~60%冠心病的发病原因,而其余40%~50%的冠心病的发病原因则归因于遗传因素[6],因此,遗传因素方面的病因也是冠心病发病机制研究热点。Shea 等[7]首次报道了白种人冠心病的易感位点基因,如CDKN2A、CDKN2B、MTAP、ANRIL等,而这些基因均为9p21染色体区域相关基因。由于易感位点基因存在人种差异,且国内尚无9p21染色体区域冠心病易感位点基因的相关报道。因此,本研究拟探讨冠心病患者9p21染色体位点CDKN2A、CDKN2B、MTAP及ANRIL的表达变化。
1 资料与方法
1.1 临床资料 选取2017年1~8月邢台市人民医院住院治疗的冠心病患者30例为病例组,以同期健康体检人群30例为对照组。病例组入选标准:①临床症状及心电图显像符合中华医学会心血管病学分会《中国心血管病预防指南》[8]冠心病诊断标准;②冠状动脉造影显示,至少有1支冠状动脉管腔狭窄至少50%;③所有入选冠心病患者无血缘关系;④所有入选冠心病患者民族均为汉族。病例组排除标准:①合并扩展型心脏病、风湿性心脏病等其它心血管疾病患者;②具有严重肝、肾疾病及全身性免疫系统疾病患者;③曾接受静脉溶栓术、心脏大型手术患者。对照组纳入排除标准:均为汉族,病史询问、体检确诊为非冠心病,且本人、家族直系三代无冠心病史。所有研究对象经知情同意后,采集空腹抗凝静脉血2 mL,本研究样本选取及实验检测均经医院伦理委员会批准同意。
1.2 外周血总RNA提取 采用TRIzol法,主要提取步骤为:①新鲜血液2 mL置于离心管内,加入6 mL红细胞裂解液混匀后,4 ℃ 12 000 r/min离心处理2 min,弃上清红色液体;②沉淀部分加入4 mL红细胞裂解液混匀后,4 ℃ 12 000 r/min离心处理2 min,弃上清红色液体;③沉淀部分加入4 mL PBS溶液混匀后,4 ℃ 12 000 r/min离心处理2 min,弃上清液体;④沉淀部分加入1 mL TRIzol溶液混匀,室温下静置10 min;⑤再加入0.2 mL氯仿混匀后,4 ℃下以12 000 r/min离心处理15 min,弃上清液体,保留底部针尖大小白色物质;⑥再加入1 mL的70%乙醇旋转洗涤,4 ℃ 12 000 r/min离心处理5 min,去除乙醇加入30 μL DEPC水融解沉淀,得到外周血总RNA样品液。
1.3 外周血cRNA合成 反转录体系配置:①RNase Free DH2O(20 μL);②Prime Script TM Buffer×5(1 μL);③Prime Script TM RT Enzyme Mix I(1 μL);④Oligo d T Primer(1μL);⑤Random 6 mer(1 μL);⑥Total RNA(500 ng)。将上述反转录体系溶液混匀后,在反应条件(37 ℃ 10 min,85 ℃ 10 s,4 ℃ 1 h)中可合成外周血cRNA。
1.4 引物设计与合成 以9p21染色体区域的CDKN2A基因、CDKN2B基因、MTAP基因和ANRIL基因作为候选基因,以β-actin作为内参基因,采用primer 5.0进行引物设计。其中候选基因β-actin正向引物:5’-ACAGTGGAAGTGGCAAGGTC-3’,反向引物:5’-GTTTCAAGTATTACACCGGC-3’,CDKN2B正向引物:5’-TCAGTCGGCTTCCGAGGTA-3’,反向引物:5’-GTTGCGTGGTTCAATGCC-3’; ANRIL正向引物:5’-GAGAGAACCTTAGGAAACTTTACAG-3’, 反向引物:5’-ACACCATCAATCCCACACCATACAC-3’; MTAP正向引物:5’-ACGAAGGAGCACGAGGAAG-3’, 反向引物:5’-CTATCCCAGGTCTCTTACCAGTC-3’; CDKN2A正向引物:5’-TCAGTCGGCTTCCGAGGTA-3’,反向引物:5’-GTTCCGTGGAGCGCGTA-3’。引物合成送宝生物公司合成。
1.5 各组mRNA表达量测定 PCR扩增体系:①PCR Reverse Primer(0.5 μL);②SYBR Premix Ex Taq TM(12.5 μL);③ROX Reference Dye Ⅱ(0.5 μL);④PCR Forward Primer(0.5 μL);⑤cRNA样品液(2 μL);⑥dH2O(9 μL)。将上述PCR扩增体系溶液混匀后,在实时荧光 PCR扩增仪中以反应条件(95 ℃ 30 s,95℃ 5 s,60 ℃ 20 s)进行反应,共循环40次后,测定两组CDKN2A、CDKN2B、MTAP和ANRIL的mRNA表达水平。
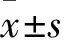
2 结果
与对照组相比,病例组的外周血9p21染色体区域的CDKN2A、CDKN2B mRNA表达水平降低(P均<0.05),MTAP mRNA表达水平升高(P<0.05),ANRIL mRNA表达水平无统计学差异(P>0.05)。见表1。
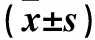
表1 两组CDKN2A、CDKN2B、MTAP和ANRIL的mRNA表达比较
3 讨论
遗传因素是当前冠心病的病因及发病机制研究中的热点方向。家族聚集性是冠心病明显的遗传病特征,即具有冠心病家族史人群的冠心病患病风险是无冠心病家族史人群的12倍,且这种高患病风险存在种族差异[9]。但是冠心病作为一种复杂的多基因遗传病,其显著特点是不符合孟德尔遗传的质量性状变异,存在着数量级差改变,即冠心病病因和发病机制可能存在多种易感基因或易感区域[10],因此冠心病遗传基因研究颇为复杂。但随着人类基因组计划的进展和单核苷酸多态性研究的不断进步,使基于候选基因策略的全基因组关联研究法成为冠心病遗传基因研究的主要方法。全基因组关联研究法又称为连锁不平衡研究法,是一种通过疾病人群和健康对照人群间遗传标记基因表达差异,来分析疾病相关致病基因的有效方法。由于全基因组关联研究法,既不需要了解致病基因在全基因组中的位置,也不需要假定疾病的相关基因,因此在冠心病遗传基因研究中的应用越来越广泛[11]。
当前,国内外学者采用候选基因策略的全基因组关联研究法,已经发现较多与冠心病病情发展相关的基因易感位点和基因易感区域,这无疑是冠心病基因研究的一个进步。而这些基因易感位点和基因易感区域主要集中在9p21染色体区域,使9p21染色体位点成为被广泛关注的热点。本世纪初,《Science》报道了美国、加拿大、英国等学者的相关报道,其研究显示9p21染色体位点是白种人群的冠心病易感位点[12,13]。之后芬兰、瑞典、法国、爱尔兰、日本等国为主的一个国际合作研究小组,分别对自己所在国冠心病人群的9p21染色体区域基因进行了研究,印证了9p21染色体区域基因与冠心病发病的关系[14]。9p21染色体位点具有高度的不平衡特性,目前研究显示,与9p21染色体位点相关联的基因主要包括CDKN2A、CDKN2B、ANRIL 和MTAP[15]。
CDKN2A和CDKN2B基因均属于9p21染色体区域基因编码周期,均属于依赖性蛋白激酶家族成员,主要在细胞周期调节中产生抑制细胞增殖作用,且与冠心病的动脉粥样硬化发病机制密切相关[16]。已经有研究显示CDKN2A和CDKN2B基因位于9p21染色体位点上游,与白种人冠心病发病密切相关[17]。ANRIL基因为9p21染色体位点的一种反义非编码RNA基因,主要表达于血管巨噬细胞、血管上皮组织细胞及动脉粥样硬化板块细胞之中,其通过转录来控制该病细胞增殖通路相关基因的表达[18]。近年研究显示,冠心病患者的单核细胞和动脉粥样硬化斑块组织中,9p21染色体位点的ANRIL 基因呈高表达,提示ANRIL 与冠心病患者动脉粥样硬化有关,推测ANRIL基因可促进9p21染色体变异,从而促进动脉粥样硬的发生[19]。MTAP 基因在甲硫腺苷磷酸化酶的代谢中起重要作用,有文献研究显示MTAP 基因多态性与缺血性脑卒中、心肌梗死的发生具有相关性[20]。因此将9p21染色体位点相关联的CDKN2A、CDKN2B、ANRIL和MTAP基因作为候选基因,探讨其与冠心病的冠心,可以为冠心病临床诊治提供新的靶向基因。本研究结果显示,与对照组相比,冠心病病例组的外周血9p21染色体区域的CDKN2A、CDKN2B mRNA表达水平明显降低,这可能是由于CDKN2A基因和CDKN2B基因作为一种具有抑制细胞增殖作用的基因,其在细胞周期调节中的抑制作用受到限制,因此导致患者冠心病发病风险的增大。MTAP mRNA表达水平明显升高,这可能是因为MTAP 基因作为一种具有促进细胞代谢功能的基因[21],作用于慢性进展性的冠心病,其表达上调加速了冠心病的进展。ANRIL mRNA表达水平无明显差异,提示ANRIL基因可能与冠心病发生发展无关,与国外研究存在差异,原因可能在于人种差异和病例样本较小。
综上所述,冠心病的发生可能与9p21染色体位点CDKN2A、CDKN2B表达下降和MTAP 表达升高有关,9p21染色体位点CDKN2A、CDKN2B和MTAP 有希望成为冠心病临床诊治的潜在靶基因。
参考文献:
[1] Mathers CD, Loncar D. Projections of global mortality and burden of disease from 2002 to 2030[J]. PLoS Med, 2016, 3(11): 442-467.
[2]卫生部心血管病防治研究中心.中国心血管病报告2012[M].北京:中国大百科全书出版社,2013,6:78-79.
[3] Marenberg ME, Risch N, Berkman LF, et al. Genetic susceptibility to death from coronary heart disease in a study of twins[J]. N Engl J Med, 2014,330(15):1041-1046.
[4] Gibbons GH, Liew CC, Goodarzi MO, et al. Genetic markers: Progress and Potential for cardiovaseular disease[J].Circulation, 2016,17(6):112-119.
[5] Nadir MA, Struthers AD. Family history of premature coronary heart disease and risk predietion[J]. Heart, 2015,97(8):684.
[6] Leander K, Hallqvist J, Reuterwall C, et al. Family history of coronary heart disease, a strong risk factor for myocardial infarction interacting with other cardiovascular risk factors: results from the Stoekholm Heart Epidemiology Program (SHEEP)[J]. Epidemiology, 2016,12(2):215-221.
[7] Shea S, Ottman R, Gabrieli C, et al. Family history as an independent risk factor for coronary artery disease[J]. J Am Coll Cardiol, 2016,4(4):793-801.
[8] 中华医学会心血管病学分会,中华心血管病杂志编辑委员会.中国心血管病预防指南[J].中华心血管病杂志,2012,36(1):322-324.
[9] 陈立娟.早发冠心病患者一级亲属心血管危险因素的筛选及其评估[J].中国临床康复,2015,31(4):190-192.
[10] Talmud PJ. Gene-environment interaction and its impact on coronary heart disease risk[J]. Nutr Metab Cardiovasc Dis, 2015, 17(2): 148-152.
[11] 汪云,朱文丽.全基因组扫描定位易感基因的策略和方法[J].中国生育健康杂志,2016,19(6):374-376.
[12] Mc Pherson R, Pertsemlidis A, Kavaslar N, et al. A common allele on chromosome 9 associated with coronary heart disease[J]. Science, 2016,316(5830):1488-1491.
[13] Helgadottir A, Thorleifsson G, Manolescu A, et al. A common variant on chromosome 9p21 affects the risk of myocardial infarction[J]. Science, 2016,316(5830):1491-1493.
[14] Ashokkumar M, Emmanuel C, Dhandapany PS, et al. Haplotypes on 9p21 modify the risk for coronary artery disease among indians[J]. DNA Cell Biol, 2015,30(2):105-110.
[15] Yoshiji Y, Sahoko I, Tamotsu N. Molecular genetics ofmyocardial infarction[J]. Genomic Med, 2047,2(4):7-22.
[16] Karvanen J, Silander K, Kee F, et al. The impact of newly identified loci on coronary heart disease, stroke and total mortality in the MORGAM prospective cohorts[J]. Genet Epidemiol, 2016,33(3):237-246.
[17] Meng W, Hughes AE, Patterson CC, et al. Chromosome 9p21.3 is associated with early-onset coronary heart disease in the Irish population[J]. Dis Markers, 2016,25(2):81-85.
[18] 郭晋,李卫,刘欣,等.染色体9p21和1p13上单核苷酸多态性位点与中国人群急性心肌梗死的关联:中国急性心梗研究[J].第二军医大学学报,2015,32(8):822-828.
[19] Bochenek G, Hsler R, El Mokhtari NE, et al. The large non-coding RNA ANRIL, which is associated with atherosclerosis, periodontitis and several forms of cancer, regulates ADIPOR1, VAMP3 and C11ORF10[J]. Hum Mol Genet, 2013,22(22):4516-4527.
[20] Ahmed W, Ali IS, Riaz M, et al. Association of ANRIL polymorphism(rs1333049: C>G) with myocardial infarction and its pharmacogenomic role in hypercholesterolemia[J]. Gene, 2015,515(2):416-420.
[21] Bataille F, Rogler G, Modes K, et al. Strong expression of methylthioadenosine phosphorylase (MTAP)in human colon carcinoma cells is regulated by TCF1/ [beta]-catenin[J]. Lab Invest, 2015,85(1):124-136.

