基于水汽凝结原理的汽凝仪的开发与应用研究
谢丽萍,钟俊杰,张旭哲,李冠孚
东北大学 中荷生物医学与信息工程学院,辽宁 沈阳 110819
引言
基因检测已成为疾病早期诊断、有害细菌或病毒检测、以及法医学应用的关键技术[1-2]。目前,已经开发出多种用于筛选基因突变的方法,包括等位基因杂交法、核苷酸掺入的引物延伸法、质谱法、核酸测序以及实时定量聚合酶链反应检测等。但是,大多数的方法需要高成本的探针标记以及昂贵的检测仪器(如激光共焦生物芯片扫描仪,CCD生物芯片扫描仪)[3-4]。此外,荧光探针容易漂白,增加了操作的复杂性。
露珠的形成是自然界中一种典型的水汽凝结现象。平时不容易看出的蜘蛛网,挂上露珠后晶莹剔透,在阳光下异常显眼。凝结的露水通过反射光线,使得蜘蛛网相比背景更亮,产生了更强的可视信号。水汽凝结产生的图案提供了一种稳定的、无标记以及无需供能的“绿色”成像方法,常用于玻璃清洁度及均一性检查[5]。随着人们对水汽凝结的兴趣的增长,很多的研究集中于形成水汽凝结的实验方法研究及理论探讨。水汽凝结方法已用于制作各种多孔材料[6],甚至设计可调控的汽凝纳米显微镜[7]。很多研究采用水汽凝结方法识别不同应用目的的自组装分子层排布[8-9]。水汽凝结是一种低成本、无损伤、无需供能的“绿色”方法,可以在短时间内(1 min)区分表面属性,并且具有高度可重复性[10]。
为了降低检测成本,Xie等[11]首次报道了利用水汽凝结信号检测p53基因的249编码区的基因突变。分析物引起的DNA扩增网络改变了检测点的湿度,水汽凝结信号读取方式将芯片的湿度变化转化为可视信号,只需哈一口气,即可实现基因突变的肉眼识别。哈气形成水汽凝结的方法虽然操作简便,但受外界环境的影响较大,在一些恶劣条件下,如高温(>40℃),空气湿度低(<30%)等条件下,哈气不易形成凝结,影响信息的读取。
本项目搭建了便携式汽凝仪,提供了基于汽凝法的基因芯片检测信号输出环境,将超声雾化的原理结合到汽凝仪中,提供了稳定的湿度条件,并可实时采集图像,对采集的图像进行分析,自动定位芯片捕获探针的阵列点,实现了基因点突变信号的读取。
1 系统硬件
1.1 结构设计
汽凝仪由控制分析模块、显示模块、图像采集模块、超声雾化模块组成,见图1。本文采用Solidworks软件设计了便携式汽凝仪的外观以及内部结构(图1a)。完成三维结构设计后,使用光敏树脂为原材料,利用3D打印机打印出仪器的整体结构。汽凝仪的外观,见图1b。特别强调的是,为防止超声雾化模块对其他模块的影响,采用了分隔设计,图像采集模块在超声雾化模块的正上方,与汽凝仪其他模块分开,通过隔板隔离(图1c),确保了超声雾化模块不会将水雾喷洒到微型控制器上而导致系统发生故障。超声雾化模块的上面设置载物台(图2a),放置待测芯片。超声雾化模块的储水箱的设计利用了蓄水的原理。从载物台的右侧注水口注入纯净水,左端插入雾化片(图2b),并采用密封胶密封,形成水雾的供给源。设置5个喷口,采用弯形喷嘴设计,以增大喷雾面积及分散喷雾强度。雾化片接触水面产生水雾,通过出雾口喷出,增加了芯片表面空气的湿度,为基于汽凝法的基因芯片的检测提供了水汽凝结的检测环境。
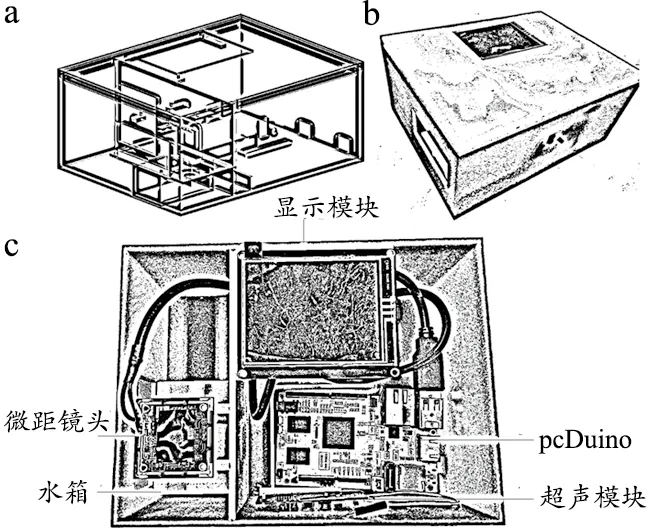
图1 汽凝仪结构示意图

图2 汽凝仪的超声雾化模块的结构示意图
1.2 硬件功能
汽凝仪的硬件部分由pcDuino微型控制器、超声雾化模块、图像采集模块和显示模块等构成(如图1c)。pcDuino3是搭载AllWinner 双核A20芯片的微型电脑[12-13],有最高达1 GB的RAM,机身自带有4 GB内存,可通过microSD存储卡扩充至32 GB,能根据项目的需求自行选择搭载Ubuntu、Android等多种操作系统。该系统完全满足了汽凝仪对硬件的功能需求。Ubuntu系统能较为容易的适配摄像模块和显示模块的相关驱动,方便后续的工作。更为重要的是,相比于树莓派等其他微信电脑,pcDuino自身搭载了Arduino开发环境,完全兼容Arduino的开发接口,便于功能模块的扩展,如加湿模块就可以使用Arduino串口方式供电或者操控,而不仅仅是依赖于pcDuino本身自带的USB接口供电。pcDuino构成了汽凝仪的核心控制模块。
汽凝仪的超声雾化模块的功能是提供一个稳定的水汽环境。本文应用家用加湿器工作方式,通过超声产生水雾环境。本文设计的超声波电路利用超声雾化片产生雾化的水汽,通过特定的通道将雾化的水汽集中引向生物芯片上方,增加了空气湿度,使水汽可以凝结在芯片表面,以便观察实验结果。超声雾化模块由陶瓷雾化片及其驱动电路以及储水箱构成。雾化片放置在储水箱中,采用密封胶密封好后,注入水,雾化片接触到水,便可以产生水雾。雾化片的驱动电路实现出雾量的控制。其驱动电路包括稳压部分、升压部分以及单片机部分。通过升压电路和5 V的稳压电路维持单片机以及雾化片正常工作电压,然后通过预设程序让单片机输出一定频率的方波,通过陶瓷雾化片的高频振荡使水雾化,出雾量稳定,操作方便。
图像采集模块采用500万像素的微距摄像头,可以较为清晰的采集到芯片上的照片。因为镜头与芯片距离固定,焦距不会变化,采用手动对焦方便快捷,故在装置安装好以后需要调好焦距再进行照片的采集。显示模块采用IPS(平面转换)电阻触摸屏显示采集的芯片图像。
2 DNA芯片图像处理算法
为了实现基因芯片的阵列点的自动识别分析,本文开发了基因芯片图像处理算法。首先通过图像预处理方式进行除噪,然后将图像中的反应点都分割出来,对分割后的图像进行判断。如果没有不需要的噪点,便可以将检测点的位置信息保存下来,将这些位置信息和预先给定的芯片反应点位置信息进行比对,识别哪些位点发生了特异性反应从而得出该芯片的实际反应结果。具体操作如下:第一步是将采集到的原始图像转化为8位的灰度图,用圆盘结构体对灰度图像进行一次腐蚀操作,接着对其进行形态学开重建,再对其进行一次膨胀操作,最后进行形态学闭重建。经过上述步骤,可得到大部分小的噪点被消除的灰度图像。尽可能消去噪点后,需要将反应点从预处理后的灰度图中提取出来。扩展极小变换是一种相对精确的求区域极小的算法,相比于直接求区域极小变换,扩展极小变换可以抑制那些与周围区域相比对比度较小(小于阈值)的极小点,本文记录下区域边界的灰度值,然后给整个区域加上一个阈值,对于加阈值后灰度值大于等于原边界灰度值的,将其置成原边界灰度,以有效改善区域极小变换过分割的情况。相比一般的分水岭算法,扩展极小变换能更好地将图像中的亮点区分开,不留下过多的噪点。在得到黑白二值化的图像后,本文会先根据留下的白点大小进行一次去噪,将明显的噪点去除。同时,因为芯片上的阵列点是呈垂直对应的关系,根据这个特征,可以先对一定数量的图像进行统计,判定出反应点之间的距离D。由于芯片距离摄像头的位置是不会改变的,故图像中反应点之间距离D是一个定值。在经过基于面积中值的去噪后,可通过对比每个点圆心为中心的x、y轴方向上距离为D的是否存在其他反应点作为判断的依据。若其在x、y轴4个垂直方向的一定距离内不存在其他对应关系的点,那么就判定这个点是噪点,再进行一次去噪。经过两次去噪,能基本全部除去原图像上的噪点,只留下芯片上反应的检测点。标记识别出的基因点突变反应点,从而完成基因点突变自动识别分析,此算法能精确的区分反应点,算法识别准确度高。
3 验证实验
由博奥生物芯片有限公司制备突变捕获探针(5 μm)阵列,各个检测点之间距离200 μm。将100 nM的待检测的基因突变片段(表1中野生序列、突变序列)、5 μm报告探针和0.8 U/μL Taq DNA连接酶组成的混合液 5 μL,加入到芯片表面,60℃下反应1 h。之后浸泡在0.01 M NaOH溶液中5 min。去除反应液,向芯片表面加入10 μM环模板、10 μM链条与0.2 μL的T4 DNA 连接酶混合液,室温反应2 h。取5 U/μL Phi29 DNA聚合酶、缓冲液以及880 μM dNTP)5 μL加入到芯片表面,30℃反应4 h。采用本文汽凝仪观察记录检测结果。
4 结果与讨论
为了验证本文汽凝仪的功能,进行了人体抑癌基因p53的220编码区的基因突变检测实验。p53是目前世界上研究较为广泛的抑癌基因。它是生物体中一种抑制正常细胞转变为癌细胞的基因,在控制细胞生长,增殖及凋亡方面充当着重要作用。p53抑癌基因突变可能会引发肿瘤产生。检测抑癌基因p53的基因点突变的序列设计,见表1。设计原则遵循:捕获探针与报告探针无互补杂交,即捕获探针存在时,不能与报告探针形成非特异性连接,保证只有目标基因存在时才能产生信号的输出。检测原理,见图3。
首先是芯片的制备,通过点样仪将捕获探针通过5’端的氨基与环氧硅烷修饰的SiO2修饰的硅片连接,每个基因点间距为200 μm,每个基因点直径约为50 μm。待分析物与报告探针一起加入到芯片上,当待分析物与捕获探针、报告探针能无差错的碱基互补配对,则将报告探针与捕获探针连接起来。若当待分析物与捕获探针、报告探针有一个碱基的错配,报告探针无法连接捕获探针。之后,向芯片上加入环(借助链条DNA使环形模板连接成环状)。利用滚环扩增技术扩增出大量的DNA[14-15]。由于DNA网络具有亲水性,特异性识别的区域的湿润度发生改变,无目标检测基因的检测点,由于无法连接报告探针,不能进行RCA扩增,检测点湿润度不发生改变。汽凝仪的超声雾化片产生的水雾遇到芯片表面,在特异性识别区域(即湿润度改变区域)形成一层水膜[16],通过光的反射现象,可以将DNA的一个碱基的基因突变事件转变为可视光线,形成可视的光斑。光斑与DNA的碱基突变事件正相关,即若待检测物中含有目标的DNA序列,芯片检测点处扩增出大量DNA,改变了此检测位点的亲水性,水汽凝结在表面形成的水膜对该位点起到了放大作用,通过微距镜头采集由显示模块显示采集结果。

表1 单链核苷酸序列
本文采用基因芯片用以检测抑癌基因p53的220编码区的基因突变(TAT > -TGT)。固定了野生基因TAT、突变基因TGT的捕获探针的芯片,通过基因特异性识别来检测相应的野生基因TAT、突变基因TGT。最后通过汽凝仪进行信号采集和输出。水汽凝结后的芯片检测结果输出,见图4a,前两行为特异性识别野生基因TAT的结果,后两行为特异性识别突变基因TGT的结果。通过基因芯片图像处理算法,可实现自动识别特异性检测区域,并准确的圈出特异性信号输出区域(图4b)。为了进一步突出显示特异性识别区域,以黑白二值化突出显示检测结果(图4c)。与芯片基底固定相应探针的位置对比,可以确切得出目标基因的出现,即使有一个基因点突变,该方法也可以特异性识别出来。汽凝仪为基于汽凝法检测基因突变提供了湿度环境,并可以采集图片,实现图片基因突变检测点的自动识别。相比于基于荧光的芯片检测仪器,大大降低了成本[17-18]。
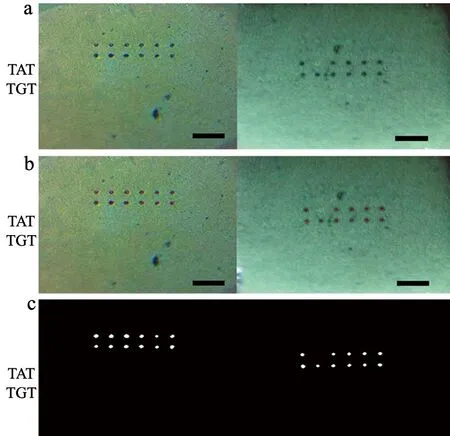
图 4 基于汽凝法的基因芯片检测的照片(标尺为400 μm)
5 结论
本文开发了一种新型的基于水汽凝结原理的汽凝仪,成本低于500元,结合RCA扩增的基因点突变识别技术,可实现基因点突变的汽凝法检测。该系统集成了控制模块、显示模块、超声雾化模块、数据采集模块,并通过3D打印技术,构建了汽凝仪的外观结构。此外,开发了用于自动识别基因点突变的检测算法。本文采用此设备检测了p53的220编码区的TAT到TGT突变识别验证实验表明了本文汽凝仪的数据采集和显示功能并成功用于芯片基因点突变检测。
[参考文献]
[1] Zhang H,Liu X,Liu M,et al.Gene Detection: An Essential Process to Precision Medicine[J].Bios Bioel,2017,99:625.
[2] Gao Y,Deng L,Yan Q,et al.Single molecule targeted sequencing for cancer gene mutation detection[J].Sci Rep,2016,6(1):26110.
[3] Tang X,Morris SL,Langone JJ,et al.Microarray and allele specific PCR detection of point mutations in Mycobacterium tuberculosis genes associated with drug resistance[J].J Microbiol Methods,2005,63(3):318-330.
[4] Zhang ZW,Zhou YM,Zhang Y,et al.Sensitive detection of SARS coronavirus RNA by a novel asymmetric multiplex nested RTPCR amplification coupled with oligonucleotide microarray hybridization[J].Methods Mol Med,2005,114(3):59-78.
[5] Aitkek J.Breath figures[J].Nature,1911,86(2172):516-517.
[6] Bai H,Du C,Zhang A,et al.Breath figure arrays: unconventional fabrications, functionalizations, and applications[J].Angew Chem,2013,52(47):1240-1255.
[7] Mcleod E,Nguyen C,Huang P,et al.Tunable vapor-condensed nanolenses[J].Acs Nano,2014,8(7):7340.
[8] Erath J,Cui J,Schmid J,et al.Phototunable Surface Interactions[J].Lang Acs J Surf Coll,2013,29(39):12138-12144.
[9] López GP,Biebuyck HA,Frisbie CD,et al.Imaging of features on surfaces by condensation figures[J].Science,1993,260(5108):647-649.
[10] Hofer RM,Textor A,Spencer ND.Imaging of surface heterogeneity by the microdroplet condensation technique[J].Langmuir,2001,17(13):4123-4125.
[11] Xie L,Wang T,Huang T,et al.Dew inspired breathing-based detection of genetic point mutation visualized by naked eye[J].Sci Rep,2014,4:6300.
[12] PCDuino User Guide (n.d.)[EB/OL].[2013-03-08].http://www.doc88.com/p-9883114996220.html.
[13] Gómez A,Cuiñas D,Catalá P,et al.Use of single board computers as smart sensors in the manufacturing industry[J].Proc Engin,2015,132:153-159.
[14] Schweitzer B,Roberts S,Grimwade BW,et al.Multiplexed protein profiling on microarrays by rolling-circle amplification[J].Nat Biotech,2002,20(4):359-365.
[15] Xie L,Cheng H,Qi H,et al.Nanostructural morphology masterregulated the cell capture efficiency of multivalent aptamers[J].Rsc Advances,2015,5(50):39791-39798.
[16] Muller PA,Vousden KH.p53 mutations in cancer[J].Nat Cell Biol,2013,15(1):2-8.
[17] Duffy MJ,Synnott NC,Crown J.Mutant p53 as a target for cancer treatment.[J].Eur J Cancer,2017,83:258-265.
[18] Wen L,Tian Y,Jiang L.Bioinspired super-wettability from fundamental research to practical applications[J].Angew Chem,2015,54(11):3387.

