海南产Ganoderma lingzhi种质的鉴定
, , , ,
(海南热带海洋学院生命科学与生态学院, 海南 三亚 572022)
灵芝是一类腐生型真菌,以腐解木质素获取营养,在分类学上将灵芝归为多孔菌目,灵芝菌科,灵芝属。野生灵芝菌盖为半圆形、圆形、扇形等,常带似漆样光泽;菌肉颜色有淡黄色、黄褐色等[1]。由于灵芝生长环境多样,造成了在生活史的不同阶段均表现出丰富的形态,因此仅通过形态观察难以对其进行鉴定,往往需要结合系统发育学分析才能有效地对菌株的分类学地位进行界定[2]。近年来,形态学结合系统发育学鉴定已广泛应用于灵芝分类。Hong等[3]采用mt SSU片段将37株灵芝分为5个类群,提出了亚洲原产赤芝类群。Cao等[4]通过ITS序列结合形态分析重新对灵芝属下的菌株进行鉴定,提出了G.lingzhi这一新的种群,明确了灵芝属下不同种的界定,同时也为今后灵芝分类提供理论依据。G.lingzhi至今已在我国台湾、辽宁、江苏、浙江、湖北、山东、安徽、河南、天津、广东、湖南、云南、四川、福建及日本发现有分布,然而据笔者了解,海南至今未见这个种的分布报道。本研究采用ITS、mt SSU序列构建系统发育树结合形态分析,对收集自海南的3株灵芝种质进行分类鉴定,明确其为G.lingzhi,进一步拓展了这个种在我国的分布范围。
1 材料与方法
1.1 灵芝的收集及形态观察
2015年5—10月于海南尖峰岭地区收集灵芝种质资源,进行形态观察,4 ℃冷藏保存。
1.2 基因组DNA提取、目的片段扩增及测序
基因组DNA提取及目的片段扩增参照Chen等[5]方法。ITS片段扩增参照White等[6]方法。采用引物CFMT (5′-TCCGTAGGTGAACCTGCGG-3′)和CRMT (5′-TCCTCCGCTTATTGATATGC-3′)扩增完整的mt SSU V 3,V 4区及部分V 2区。PCR产物委托北京华大基因科技股份有限公司测序。
1.3 系统发育学分析
根据ITS、mt SSU片段测序结果,Gen Bank中进行Blast比对,搜索同源序列。选取不同种的代表性菌株进行系统发育学分析。采用Clustalx 1.83[7]将序列对齐,利用MEGA 5.05[8]软件以最大简约法构建系统发育树,并以自展法(bootstrap)进行检测,循环1 000次。
2 结 果
本研究中3个菌株在形态上菌盖呈半圆形,深红色或紫红色,菌盖表面光滑(图1);有菌柄;担孢子长宽分别为8.3~8.7μm和6.4~9.4μm,这些形态特征与G.lingzhi相似(表1)。系统发育学分析表明,根据ITS序列和mt SSU序列构建的最大简约树具有相似结构。ITS最大简约树:步长=107;一致性指数(CI)=0.687;总留存指数(RI)=0.908;mt SSU最大简约树:步长=41;一致性指数(CI)=0.827;总留存指数(RI)=0.925。除了模式菌种G.lucidum遗传关系比较分散外,不同种灵芝在系统发育树上具有明显聚类,同种灵芝聚为一支。其中在基于ITS序列构建的系统发育树上,菌株Jfl 1502、Jfl 1513、Jfl 1515与G.lingzhi聚为一支,自展值为92;在基于mt SSU序列构建的系统发育树上,菌株Jfl 1502、Jfl 1513、Jfl 1515与G.lingzhi聚为一支,自展值为62;显示了这3个菌株与G.lingzhi具有较近的亲缘关系。

注:a为Jfl 1502;b为Jfl 1513;c为Jfl 1515。图1 灵芝子实体的形态特征
表1 灵芝的形态特征

菌株菌盖颜色菌柄颜色孔口颜色 菌盖特征担孢子(μm)Ganodermasp.Jfl1502深红色深红色淡黄色表面光滑、半圆形(8.3~8.7)×(6.4~9.4)Ganodermasp.Jfl1513深红色深红色淡黄色表面光滑、半圆形(8.5~9.3)×(5.7~6.2)Ganodermasp.Jfl1515深红色深红色淡黄色表面光滑、半圆形(9.0~9.6)×(5.6~8.1)Ganodermalingzhi淡黄色、黄色、土黄色黄色、紫红色淡黄色、稻草黄表面光滑、半圆形、扇形[(8~9)~(10.7~12)]×[(5.2~5.8)~(7~7.5)]
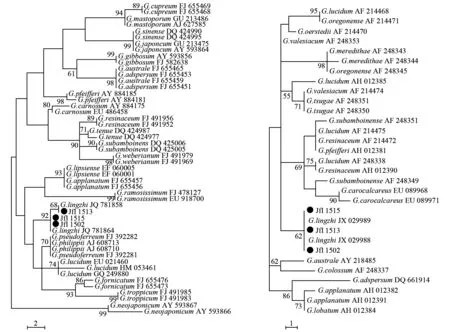
注:1 000次重复,自展值>50%。图2 根据ITS和mt SSU序列构建的灵芝最大简约树
3 讨 论
本研究中3个灵芝子实体形态相似,菌盖深红色、表面光滑、半圆形,这些特征与G.lingzhi菌盖形状相似,但与其常见的淡黄色至土黄色菌盖颜色有差异[4]。然而灵芝生长在森林中,气候多样,这可能造成灵芝菌盖颜色的差异[9]。本研究中灵芝来源于海南尖峰岭地区,雨水充足,而与至今在海南以外省份发现的G.lingzhi生长环境存在差异,这些也可能造成了菌盖颜色的不同[4]。系统发育学分析显示,根据ITS序列和mt SSU序列构建的系统发育树结构相似,不同种灵芝分别独立聚类,其中3株菌株Jfl 1502、Jfl 1513、Jfl 1515与G.lingzhi聚为一支,并具有较高的自展值支持,反映了3株菌株与G.lingzhi亲缘关系紧密。结合系统发育树和形态特征的结果表明,这3个菌株在分类地位上属于G.lingzhi。这个结果也反映了G.lingzhi北至辽宁,南至海南均有分布,展现了其分布的广泛性。
参考文献:
[1]吴兴亮,戴玉成.中国灵芝图鉴[M].北京:科学出版社,2005:1-14.
[2]Alxandra M G,Jorge E W.Taxonomy ofGanodermafrom southern South America:subgenusGanoderma[J].Microbiological Research,1999,103(6):661-673.
[3]Hong S G,Jung H S.Phylogenetic analysis ofGanodermabased on nearly complete mitochondrial small-subunit ribosomal DNA sequences[J].Mycologia,2004,96(4):742-755.
[4]Cao Y,Wu S H,Dai Y C.Species clarification of the prize medicinalGanodermamushroom “Lingzhi”[J].Fungal Diversity,2012,56:49-62.
[5]Chen Y G,Ji Y L,Yu H S,et al.A newNeotyphodiumspecies fromFestucaparviglumaSteud.grown in China[J].Mycologia,2009,101(5):681-685.
[6]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetic.In:Innis MA,Gelfand DH,Sninsky JJ,White TJ (eds)PCR protocols:A guide to methods and applications[M].Academic Press,New York.1990:315-322.
[7]Thompson J D,Higgins D G,Gibson T J.Clustal W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Res,1994,22:4 673-4 680.
[8]Tamura K,Peterson D,Peterson N,et al.MEGA 5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods[J].Mol Biol Evol,2011,28:2 731-2 739.
[9]Pilotti C A,Sanderson F R,Aitken E A B,et al.Morphological variation and host range of twoGanodermaspecies from Papua New Guinea[J].Mycopathologia,2004,158:251-265.
各类文献规范数据选项表
注:“√”为必备项,“0”为任选项,“*”为有则加项,“-”为不要求项。

