峨眉山区桑科药用植物ITS2条形码序列鉴定*
任瑶瑶,张 良,谢博文,江南屏,刘睿颖,谭 睿
(西南交通大学生命科学与工程学院 成都 610031)
峨眉山位于四川盆地边缘亚热带季风气候区,峨眉山最高海拔为3 099 m,山顶山麓相对高差近2 600 m,气候悬殊,山顶和山麓温差大,气温垂直变化显著,因而其植被分布具有明显的垂直分布性,加之峨眉山区降雨量丰富,空气相对湿度达80%以上,使得该区域植物资源极为丰富,多种自然要素的交汇,形成了峨眉山丰富的植物种类和复杂的区系成分[1,2]。
桑科(Moraceae)植物约有53属1 400种,多产于热带、亚热带,少数分布在温带地区。我国约有12属153种和亚种,并有变种及变型59个[3]。桑科植物中具有药用价值的桑科植物约55种,在我国国民经济中具有重大意义[4]。构属(Broussonetia)药用植物具有增强免疫[5]、消除自由基[6]、降血脂等作用[7];桑属(Morus)植物降血糖作用[8]、降压降血脂作用、抗氧化作用[9]抗菌和抗病原微生物作用[10];榕属(Ficus Linn)植物有降血糖[11]、镇痛抗炎[12]、抗菌[13,14]、增强免疫系统的作用。峨眉山有丰富的桑科药用资源,桑科中许多的用植物具有药食两用价值,峨眉山常见的桑科药用植物有桑(Morus alba)、地果(Ficus tikoua)、黄葛树(Ficus virens)、冠毛榕(Ficus gasparriniana)、异叶榕(Ficus heteromorpha)、葎草(Humulus scandens)、小构树(Broussonetia kazinoki)、构树(Broussonetia papyrifera)等,且分布广泛,资源较为丰富。
运用DNA条形码技术对药用植物进行鉴定是现在中药鉴定研究的热点[15]。陈士林等[16]首次提出将ITS2序列作为药用植物鉴定的通用条形码序列。本实验采集了峨眉山区不同地区的桑科植物样本,并从Genbank下载同科或同属ITS2序列,以ITS2序列作为DNA条形码,对所有序列进行遗传距离、barcoding gap、Neighbor Joining(NJ)系统进化树和ITS2序列二级结构分析,对桑科药用植物进行鉴定。通过DNA条形码技术对桑科植物的分类进行研究,对具有药用价值的桑科植物的研究提供基础,同时也为进一步对峨眉山药用植物资源的分布环境、种群量、种群动态等方面的调查分析奠定基础。
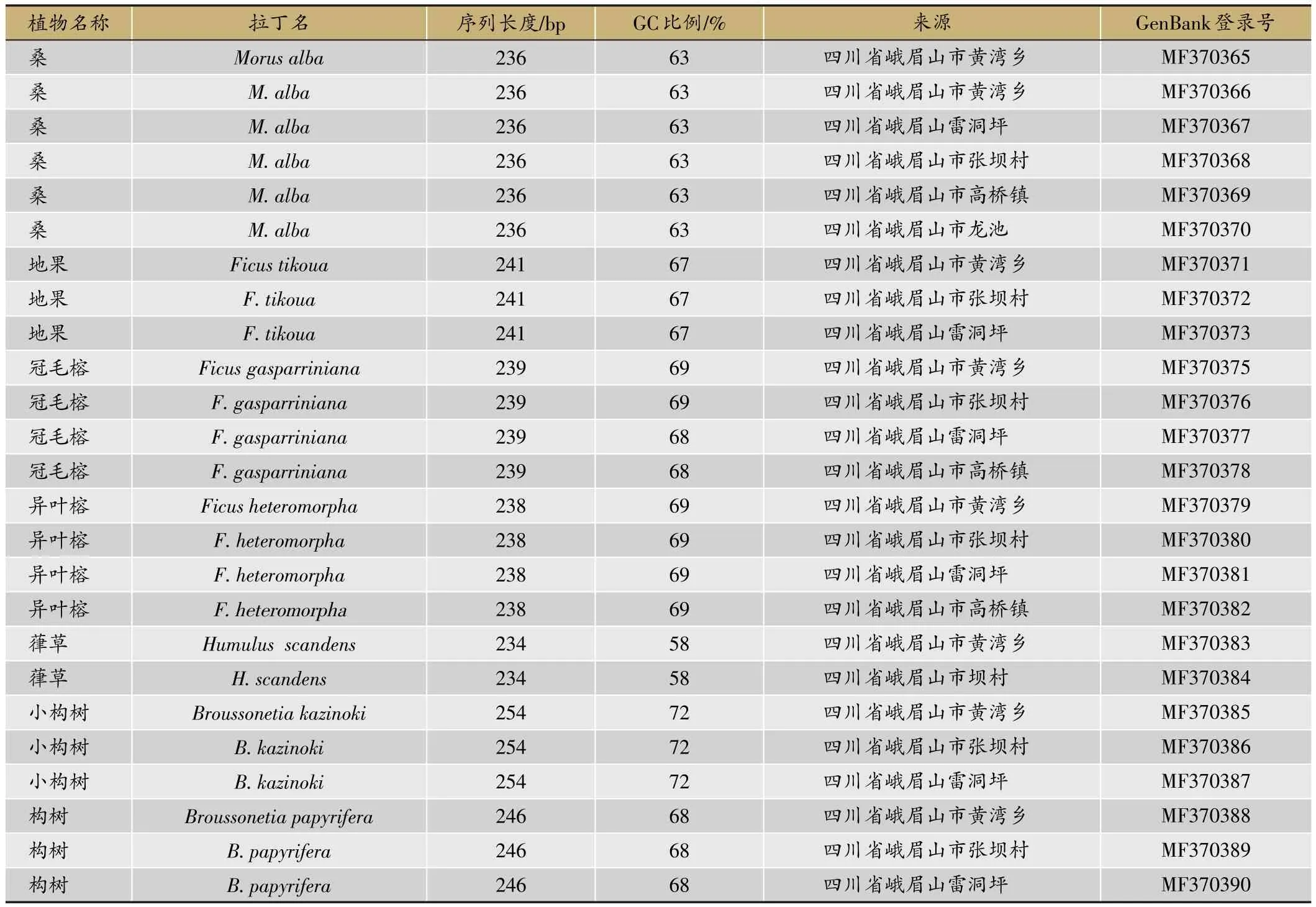
表1 桑科植物样本信息
1 材料
从四川省峨眉山区采集了25份桑科植物样本,经西南交通大学生命科学与工程学院宋良科副教授鉴定,其中桑样品6份、地果样品3份、冠毛榕样品4份、异叶榕4份样品、葎草样品2份、小构树样品3份、构树样品3份样品信息见表1。标本保存于西南交通大学标本室。同时在GenBank数据库中下载桑科植物ITS2序列23条,见表2。
2 方法
2.1 样品DNA提取、PCR扩增及测序
参照任瑶瑶等[18]文章中样品DNA提取、测序等方法,对所采集的样本进行基因组DNA提取、ITS2片段PCR扩增及测序。
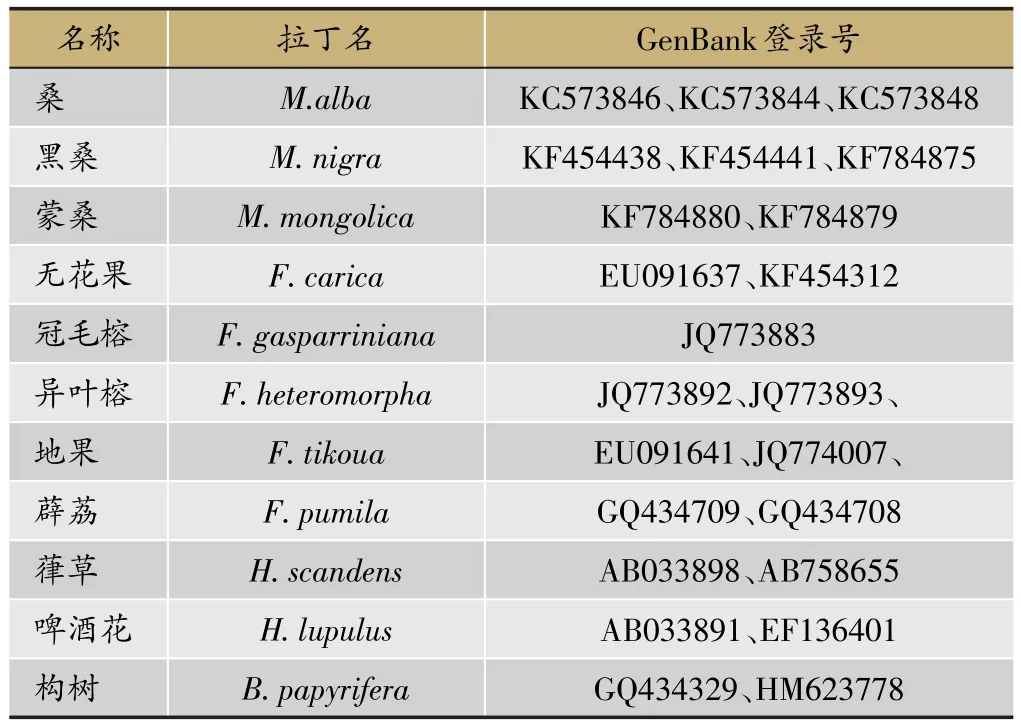
表2 GenBank下载桑科植物ITS2序列
2.2 数据处理
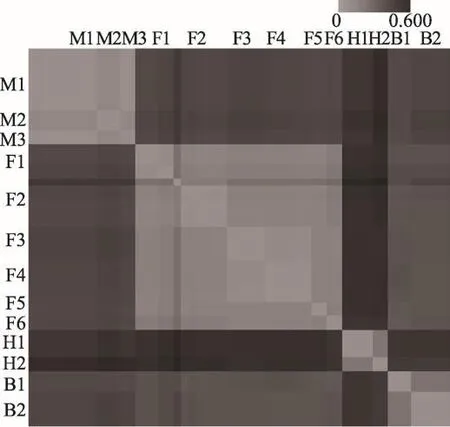
图1 桑科植物样本种间和种内距离
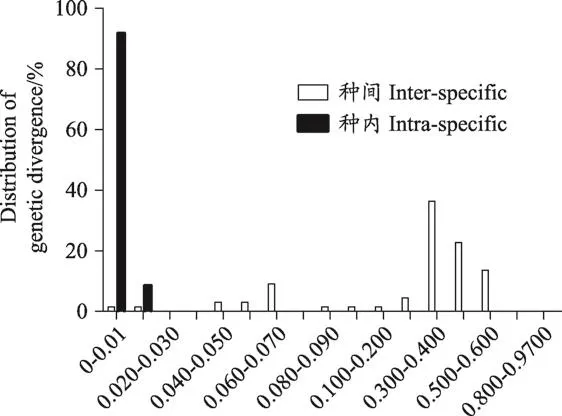
图2 桑科药用植物样本ITS2序列种内和种间变异分布
采用CodonCode Aligner 6.0.2软件对所得序列峰图进行校对拼接,去除引物区和低质量的序列,由基于隐马尔可夫模型(HMMer)的注释方法切除ITS2两端的5.8 S和28 S区域,获得标准的ITS2间隔区序列[19]。所有ITS2序列用MEGA 6.0比对[20]进行种内、种间的遗传变异分析[21]、计算K2P(Kimura 2-parameter)遗传距离、相邻结合法(Neighbor Joining,NJ)构建系统进化树[22],利用Booststrap(1000重复)检验各分支的支持率。利用TAXON DNA 1.8和DNAMAN 8.0软件对所有样本种内、种间遗传距离进行分布频度及样本序列的变异程度进行分析,评价Barcoding gap。根据Koetschan等[23]建立的ITS2数据库网站(http://its2.bioapps.biozentrum.uniwuerzburg.de)预测不同种属样本ITS2序列的二级结构。
3 结果
3.1 变异位点及种内、种间K2P遗传距离分析
所有ITS2序列长度为234-254 bp、GC含量58%-72%(见表1)。应用MEGA6.0软件对所有样品及下载序列进行多序列比对,全序列排序比对后序列长度为274 bp,种内变异位点少,种间变异位点多,其中变异位点有185个,简约性信息位点166个,分别占总序列长度的67.52%和60.58%。
物种间种内距离越小、种间距离越大越适合条形码鉴定[18]。参照陈士林等方法[16],应用MEGA 6.0软件基于K2P距离模型计算样本种内、种间变异距离值,结果见图1。DNAMAN软件对比序列发现:M.mongolica与M.alba种类序列相似度为100%,种间序列相似度为99.89%,两者种间遗传变异较小;而F.tikoua种内序列相似度为99.50%,其种内变异程度(K2P 0.012)大于M.mongolica与M.alba种间变异程度(K2P 0.005)。此外所有样本种内最大变异值为0.0049,所有样本种间最小遗传距离为0.041;由热图可见本实验中桑科样品种内、属内颜色浅,其K2P遗传距离较小;各属之间遗传距离较大,为图中颜色较深区域;葎草属与其他属样本种间遗传变异值较大,在热图中所对应的区域颜色最深。分析统计样本种内和种间遗传距离分布(图2)发现:样本间存在较为明显的barcoding gap,说明ITS 2序列对桑科样本在种水平上鉴定能力较强;种间变异距离值主要集中在0.300-0.600区间,主要是构属、葎草属、榕属之间的遗传变异距离值组成。
3.2 NJ树分析
利用MEGA 6.0软件根据Kimura 2-parameter model对所有ITS2序列样本构建Neighbor-joining(NJ)系统聚类树(图3),从NJ进化树图可以看出,各样本属内各单据一支,分别为榕属、桑属、构属、葎草属,分支之间靴带检验法(bootstrap)支持率在90%以上;各属内种内之间又各聚为小枝,支持率在85%以上;由NJ进化树可见,ITS2序列能将桑科样本在种水平上很好地分开,鉴定成功率为100%。
3.3 ITS2二级结构的分析
桑科植物样本ITS2序列的二级结构如图4所示,从样本的二级结构中可以看出,ITS2二级结构相互之间均有差异,其均为一个中心环及4个螺旋区(Helix)组成,中心环大小、形状形成差异,在螺旋区茎环(Loop)数量、形状及大小形成差异,四个螺旋区的夹角也有明显差异。F.pumila、H.scandens、M.alba、B.papyrifera的中心环为异形,四者二级结构主要差异在于Ⅱ螺旋区茎环的大小数量差异。因此通过ITS2二级结构可将桑科样本中各个种较为直观地鉴别开来。
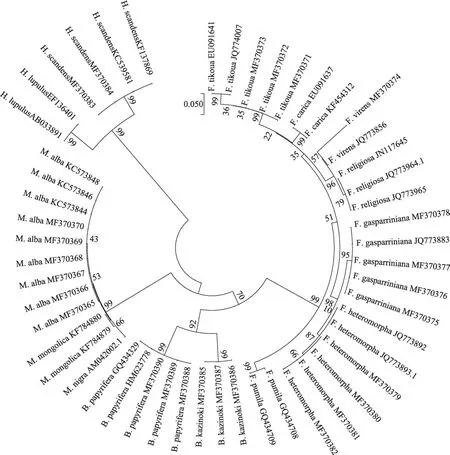
图3 基于ITS序列构建的桑科植物样本的NJ树
4 结论
DNA条形码技术旨在建立一种准确、方便、快捷且对技术要求不高的一种生物分类鉴定方法,ITS2序列对植物具有良好的扩增成功率和物种水平的鉴定成功率[24,25],以ITS2序列作为DNA条形码对桑科植物的鉴定进行研究可以达到即快速又准确。本研究将峨眉山区不同地方的25份桑科植物样本的ITS2序列分析,桑科样本ITS2序列变异位点和简约性信息位点在全序列中占比67.52%和60.58%,说明ITS2序列中存在大量的物种分类及进化信息,在对K2P遗传距离分析通过桑科植物样本种间和种内距离的热图可以看出种内颜色较浅、属内颜色次之,种内和种间变异分布有明显的barcoding gap,NJ树可以看出桑科不同属亲缘关系较远各聚一大支,而同属之间亲缘关系比较近,但是不同种也很明显的各聚一小支。综合所有样本ITS2序列种内、种间的变异距离值、barcoding gap分析、NJ树构建及二级结构预测等多种方法从不同方面对考察了ITS2条形码序列对桑科植物鉴定能力,结果均表明,以ITS2序列为DNA条形码对桑科药用植物种水平上的鉴定能力强。
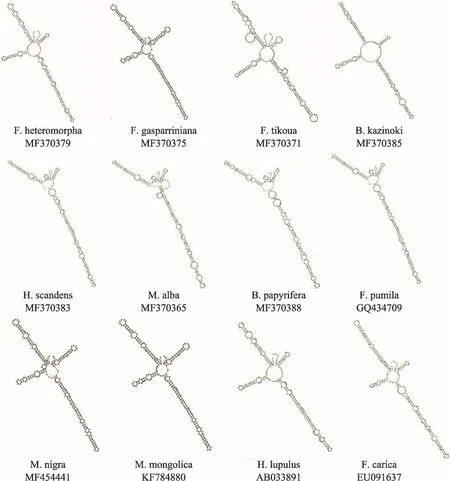
图4 桑科ITS2序列的二级结构
峨眉山资源植物品类齐全,尤其是在药用、食用等方面的植物资源最为丰富多样,是我国资源植物的重要野外基因库,为当地的社会与经济发展提供了独特的、重要的支撑[26]。利用DNA条形码技术,对资源植物进行快速、准确的鉴定,对峨眉山地区药用植物资源进行广泛地调查和分析,并对其进行认识、保护和合理开发利用。药用植物资源的鉴定工作固然重要,但资源管理也相当重要,将二维DNA条形码应用于中药材鉴定,对中药材流通的数字化监管[27]的思路可借鉴到对药用资源的调查中,结合分布环境,弄清种群量和种群动态等问题,正确的评价资源植物当前的状况,在此基础上制订出切合实际的保护及合理开发利用的措施。
1 胡霞,曾沙.峨眉山不同植被类型冬季大型土壤动物群落结构.重庆师范大学学报,2017,34(2):96-101.
2 胡霞,蔡霜,廖金花,等.峨眉山不同海拔森林土壤微生物和酶活性特征.重庆师范大学学报,2016,33(1):109-114.
3 中国科学院中国植物志编辑委员会.中国植物志第二十三卷第一分册.北京:科学出版社,1998.
4 郑汉臣,蔡少青.药用植物学与生药学(第四版).北京:人民卫生出版社,2003.
5 渠桂荣,张倩,李彩丽.构树的药理与临床作用研究述略.中国药学刊,2003,21(11):1810-1811.
6 Adhikrao V Y,Vandana S N.Anti-dopaminergic effect of the methanolic extract of MorusalbaL.leaves.Indian J Pharmacol,2008,40(5):221-226.
7 张尊祥,戴新民,杨然,等.楮实对老年痴呆血液LPO、SOD和脂蛋白的影响.解放军药学学报,1999,15(4):5-7.
8 陈福君,卢军,张永煜.桑的药理研究-桑叶降血糖有效组分对糖尿病动物糖代谢的影响.沈阳药科大学学报,1996,13(1):24-27.
9 Chulaluck C,Plernchai T,Saowaluk R,et al.Application of Mulberry(MorusalbaL.)for Supplementing Antioxidant Activity in Extruded Thai Rice Snack.Kasetsart J,2008,42:79-87.
10 Tankasugi M,Anetaki M,Masamunet T,et al.Studies on phytoalexins of the Moraceae.5.Broussonins A and B,new phytoalexins from diseased paper mulberry.Chem Lett,1980,(3):339-340.
11 Ghosh R,Sharatchandra K H,Rita S,et al.Hypoglycemic Activity of Ficus hispida(bark)in normal and diabetic albino rats.Indian J Pharmacol,2004,36(4):222-225.
12程辉,莫石林,韦春,等.细叶榕水提物镇痛抗炎作用的实验研究.右江民族医学院学报,2007,5:695-697
13 Tanvir S,Ruksana R,Kiran B,et al.Anti-Bacterial activity of Ficus racemosa Linn.Leaveson Actinomyces viscosus.J.Pharm.Sci.&Res,2010,2(1):41-44.
14 Sivaraman D,Muralidharan P.Anti-ulcerogenic evaluation of root extract of Ficus hispida Linn.In aspirin ulcerated rats.Afr J Pharm Pharmacol,2010,4(2):79-82.
15赵莎,庞晓慧,宋经元.应用ITS2条形码鉴定中药材合欢皮、合欢花及其混伪品.中国中药杂志,2014,39(12):2164-2168.
16 Chen S L,Yao H,Han J P,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species.PLoS ONE,2010,5(1):e8613.
17任瑶瑶,江南屏,刘睿颖,等.藏药臭蒿及其近缘种药材的ITS2 DNA条形码鉴别.中国中药杂志,2017,42(7):1395-1400.
18陈士林.中药DNA条形码分子鉴定(第一版).北京:人民卫生出版社,2012年.
19 Keller A,Schleicher T,Schultz J,et al.5.8S-28S rRNA interaction and HMM-based ITS2 annotation.Gene,2009,430:50-57.
20 Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Mol Biol Evol,2011,28:2731-2739.
21 Liao B S,Chen X C,Han J P,et al.Identification of commercial Ganoderma(Lingzhi)species by ITS2 sequences.Chinese medicine,2015,10:1-9.
22 Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees.Mol Biol Evol,1987,4(4):406-425.
23 Koetschan C,Förster F,Keller A,et al.(2010)The ITS2 database III—sequences and structures for phylogeny.Nucleic Acids Res,38(suppl 1):D275-D279.
24 Gao T,Yao H,Song J Y,et al.Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2,Elsevier Science,2010,130:116-121.
25罗焜,陈士林,陈科力.基于芸香科的植物通用DNA条形码研究.中国科学,2010,40(4):342-351.
26甘书龙.四川经济动植物资源开发.成都:四川社会科学出版社,1998:15-248,404-503.
27辛天怡,李西文,姚辉,等.中药材二维DNA条形码流通监管体系研究.中国科学,2015,45(7):695.

