基于子类问题分类能力度量的特征选择方法
刘 磊,郑陶然,赵晨飞,刘 林,王淑琴,何茂伟
(1.天津师范大学 计算机与信息工程学院,天津300387;2.天津工业大学 计算机科学与软件学院,天津 300387)
特征选择与特征抽取是维数约简中2个主要的方法[1],是构造分类器中关键的数据预处理步骤,其结果直接影响分类器的准确率.在当今大数据时代,数据呈数量巨大且纷繁复杂的特点,使得特征选择方法的研究尤为重要[2].特征选择是指根据一些评价标准在原有的特征集合上选择对分类有意义的特征子集,而去除无关或冗余特征,从而将原空间的维数降至远小于原维数的m维.特征选择方法分为Wrapper、Filter和Embedded 3类[3-4].Wrapper和Embedded方法通常用分类器的准确率来评价选择的特征子集[5-7],而Filter方法不依赖于分类器,只考虑特征的分类能力.Filter方法运算速度快,适用于大数据.Filter方法可以分为对各个特征单独评价和对特征子集评价2类[8],前者可以获得所有特征的得分排序,而后者根据某种搜索策略获得特征子集,本文方法属于前者.
近年来,国内外学者对特征选择方法做了大量的研究.大多特征选择方法有一个共同之处,即各种分类能力度量方法都是针对一个特征或特征子集给出描述该特征或特征子集的分类能力大小的一个分值,如卡方检验、信息增益、互信息、增益比、Relief、相关性、Fisher评分等指标[9-13].这些方法通常认为分值大的特征比分值小的特征的分类能力强,因而分值大的特征也就会被优先选择.然而,一些研究已经表明一些分值小的特征也应该被选择,而且一些有较高分类能力值的特征组合也不总是能得到好的分类结果[8,14-15].以单一值表示特征分类能力大小仅仅是对这个特征分类能力的综合评价,而忽略了特征对于不同类别的分类能力评价.
针对上述问题,本文既考虑各个特征对不同子类问题的分类能力,又考虑各特征总的分类能力,进而提出了一个新的特征分类能力排序方法.该方法既能确保总分类能力强的特征被选择,也能确保对子类问题分类能力强但总分类能力不强的特征被选择,从而获得特征分类能力更合理的排序,以提高分类准确率.
1 基于子类问题分类能力度量的特征选择方法
本文提出的基于子类问题分类能力度量的特征选择方法,简记为RRSPFS(Round-Robin and subproblem based feature selection).首先计算所有特征对各子类问题的分类能力,并按分类能力降序排列所有特征;然后采用Round-Robin[16]方法计算各子类问题中特征的并集.
1.1 相关定义
2个特征X和Y的信息增益IG(X,Y)为
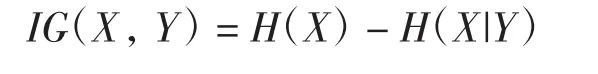
2个特征X和Y的信息增益比GR(X,Y)为

给定具有m个特征k类n个样本的分类问题D=(F,C),F={f1,f2,…,fm}为特征集合,C={c1,c2,…,cn}为类别特征,ci∈{1,2,…,k}.采用 1-vs-1 形式将其转化为由任意两类组成的s个二分类子问题,s=k(k-1)/2,其中每个二分类子问题称为子类问题或子问题.
本文采用特征对类别特征的信息增益比作为特征的分类能力值,特征fi对第j个子问题的分类区分能力 fca(i,j)为
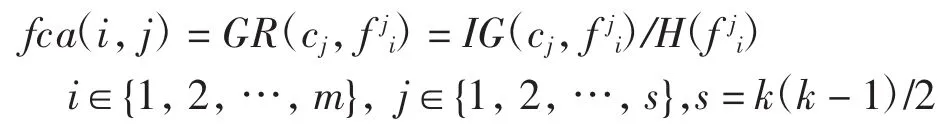

其中:Nj为第j个子问题中所含样本的个数.特征fi的总分类能力R(f)i是它对各个子问题的分类能力的加权平均.
1.2 RRSPFS特征选择方法
对于分类问题D=(F,C),计算每个特征fi对第j个子问题的分类区分能力fca(i,j),获得各个特征对各个子问题的分类能力矩阵(fac(i,j))m×s,将每个子问题中的所有特征按照其分类能力进行降序排列,即可得到各子问题中分类能力由高到低的特征的排列.
采用Round-Robin方法计算各子问题中特征的并集,即首先依次选择各子问题中排在第一且未被选择的特征,再选择排在第二且未被选择的特征,依此类推,直到所有子问题中特征都被选择.其中对于属于各子问题同一等级的特征的选择次序,按照其总分类能力降序进行.
按照上述方法就得到了各特征的分类能力的降序序列.具体算法流程如下.
算法:特征选择方法RRSPFS.
输入:具有n个样本、m个特征、k类的训练集D=({fi1,…,fim,c)i}ni=1,F={f1,…,fm},ci∈{1,2,…,k}.
输出:按分类能力降序排列的特征集合Fm.
令s=k(k-1)/2,Fm= ,T= ;对F中的每一个特征 fi和每个子问题 j,计算 fca(i,j);对每个子问题j,按分类能力 fac(i,j)降序排列所有特征.

对集合T中的特征按照R(f)i降序排列;

2 结果与分析
2.1 数据集
为了验证RRSPFS算法的正确性和有效性,在4个数据集 Breast、Cancers、GCM 和 Leukemia3 上进行了实验,它们均下载自http://www.ccbm.jhu.edu/[17].表1给出了这些数据集中含有的类别数(numberofclassifications)k、特征数(number of features)m 及样本数(number of samples)n.采用传统的客观评价指标,即分类预测准确率测试算法的性能.分类预测准确率是将选择的特征子集作为分类器的输入获得的准确率.将本方法与现有的特征分类能力排序算法InfoGain、GainRation、ReliefF进行比较,并使用朴素贝叶斯(NB)、支持向量机(SVM)、K近邻(KNN)、决策树(C4.5)、随机森林(RandomForest)、简单逻辑回归(Simple Logistic)和简单分类与回归树(SimpleCart)等 7种分类器进行分类预测.

表1 多类基因表达数据集Tab.1 Multiclass gene expression datasets
2.2 实验结果及分析
为合理比较这些方法的实验结果,选择每种方法获得的特征排序结果中相同个数的特征,分别使用上述7种分类器在4个数据集上进行分类预测,相应实验结果见图1~图4,横坐标为排在前面的特征的个数(m),纵坐标为选择特征后使用上述7种分类器进行分类预测获得的准确率的平均值(average of accuracies,AACC).
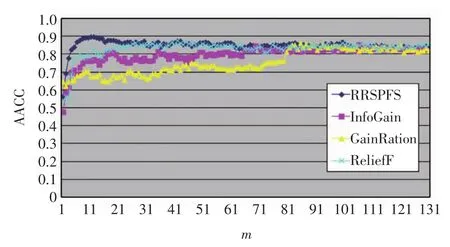
图1 Breast数据集上的准确率比较Fig.1 Accuracy comparison on Breast dataset

图2 Cancers数据集上的准确率比较Fig.2 Accuracy comparison on Cancers dataset

图3 GCM数据集上的准确率比较Fig.3 Accuracy comparison on GCM dataset

图4 Leukemia3数据集上的准确率比较Fig.4 Accuracy comparison on Leukemia3 dataset
由图1~图4可以看出,RRSPFS与其他3种算法相比,不仅在多数情况下获得的分类预测准确率更高,而且也能最快获得最高的准确率.4种算法获得最高准确率(the highest accuracy,HACC)及其选择的特征数比较见表2.这些结果表明RRSPFS优于另外3种,能获得更准确的特征分类能力的排序,并提高了分类器的预测准确率.
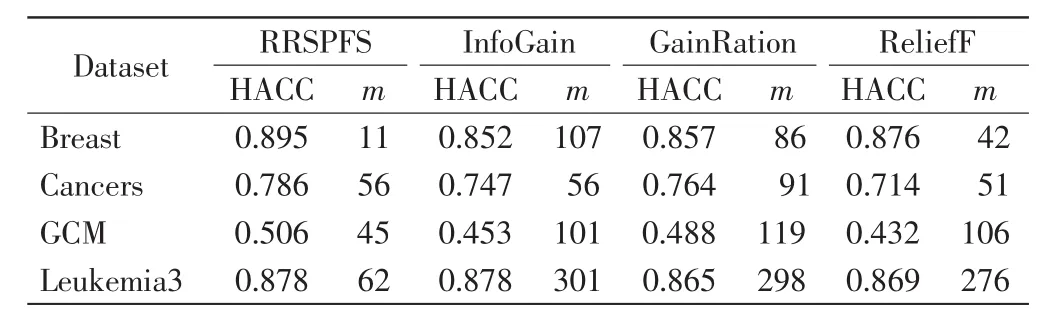
表2 最高准确率(HACC)及其选择的特征数(m)比较Tab.2 Comparison of highest accuracy(HACC)and number of features(m)selected with highest accuracy
记录了RRSPFS在Breast数据集上获得的前21个特征的编号,以及这些特征在另3种算法中的相应排位,表3给出了这21个特征中在另3种算法排名靠后的部分特征.由表3可以看出,有些特征在另3种算法中排序在1000位以后,但它们对子问题的分类能力很强,因此被RRSPFS排在了前面,如特征44、17、115、632、124 等.由图 1 可以看出,排序在前 21的特征的准确率都高于其他算法,这说明了总分类能力不强但子问题分类能力强的特征应该被排在前面.

表3 Breast数据集中部分特征在各个方法中的排名比较Tab.3 Comparison of ranking of features selected by each method on Breast dataset
表4给出了Leukemia3数据集上RRSPFS排名前58但在其他算法中排名靠后的部分特征,可以看到特征 3 035、4 007、11 005、6 796、5 009、2 444、2 701 在其他3种算法中排名比较靠后,有的甚至在9 000位之后,而图4显示RRSPFS的特征排序得到了更好的准确率,这也表明RRSPFS获得的特征排名更合理.

表4 Leukemia3数据集中部分特征在各个方法中的排名比较Tab.4 Comparison of ranking of features selected by each method on Leukemia3 dataset
参考文献:
[1]JAIN A K,DUIN R P W,MAO J C.Statistical pattern recognition:A review[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,2000,22(1):4-37.
[2]BOLÓN-CANEDO V,SÁNCHEZ-MARON~O N,ALONSO-BETANZOS A.Recent advances and emerging challenges of feature selection in the contextofbigdata[J].Knowledge-BasedSystems,2015,86(C):33-45.
[3]GUYON I,GUNN S,NIKRAVESH M,et al.Feature Extraction:Foundations and Applications(Studies in Fuzziness and Soft Computing)[M].New York:Springer,2006.
[4]SAEYS Y,INZA I,LARRAN~AGA P.A review of feature selection techniques in bioinformatics[J].Bioinformatics,2007,23(19):2507-2523.
[5]CHEN G,CHEN J.A novel wrapper method for feature selection and its applications[J].Neurocomputing,2015,159(C):219-226.
[6]RODRIGUES D,PEREIRA L A M,NAKAMURA R Y M,et al.A wrapper approach for feature selection based on Bat Algorithm and Optimum-Path Forest[J].Expert Systems with Applications,2014,41(5):2250-2258.
[7]WANG A G,AN N,CHEN G L,et al.Accelerating wrapper-based feature selection with K-nearest-neighbor[J].Knowledge-Based Systems,2015,83(C):81-91.
[8]YU L,LIU H.Efficient feature selection via analysis of relevance and redundancy[J].Journal of Machine Learning Research,2004,5(12):1205-1224.
[9]PENG H C,LONG F H,DING C.Feature selection based on mutual information:Criteria of max-dependency,max-relevance,and minredundancy[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,2005,27(8):1226-1238.
[10]KONONENKO I.Analysis and extension of Relief[C]//Proceedings of theEuropeanConferenceonMachineLearning,Berlin:Springer,1994:171-182.
[11]FENG J,JIAO L C,LIU F,et al.Unsupervised feature selection based on maximum information and minimum redundancy for hyperspectral images[J].Pattern Recognition,2016,51(C):295-309.
[12]KOPRINSKA I,RANA M,AGELIDIS V G.Correlation and instance based feature selection for electricity load forecasting[J].Knowledge-Based Systems,2015,82:29-40.
[13]WANG J,WEI J M,YANG Z,et al.Feature selection by maximizing independent classification information[J].IEEE Transactions on Knowledge and Data Engineering,2017,29(4):828-841.
[14]WANG J Z,WU L S,KONG J,et al.Maximum weight and minimum redundancy:A novel framework for feature subset selection[J].Pattern Recognition,2013,46(6):1616-1627.
[15]WANG S Q,WEI J M.Feature selection based on measurement of ability to classify subproblems[J].Neurocomputing,2017,224(C):155-165.
[16]FORMAN G.A pitfall and solution in multi-class feature selection for text classification[C]//Proceedings of the 21st International Conference on Machine Learning,New York:ACM,2004:38-46.
[17]TAN A C,NAIMAN D Q,XU L,et al.Simple decision rules for classifying human cancers from gene expression profiles[J].Bioinformatics,2005,21(20):3896-3904.

