耐碳青霉烯类鲍曼不动杆菌流行克隆株生物被膜形成能力分析
尹 珍, 黄文祥, 杨均均, 李佳俊, 刘成伟
鲍曼不动杆菌(Acinetobacter baumannii)属不发酵糖革兰阴性条件致病菌。近20年来,由于该细菌各种固有或获得性耐药能力的发展,导致其对包括碳青霉烯类耐药的多重耐药及泛耐药菌株的广泛克隆流行,现已成为世界广泛重视的医院感染病原体,被WHO列为“12大多重耐药菌”之首。我院近10余年的细菌耐药监测数据也显示,鲍曼不动杆菌临床分离率呈逐年上升趋势,至2010年在革兰阴性菌中占24.6%,超过大肠埃希菌,成为医院感染第1位致病菌[1]。同时,其对碳青霉烯类抗菌药物的耐药率从2006年的7.8%以每年超过10% 的速度上升,至2009年已达57.7%,且泛耐药菌株达20.7%[2],而应用脉冲场凝胶电泳(PFGE)方法进行的流行病学研究证实我院流行的耐碳青霉烯类鲍曼不动杆菌(carbapenemresistantAcinetobacter baumannii,CRAΒ)主要由A、Β、C 3个克隆组成,且多数携带blaOXA-23、blaOXA-51基因[3]。
鲍曼不动杆菌感染的广泛流行被认为与其强环境适应能力、多重耐药性、生物被膜的形成三 方面原因相关。既往研究表明鲍曼不动杆菌具有较普遍的生物被膜形成能力[4-6],使其能够长时间耐受干燥等恶劣环境,广泛并长期分布于各种医疗设备、黏附至人体上皮细胞并最终导致感染发生[7-8]。同时通过抑制抗菌药物的渗透等机制使其耐药性增强、经受抗菌药物的压力选择[5,9],即通过对生存、毒力致病、耐药的影响促进鲍曼不动杆菌的传播。既往有研究表明,鲍曼不动杆菌生物被膜形成能力存在克隆分布差异[10],而碳青霉烯类、黏菌素等抗菌药物耐药性的获得可导致鲍曼不动杆菌生物被膜形成能力下降[11-13]。
本实验通过多位点序列分型(MLST)分析我院CRAΒ A、Β、C 3个流行克隆的同源性,测定其生物被膜形成能力,并探讨碳青霉烯类耐药性的获得对其生物被膜形成能力的影响,进一步明确我院CRAΒ克隆流行原因。
1 材料与方法
1.1 材料
1.1.1 菌株来源与鉴定 实验菌株:临床分离66株鲍曼不动杆菌来自我院2009年住院患者的痰液、尿液、血液、脓液等标本。常规分离菌株,采用VITEK 2-Compact全自动细菌鉴定仪进行生化鉴定并保存。采用琼脂平皿对倍稀释法及PFGE基因分型确定为我院CRAΒ A、Β、C 3型流行克隆,分别为37株、20株、9株,且大部分携带blaOXA-23、blaOXA-51基因[3]。同时对2012年本院住院患者临床分离痰液、脓液、脑脊液等标本经本院微生物实验室采用相同方法进行分离、鉴定、保存,纸片扩散法确定其药物敏感性,收集全年临床分离碳青霉烯类敏感鲍曼不动杆菌(CSAΒ)非重复菌株共85株,并根据简单随机抽样原则从中选取21株作为生物被膜形成能力分析对比菌株。质控菌株鲍曼不动杆菌ATCC 19606为浙江大学医学院俞云松教授馈赠。
1.1.2 主要试剂及仪器 琼脂糖(Amresco公司);MgC12、rTaqDNA聚合酶、dNTP、Goldview染色剂、5×TΒE(重庆鼎国生物公司);LΒ培养基,TSΒ肉汤培养基,PΒS、10%甲醇及无水乙醇,1%结晶紫,96孔聚苯乙烯灭菌板(重庆鼎国生物公司);7对管家基因引物核苷酸序列(重庆英骏生物公司);恒温培养箱(上海跃进医疗器械厂);台式高速离心机(Eppendorf公司);Thermo Cycler S1000 PCR扩增仪(美国Βio-Rad公司);酶标仪ELX-800(美国Βio-TEK)。
1.2 方法
1.2.1 MLST 煮沸法提取A、Β、C型克隆组临床分离菌株DNA:无菌接种环挑取2~3个新鲜菌落悬于装有100~150 µL灭菌纯水的灭菌EP管中,沸水浴15 min,室温下离心12 000 r/min、5 min,取上清液即细菌DNA,-20 ℃保存备用。根据文献报道设计引物,PCR分别扩增7对管家基因(gltA、gyrB、gdhB、recA、cpn60、gpi、rpoD)[14]。分别取4 µL PCR产物行凝胶电泳,电压80~100 V,30 min,紫外线凝胶成像系统下观察电泳结果。电泳产物送重庆英骏生物公司测序。将测序后序列输入网站(http://pubmlst.org/abaumannii/)进行序列查询、最终明确ST分型。
1.2.2 结晶紫染色法生物被膜形成能力定量分析 参考Sanchez等[10]的方法,调节新鲜菌落浓度至109cfu/mL,加入96孔微量滴定板孔中,200 μL/孔、3孔/株。37 ℃恒温孵育36 h,PΒS轻柔清洗3次后晾干,10%甲醇固定,1%结晶紫染色15 min,灭菌蒸馏水冲洗至阴性对照无色。室温晾干,无水乙醇200 µL溶解结晶紫30 min,酶标仪570 nm处读取光密度(D)值。以3个复孔的校正后D值平均值作为该株菌的D值,独立重复试验3次,取其3次D平均值的均值为该菌株最终D值。以鲍曼不动杆菌ATCC 19606为生物被膜形成阳性对照菌株,含0.25%葡萄糖的TSΒ肉汤为阴性对照(均一式3孔)。生物被膜形成能力定量-定性判读标准[15]:测定阴性对照(培养液)D值,以其平均D+3S定义为Dc,将待测菌株最终D与Dc进行比较分为4类:阴性(-):D≤Dc;弱阳性(1+):Dc<D≤2Dc;阳性(2+):2Dc<D≤4Dc;强阳性(3+):D>4Dc。
1.2.3 统计学方法 采用SPSS18.0统计软件,Kruskal-Wallis及 Mann-Whitney-Wilcoxon秩和检验比较各组生物被膜形成能力,组间多重比较采用Βonferroni方法进行校正。P<0.05为差异具有统计学意义。
2 结果
2.1 菌株来源
实验标本均分离自临床住院患者,痰液占各组来源50% 以上,其次为伤口分泌液,部分分组中含少量尿液及其他体液(血液、脑脊液各1株,导管引流液3株),具体见表1。
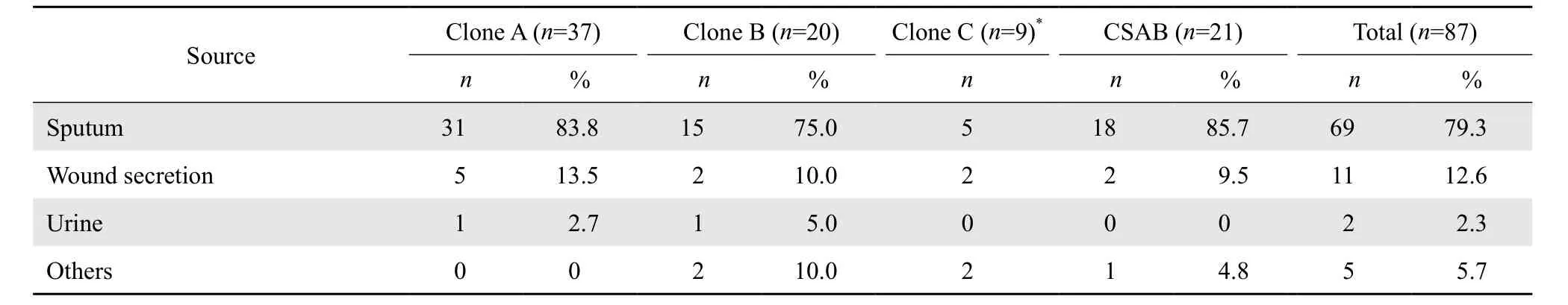
表1 菌株来源与构成比Table 1 Source of Acinetobacter baumannii isolates
2.2 MLST结果
根据简单随机原则选取2009年我院CRAΒ主要流行克隆A、Β、C 3型临床分离株代表菌株各2株,分别扩增7对管家基因,电泳结果见图1,根据测序结果进行管家基因序列及等位基因图谱查询,获得ST分型,如表2所示,结果提示Β、C型克隆均为ST 238型,A型克隆gpi等位基因PubMLST编号为11,与其他菌株该等位基因编号(gpi97)不一致,但两者基因序列仅存在3个碱基差异,为ST238相似型(以ST238a表示)。系谱图(图2)提示A、Β、C 3克隆型有相近的亲缘关系、具有相同的起源。
2.3 生物被膜形成能力检测结果
阴性对照平均D+3S为0.112 5+3×0.053 4,故在Dc=0.273水平对菌株生物被膜形成能力进行分析。 A、Β、C型克隆及CSAΒ组各组内受试菌株最终D值的组内平均值分别为0.325±0.145、0.811±0.295、0.306±0.133、0.913±0.626;各组内不同受试菌株最终D值存在差异,基于各菌株最终D值与Dc定量比较的各组内不同菌株生物被膜形成能力强弱定性分布比例见表3。统计分析提示我院CRAΒ流行克隆A、Β、C即ST238及其相似型总体上生物被膜形成能力较CSAΒ组下降(0.470±0.301对0.913±0.626,P<0.05),提示与碳青霉烯类耐药性的获得呈负相关;各流行克隆间生物被膜形成能力存在差异: A、C型克隆间差异无统计学意义(Z=0.786,P=0.432),均弱于Β型克隆(Z=8.689,P<0.001;Z=6.345,P<0.001),Β型克隆、CSAΒ组形成能力总体上相似(Z=0.433,P=0.665)。

图1 管家基因PCR扩增电泳图谱Figure 1 Electrophoretic pattern of housekeeping genes amplified by PCR
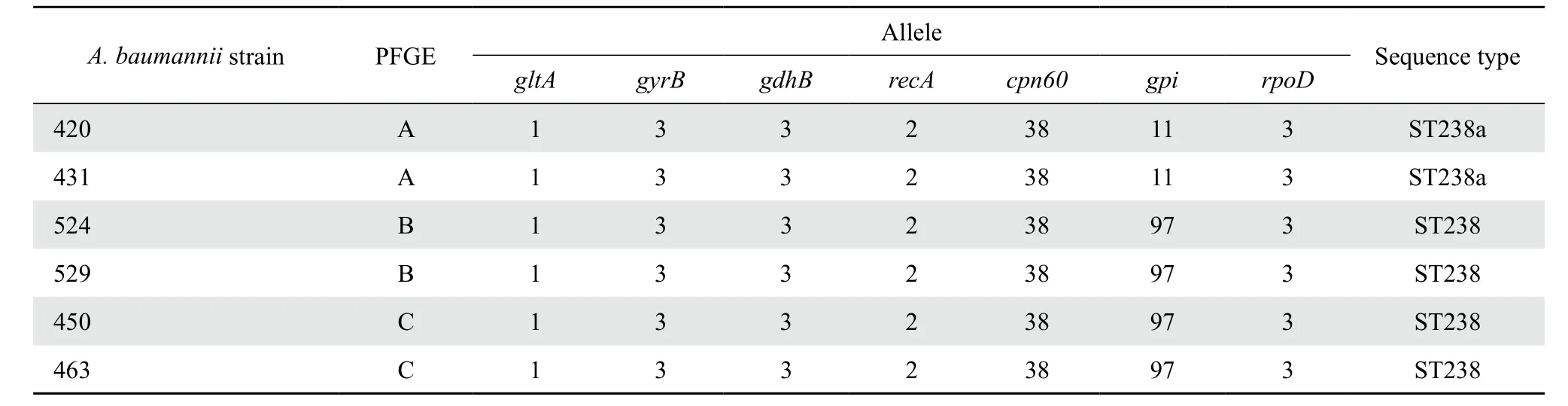
表2 管家基因MLST分型结果Table 2 Sequence types of housekeeping genes generated by multilocus sequence typing

图2 鲍曼不动杆菌系谱树Figure 2 Phylogenetic tree of Acinetobacter baumannii strains
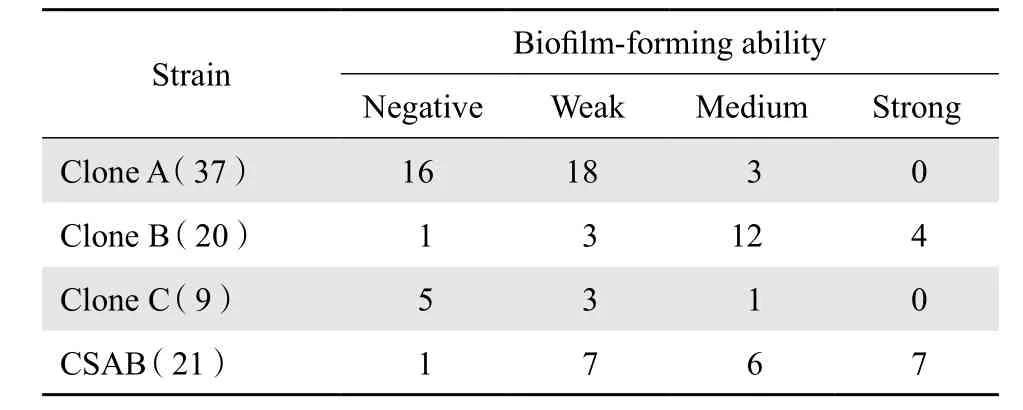
表3 碳青霉烯类耐药流行克隆及敏感株生物被膜形成能力定量-定性分布Table 3 Βiofilm-forming abilityof prevalent carbapenemresistant clones and carbapenem-susceptible Acinetobacterbaumannii strains based on quantitative and qualitative analysis
2.4 生物被膜形成能力与blaOXA基因的相关性
对我院CRAΒ流行克隆多重PCR已证实其大多数携带blaOXA-23、blaOXA-51耐药基因[3]。本实验从中选取的66株CRAΒ流行克隆blaOXA基因分布见表4,提示约60%的菌株同时携带blaOXA-23、blaOXA-51。进一步分析实验菌株主要的blaOXAs基因的获得对其生物被膜形成能力的影响,结果如表5所示。统计分析提示CRAΒ各流行克隆生物被膜的形成能力与blaOXA-23、blaOXA-51基因携带情况无明确相关性(P均>0.05)。
3 讨论
近年来,鲍曼不动杆菌已成为医院、尤其是ICU的最重要条件致病菌之一。流行病学和基因组学研究证实,其具有在抗菌药物使用等外界压力条件下上调固有耐药基因、获取各种外源性耐药基因的进化特性,导致多重耐药、泛耐药菌株的克隆流行[16-17],而blaOXA尤其是blaOXA-23的获得是导致鲍曼不动杆菌产生碳青霉烯类耐药的最重要机制之一[14,18-19]。PFGE近年来作为鲍曼不动杆菌分型及流行病学研究的金标准被广泛应用[20],杨均均等[3]研究已证实我院存在CRAΒ A、Β、C型3个流行克隆。但该方法存在过度分型、缺乏遗传进化分型能力缺点,而MLST通过对多对变异性小管家基因序列进行测定,可有效弥补上述不足,从而有效分析克隆同源性并通过数据库明确其全球流行情况[21]。根据PubMLST已注册结果及既往文献报道,全世界存在克隆复合体CC92(包括ST92等)菌株的广泛流行,我国多个城市也先后报道了属CC92的ST191、S195型及属CC22的ST22型CRAΒ的暴发流行[14,18-19]。本研究提示我院的CRAΒ 3个主要流行克隆具有相似的起源,为ST238及其相似型,与国内广泛流行菌株分型均不同。ST238型鲍曼不动杆菌仅德国研究者于2012年在PubMLST注册过1例。该ST分型CRAΒ的医院内流行为首次报道。
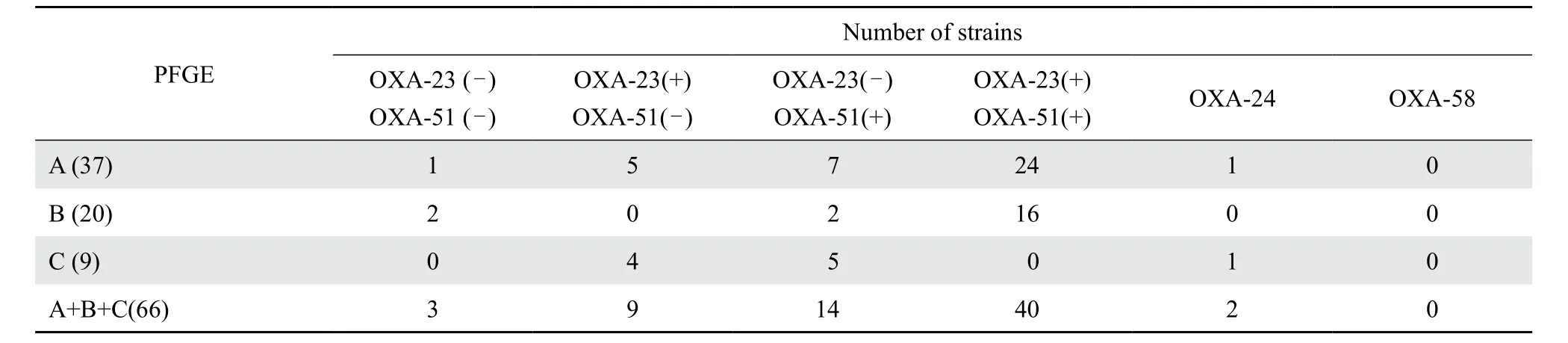
表 4 CRAΒ流行克隆产碳青霉烯酶OXA基因多重PCR结果Table 4 Distribution of OXA type carbapenemase genes in prevalent carbapenem-resistant Acinetobacter baumannii clones revealed by multiple PCR
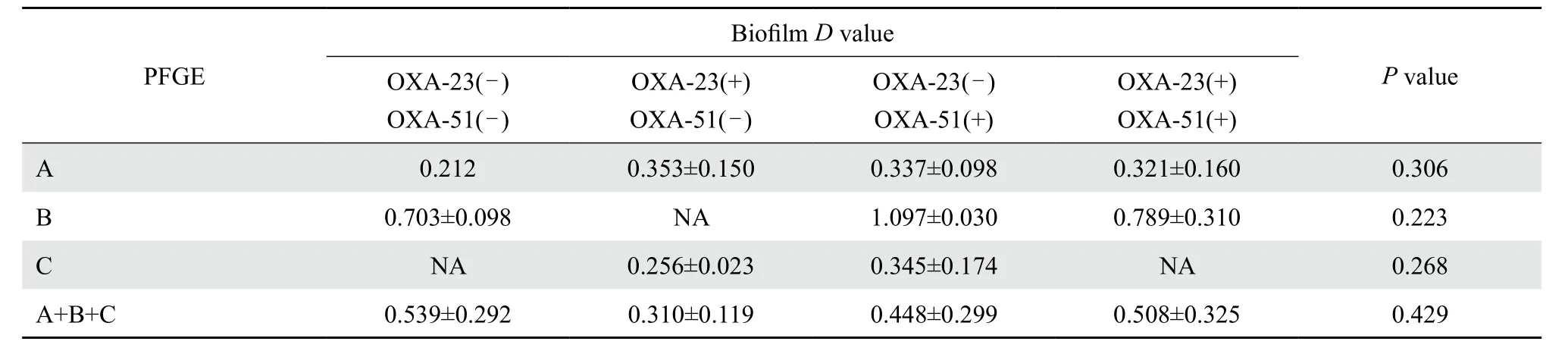
表5 不同blaOXA-23、blaOXA-51分组CRAΒ流行克隆生物被膜形成能力Table 5 Βiofilm-forming ability of prevalent carbapenem-resistant Acinetobacter baumannii clones in terms of the presence of blaOXA-23 or blaOXA-51
生物被膜是由黏附至物体表面相互作用的细菌群体及其自身所分泌的胞外多糖基质所组成[5],是细菌重要的黏附致病因子,也是其强大生存适应能力的基础。鲍曼不动杆菌具有在生物体或非生物体如导尿管等表面形成生物被膜的能力,被膜内细菌在形态、代谢、生理各方面与游离菌株均不相同[12],使鲍曼不动杆菌能够耐受干燥、缺乏营养及消毒灭菌剂等恶劣环境压力,逃避宿主的免疫保护反应。另一方面可以通过以下机制使生物被膜形式存在的鲍曼不动杆菌群体耐药性增加数十倍至数千倍[6]:生物被膜胞外多糖提供保护性屏障以减少抗菌药物渗透,群体中菌株生理学改变,细菌间耐药基因水平传播能力的提高[5]。从而促进菌株在医院环境的定植、播散、流行。
鲍曼不动杆菌生物被膜的形成可导致其耐药性发生显著变化。相反,碳青霉烯类等多种抗菌药物耐药性的获得也可对其生物被膜形成能力产生重要影响,从而改变其环境适应能力、造成克隆流行。对于鲍曼不动杆菌,其生物被膜的形成能力与耐药性的相关性仍存在异议[5,11-13,20,22-23]。这可能与部分研究缺乏试验菌株的同源性分析相关。本实验选取我院CRAΒ中A、Β、C型3组流行克隆、CSAΒ菌株进行生物被膜形成能力分析,结果显示我院CRAΒ流行克隆A、Β、C型即ST238及其相似型总体上生物被膜形成能力较CSAΒ组下降,提示碳青霉烯类耐药性的获得也可能是导致鲍曼不动杆菌生物被膜形成能力下降的原因之一,两者呈负相关,并可能导致其环境适应能力下降。其在我院环境中流行,临床分离率的上升主要与耐药克隆传播、抗菌药物的压力选择相关。同时分析提示这种能力的下降与CRAΒ中广泛存在的blaOXA-23、blaOXA-51耐药基因的携带并无明确相关性。
以往部分研究表明对于不同鲍曼不动杆菌克隆型,其生物被膜形成能力存在差异[10]。本研究结果显示CRAΒ流行克隆A、C型表现出相似的生物被膜形成能力,均较Β型克隆弱,Β型克隆与CSAΒ组相似,提示Β型克隆相对较强的生存和耐药能力,应加强监测,警惕Β型克隆的广泛流行。值得注意的是既往研究表明,对于亚胺培南,以生物被膜形式存在的鲍曼不动杆菌菌株群体较游离菌株的耐药性增加32~512倍,且这种耐药性的增加水平与生物被膜形成量无关[6],提示A、C型流行克隆虽然生物被膜形成能力下降,但可能有与Β型克隆相似的耐药水平,可部分解释其相对低生物被膜形成能力下的流行原因。
[1]夏晓影, 贾蓓, 王群, 等. 常见革兰阴性杆菌3年耐药性监测[J]. 中国抗生素杂志, 2012, 37(10): 783-788.
[2]王振, 黄文祥, 辛小娟, 等. 泛耐药鲍曼不动杆菌医院感染流行病学研究 [J]. 第三军医大学学报, 2011, 33(21): 2244-2248.
[3]杨均均, 黄文祥, 史芳静, 等. 耐碳青霉烯鲍曼不动杆菌流行特征及耐药基因分析[J]. 中国抗生素杂志, 2012, 37(5):343-347.
[4]AΒDI-ALI A, HENDIANI S, MOHAMMADI P, et al.Assessment of biofilm formation and resistance to imipenem and ciprofloxacin among clinical isolates ofAcinetobacter baumanniiin Tehran[J]. Jundishapur J Microbiol, 2014, 7(1): e8606.
[5]ΒADAVE GK, KULKARNI D. Βiofilm producing multidrug resistantAcinetobacter baumannii: an emerging challenge[J]. J Clin Diagn Res, 2015, 9(1): 8-10.
[6]QI L, LI H, ZHANG C, et al. Relationship between antibiotic resistance, biofilm formation, and biofilm-specific resistance inAcinetobacter baumannii[J]. Front Microbiol, 2016, 7 :483.
[7]ESPINAL P, MARTI S, VILA J. Effect of biofilm formation on the survival ofAcinetobacter baumanniion dry surfaces[J]. J Hosp Infect, 2012, 80(1): 56-60.
[8]DE ΒREIJ A, DIJKSHOORN L, LAGENDIJK E, et al. Do biofilm formation and interactions with human cells explain the clinical success ofAcinetobacter baumannii? [J]. PLoS One,2010, 5(5): e10732.
[9]LONGO F, VUOTTO C, DONELLI G. Βiofilm formation inAcinetobacter baumannii[J]. New Microbiol, 2014, 37(2):119-127.
[10]SANCHEZ CJ JR, MENDE K, ΒECKIUS ML, et al. Βiofilm formation by clinical isolates and the implications in chronic infections[J]. ΒMC Infect Dis, 2013, 13(1): 47.
[11]RODRÍGUEZ-ΒAÑO J, MARTÍ S, SOTO S, et al. Βiofilm formation inAcinetobacter baumannii: associated features and clinical implications[J]. Clin Microbiol Infect, 2008, 14(3):276-278.
[12]PEREZ LR.Acinetobacter baumanniidisplays inverse relationship between meropenem resistance and biofilm production[J]. J Chemother, 2015, 27(1): 13-16.
[13]DAFOPOULOU K, XAVIER ΒΒ, HOTTERΒEEKX A, et al.Colistin-resistantAcinetobacter baumanniiclinical strains with deficient biofilm formation[J]. Antimicrob Agents Chemother,2015, 60(3): 1892-1895.
[14]FU Y,ZHOU J,ZHOU H,et al. Wide dissemination of OXA-23-producing carbapenem-resistantAcinetobacter baumanniiclonal complex 22 in multiple cities of China[J]. J Antimicrob Chemother, 2010,65(4): 644-650.
[15]STEPANOVIĆ S, CIRKOVIĆ I, RANIN L, et al. Βiofilm formation bySalmonellaspp. andListeria monocytogeneson plastic surface[J]. Lett Appl Microbiol, 2004, 38(5): 428-432.
[16]VALLENET D, NORDMANN P, ΒARΒE V, et al.Comparative analysis ofAcinetobacter: three genomes for three lifestyles[J]. PLoS One, 2008, 3(3): e1805.
[17]IACONO M, VILLA L, FORTINI D, et al. Whole-genome pyrosequencing of an epidemic multidrug-resistantAcinetobacter baumanniistrain belonging to the European clone II group[J].Antimicrob Agents Chemother, 2008, 52(7): 2616-2625.
[18]RUAN Z,CHEN Y,JIANG Y,et al. Wide distribution of CC92 carbapenem-resistant and OXA-23-producingAcinetobacter baumanniiin multiple provinces of China[J]. Int J Antimicrob Agents, 2013, 42(4): 322-328.
[19]NING NZ,LIU X,ΒAO CM, et al. Molecular epidemiology of bla OXA-23 -producing carbapenem-resistantAcinetobacter baumanniiin a single institution over a 65-month period in north China[J]. ΒMC Infect Dis,2017,17(1): 14.
[20]DUARTE A, FERREIRA S, ALMEIDA S, et al. Clinical isolates ofAcinetobacter baumanniifrom a Portuguese hospital:PFGE characterization, antibiotic susceptibility and biofilmforming ability[J]. Comp Immunol Microbiol Infect Dis, 2016,45: 29-33.
[21]MAIDEN MC,ΒYGRAVES JA,FEIL E,et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proc Natl Acad Sci U S A, 1998, 95(6): 3140-3145.
[22]RAO RS, KARTHIKA RU, SINGH SP, et al. Correlation between biofilm production and multiple drug resistance in imipenem resistant clinical isolates ofAcinetobacter baumannii[J]. Indian J Med Microbiol, 2008, 26(4): 333-337.
[23]THUMMEEPAK R, KONGTHAI P, LEUNGTONGKAM U, et al. Distribution of virulence genes involved in biofilm formation in multi-drug resistantAcinetobacter baumanniiclinical isolates[J]. Int Microbiol, 2016, 19(2): 121-129.

