大豆抗豆卷叶螟的转录组和蛋白质组关联分析
曾维英,孙祖东,赖振光,蔡昭艳,陈怀珠,杨守臻,唐向民
大豆抗豆卷叶螟的转录组和蛋白质组关联分析
曾维英,孙祖东,赖振光,蔡昭艳,陈怀珠,杨守臻,唐向民
(广西农业科学院经济作物研究所/农业部西南玉米大豆间套作区农业科学观测实验站,南宁 530007)
【】通过对大豆受豆卷叶螟幼虫胁迫下的转录组和蛋白质组结果进行联合分析,筛选出一些与大豆抗豆卷叶螟相关的候选基因,为深入认识大豆抗豆卷叶螟的分子调控机制奠定基础。【】以高抗材料赶泰-2-2(HR)和高感材料皖82-178(HS)为研究对象,运用RNA-Seq技术和iTRAQ技术鉴定出豆卷叶螟幼虫取食诱导0和48 h时样品间的差异表达基因(DEGs)和差异表达蛋白(DEPs),将在蛋白水平和转录水平关联到的所有可定量数据进行关联分析,计算蛋白水平和转录水平间的相关系数。【】蛋白质鉴定结果表明,HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组中分别鉴定出236、250、213、211个DEPs;转录组鉴定结果表明,HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组中分别鉴定出1 064、680、605、468个DEGs。定量关联分析结果表明,HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组的相关系数分别为0.156、0.2687、0.1149和0.035;HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组中分别关联到11、9、3和4个DEPs/DEGs,其中蛋白和mRNA表达趋势相同的基因分别有11、9、2和3个,蛋白和mRNA表达趋势相反的基因分别有0、0、1和1个。生物信息学分析结果表明,这些差异蛋白功能涉及代谢途径、核糖体、类黄酮生物合成、亚油酸代谢、氨基糖-核苷酸代谢、氨酰生物合成、亚麻酸代谢、次生代谢产物的生物合成、苯基丙酸生物合成、RNA运转、谷胱甘肽代谢、抗坏血酸盐和aldarate代谢等。功能关联分析结果表明,HR48/HR0比较组中有27条Pathway通路共关联到101个DEPs和23个DEGs,HS48/HS0比较组中有15条Pathway通路共关联到147个DEPs和16个DEGs,HR0/HS0比较组中有18条Pathway通路共关联到82个DEPs和10个DEGs,HR48/HS48比较组中仅有1条Pathway通路关联到71个DEPs和2个DEGs。同时关联发现胰蛋白酶抑制剂A、K型胰蛋白酶抑制剂、查尔酮异构酶4、脂氧合酶9、α-加氧合酶1、9S-脂氧合酶、植物凝集素前体、POD12、应激蛋白SAM22和cAPX1等可能是大豆抗豆卷叶螟潜在的靶标蛋白(基因)。qRT-PCR表达分析结果表明,5个DEPs/DEGs在HR48/HR0比较组的表达趋势与它们的RNA-Seq和iTRAQ结果基本一致。【】确定了一些与豆卷叶螟的抗性相关蛋白(基因)和代谢通路,这些差异蛋白可能直接或间接参与大豆对豆卷叶螟的抗虫反应。
大豆;豆卷叶螟;转录组;蛋白质组;关联分析
0 引言
【研究意义】大豆是世界主要的油料作物,广泛应用于食品、饲料、工业生产以及其他产业领域[1]。然而,虫害大大増加了大豆生产的成本。豆卷叶螟属鳞翅目螟蛾科,以幼虫卷曲大豆叶片并潜伏其中取食大豆叶片组织,影响光合作用,使植株不能正常生长,是重要的大豆食叶性害虫[2]。在吉林省、辽宁省以南、四川省以东地区均有发生,在南方大豆产区一年可发生多代,在发生危害严重的年份,叶片被取食后只剩叶脉和叶柄,造成产量的严重损失[3]。因此,挖掘出大豆抗豆卷叶螟相关的功能基因,对开展大豆抗豆卷叶螟分子育种具有重要意义。【前人研究进展】孙祖东等[4]研究表明卷包密度和虫口密度可作为鉴定大豆种质抗豆卷叶螟的鉴定指标,并且筛选出高抗品种赶泰-2-2、牛黄豆、PI227687等,高感品种皖82-178、金龙黑豆、Mosoy等。邢光南等[5]研究表明豆卷叶螟引起的虫包数和卷叶率与叶片、叶柄茸毛角度及叶片茸毛长度极显著正相关,而与叶片茸毛密度显著负相关,与茸毛末端形态无关。叶片茸毛角度与抗虫性指标相关性最强,是大豆抗豆卷叶螟的重要因子。邢光南等[6]、李广军等[7]研究表明大豆对豆卷叶螟抗性符合2对主基因+多基因的混合遗传模式。李广军等[8]对豆卷叶螟抗性进行QTL定位,检测到位于D1b和K连锁群上有2个QTL、位于A2、D1b、K和N连锁群上有4个QTL和6个互作QTL。XING等[9]研究表明大豆抗豆卷叶螟的4个RIL群体中KY群体检测到8个QTL、WT群体检测到10个QTL、XG和SX群体各检测到1个加性QTL,其中和是主效QTL。【本研究切入点】因蛋白质组与转录组分别从2个不同层面反映了基因的表达情况,所以要了解转录组与蛋白质组的相互调控作用,需要对mRNA与蛋白质的表达进行同步监测。关联分析的目的之一是实现数据互补,得到生物体更加完整的表达信息。关于大豆抗豆卷叶螟的转录组和蛋白质组关联分析未见报道。【拟解决的关键问题】本研究以大豆高抗豆卷叶螟材料(赶泰-2-2)和高感豆卷叶螟材料(皖82-178)为研究对象,以iTRAQ分析的蛋白质组结果为基础,与RNA-Seq测序获得的转录组结果进行定量和功能关联分析,以期筛选出一些可能作为大豆抗豆卷叶螟的潜在靶标蛋白(基因),为大豆抗豆卷叶螟的分子调控机制深入研究提供线索。
1 材料与方法
1.1 试验材料
供试大豆品种为赶泰-2-2(高抗材料)和皖82-178(高感材料)。试验材料于2015年7月种植在广西农业科学院试验田防虫网室中,3行区播种,每行10株,在大豆整个生育期间不喷施农药和肥料。待植株长至10片复叶时,按照豆卷叶螟4龄幼虫每苗接5虫的密度分别接虫。接虫0 h(未接虫,对照)和48 h后对样品进行取样,每个处理2个生物学重复。分别命名为HR0、HR48(抗性基因型)和HS0、HS48(感性基因型),其中数字0和48代表处理时间。每个样品取5株混合,用液氮迅速冷冻并储存在-80℃冰箱备用。
1.2 蛋白质组学研究方法
采用丙酮沉淀法提取叶片蛋白质[10]。蛋白质浓度通过Bradford测定法以牛血清蛋白(BSA)系列浓度为标准来测定[11],利用SDS-PAGE检测蛋白质量和浓度。获得的蛋白样本保存在-80℃冰箱中备用。iTRAQ试验及其结果分析由深圳华大基因研究院完成。赶泰-2-2在豆卷叶螟取食前的酶标是113和115标记、取食48 h的酶标是117和119标记,皖82-178在豆卷叶螟取食前的酶标是114和116标记、取食48 h的酶标是118和121标记。将标记后的各组肽段混合,真空抽干后用SCX柱进行液相分离,再进行基于Triple TOF 5600的LC-ESI-MSMS分析。对质谱下机的原始文件进行峰识别以获取峰列表,建立参考数据库,进行肽段及蛋白质的鉴定。利用Proteome Discoverer 2.2软件将从Orbitrap获得的质谱原始文件转换成MGF文件格式并进行搜索数据库。使用Mascot搜索引擎将原始数据比对到NCBI中包含65,681个序列的大豆数据库(http://www.ncbi.nih.gov/genomes/Glycine_ max/protein)进行搜库鉴定蛋白质。为了降低假阳性多肽识别概率,通过Mascot概率分析时仅鉴定具有95%置信度的多肽。每个确定的识别蛋白至少具有1个Unique peptide。对于蛋白质的定量,必须至少包含2个Unique peptide。蛋白定量比例通过Mascot的中位比例进行加权和标准化,本研究中以“|Fold Change|≥1.2和≤0.05”作为阈值来判断蛋白丰度差异的显著性。
1.3 转录组学研究方法
利用TRIzol Kit(Invitrogen,Carlsbad,CA,USA)分别提取高抗材料与高感材料叶片的总RNA(5 μg)。利用超微量分光光度计NanoDrop2000(Thermo Fisher Scientific,Waltham,MA,USA)检测总RNA的浓度与纯度,利用生物学分析仪Agilent 2100 Bioanalyzer(Agilent,Santa Clara,CA,USA)检测RNA的完整性。利用TruseqTMRNA样本制备试剂盒(Illumina,SanDiego,CA,USA)进行mRNA纯化和cDNA文库构建,对获得的cDNA文库进行PCR扩增富集,2%琼脂糖凝胶电泳检测,回收目的片段。利用微型荧光计TBS380(QuantiFluoTMST/P,Promega,Madison,WI,USA)对cDNA文库进行定量。最后,使用Illumina HiseqTM2000测序仪进行测序(Hiseq2000 Truseq SBS Kit v3-HS(200cycles),Illumina)。以上试验均由深圳华大基因科技服务有限公司完成。经Base calling转化为Raw data或Raw reads,随后对Raw reads进行质控(QC)[12]。利用SOAPfuse软件[13]对原始测序数据进行去除处理得到Clean reads。利用Tophat将Clean reads比对到大豆参考基因组ftp://ftp.jgi-psf.org/ pub/compgen/phytozome/v9.0/early_release/ Gmax_275 _Wm82.a2.v1/, version Glyma2.0),允许有2个碱基的错配[14-15]。表达定量的结果以FPKM(Fragments per kilobase of exon model per million mapped fragments)为单位,在得到差异检验的FDR值同时,根据基因的表达量(FPKM值)计算该基因在不同样本间的差异表达倍数。以“FDR≤0.001和|log2Ratio(FC,Fold Change)|≥2”作为阈值来判断基因差异表达的显著性[16]。
1.4 生物信息学分析
利用Blast2go_v2.5软件对被鉴定的差异基因/蛋白进行GO注释分析,计算得到的-value通过Bonferroni校正之后,以Corrected-value≤0.05为阈值。利用Blast_v2.2.26软件在KEGG pathway数据库对所鉴定到的差异基因/蛋白进行Pathway富集分析,计算得到的-value通过Bonferroni校正之后,以Corrected-value≤0.05为阈值。
1.5 关联分析
将在蛋白水平和转录水平关联到的所有可定量数据进行关联分析。基于蛋白质组鉴定结果,筛选出符合条件的可定量蛋白质,筛选条件为:该蛋白可定量Unique peptide≥2时,计算Peptide ratio,以Peptide ratio的中值代表该蛋白的Ratio,然后与转录组测序结果中基因的Ratio比值进行关联分析,计算蛋白水平和转录水平间Person相关系数[17],确定蛋白和mRNA的相关性的强弱。
1.6 实时荧光定量PCR(qRT-PCR)分析
选取了5个与抗豆卷叶螟相关潜在功能基因进行qRT-PCR验证。参考unigene序列,引物序列用软件Primer Premier 5.0(premier biosoft international,palo alto,CA)设计。用带有gDNA消除剂的PrimeScript®RT试剂盒安装厂家说明书对经过DNA酶纯化的RNA样本反转录合成cDNA。qRT-PCR反应混合液(25 μl)包含SybrGreen qPCR Master Mix 12.5 μl、正反引物(10 μmol·L-1)各0.5 μl、cDNA模板2 μl和ddH2O 9.5 μl。qRT-PCR反应程序为95℃2 min;95℃ 10 s,60℃ 40 s,共40个循环。qRT-PCR分析由ABI7500FAST型实时-荧光定量PCR仪完成。以作为内参基因,利用2-ΔΔct法计算基因的相对表达量。
2 结果
2.1 蛋白质组和转录组数据分析
2.1.1 蛋白质组数据分析 利用iTRAQ技术分析抗感材料受豆卷叶螟取食前后叶片的蛋白质差异,结果共产生354 049张图谱。利用Mascot软件分析发现共45 454张谱图匹配到大豆参考基因组的已知谱图上,其中28 525张匹配到Unique图谱。鉴定到15 264个肽段,11 068个Unique肽段,最终鉴定出4 073个蛋白质。
当蛋白质丰度比达到1.2倍以上,且经统计检验其-value≤0.05时,认为此蛋白为两样品间的差异表达蛋白(DEPs)。结果表明,HR48/HR0比较组中鉴定出236个DEPs,其中120个上调表达,116个下调表达;HS48/HS0比较组中鉴定出250个DEPs,其中140个上调表达,110个下调表达;HR0/HS0比较组中鉴定出213个DEPs,其中109个上调表达,104个下调表达;HR48/HS48比较组中鉴定出211个DEPs,其中125个上调表达,86个下调表达。
2.1.2 转录组数据分析 利用Illumina HiseqTM2000测序仪对抗感材料受豆卷叶螟取食诱导前后叶片转录组进行高通量测序。结果表明,8个测序样品共产生458 758 338条100 bp的Raw reads,经过去除低质量Reads,共剩余442 422 398条100 bp的高质量Clean reads。通过Tophat软件将获得的所有Clean reads比对到大豆参考基因中,匹配比例范围为84.81%—87.67%。
当FDR≤0.001,且|log2Ratio(FC,Fold Change)|≥2时,认为此基因是两样品间的差异表达基因(DEGs)。结果表明,HR48/HR0比较组中鉴定出1 064个DEGs,其中894个上调表达,170个下调表达;HS48/HS0比较组中鉴定出680个DEGs,其中495个上调表达,185个下调表达;HR0/HS0比较组中鉴定出605个DEGs,其中192个上调表达,413个下调表达;HR48/HS48比较组中鉴定出468个DEGs,其中202个上调表达,266个下调表达。
2.2 定量关联性分析
2.2.1 转录组和蛋白质组数量关联关系 首先,对赶泰-2-2与皖82-178在豆卷叶螟取食前后获得的转录组测序结果被用于CDS(coding sequence)预测,从而获得这些基因的蛋白质序列。然后,将获得的蛋白质序列作为搜索数据库。为了避免假阳性的出现,转录组和蛋白质组结果再次进行统一参考基因组数据库(大豆参考基因组)搜索比对。当某一蛋白被鉴定并且在转录组水平有表达量信息时,被认为关联。
在鉴定、定量和显著差异3个范围中,能关联的蛋白质和基因数量见表1。HR48/HR0比较组中有3 973个基因在蛋白质和mRNA水平均被鉴定,并定量2 128个基因,有11个DEPs同DEGs关联;HS48/HS0比较组中有3 976个基因在蛋白质和mRNA水平均被鉴定,并定量2 127个基因,有9个DEPs同DEGs关联;HR0/HS0比较组中有3 977个基因在蛋白质和mRNA水平均被鉴定,并定量2 124个基因,有3个DEPs同DEGs关联;HR48/HS48比较组中有3 973个基因在蛋白质和mRNA水平均被鉴定,并定量2 132个基因,有4个DEPs同DEGs关联。
2.2.2 转录组和蛋白质相关性分析 对4个比较组中鉴定出的蛋白质水平和mRNA水平的相对表达量进行关联分析,且将蛋白和mRNA表达量的变化分为4种类型:蛋白和mRNA表达趋势相同、蛋白和mRNA表达趋势相反、蛋白表达有差异而mRNA表达无差异和蛋白表达无差异而mRNA表达有差异。结果表明,HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组的相关系数分别为0.1565、0.2687、0.1149和0.035(图1)。HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组中关联到蛋白和mRNA表达趋势相同的基因分别有11、9、2和3个,说明在转录后或翻译水平蛋白不受调控或调控较少的基因;蛋白和mRNA表达趋势相反的基因分别为0、0、1和1个;蛋白表达有差异而mRNA表达无差异的基因分别有225、241、210和207个,可能样本间的蛋白翻译修饰有差异;蛋白表达无差异而mRNA表达有差异的基因分别有35、26、10和8个,可能mRNA受到转录后调控(表2)。
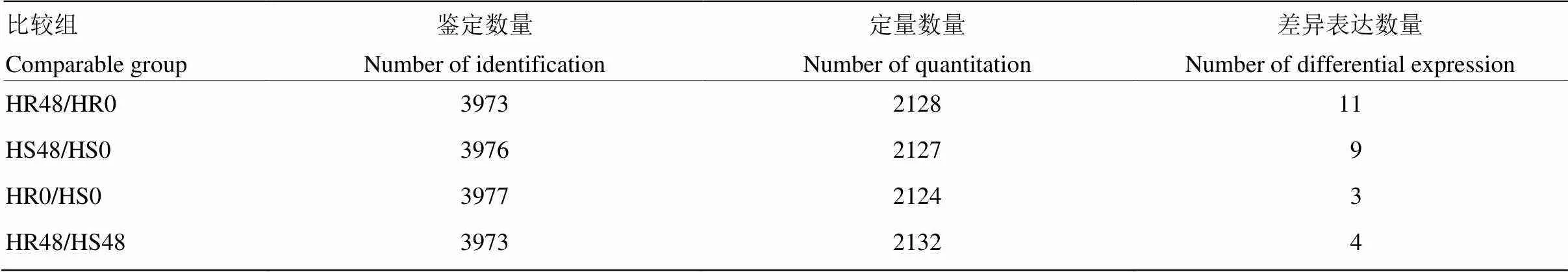
表1 在鉴定、定量、显著差异3个范围中,能被关联的蛋白质和基因数量关系
通过关联分析,发现一些可能与大豆抗豆卷叶螟胁迫相关的靶标蛋白(表3),如胰蛋白酶抑制剂A、查尔酮异构酶4、醌氧化还原酶蛋白质At1g23740、茎31 kD糖蛋白前体、橡胶蛋白、脂氧合酶9、α-加氧合酶1、植物凝集素前体、POD12、茎28 kD糖蛋白前体和应激蛋白SAM22等受豆卷叶螟胁迫后受均上调表达,可能在应答豆卷叶螟胁迫中扮演重要角色。而cAPX1在豆卷叶螟取食前后,在高抗材料中的表达量始终高于高感材料,可能是贮存在植物体内的抑制昆虫聚集的种间感应的化合物,是一个组成型防御蛋白。这些DEPs与DEGs的表达丰度不一致,如蛋白表达丰度较mRNA低或高,可能是在转录后或翻译水平调控,miRNA调控靶基因导致抑制蛋白翻译或积累蛋白。
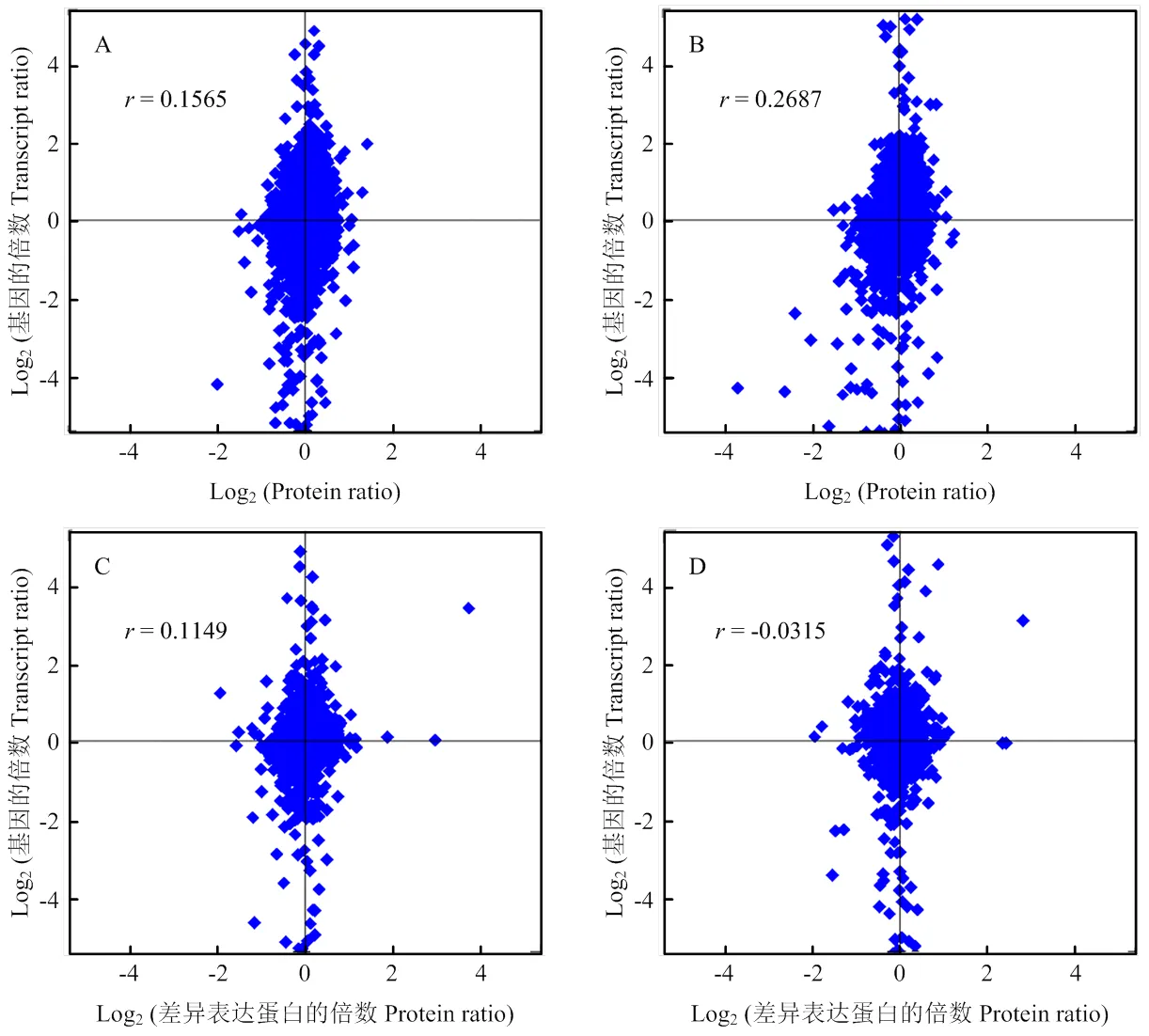
A:HR48-VS-HR0;B:HS48-VS-HS0;C:HS0-VS-HR0;D:HS48-VS-HR48

表2 4种蛋白和mRNA表达量变化类型
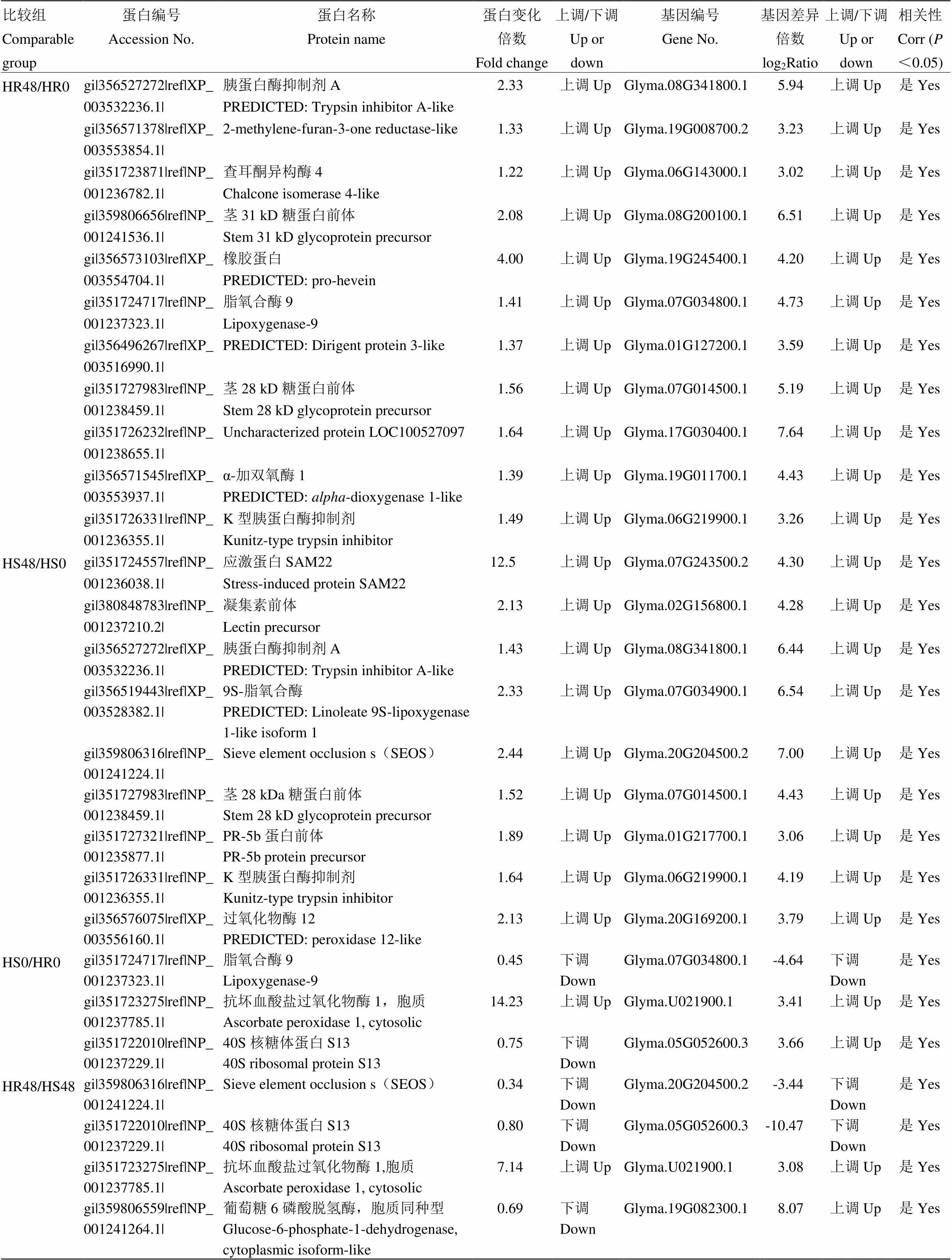
表3 基因和蛋白质水平上鉴定出的表达模式相同或相反的共有DEPs/DEGs
2.2.3 表达趋势相同的DEPs/DEGs生物信息学分析 对关联到的蛋白和mRNA表达趋势相同的DEPs/ DEGs进行GO功能显著性富集分析,以确定差异表达基因行使的主要功能。结果表明,HR48/HR0比较组中关联到的11个DEPs/DEGs生物学过程有代谢过程、刺激响应、细胞过程、多生物过程和免疫系统过程,参与细胞组分主要有细胞部分和细胞,分子功能主要有结合、催化活性、库营养活动、酶调节活性;HS48/HS0比较组中关联到的9个DEPs/DEGs生物学过程有代谢过程、刺激响应、细胞过程,参与细胞组分有细胞部分、细胞、细胞器和膜,分子功能主要有结合、催化活性、库营养活动、酶调节活性和抗氧化活性;HR0/HS0比较组中关联到的2个DEPs/DEGs生物学过程有代谢过程、细胞过程和刺激响应过程,参与细胞组分有细胞部分和细胞,分子功能主要有结合、催化活性、抗氧化活性;HR48/HS48比较组中关联到的3个DEPs/DEGs生物学过程有代谢过程、细胞过程和刺激响应过程,参与细胞组分有细胞部分、细胞、细胞器、细胞器部分、复杂大分子,分子功能主要有结合、催化活性、结构分子活性和抗氧化活性。当大豆受到豆卷叶螟危害以后,植物体内的防御系统会立即响应刺激,适当地增加体内的代谢活动,产生防御物质,而且增强各种酶的活性来促进防御。
利用KEGG pathway数据库对共有DEPs/DEGs进行了Pathway富集分析,确定蛋白质参与的主要生化代谢途径和信号转导途径。结果表明,HR48/HR0比较组中关联到的11个DEPs/DEGs中有9个Pathway到8条代谢通路中,为代谢途径、核糖体、类黄酮生物合成、亚油酸代谢、氨基糖和核苷酸糖代谢、氨酰生物合成、亚麻酸代谢、次生代谢产物的生物合成;HS48/HS0比较组关联到的9个DEPs/DEGs中有7个Pathway到6条代谢通路中,为代谢途径、亚油酸代谢、核糖体、苯基丙酸生物合成、RNA运转、次生代谢产物的生物合成。HR0/HS0比较组中关联到的2个DEPs/DEGs Pathway到3条代谢通路中,为亚油酸代谢、抗坏血酸盐和aldarate代谢、谷胱甘肽代谢;HR48/HS48比较组中关联到的3个DEPs/DEGs Pathway到3条代谢通路中,为谷胱甘肽代谢、核糖体、抗坏血酸盐和aldarate代谢。
2.3 功能方面关联分析
有些DEPs和DEGs虽然在定量上没有被关联,但它们可能同时参与某条Pathway通路,从而使该条通路发生变化。因此利用KEGG数据库对转录组和蛋白组数据同时进行Pathway注释,把这些DEPs和DEGs进行功能关联,将转录组和蛋白组表达数据在一张通路图中展示,直观展示一条通路中的所有转录组和蛋白质组鉴定和定量到的数据,从生物学代谢通路的角度进一步说明蛋白组和转录组富集关联情况。
功能方面关联到的代谢通路中涉及到的差异蛋白直接或间接参与大豆对豆卷叶螟的抗虫反应,在同一代谢途径中显示出mRNA上调表达,蛋白上调表达,mRNA和蛋白同时改变或者一方改变,mRNA和蛋白同时不变等情况。结果表明,HR48/HR0比较组有27条Pathway通路共关联到101个DEPs和23个DEGs,如:代谢途径中关联到64个DEPs和13个DEGs、次生代谢产物的生物合成中关联到44个DEPs和14个DEGs、核糖体中关联到23个DEPs和1个DEGs、淀粉和蔗糖代谢中关联到7个DEPs和1个DEGs、糖酵解/糖异生中关联到6个DEPs和1个DEG、苯基丙酸生物合成中关联到6个DEPs和7个DEGs等(表4),且这些DEPs和DEGs均上调表达。如在糖酵解/糖异生代谢途径中gi|356496743|ref|XP_003517225.1|、gi|356515641|ref|XP_003526507.1|、gi|356572486|ref| XP_003554399.1|、gi|356576165|ref|XP_003556204.1||、gi|363808320|ref|NP_001241992.1|、gi|356516690|ref| XP_003527026.1|上调表达(差异表达蛋白)和gi|356497822|ref|XP_003517756.1|上调表达(差异表达基因)能调控醋酸、乙醛等物质的合成;在甘油酯代谢途径中gi|356516690|ref|XP_003527026.1|上调表达(蛋白)和gi|356497822|ref|XP_003517756.1|上调表达(基因)都能调控D-甘油酸酯、D-甘油醛等物质的合成;在苯丙素生物合成途径中gi|351727703|ref| NP_001236658.1|、gi|356525622|ref|XP_003531423.1|、gi|356534700|ref| XP_003535890.1|、gi|356539666|ref| XP_003538316.1|、gi|358248528|ref|NP_001240152.1|、gi|359807592|ref| NP_001240903.1|上调表达(差异表达蛋白)和gi|351726279|ref|NP_001237377.1|、gi|356498192|ref| XP_003517937.1|、gi|356568192|ref| XP_003552297.1|、gi|356568196|ref|XP_003552299.1|、gi|356576075|ref| XP_003556160.1|、gi|363807568|ref| NP_001241894.1、gi|571514246|ref|XP_006597070.1|上调表达(差异表达基因)都能调控P-羟基苯基木质素、5-羟基苯基木质素、愈创木基木质素、紫丁香木质素等物质的合成;在丙酮酸代谢途径中gi|356495891|ref|XP_003516804.1|、gi|356516690|ref| XP_003527026.1|、gi|356555674|ref|XP_003546155.1|、gi|356569869|ref|XP_003553117.1|、gi|356576165|ref| XP_003556204.1|上调表达(差异表达蛋白)和gi|356497822|ref|XP_003517756.1|上调表达(差异基因)都能调控醋酸的合成(图2)。HS48/HS0比较组有15条Pathway通路共关联147个DEPs和16个DEGs,如:代谢途径中关联94个DEPs和9个DEGs、次生代谢产物的生物合成中关联59个DEPs和8个DEGs、核糖体中关联22个DEPs和1个DEGs等(表5),且这些DEPs和DEGs均上调表达。HR0/HS0比较组有18条Pathway通路共关联82个DEPs和10个DEGs,如:代谢途径中关联65个DEPs和4个DEGs、次生代谢产物的生物合成中关联44个DEPs和3个DEG、苯基丙酸生物合成中关联6个DEPs和1个DEGs等(表6);HR48/HS48比较组仅代谢途径中关联到71个DEPs和2个DEGs(表7)。
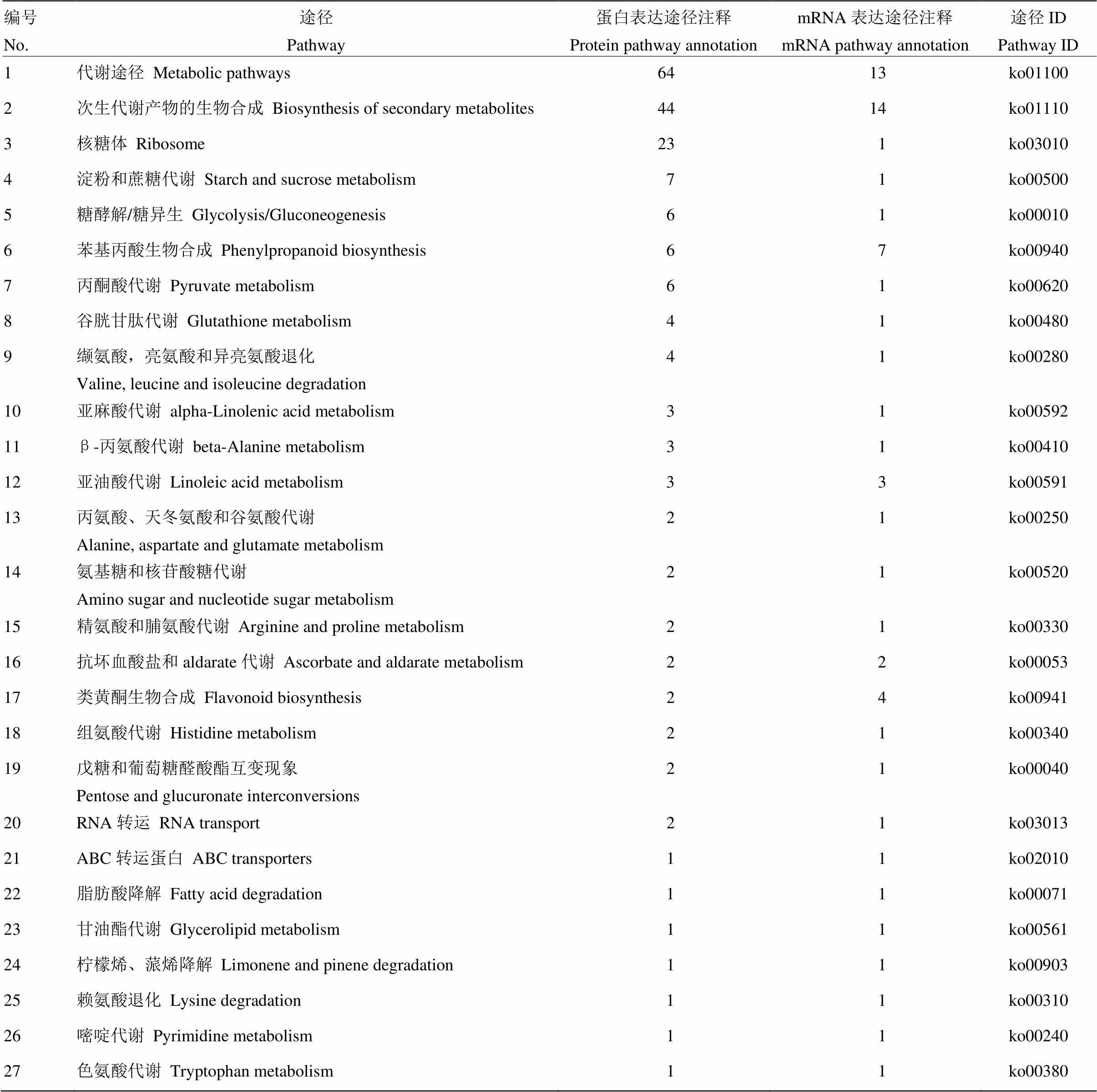
表4 HR48/HR0比较组差异表达蛋白与差异表达基因的功能关联分析
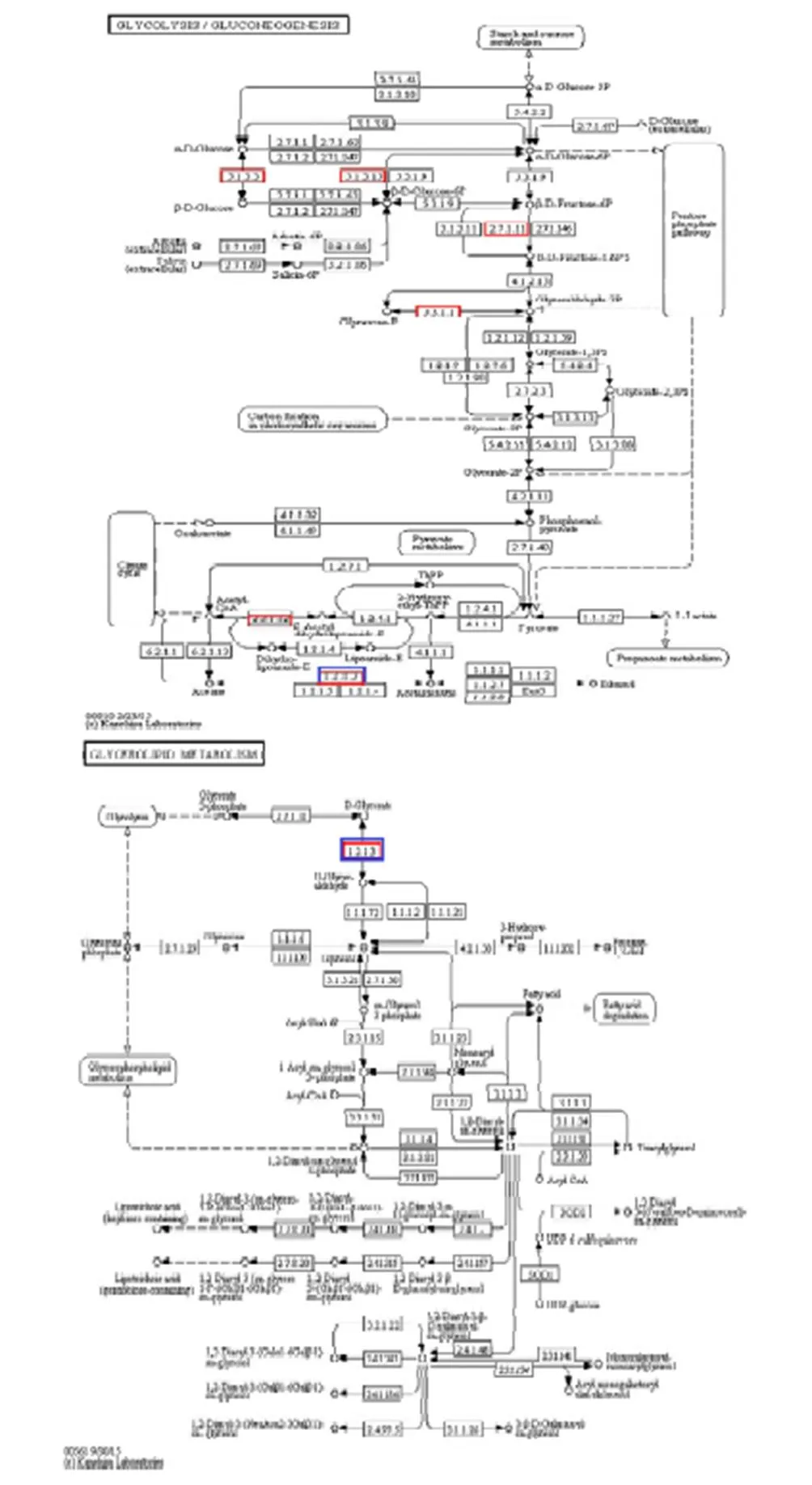
红色框表示蛋白表达有差异而mRNA表达无差异,蓝色框表示蛋白表达无差异而mRNA表达有差异
Red box showed protein_expression_mRNA_0, blue box showed protein _0_mRNA_expression
图2 HR48/HR0中仅蛋白表达有差异与仅mRNA表达有差异的基因的功能关联图
Fig. 2 Association with the function of the protein_expression_mRNA_0 or protein _0_mRNA_expression in the HR48/HR0
2.4 qRT-PCR验证
为了验证转录组和蛋白质组关联分析结果的可信度,对HR48/HR0中5个具有相同表达模式的DEGs/DEPs进行了qRT-PCR表达分析。发现5个DEGs/DEPs的qRT-PCR表达模式与其RNA-Seq和iTRAQ结果一致(图3)。
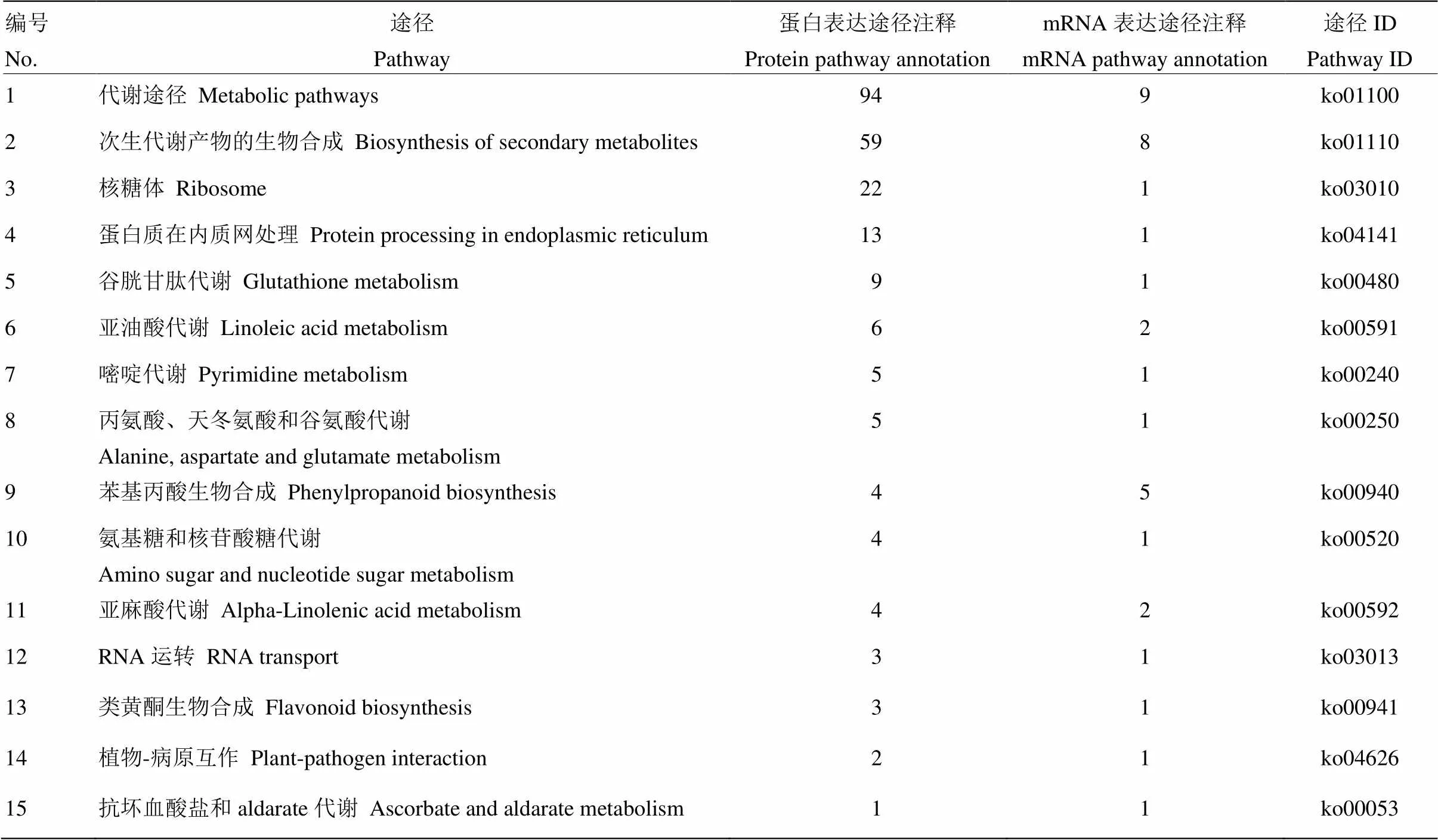
表5 HS48/HS0比较组中差异表达蛋白与差异表达基因的功能关联分析
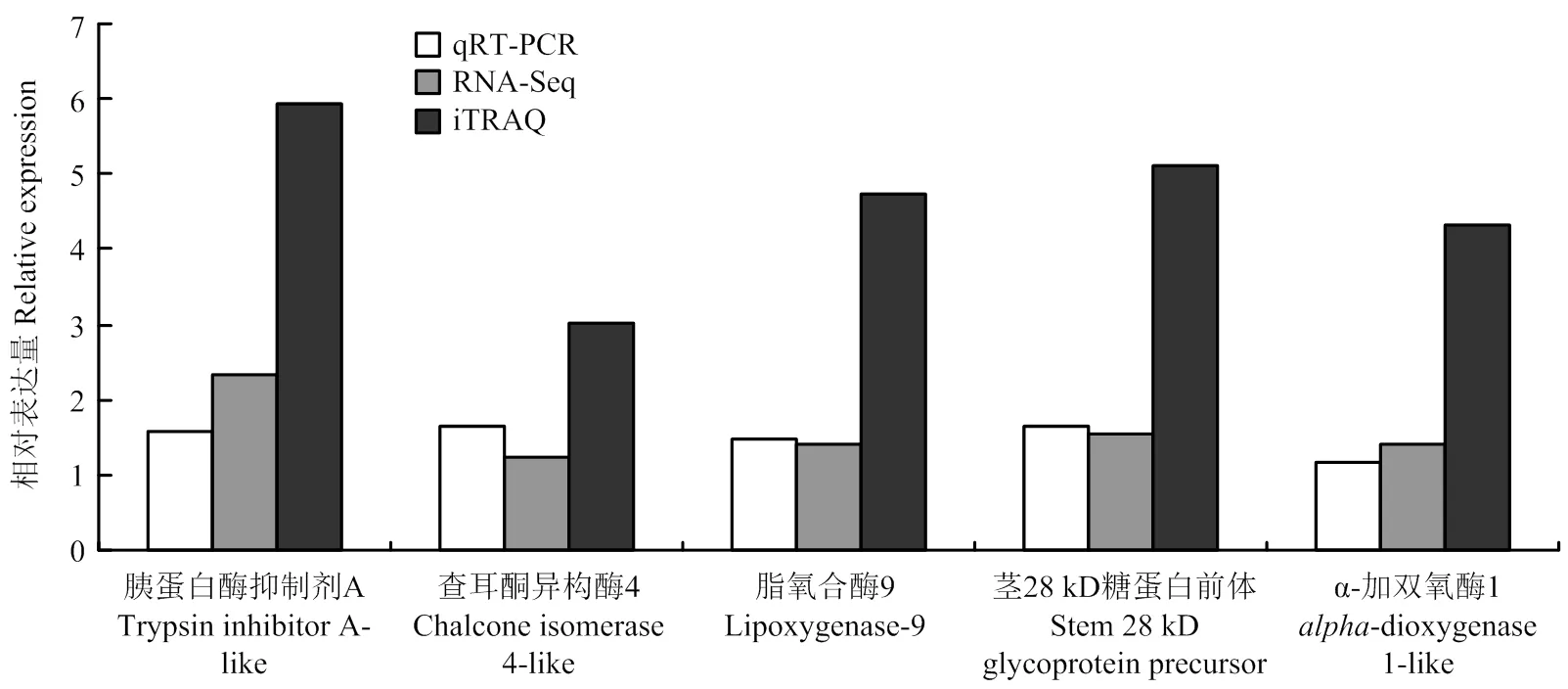
图3 实时荧光定量PCR分析

表6 HR0/HS0比较组中差异表达蛋白与差异表达基因的功能关联分析

表7 HR48/HS48比较组中差异表达蛋白与差异表达基因的功能关联分析
3 讨论
受到蛋白质组学技术的局限性,大多数研究主要集中在转录组水平,并认为蛋白质表达的多 少在很大程度上依赖于其对应的mRNA水平。由于蛋白质组和转录组关联分析可以深入了解蛋白质和基因的内在联系,能揭示基因表达的转录后调控状态,因此,蛋白质组和转录组间的关系研究可能是未来的系统生物研究中不可忽略的一部分[18]。Wu等[19]对开花后的晚熟型甜橙突变种和其野生种果肉的转录组和蛋白质组进行关联分析,与转录组关联上的差异蛋白数为54个,且其中许多为柑橘成熟调控网络相关的差异基因和蛋白。陈全助[20]对接菌后桉树叶片的转录组和蛋白质组进行关联分析,研究发现接菌诱导12 h比较组中蛋白组与转录组的相关系数=0.2935,而24 h的相关系数=0.6985,且多数关联差异蛋白参与了植物的抗逆响应,如几丁质酶、苯丙氨酸解氨酶、病程相关蛋白和过氧化物酶等。苏亚春[21]对甘蔗崖城05-179和“ROC”22黑穗病菌接种48 h后蔗芽的转录组和蛋白质组进行关联分析,研究发现崖城05-179比较组中蛋白组与转录组的相关系数=0.1502,“ROC”22的相关系数=0.2466。本研究通过对高抗品种赶泰-2-2和高感品种皖82-178受豆卷叶螟取食胁迫48 h时的叶片蛋白质组和转录组进行关联分析。关联分析结果显示,HR48/HR0、HS48/HS0、HR0/HS0和HR48/HS48比较组中蛋白质组与转录组的相关系数分别为0.1565、0.2687、0.1149和0.035。可见4个比较组的相关性并不是很高,因DNA转录成mRNA以及mRNA翻译成蛋白质的各个步骤会受到各种因子对转录、翻译过程的调节及翻译后调控[22],使mRNA转录子的数目、蛋白质位置、数量和功能发生改变,从而导致mRNA与相对应蛋白质之间相关性的丢失[23],且植株中多数基因和蛋白非差异表达。且本研究中各比较组鉴定出的DEPs较多,其中HR48/HR0比较组鉴定出236个DEPs,HS48/HS0比较组鉴定出250个DEPs,HR0/HS0比较组鉴定出213个DEPs,HR48/HS48比较组鉴定出211个DEPs,不易筛选出与抗豆卷叶螟相关的目标蛋白。通过转录组与蛋白质组关联分析的手段,比较容易找出与与豆卷叶螟的抗性相关基因和蛋白及抗虫相关代谢通路。如定量关联分析表明,HR48/HR0和HS48/HS0比较组中筛选出的与转录组关联的差异蛋白分别为11个和9个,且表达趋势相同均为上调表达,关联到差异蛋白主要与植物抗逆性密切相关,如胰蛋白酶抑制剂A、植物凝集素、橡胶蛋白、脂氧合酶、过氧化物酶12、α-加双氧酶1、病程相关蛋白PR-5b、应激蛋白SAM22、查尔酮异构酶4等,可能在应答豆卷叶螟胁迫中扮演重要角色。HR0/HS0和HR48/HS48比较组中与转录组关联的差异蛋白分别为3个和4个,其中与差异基因表达趋势相同的差异蛋白数量分别为2个和3个,其中与植物抗逆相关的抗坏血酸盐过氧化物酶1(cAPX1)上调表达,即豆卷叶螟取食前后其在赶泰-2-2中的含量始终高于皖82-178,可能是贮存在植物体内的抑制昆虫聚集的种间感应的化合物,是一种组成型防御蛋白。功能方面关联分析表明,HR48/HR0比较组有27条Pathway通路共关联到101个DEPs和23个DEGs,包括代谢途径、次生代谢产物的生物合成、核糖体、苯基丙酸生物合成等;HS48/HS0比较组有15条Pathway通路共关联到147个DEPs和16个DEGs,包括代谢途径、次生代谢产物的生物合成、核糖体等;HR0/HS0比较组有18条Pathway通路共关联到82个DEPs和10个DEGs,包括代谢途径、次生代谢产物的生物合成、苯基丙酸生物合成等;HR48/HS48比较组仅有代谢途径关联到71个DEPs和2个DEGs。且所有DEPs和DEGs均上调表达,在同一代谢通路共同协调或相互协调作用调控与抗虫相关的基因合成,功能方面关联的代谢通路中涉及到的差异蛋白直接或间接参与大豆对豆卷叶螟的抗虫反应。
大豆胰蛋白酶抑制剂能显著抑制昆虫肠中蛋白酶活性,阻碍昆虫消化蛋白质,尤其对鳞翅目昆虫有较强的抑制作用[24-26]。赤拟谷盗[27]、甜菜夜蛾[28]、美洲棉铃虫[29]、四纹豆象[29]和蜜蜂[30]等昆虫喂养胰蛋白酶抑制剂后出现营养不良甚至死亡率升高的现象。植物凝集素是至少具有一个可与单糖或寡聚糖特异可逆结合的非催化结构域的蛋白,当凝集素随食物进入昆虫肠道中诱发局部或系统性毒害效应后,可能引起昆虫的拒食、生长停滞甚至死亡[31]。已有研究表明烟草夜蛾幼虫[32]、桃蚜[33]、番茄夜蛾[34]等昆虫取食转凝集素基因的植物后,其生存率、繁殖率等都大大降低。豆卷叶螟取食诱导后,胰蛋白酶抑制剂A在抗感材料中均上调表达,凝集素前体在皖82-178中上调表达。推测豆卷叶螟侵害大豆后,胰蛋白酶抑制剂A、K型胰蛋白酶抑制剂和凝集素前体从大豆受害细胞中释放出来,影响昆虫营养物质的正常吸收,使昆虫生长发育受到抑制甚至死亡,从而达到杀虫防御虫害的目的。
茉莉酸(Jasmonic acid,JA)信号途径在植物抗虫反应中起重要作用,参与机械损伤和抗虫防御信号转导,调控着植物下游防御反应基因的表达,显著诱导植物体内防御系统响应,减少虫害,是病虫害和机械伤害诱导的主要途径[35-38]。脂氧合酶(LOX)是JA合成过程中的关键酶类[39],且是植物诱导防御途径中的重要信号因子[40]。-加双氧酶(-DOX)也能够催化脂肪酸氧化,其产物2-氢过氧化脂肪酸是一种新型的氧化脂肪酸,也是JA途径中的一个酶[41]。豆卷叶螟取食诱导后,脂氧合酶-9在赶泰-2-2中上调表达,9S-脂氧合酶和α-加双氧酶在皖82-178中上调表达。说明豆卷叶螟诱导激活了大豆JA信号途径,JA信号途径在不同基因型大豆抗虫防御过程中均起重要作用。
过氧化物酶(Peroxidase,POD)是植物体内重要的防御酶系,能催化H2O2的分解来清除细胞受胁迫时产生的ROS[42-43]。POD在小麦[44-45]、高粱[46]、黄瓜[47]和水稻[41, 48]等作物中均能被昆虫诱导。豆卷叶螟诱导后,氧化物酶-12在皖82-178中上调表达,推测该基因在高感材料中上调表达有助于降低由虫害引起的ROS对大豆造成的伤害,维持大豆正常代谢功能。
存在于植物叶绿体和细胞质中的抗坏血酸盐过氧化物酶(APX)是H2O2解毒体系中的关键酶,它通过催化抗坏血酸-谷胱甘肽循环来发挥这一关键作用[49-50]。高等植物的APX同工酶主要分为胞质APX(cAPX)和叶绿体APX(chlAPX),其中cAPX是植物应答恶劣环境的主要酶类[51]。CALDWELL等[52]研究表明大豆中cAPX的转录、翻译和翻译后调控能增强其抵抗环境胁迫的能力。Yoshimura等[53]研究表明缺少cAPX1会导致叶绿体H2O2清除系统的崩溃。豆卷叶螟取食前后,cAPX1在赶泰-2-2中表达量始终高于皖82-178,说明cAPX1的表达能保护重要的细胞区域免受氧化胁迫,严格控制细胞内H2O2水平,从而抵制虫害入侵。
查尔酮异构酶是黄酮类化合物生物合成过程中的关键酶,查尔酮在查尔酮异构酶作用下形成类黄酮,是合成黄烷酮、黄酮、黄酮醇及花色素苷等物质所必需的酶,涉及了苯丙氨酸代谢途径中各种具有防御性产物的生物合成,在植物的抗胁迫逆境胁迫方面也起着重要的作用[54-55]。豆卷叶螟诱导后,查尔酮异构酶4在赶泰-2-2中上调表达,推测其在高抗材料中能应答豆卷叶螟的防御反应。
PR-5蛋白是PRs蛋白家族中的一类,在抵抗逆境胁迫中起着非常重要的作用[56]。机械损伤或外施SA、甲基紫精、真菌诱导物、过氧化氢、磷酸钠均能诱导大豆的应激蛋白SAM22表达[57]。豆卷叶螟诱导后,PR-5蛋白和应激蛋白SAM22在皖82-178中上调表达,推测其可能参与了高感材料的防御反应。
4 结论
HR48/HR0和HS48/HS0比较组中分别鉴定出11个和9个DEPs/DEGs且表达模式相同,HR0/HS0和HR48/HS48比较组中分别鉴定出3个和4个DEPs/ DEGs,表达模式相同的分别有2个和3个。这些差异蛋白与代谢途径、核糖体、类黄酮生物合成、亚油酸代谢、氨基糖-核苷酸代谢、氨酰生物合成、亚麻酸代谢、次生代谢产物的生物合成、苯基丙酸生物合成、RNA运转、谷胱甘肽代谢、抗坏血酸盐和aldarate代谢等相关。同时,胰蛋白酶抑制剂A、K型胰蛋白酶抑制剂、查尔酮异构酶4、脂氧合酶9、α-加氧合酶1、植物凝集素前体、POD12、应激蛋白SAM22和cAPX1等可能是大豆抗豆卷叶螟潜在的靶标蛋白。
[1] Stacey G..: New York: Verlag New York Inc, 2008: 3-15.
[2] 中国农作物病虫图谱编写组. 中国农作物病虫图谱: 第五分册, 油料病虫(一). 北京: 中国农业出版社, 1982: 136-137.
Editorial committee of plate of Chinese diseases and insects on crop.. Beijing: China Agriculture press, 1982: 136-137. (in Chinese)
[3] 崔章林, 盖钧镒, 吉东风, 任珍静. 大豆种质资源对食叶性害虫抗性的鉴定. 大豆科学, 1997, 16(2): 93-102.
CUI Z L, GAI J Y, JI D F, REN Z J. A study on leaf-feeding insect species on soybeans in Nanjing area., 1997, 16(2): 93-102. (in Chinese)
[4] 孙祖东, 杨守臻, 陈怀珠, 李初英, 龙丽萍. 大豆对豆卷叶螟的抗性鉴定. 中国油料作物学报, 2005, 27(4): 69-71.
SUN Z D, YANG S Z, CHEN H Z, LI C Y, LONG L P. Identification of soybean resistance to bean pyralid (Fabricicus) and oviposition preference of bean pyralid on soybean varieties., 2005, 27(4): 69-71. (in Chinese)
[5] 邢光南, 谭连美, 刘泽稀楠, 岳汉, 张寒竹, 史鸿飞, 赵团结, 盖钧镒. 大豆地方品种叶片叶柄茸毛性状的形态变异及其与豆卷叶螟抗性的相关分析. 大豆科学, 2012, 5(31): 691-696.
XING G N, TAN L M, LIU Z X N, YUE H, ZHANG H Z, SHI H F, ZHAO T J, GAI J Y. Morphological variation of pubescence on leaf blade and petiole and their correlation with resistance to bean pyralid(Fabricius) in soybean landraces., 2012, 5(31): 691-696.(in Chinese)
[6] 邢光南, 赵团结, 盖钧镒. 大豆对豆卷叶螟(Fabricius)抗性的遗传分析. 作物学报, 2008, 34(1): 8-16.
XING G N, ZHAO T J, GAI J Y. Inheritance of resistance to(Fabricius) in soybean., 2008, 34(1): 8-16.(in Chinese)
[7] 李广军, 程利国, 张国政, 何小红, 智海剑, 章元明. 大豆对豆卷叶螟抗性的主基因+多基因混合遗传. 大豆科学, 2008, 27(1): 33-36, 41.
LI G J, CHENG L G, ZHANG G Z, HE X H, ZHI H J, ZHANG Y M. Mixed major-gene plus polyegens inheritance analysis for resistance in soybean to bean pyralidFabricius)., 2008, 27(1): 33-36, 41. (in Chinese)
[8] 李广军, 李河南, 程利国, 章元明. 大豆对豆卷叶螟抗性的QTL定位. 中国油料作物学报, 2009, 31(3): 365-369.
LI G J, LI H N, CHENG L G, ZHANG Y M. Mapping quantitative trait loci for resistance in soybean to bean pyralid (Fabricius)., 2009, 31(3): 365-369.(in Chinese)
[9] XING G N, ZHOU B, WANG Y F, ZHAO T J, YU D Y, CHEN S Y, GAI J Y. Genetic components and major QTL confer resistance to bean pyralid (Fabricius) under multiple environments in four RIL populations of soybean., 2012, 125: 859-875.
[10] 陈晶, 韩贵清, 尚晨, 张海玲, 李佶恺, 刘慧莹, 张月学. 去除紫花苜蓿叶片高丰度蛋白的方法及其应用. 草业学报, 2015, 24(7): 131-138.
CHEN J, HAN G Q, SHANG C, ZHANG H L, LI J K, LIU H Y, ZHANG Y X. Proteomic methods for removing high-abundance proteins in alfalfa leaf., 2015, 24(7): 131-138. (in Chinese)
[11] BRADFORD B M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding., 1976, 72: 248-254.
[12] MORTAZAVI A, WILLIAMS B A, MCCUE K, SCHAEFFER L, WOLD B. Mapping and quantifying mammalian transcriptomes by RNA-Seq., 2008, 5(7): 621-628.
[13] JIA W, QIU K, HE M, SONG P, ZHOU Q, ZHOU F, YU Y, ZHU D, NICKERSON M L, WAN S, LIAO X, ZHU X, PENG S, LI Y, WANG J, GUO G. SOAPfuse: an algorithm for identifying fusion transcripts from paired-end RNA-Seq data., 2013, 14: R12.
[14] TRAPNELL C, PACHER L, SALZBERG S L. TopHat: discovering splice junctions with RNA-Seq., 2009, 25(9): 1105-1111.
[15] TRAPELL C, ROBERTS A, GOFF L, PERTEA G, KIM D, KELLEY D R, PIMENTAL H, SALZBERG S L, RINN J L, PACHTER L. Differential gene and transcript expression analysis of RNA-Seq experiments with TopHat and Cufflinks., 2012, 7(3): 562-578.
[16] BENJAMINI Y, YEKTIELI D. The control of the false discovery rate in multiple testing under dependency., 2001, 29(4): 1165-1188.
[17] LAN P, LI W F, SCHMIDT W. Complementary proteome and transcriptome profiling in phosphate-deficientroots reveals multiple levels of gene regulation., 2012, 11(11): 1156-1166.
[18] KITANO H. Systems biology: a brief overview., 2002, 295(5560): 1662-1164.
[19] WU J, XU Z, ZHANG Y, CHAI L J, YI H L, DENG X X. An integrative analysis of the transcriptome and proteome of the pulp of a spontaneous late-ripening sweet orange mutant and its wild type improves our understanding of fruit ripening in citins., 2014, 65(6): 1651-1671.
[20] 陈全助. 福建桉树焦枯病菌鉴定及其诱导下桉树转录组和蛋白组学研究[D]. 福州: 福建农林大学, 2013.
CHEN Q Z. Pathogenic identification of cylindrocladium leaf blight collected from Fujian province and studies on transcriptomics and proteomics of eucalyptus induced by[D]. Fuzhou: Fujian Agriculture and Forestry University,2013.(in Chinese)
[21] 苏亚春. 甘庶应答黑穗病菌侵染的转录组与蛋白组研究及抗性相关基因挖掘[D]. 福州: 福建农林大学, 2014.
SU Y C. Transcriptomics and proteomics of sugarcane response toinfection and mining of resistance-related genes[D]. Fuzhou: Fujian Agriculture and Forestry University, 2014. (in Chinese)
[22] DERIBE Y L, PAWSON T, DIKIC T. Post-translational modifications in signal integration., 2010, 17(6): 666-672.
[23] KARAS M, HILLENKAMP E. Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons., 1988, 60(20): 2299-2301.
[24] TAKEHIKO K, TOKUJI I, SUSUMU T. Studies on soybean trypsin inhibitors., 1973, 32(3): 401-407.
[25] SONG S L, KIM C H, BAEK S J, CHOI Y D. Nucleotide sequences of cDNAs encoding the precursors for soybean () trypsin inhibitors (Kunitz type)., 1993, 101(4): 1401-1402.
[26] GATEHOUSE A M R, SHI Y, POWELL K S, BROUGH C, HILDER V A, HAMILTON W D O, NEWELL C A, MERRYWEATHER A, BOULTER D, GATEHOUSE J A. Approaches to insect resistance using transgenic plants., 1993, 342(1301): 279-286.
[27] BIRK Y. Purification and some properties of a highly active inhibitor of trypsin and α-chymotrypsin from soybeans., 1962, 54(2): 378-381.
[28] BROADWAY R M, DUFFEY S S, BROADWAY R M, DUFFEY S S. Plant proteinase inhibitors mechanism of action and effect on the growth and digestive physiology of larvaland Spodoptera exiqua., 1986, 32(10): 827-833.
[29] MACEDO M L R, MELLO G C, FREIRE M D G M, NOVELLO J C, MARANGONI S, DE MATOS D G G. Effect of a trypsin inhibitor fromseeds on the development of., 2002, 40(10): 891-898.
[30] SAGILI R R, TANYA P, KEYAN Z S. Effects of soybean trypsin inhibitor onhypopharyngeal gland protein content total midgut protease activity and survival of the honey bee(L.)., 2005, 51: 953-957.
[31] PEUMANS W J, VAN DAMME E J M. Lectins as plant defense proteins., 1995, 109(2): 347-352.
[32] BOULTER D, EDWARDS G A, GATEHOUSE A M R, GATEHOUSE J A, HILDER V A. Additive protective effects of incorporating two different higher plant insect resistance genes in transgenic tobacco plants., 1990, 9(5): 351-354.
[33] HILDER V A, POWELL K S, GATEHOUSE A M R, GATEHOUSE J A, GATEHOUSE L N, SHI Y, HAMILTON W D O, MERRYWEATHER A, NEWELL C A, TIMANS J C, PEUMANS WJ, EAMME E V, DOULTER D. Expression of tobacco plants results in added snow drop lectin transgenic protection against aphids., 1995, 4(1): 18-25.
[34] VAN DAMME E J M, PEUMANS W J, PUSZTAI A, BARDOCZ S.. New York : John Willey & Sons, 1998, 20-31.
[35] REYMOND P, FARMER E E. Jasmonate and salicylate as global signals for defense gene expression., 1998, 1(1): 404-411.
[36] REYMOND P, WEBER H, DAMOND M, FANNER E E. Differential gene expression in response to mechanical wounding and insect feeding in., 2000, 12(5): 707-719.
[37] ZHOU G X, WANG X,YAN F, WANG X, LI R, CHENG J A, LOU Y G. Genome-wide transcriptional changes and defense-related chemical profiling of rice in response to infestation by the rice striped stem borersuppressalis., 2011, 143(1): 21-40.
[38] BIRKETT M A, CAMPBEEL C A, CHAMBERLAIN K, GUERRIERI E, HICK A J, MARTN J L, MATHES M, NAPIER J A, PICKETT J A, POPPY G M, POW E M, PYE B J, SMART L E, WADHAMS G H, WADHAMS L J, WOODCOCK C M. New roles for cis-jasmone as an insect semiochemical and in plant defense., 2000, 97(97): 9329-9334.
[39] SCHALLER F, SCHALLER A, STINTZI A. Biosynthesis and metabolism of jasmonates., 2004, 23(3): 179-199.
[40] GARDNER W H. Biological roles and biochemistry of the lipoxygenase pathway., 1995, 30(2): 197-205.
[41] WEI Z, HU W, LIN Q S, CHENG X Y, TONG M J, ZHU L L, CHEN R Z, HE G C. Understanding rice plant resistance to the brown plant hopper (): A proteomic approach., 2009, 9(9): 2798-2808.
[42] CHRISTENSEN J H, BAUW G, WELINDER K G, VAN M M, BOERJAN W. Purification and characterization of peroxidases correlated with lignification in poplar xylem., 1998, 118(1): 125-135.
[43] VITALI A, BOTTA B, DELLE M G, ZAPPITELLI S, RICCIARDI P, MELINO S, PETRUZZELLI R, GIARDINA B. Purification and partial characterization of a peroxidase from plant cell cultures of Cassia didymobotrya and biotransformation studies., 1998, 331(2): 490-518.
[44] LESZCZYNSKI B. Changes in phenols content and metabolism in leaves of susceptible and resistant wheat cultivars infested by(L.).,1985, 100(4): 343-348.
[45] NI X, QUISENBERRY S S, HENG-MOSS T, MARKWELL J, SARATH G, KLUCAS R, BAXENDALE F. Oxidative responses of resistant and susceptible cereal leaves to symptomatic and nonsymptomatic cereal aphid () feeding., 2001, 94(3): 743-751.
[46] HUANG Y H. Phloem feeding regulates the plant defense pathways responding to both aphid infestation and pathogen infection[C]. Beijing:2006: 215-219.
[47] ZHANG S Z, ZHANG F, HUA B Z. Enhancement of phenylalanine ammonia lyase, polyphenoloxidase, and peroxidase in cucumber seedlings by(Gennadius) (Hemiptera: Aleyrodidae) infestation., 2008, 7(1): 82-87.
[48] DU B, WEI Z, WANG Z Q, WANG X X, PENG X X, DU B, CHEN R Z, ZHU L L, HE G C. Phloem-exudate proteome analysis of response to insect brown plant-hopper in rice., 2015, 183: 13-22.
[49] KHATOON A, REHMAN S, Oh M W, WOO S H, KOMATSU S. Analysis of response mechanism in soybean under low oxygen and flooding stresses using gel-base proteomics technique., 2012, 39(12): 10581-10594.
[50] ASADA K. Ascorbate peroxidase: A hydrogen peroxide scavenging enzyme in plants., 1992, 85(2): 235-241.
[51] SHIGEOKA S, ISHIKAWA T, TAMOI M, MIYAGAWA Y, TAKEDA T, YABUTA Y, YOSHIMURA K. Regulation and function of ascorbate peroxidase isoenzymes.,2002, 53(372):1305-1319.
[52] CALDWELL R C, TURANO J F, MCMAHON B M. Identification of two cytosolic ascorbate peroxidase cDNAs from soybean leaves and characterization of their products by functional expression., 1998, 204: 120-126.
[53] YOSHIMURA K, YABUTA Y, ISHIKAWA T, SHIGEOKA S. Expression of spinach ascorbate peroxidase isoenzymes in response to oxidative stresses., 2000, 123(1): 223-234.
[54] KOES R E, QUATTROCCHIO F, MOLIN M. The flaonoivd biosynthetic pathway in plants: Function and evolution., 1994, 16: 123-132.
[55] MUIR S R, COLLINS G J, ROBINSON S, HUGHES S, BOVY A, RIC DE VOS C H, VAN TUNEN A J, VERHOEYEN M E. Overexpression of petunia chalcone isomerase in tomato results in fruit containing increased levels of flavonols., 2001, 19(5): 470-474.
[56] LOON L C V. The nomenclature of pathogenesis-related protein., 1990, 37(3): 229-230.
[57] CROWEEL D N, JOHN M E, RUSSELLl D, AMASINO R M. Characterization of a stress-induced, developmentally regulated gene family from soybean., 1992, 18(3): 459-466.
(责任编辑 李莉)
Correlation Analysis on Transcriptomic and Proteome of Soybean Resistance to Bean pyralid()
ZENG WeiYing, SUN ZuDong, LAI ZhenGuang, CAI ZhaoYan, CHEN HuaiZhu, YANG ShouZhen, TANG XiangMin
(Institute of Economic Crops of Guangxi Academy of Agricultural Sciences/MOA Southwest Experimental Station of Maize-Soybean Intercrop, Nanning 530007)
【】In order to lay a solid basis for a holistic understanding of the defense molecular mechanism of soybean resistance toin soybean, the correlation analysis of the transcriptomic and proteomic of soybean leaves (the highly resistant line (Gantai-2-2) and the highly susceptible line (Wan 82-178)) underlarva feeding for 0 h and 48 h was conducted. 【】In this study, soybeans with high resistance and susceptibility towere selected as the research objects. The gene and protein expression abundance was analyzed for the soybean following thefeedings using RNA-Seq and iTRAQ technology, respectively. Then the correlation value between the protein level and the transcription level was calculated.【】There were 236 DEPs and 1 064 DEGs, 250 DEPs and 680 DEGs, 213 DEPs and 605 DEGs, 211 DEPs and 468 DEGs identified in HR48/HR0, HS48/HS0, HR0/HS0 and HR48/HS48. The results indicated that the correlations between the quantitative protein and mRNA were 0.156, 0.2687, 0.1149, and 0.035 in HR48/HR0, HS48/HS0, HR0/HS0 and HR48/HS48, and 11, 9, 3 and 4 DEPs/DEGs were identified, of which 11, 9, 2 and 3 with same expression trend, respectively. These differential expression proteins were revealed to be involved in metabolic pathways, ribosome, flavonoid biosynthesis, linoleic acid metabolism, amino sugar and nucleotide sugar metabolism, aminoacyl-RNA biosynthesis, alpha-linoleic acid metabolism, biosynthesis of secondary metabolism, phenylpropanoid biosynthesis, RNA transport, glutathione metabolism, ascorbate andaldarate metabolism. The function correlation analysis results showed that 27 pathways correlated to 101 DEPs and 23 DEGs were identified in HR48/HR0, 15 pathways correlated to 147 DEPs and 16 DEGs were identified in HS48/HS0, 18 pathways correlated to 82 DEPs and 10 DEGs were identified in HR0/HS0, and 1 pathway correlated to 71 DEPs and 2 DEGs were identified in HR48/HS48. Candidate differential expression proteins closely related to insect resistance, such as trypsin inhibitor A-like, kunitz-type trypsin inhibitor, chalcone isomerase 4-like, lipoxygenase-9, alpha-dioxygenase 1-like, linoleate 9S-lipoxygenase 1-like isoform 1, lectin precursor, peroxidase 12-like, stress-induced protein SAM22, scorbate peroxidase 1, cytosolic, and so on. Finally, qRT-PCR analysis confirmed that the two kinds of “omics” results were reliable. 【】In this study, some proteins and metabolic pathways were related to insect-resistant, these differential proteins were directly or indirectly involved in soybean response to.
soybean;; transcriptomic; proteomic; correlation analysis
2017-11-03;
2017-12-18
国家重点研发计划(2017YFD0101504)、广西自然科学基金(2017GXNSFDA198037,2016GXNSFAA380238)、广西农业科学院科技发展基金(桂农科2016JM03)
曾维英,E-mail:zengweiying_1981@163.com。
孙祖东,E-mail:sunzudong639@163.com

