玉米籽粒淀粉粒密度基因tw1的精细定位
孙粲然,张雪海,马指挥,郭战勇,汤继华,付志远
玉米籽粒淀粉粒密度基因的精细定位
孙粲然,张雪海,马指挥,郭战勇,汤继华,付志远
(河南农业大学农学院,郑州 450002)
【】淀粉粒密度影响籽粒容重,通过对一个玉米籽粒淀粉粒密度突变体进行鉴定和精细定位,为容重相关基因的克隆和功能验证奠定基础。【】以育种选系过程中发现的一个淀粉粒密度突变体为材料,利用近红外光谱分析仪检测其籽粒内部化学成分的变化,用扫描电镜观察授粉后18—45 d正常籽粒和突变籽粒中淀粉粒形态的差异;于2014—2016年分别在河南郑州和原阳以及海南三亚种植与B73的杂交组合及F2和BC1分离群体,并对其进行遗传分析;使用来自maizeGDB(http://www.maizegdb.org)的覆盖全基因组的1 000对SSR引物,通过集团分离分析法(bulked segregation analysis,BSA)筛选与目的基因紧密连锁的标记,实现目的基因的初步定位;并在该定位区间内开发新的标记,对从38 000 BC1分离群体中筛选出的交换单株进行基因型分析,实现目的基因的精细定位;通过候选基因序列分析、功能预测和等位性测验确定首选候选基因。【】该突变体籽粒较正常籽粒体积变小,比重增加;细胞学和化学组份分析结果表明,与野生型籽粒相比,突变体籽粒中的粗蛋白含量降低,粗淀粉含量没有显著变化,淀粉粒形状不规则且变小、密度增加,可能是导致籽粒容重变大的原因;对授粉后不同天数籽粒内部淀粉粒结构的观察显示,突变体籽粒淀粉粒的密度比正常籽粒密度大,并随发育进程不断增加;对与B73的F2及测交后代分离群体的遗传分析结果表明,籽粒突变是由单隐性基因(命名为)控制的;该基因首先被定位在第6染色体的SSR标记umc1105和bnlg1154之间,物理距离为22 Mb;利用上述2个标记对BC1群体进行交换单株筛选,并开发标记,将该基因定位于SSR标记B3和A47之间,物理距离为0.2 Mb;在该候选区段内有包含在内的3个候选基因,等位性测验结果表明,与不是等位基因;候选基因序列分析和功能预测结果表明GRMZM2G042607编码的蛋白具有碳水化合物识别结构域,在种子中对碳水化合物的储藏起沉积作用,是最可能的候选基因。【】实现了籽粒淀粉粒密度突变性状基因的精细定位,并确定了候选基因为编码一种β-1,3半乳糖基转移酶的GRMZM2G042607。
玉米;容重;淀粉粒密度;;精细定位
0 引言
【研究意义】玉米是中国第一大粮食作物,对于保证国家粮食安全具有极其重要的意义。2010年起,中国已由玉米出口国转变为玉米进口国,玉米供需矛盾日益突出。销售市场竞争的日益加剧对玉米的商品品质和加工品质提出了越来越高的要求。容重能够反映玉米的使用价值和商品价值[1-3],是检验籽粒商品品质的重要指标之一。在国际贸易中,容重已成为国际上玉米质量定级的重要参考[4]。此外,容重还是玉米产量的一个次级构成因子,与玉米产量密切相关[5]。影响玉米容重的诸多因素中,如玉米的形状、胚乳形态[6]、灌浆期[7]、生育期[8]等,遗传因素占主导地位[9]。但容重性状的量化测定需要的种子量较大,难以用于群体单株果穗籽粒容重的遗传分析中。比重是种子绝对重量与绝对体积的比值(g·ml-1),与容重相比,种子的比重排除了籽粒之间空隙以及杂物的影响,与种子的化学成分密切相关,是影响容重的主要因子,因此,遗传分析中多用籽粒比重的大小来反映容重的大小。【前人研究进展】目前,玉米籽粒容重的遗传研究主要集中在相关性状的QTL定位方面。Ding等[10]利用玉米的F2:3群体定位到16个分布在第1、2、3、4、5、7染色体上的容重QTL。许理文等[6]用SNP芯片对240个DH系群体进行基因型分析,在4个环境下检测到5个控制玉米容重的QTL,共解释23.61%的表型变异,其中位于第1染色体的和第9染色体的贡献率最高。Beavis等[11]利用96个RFLP标记对B73和Mo17构建的F2:4群体的容重性状进行QTL分析,鉴定出分布在玉米第3、5、7、10染色体上的4个QTL。Ajnone-marsan等[12]利用B73和A7构建的F2:3群体,定位到6个容重相关QTL,分布在玉米的第1、2、3、5、9染色体上。Peng等[13]利用F2:3分离群体,结合两年三点的试验数据,共定位到4个籽粒容重相关的QTL,其中只有位于第1染色体umc1298—bnlg1671标记之间的QTL在6个环境中都被检测到,并且表现为稳定遗传的加性效应,对表型的贡献率达到7%;白光红等[14]利用综3和87-1的294个F9:10家系,鉴定出7个与玉米籽粒比重有关的QTL,其中对表型的贡献率达到14.06%。【本研究切入点】尽管玉米籽粒容重相关性状的QTL定位研究较多,但相关基因的精细定位和克隆尚未见报道。【拟解决的关键问题】本研究对育种选系过程中发现的导致玉米籽粒容重变化的淀粉粒密度突变体及其与常规自交系B73组配的F2和BC1分离群体进行细胞学和遗传学分析,通过标记开发和交换单株筛选实现突变基因的精细定位,为相应基因的克隆和功能验证奠定基础。
1 材料与方法
1.1 试验材料与作图群体
所用材料为育种选系过程中发现的淀粉粒密度变化的自然突变体和常规自交系B73,2014年夏在河南省郑州市河南农业大学科教园区组配了B73×的杂交组合,同年冬季在海南省三亚市河南农业大学南繁基地种植淀粉粒密度自然突变体和F1种子,通过自交和回交获得了F2果穗(15穗)和BC1代果穗(300穗)。2015年春在河南省郑州市河南农业大学科教园区种植了来自同一F2果穗的所有420个籽粒和来自同一个BC1果穗上的所有475个籽粒,并对其相应的F2单株和BC1单株进行自交。2015年夏在河南省郑州市河南农业大学科教园区种植2 800个在两侧标记发生交换的BC1正常籽粒,并通过自交进一步验证是否为交换单株。2016年夏在河南省原阳县河南农业大学科教园区种植了突变体和4个来自玉米突变体库(http://maizecoop.cropsci.uiuc.edu/)的突变体,用于二者的等位性测验(表1)。

表1 su2突变体来源
⊕代表自交
⊕means selfing
1.2 突变体籽粒的细胞学观察
为明确淀粉粒密度突变体籽粒内部性质的变化,利用日立S-3400扫描电镜对授粉后18、21、24、27、30和45 d的野生型和突变体籽粒胚乳中的淀粉粒形态进行观察;利用近红外光谱分析仪对成熟籽粒的粗淀粉和粗蛋白含量进行测定。
1.3 淀粉粒密度基因的定位
将F2群体中的15个正常籽粒的单株DNA等摩尔数混合成显性池,15个突变籽粒的纯合单株DNA等摩尔数混合成隐性池[15]。利用均匀覆盖玉米全基因组的1 000对SSR引物(http://www.maizegdb.org)对双亲、显性池和隐性池进行标记分析,筛选差异标记。筛选到的差异标记用于分析已知基因型的420个F2单株,验证差异标记与目的基因是否连锁。与目的基因紧密连锁的标记用于分析38 000个BC1正常籽粒胚乳,对可能的交换单株进行自交鉴定,根据交换单株自交果穗的籽粒表型确定单株的基因型,从而确定与目的基因连锁的标记以及连锁标记与目的基因的相对位置,实现目的基因的精细定位。
采用改良的CTAB法提取[16]亲本DNA、BSA混池所用的单株DNA及关键交换单株DNA,采用碱煮法提取[17]大群体籽粒样品胚乳DNA。开发SSR标记时,参考序列为B73(https://maizegdb.org/,B73 reference sequence Version3),SSR序列查找利用SSRHunter软件,SSR所在的单拷贝序列在NCBI(https://www.ncbi.nlm.nih.gov/)网站进行blast分析,SSR引物设计采用primer premier 5.0软件。引物设计原则:PCR产物长度100—250 bp,GC含量在40%—60%,引物无错配,引物3’端无发夹结构,退火温度在55—62℃,引物长度18—24 bp。由于切胚乳提取DNA的方法易对种子造成创伤,为了提高种子的成活率,通常会用3%的H2O2(ddH2O)对种子处理15 min进行消毒,并通过育苗移栽提高其成株率。
1.4 等位性测验和候选基因确定
精细定位的候选区段内包含前人已克隆的,为明确本研究的突变基因是否是的等位基因,用突变体和4个来自玉米突变体库的不同背景的突变体进行等位性测验。通过候选基因序列分析和功能预测筛选最可能的目的基因。
2 结果
2.1 突变体表型鉴定
与野生型籽粒相比,淀粉粒密度自然突变体籽粒体积变小,粒长没有变化籽粒能够正常发育,且籽粒顶部较黄(图1)。比重增加(正常1.10 g·ml-1,突变1.25 g·ml-1),近红外分析表明突变体籽粒的粗蛋白含量降低(正常3.37%,突变2.83%),但粗淀粉含量没有显著变化(正常66.22%,突变66.68%)。进一步对其籽粒内部淀粉粒进行扫描电镜分析,发现突变体籽粒的淀粉粒变小(正常13.27 μm,突变8.52 μm),密度增加(图2)。授粉后不同时期突变体与野生型的籽粒淀粉粒观察结果表明,授粉后18 d开始,突变体籽粒出现不规则形状的淀粉粒,淀粉粒密度随着发育进程不断增加(图3)。
2.2 突变体的遗传分析
淀粉粒密度突变体和B73的杂交F1果穗、F2果穗及分离群体和BC1代果穗的自交群体的表型分析结果显示,F1果穗籽粒正常且无分离,F2果穗中正常籽粒和突变籽粒符合3﹕1(1.784<χ20.05)的分离比;420个F2单株自交后,100个单株的自交果穗与野生型相同,213个单株的自交果穗出现正常籽粒与突变籽粒3﹕1(1.784<χ20.05)的分离比,107个单株的自交果穗与突变型相同,表现1﹕2﹕1的野生型穗:分离穗:突变型穗的分离比(0.162<χ20.05);BC1代果穗中正常籽粒和突变籽粒符合1﹕1(0.162<χ20.05)的分离比;475个BC1单株自交后,249个单株的自交果穗出现正常籽粒与容重突变籽粒3﹕1(0.689<χ20.05)的分离比,226个单株的自交果穗与突变体表型一致,符合1﹕1(0.557<χ20.05)的分离穗:突变型穗的分离比(表2和表3)。遗传分析结果表明籽粒淀粉粒密度突变体的突变是由1对隐性基因控制的,将该基因命名为。

a:突变型果穗;b:B73果穗;c:F1果穗;d:BC1分离穗;e:突变籽粒和正常籽粒

图2 突变型(a)和野生型(b)籽粒淀粉粒

a、c、e、g、i:授粉后18、21、24、27和30 d野生型籽粒的淀粉粒;b、d、f、h、j:授粉后18、21、24、27和30 d突变型籽粒的淀粉粒
a, c, e, g, i: Starch granules of wild grain phenotype in 18, 21, 24, 27, 30 DAP; b, d, f, h, j: Starch granules of mutant grain phenotype in 18, 21, 24, 27, 30DAP
图3 授粉后不同天数AA和aa淀粉粒的变化
Fig. 3 The change of starch granules for AA and aa in different days after pollination (DAP)

表2 (Mrd×B73)F2和BC1群体分离情况
χ20.05=3.84
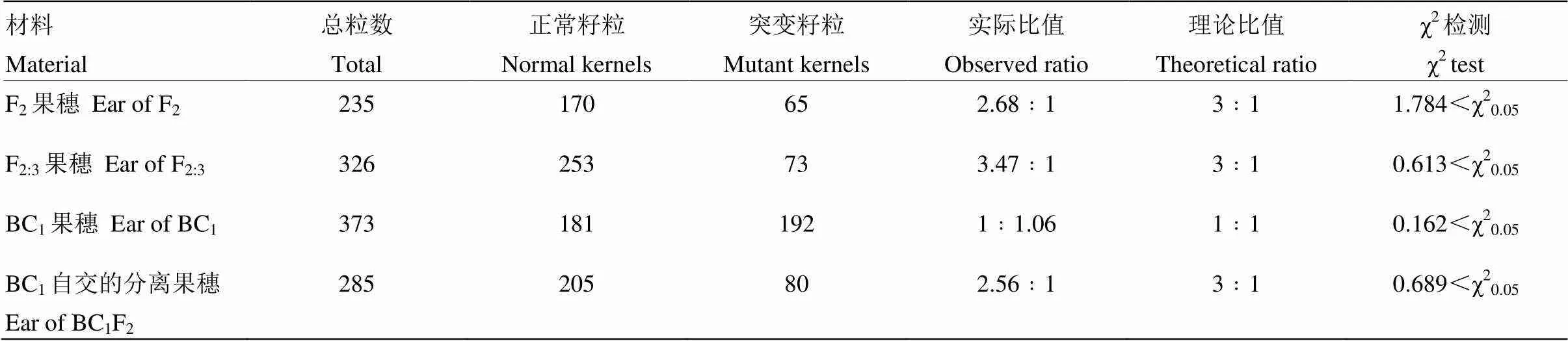
表3 (Mrd×B73)F2和BC1代果穗和群体中单株果穗的表型分析
χ20.05=3.84
2.3 籽粒淀粉粒密度突变基因的精细定位
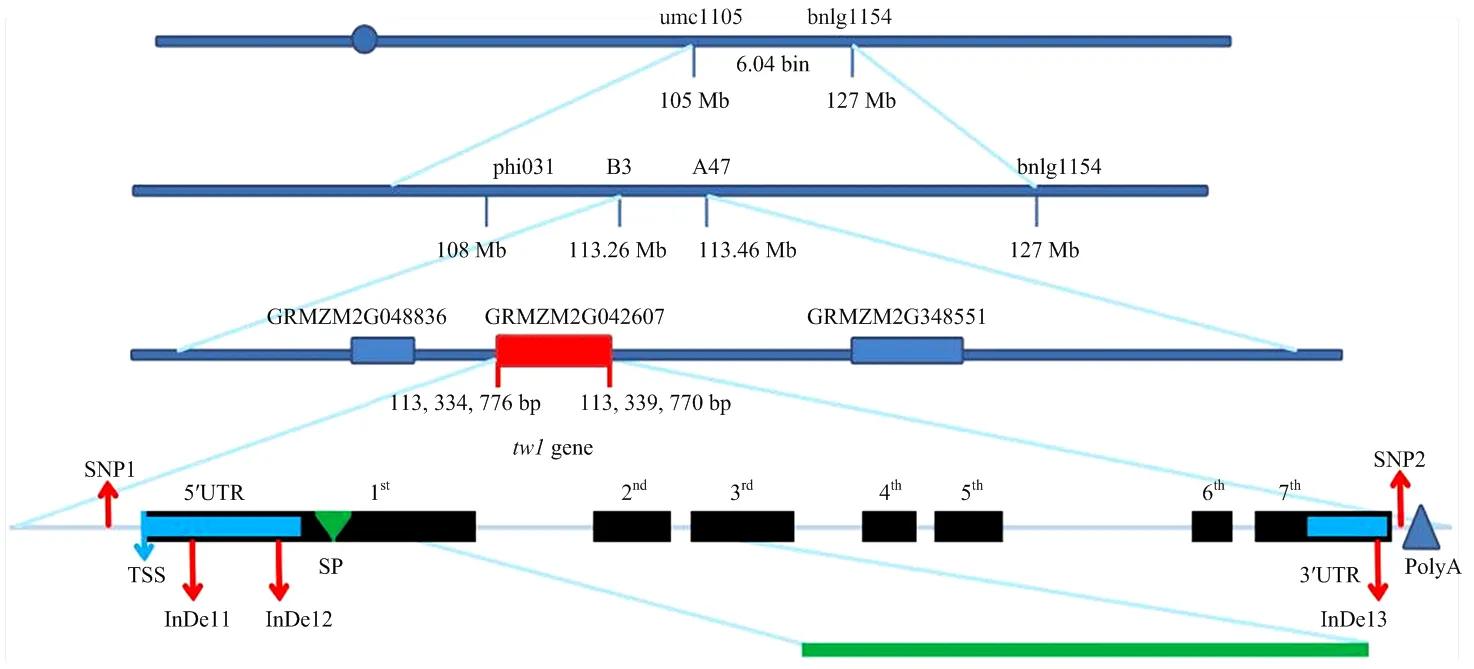
利用BSA方法在1 000对SSR标记中筛选到43对差异标记,其中位于bin6.04区的7对SSR标记利用包含50个单株的小群体验证后是与目的基因紧密连锁的。利用与目的基因紧密连锁的SSR标记对420个F2单株DNA进行标记分析,根据F2单株自交果穗的表现型确定交换单株,初步将目的基因定位于SSR标记umc1105和bnlg1154之间,物理距离22 Mb。在SSR标记umc1105和bnlg1154之间开发了349对SSR标记,并利用靠近目的基因左右两端的两对引物对BC1果穗中的3.8万个正常籽粒胚乳DNA进行标记分析,对筛选到的交换单株在田间自交进行验证其表型,同时对交换单株的DNA再次进行标记分析验证。通过多步交换单株筛选和标记分析,将目的基因定位于新开发的SSR标记B3(Chr.6:113 266 416)和A47(Chr.6:113 466 703)之间,物理距离200 kb(表4)。B73基因组显示该物理区间内有3个蛋白编码基因,GRMZ M2G048836、GRMZM2G042607和GRMZM2G348551()(图4)。生物信息学分析结果表明,GRMZ M2G048836编码叶绿素中ATP依赖的锌蛋白酶;GRMZM2G042607编码β-1,3半乳糖基转移酶;GRMZM2G348551是,编码可溶性淀粉合成酶,参与淀粉和蔗糖的代谢(表5)。

表4 多态性标记信息
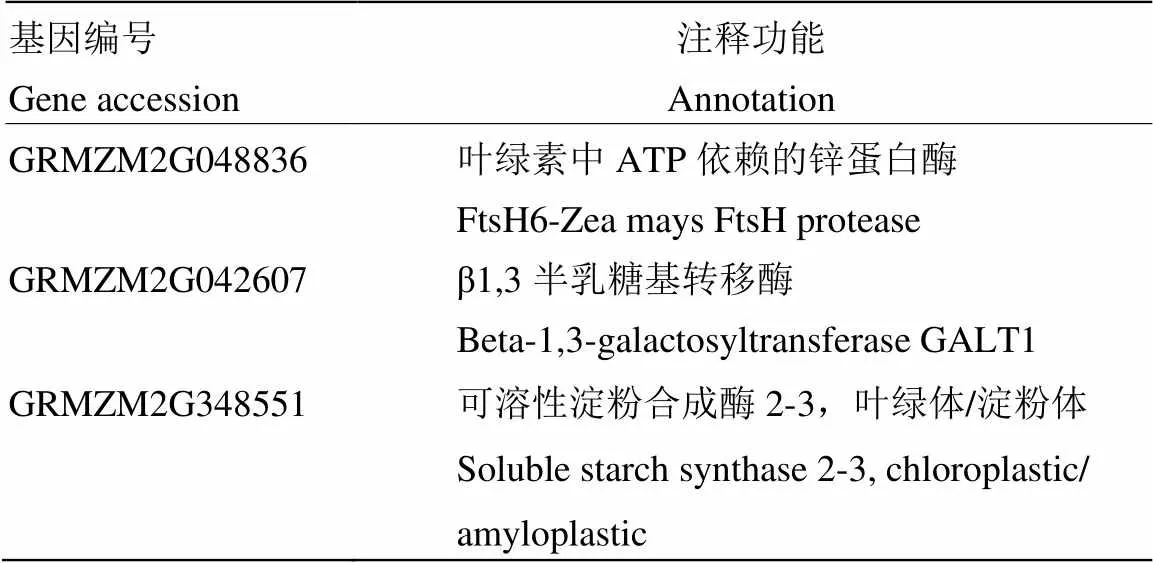
表5 候选基因预测
2.4 候选基因确定
候选区段内的4个不同遗传背景的突变体与的等位性测验结果表明,杂交当代果穗籽粒正常且无分离,说明突变体不是的等位突变引起的(图5)。生物信息学分析显示GRMZM2G042607基因162—359个氨基酸位置是其对碳水化合物的识别结构域,具有碳水化合物结合能力,在种子中对碳水化合物的储藏起沉积作用(图4),是本研究突变的首选目的基因。对GRMZM2G042607基因的测序结果显示目的基因的调控序列在突变体和野生型之间有5个多态性位点,启动子区的SNP1(A/G),5′UTR的InDel1(7T/8T)和InDel2(-------/GTGAAGA),3′UTR的InDel3(8T/9T),Poly A尾巴前的SNP2(G/T)。为验证GRMZM2G042607基因的效应,基于540份自然群体胚乳细胞和淀粉粒大小的分析结果,对相应淀粉粒大小的2个极端类型的自交系的GRMZM2G042607基因进行测序,结果表明,这5个多态位点都不特异,因此在下一步的试验中需要进一步分析其启动子序列。
3 讨论
3.1 淀粉粒大小影响籽粒容重
容重作为重要的玉米籽粒商品品质衡量指标,在品种选育过程中受到育种工作者的重视。玉米籽粒容重与籽粒硬度、淀粉含量、淀粉颗粒大小等因素密切相关。玉米籽粒内含物中淀粉含量占75%以上,淀粉含量与籽粒容重显著正相关[18-20]。淀粉粒是淀粉的主要存在形式,不同粒度的淀粉粒对籽粒容重的贡献不同,籽粒中2—10 μm的淀粉粒体积百分比与容重正相关,10—20 μm的淀粉粒体积百分比与容重负相关[21]。此外,小淀粉粒的多少决定了淀粉粒的总表面和结合的蛋白量,影响胚乳的硬度,而籽粒硬度又与籽粒容重极显著正相关[22-24]。可见,籽粒中淀粉粒变小,密度增加,结合的蛋白增多,籽粒容重也会相应增加。本研究中的突变体籽粒的淀粉粒变小(8.52 μm),密度增加,淀粉粒之间的间隙变小,容重变大,而且该突变体的总淀粉含量并没有显著变化,因而可以在不改变淀粉含量的前提下提高玉米商品品质。
3.2 候选基因的确定
大量的研究表明,当籽粒中碳水化合物的合成受到影响时,玉米籽粒中淀粉的形态和含量会发生变化,从而导致容重及品质发生改变[18-20]。编码一种异淀粉酶,该基因突变在乳熟期阻止糖分向淀粉转化,籽粒中的还原糖和蔗糖的含量显著增高,导致突变体籽粒胚乳变小,干重降低,容重变小[25-26]。编码水溶性淀粉合成酶,导致突变体胚乳中碳水化合物的性质发生改变,在籽粒发育过程中糖类浓度高,淀粉含量低[27]。()编码内在包膜小蛋白BT1[28],负责把糖元从细胞质运输到淀粉体内作为糖基供体用于淀粉的合成,在乳熟期,的突变导致籽粒还原糖和蔗糖含量高而水溶性多糖和淀粉含量低[29]。本研究中,(GRMZM2G042607)基因编码的β-1,3半乳糖基转移酶具有碳水化合物结合能力,在种子中对碳水化合物的储藏和转运起沉积作用。前人研究表明碳水化合物结合模块中的淀粉结合域数目增加,对淀粉的亲和力也相应增加,但淀粉粒变小而淀粉特性不受影响[30]。该基因可能是导致突变体淀粉粒变小,密度增加,容重变大的主要原因。

图4 tw1的精细定位
a:Mrd穗子t1、t1*su2-1、T2*su2-1、su2-1、su2-1⊕和su2-1⊕;b:Mrd穗子T2、T2*su2-2、T2*su2-2、su2-2、su2-2⊕和su2-2⊕;c:Mrd穗子T3、T3*su2-3、su2-3和su2-3⊕;d:Mrd穗子T4、T4*su2-4、su2-4和su2-4⊕
糖基化对于真核生物中蛋白质的转录修饰是十分重要的,是植物细胞维持代谢平衡的重要机制之一[31]。在水稻中,类黄酮糖基化的改变,会造成可溶性糖分的增加以及淀粉合成的减少,导致水稻结实率下降[32]。N聚糖是植物糖基化中需要的重要的低糖,水稻α-1,3-岩藻糖转移酶的缺乏造成藻糖无法参与N-糖基合成,影响水稻的分蘖角、节间和穗节长度,增加形状不规则的白垩籽粒数[33]。昆虫细胞中,β-1,3半乳糖基转移酶可以将β-1,3半乳糖转移到N聚糖中,随后参与糖基化[34]。人类中,β-1,3半乳糖基转移酶通常与细胞增殖有关,可能会导致恶性肿瘤的形成[35-36]。植物中,β-1,3半乳糖转移酶在N聚糖的延伸中起作用,能够转移β-1,3半乳糖,并在α-1,4-岩藻糖参与下形成三糖复合体Lewis a结构[37-38],参与糖基化。本研究中的目的基因编码了一个β-1,3半乳糖基转移酶GALT1。目前还没有β-1,3半乳糖基转移酶和相应糖基化对于玉米籽粒发育相关的报道,但是根据前人的研究,本文中GRMZM2G042607基因编码的β-1,3半乳糖基转移酶GALT1可能会影响N聚糖的合成,有待进一步研究。
4 结论
利用籽粒淀粉粒密度突变体定位到一个淀粉粒密度基因,该基因的候选基因是编码β-1,3半乳糖基转移酶的GRMZM2G042607。
[1] Rush I, Weichenthal B, Van P B. Feeding value of light-test weight corn for growing and finishing steers., 1996, 489: 54-55.
[2] Kirleis A W, Stroshine R L. Effects of hardness and drying air temperature on breakage susceptibility and dry-milling characteristics of yellow dent corn., 1991, 67(6): 523-528.
[3] Fox S R, Johnson L A, Crjr H, Dorseyredding C, Bailey T B. Relations of grain proximate composition and physical properties to wet-milling characteristics of maize., 1992, 69(2): 191-197.
[4] 郭淑春, 吴月红, 钱丽燕, 李学军. 用容重评定玉米质量方法的研究. 粮食储藏, 1996(5): 33-44.
GUO S C, WU Y H, QIAN L Y, LI X J. The study of the method to assess quality of corn with test weight., 1996(5): 33-44. (in Chinese)
[5] Gravois K A. Genetic effects determining rice grain weight and grain density., 1992, 64(3): 161-165.
[6] 许理文, 段民孝, 田红丽, 宋伟, 王凤格, 赵久然. 基于SNP标记的玉米容重QTL分析. 玉米科学, 2015, 23(5): 21-25.
XU L W, DUAN M X,TIAN H L, SONG W, WANG F G, ZHAO J R. QTL identification for test weight based on SNP mapping in maize., 2015, 23(5): 21-25. (in Chinese)
[7] 高荣岐, 董树亭, 胡昌浩, 王群瑛. 夏玉米籽粒发育过程中淀粉积累与粒重的关系. 山东农业大学学报(自然科学版), 1993(1): 42-48.
GAO R Q, DONG S T, HU C H, WANG Q Y. Relationship between starch accumulation and grain weight in summer maize kernel development., 1993(1): 42-48. (in Chinese)
[8] Rumbaugh M D. Test weight and maturity of corn., 1959(5): 307.
[9] Yamazaki W T, Briggle L W, Petersen H D. Effect of plant density upon soft wheat quality characteristics., 1969, 9(1): 35-37.
[10] Ding J Q, Jin-Liang M A, Zhang C R, Dong H F, XI Z Y, Xia Z L. QTL mapping for test weight by using F2:3, population in maize., 2011, 90(1): 75-80.
[11] Beavis W D, Smith O S, Grant D, FINCHER R. Identification of quantitative trait loci using a small sample of topcrossed and F4progeny from maize., 1994, 34(4): 882-896.
[12] Ajnone-marsan P, Monfredini G, Ludwig W F, Melchinger A E, Franceschini P, Pabnotto G, Motto M. In an elite cross of maize a major quantitative trait locus controls one-fourth of the genetic variation for grain yield..1995, 90(3/4): 415-424.
[13] Peng B, Li Y, Wang Y, Liu C, Liu Z, Tan W, Zhang Y, Wang D, Shi Y, Sun B, Song Y, Wang T, Li Y. QTL analysis for yield components and kernel-related traits in maize across multi-environments.2011, 122(7): 1305-1320.
[14] 白光红, 杨小红, 李林,任元, 高玉峰, 章建新. 玉米子粒体积和比重的QTL分析. 玉米科学, 2010, 18(5): 19-22.
BAI G H, YANG X H, LI L, REN Y, GAO Y F, ZHANG J X. QTL mapping for kernel volume and specific gravity with molecular markers in maize., 2010, 18(5): 19-22. (in Chinese)
[15] Michelmore R W, Paran I, Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations., 1991, 88(21): 9828-9932.
[16] Zhang X, Wang L. Modified CTAB method for extracting genomic DNA from wheat leaf., 2013, 14(7): 946-949.
[17] 吴向远, 丁冬, 宋桂良, 付志远. 玉米基因组DNA快速提取方法. 河南农业大学学报, 2012(1): 7-10.
WU X Y, DING D, SONG G L, FU Z Y. Rapid methods of genomic DNA extraction from maize., 2012(1): 7-10. (in Chinese)
[18] 徐富贤, 郑家奎, 朱永川, 王贵雄. 川东南高温伏旱区杂交中稻品种库源结构对稻米整精米率与垩白粒率的影响. 作物学报, 2004, 30(5): 432-437.
XU F X, ZHENG J K, ZHU Y C, WANG G X. Effect of ratio source to sink on percentage of head milled rice and chalky rice of combinations of mid-season hybrid rice in the south-east districts of sichuan province under high temperature and summer drought., 2004, 30(5): 432-437. (in Chinese)
[19] 张丽, 董树亭, 刘存辉,王空军, 张吉旺, 刘鹏. 玉米籽粒容重与产量和品质的相关分析. 中国农业科学, 2007, 40(2): 405-411.
ZHANG L, DONG S T, LIU C H, WANG K J, ZHAGN J W, LIU P. Correlation analysis on maize test weight, yield and quality., 2007, 40(2): 405-411. (in Chinese)
[20] Ellis R P, Cochrane M P, Dale M F B, Duffus C M, Lynn A, Morrison L M, Pretice R D M, Swanston J S, Tiller S A. Starch production and industrial use., 2010, 77(3): 289-311.
[21] 张丽, 张吉旺, 刘鹏, 董树亭. 不同淀粉含量玉米籽粒淀粉粒度的分布特性. 中国农业科学, 2011, 44(8): 1596-1602.
ZHANG L, ZHANG J W, LIU P, DONG S T. Starch granule size distribution in grains of maize with different starch contents., 2011, 44(8): 1596-1602. (in Chinese)
[22] Lee K M, Herrman T J, Lingenfelser J, Jackson D S. Classification and prediction of maize hardness-associated properties using multivariate statistical analyses., 2005, 41(1): 85-93.
[23] Chiremba C, Rooney L W, Taylor J R N. Relationships between simple grain quality parameters for the estimation of sorghum and maize hardness in commercial hybrid cultivars., 2011, 88(6): 570-575.
[24] Dorsey-Redding C, Hurburgh C R, Johnson L A, Fox S R. Relationships among maize quality factors., 1991, 68: 602-605.
[25] Myers A M, James M G. Characterization of SU1 isoamylase, a determinant of storage starch structure in maize., 1998, 117(2): 425-435.
[26] Burton R A, Jenner H, Carrangis L. Starch granule initiation and growth are altered in barley mutants that lack isoamylase activity., 2002, 31(1): 97-112.
[27] Davis J H, Kramer H H, Whistler R L. Expression of the gene du in the endosperm of maize., 1962(5): 232-235.
[28] Sullivan T D, Kaneko Y. The maize brittle 1 gene encodes amyloplast membrane polypeptides., 1995, 196(3): 477-484.
[29] Shannon J C, Pien F M, Cao H, Liu K C. Brittle-1, an Adenylate translocator, facilitates transfer of extraplastidial synthesized ADP-Glucose into amyloplasts of maize endosperms., 1998, 117(4): 1235-1252.
[30] Ji Q, Oomen R J F J, Vincken J P, Bolam D N, Gilbert H J, Suurs L C J M, Visser R G F. Reduction of starch granule size by expression of an engineered tandem starch-binding domain in potato plants., 2004, 2: 251-260.
[31] Weis M, Lim E K, Bruce N C, Bowles D J. Engineering and kinetic characterisation of two glucosyltransferases from., 2008, 90(5): 830-834.
[32] Zhan X, Shen Q, Wang X, Hong Y. The sulfoquinovosyltransferase- like enzyme SQD2.2 is involved in flavonoid glycosylation, regulating sugar metabolism and seed setting in rice., 2017, 7(1): 4685.
[33] Harmoko R, Yoo J Y, Ko K S, Ramasamy N K Hwang B Y, Lee, E J, Kim H S, Lee K J, Oh D B, Kim D Y, Lee S, Li Y, Lee S Y, Lee K O. N‐glycan containing a core α1,3‐fucose residue is required for basipetal auxin transport and gravitropic response in rice ()., 2016, 212(1): 108-122.
[34] Strasser R, Bondili J S, Vavra U, Schoberer J, Svoboda B, Glössl J, Léonard R, Stadlmann J, Altmann F, Steinkellner H, Mach L. A unique β1,3-galactosyltransferase is indispensable for the biosynthesis of N-glycans containing lewis a structures in., 2007, 19(7): 2278-2292.
[35] Ono M, Handa K, Sonnino S, Withers D A, Nagai H, Hakomori S. GM3 ganglioside inhibits CD9-facilitated haptotactic cell motility: coexpression of GM3 and CD9 is essential in the downregulation of tumor cell motility and malignancy., 2001, 40(21): 6414-6421.
[36] Furukawa K, Ohkawa Y, Yamauchi Y, Hamamura K, Ohmi Y, Furukawa K. Fine tuning of cell signals by glycosylation., 2012, 151(6): 573-578.
[37] Fitchette L A C, Gomord V, Cabanes M, Michalski J, Macary M S, Foucher B, Cavelier B, Hawes C, Lerouge P, Faye L. N‐glycans harboring the Lewis a epitope are expressed at the surface of plant cells., 1997, 12(6): 1411-1417.
[38] Melo N S, Conradt H S, Fevereiro P S, Costa J, Nimtz M. Identification of the human Lewis(a) carbohydrate motif in a secretory peroxidase from a plant cell suspension culture (L.)., 1997, 415(2): 186-191.
(责任编辑 李莉)
Fine mapping of Grain test weight gene
SUN Canran, ZHANG Xuehai, MA Zhihui, GUO Zhanyong, Tang Jihua, FU Zhiyuan
(College of Agronomy, Henan Agricultural University, Zhengzhou 450002)
【】Starch grain density affects maize kernel test weight. In this study, we use a mutantof maize to identify and fine map the gene controlling starch grain density, which are helpful for the cloning and function verification of the related grain test weight gene.【】Theis a starch grain density mutant which was identified during maize breeding practices. Scanning electron microscopy and near infrared spectroscopy (NIR) analyzer were used to observe changes of chemical composition in thekernels. The segregation population F2and BC1were derived from the cross ofand B73, which were planted in Zhengzhou and Yuanyang, Henan Province and Sanya, Hainan Province, and used for genetic analysis. BSA (Bulked Segregation Analysis) was used to identify linkage markers selected from 1 000 pairs of SSR primers from maizeGDB (http://www.maizegdb.org) of target gene. The BC1segregation population of 38 thousand individuals were used to fine map the target gene. The candidate genes were sequenced and functional predicted by bioinformatics. Allelism test was performed forand. 【】Compared with the normal seed, thehad smaller grain size and no change in grain length and increased specific gravity.The crude protein content in mutants decreased, the content of crude starch did not change significantly, but thehas irregular shape and smaller starch grain, increased density, and increased grain test weight. Observations of the starch grain structure inside the kernel in different days after pollination showed that the density of the mutant kernel starch kernel increased with the progress of development and was always higher than that of the normal kernel. The genetic analysis of F2and BC1population showed that grain test weight mutation was controlled by a single recessive genewhich was firstly located on chromosome 6 between SSR marker umc1105 and bnlg1154. After screening and analysis the recombinant from the BC1segregation population, the gene was located between B3 and A47. There are three protein-coding genes in the 0.2 Mb candidate physical interval. Allelism test excludedgene and sequence analysis of the other two candidate genes verified that GRMZM2G042607 should be the primary candidate gene of, which encodes protein with a carbohydrate recognition domain and deposits deposition of carbohydrates in the seed. 【】Thegene is fine mapped and find the candidate gene, GRMZM2G042607,encodes beta-1,3 galactosyltransferase.
maize; test weight; starch grain density;; fine mapping
2017-11-03;
2018-01-30
国家自然科学基金重大研究计划集成项目(91735306)
孙粲然,E-mail:suncanran123@163.com。
付志远,E-mail:fuzhiyuan2004@163.com

