干旱胁迫下复苏植物牛耳草基因组DNA甲基化分析
刘 杰, 张潇雅, 邓 馨
(1.潍坊科技学院贾思勰农学院设施园艺实验室,山东 寿光 262700;2.中国科学院植物研究所北方资源植物重点实验室,北京 100093)
随着全球气候变化,大范围持续性严重干旱已成为农业生产的最严重威胁.21世纪以来,重大干旱在我国主要粮食产区频繁发生,对尚在营养生长阶段的幼苗造成严重伤害,导致其光合受抑、生长停滞、干枯甚至死亡,产量严重降低甚至绝收,造成了巨大的经济损失,对农业和生态环境造成威胁.与大多数植物不同,一些植物因为在特殊生境中长期进化获得了特有的抵御某些逆境的能力,例如全球仅有的大约200种“复苏被子植物”,它们的营养组织可以长期忍耐干旱脱水,且在遇水后仍可迅速恢复正常生长[1].苦苣苔科旋蒴苣苔属植物牛耳草(Boeahygrometrica),即是一种广泛分布于东南亚的复苏被子植物,植株耐干旱脱水,甚至其离体叶片或叶片的一部分均耐旱,并在遇水后复苏,几天内即可恢复正常生活状态.研究已证实这类植物中可能蕴藏着一些特殊的逆境保护与调控机制,因其本身属于种子植物,与农作物和经济作物结构和发育过程甚至分子调控机制相同,故此研究复苏植物的这些特殊机制可为提高作物对水分胁迫的抗性提供新思路和新技术.
近年来,植物表观遗传学成为研究热点,研究表明表观遗传学在植物生长发育过程中起着极其重要的作用,主要是通过DNA甲基化、蛋白质共价修饰、非编码RNA的调控、染色质重塑和基因印记等机制调控植物的生长发育.而DNA甲基化是表观遗传学中发现最早、研究最深入的内容之一,它不仅参与植物个体发育和系统发育的调控, 而且还是基因组防御、亲本印迹、副突变、转基因沉默等表观遗传现象的主要机制[2,3].植物体内DNA甲基化动态也受到植物生理生态、发育阶段及环境刺激等多种因素的影响[2].甲基化状态的改变可启动植物体内与逆境相关基因的表达以及某些转座子的转座,从而提高植物对胁迫的抗性.DNA甲基化参与植物适应干旱基因表达的调控已在模式植物和作物中被证实[4-6],但DNA甲基化是否参与调控复苏植物干旱复苏过程还未见研究,因此,本研究利用MSAP技术分析牛耳草干旱复苏过程中的DNA甲基化水平和状态的变化及规律,为深入探讨DNA甲基化在牛耳草干旱复苏应答过程中的作用提供依据.
1 材料与方法
1.1 材料
复苏植物旋蒴苣苔(Boeahygrometrica),俗名牛耳草,种子采自北京植物园樱桃沟.取种子进行组织培养,待植株生长到一定阶段移至土中培养,培养室温度为25 ℃±2 ℃,相对湿度为50%,光照周期为16 h光照/8 h黑暗.
1.2 方法
1.2.1不同处理条件下牛耳草表型观察干旱胁迫处理:挑选生长均匀一致的牛耳草12盆(每盆1株),平均分为4组,每组3个重复.其中Ⅰ组作为对照组(F),Ⅱ组干旱处理5 d(SD5),Ⅲ组干旱处理14 d(SD14),Ⅳ组干旱处理14 d后复水3 d(A),每组在干旱处理前1 d均充分浇足水.处理过程中时刻观察牛耳草的生长状态与表型差异并拍照.
1.2.2基因组DNA提取从各处理组的3株牛耳草上取叶片,混合后,迅速置于液氮中备用.采用改良的CTAB法提取基因组DNA[7].用1%的琼脂糖凝胶检测提取的牛耳草基因组DNA的质量,用NanoDrop仪(NanoDrop 2000, Thermo Scientific)检测提取的基因组DNA的浓度与纯度,剩余DNA于-20 ℃冰箱保存备用.
1.2.3甲基化敏感扩增多态性(MSAP)分析MSAP主要步骤及体系:实验采用的接头引物、预扩增引物及选择性扩增引物见表1.接头和引物序列由上海生工合成.酶切反应体系25 μL(2.5 μL 10×NEBuffer,5UMspⅠ/HpaⅡ,5UEcoRⅠ,500 ng的基因组DNA),37 ℃下酶切7 h,80 ℃下孵育30 min,使酶失活.接头连接体系28 μL(双酶切产物20 μL,EcoRⅠ接头EA1、EA2各5×10-6μmol,MspⅠ/HpaⅡ接头H/MA1、H/MA2各5×10-5μmol,T4 DNA ligase 0.8 U,10×T4 Ligation buffer 2.8 μL),16 ℃连接12 h.将连接产物稀释10倍作为预扩增反应的模板,反应体系为25 μL (10 μL 2×TaqPCR MasterMix,3 μL连接产物,5×10-6μ mol预扩增引物E0,5×10-6μmol预扩增引物H/M0),扩增程序为94 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,共30个循环;72 ℃ 10 min.预扩增产物稀释20倍用于选择性扩增模板,反应体系与预扩增反应相同,引物为表1中的选扩增引物组合(共25对引物组合).扩增程序为:94 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s(每循环降低0.7 ℃),72 ℃ 1 min,共13个循环;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min, 23个循环;72 ℃ 10 min.选择扩增产物变性后进行8%聚丙烯酰胺凝胶电泳分离,硝酸银染色后进行H(EcoRⅠ/HpaⅡ)和M(EcoRⅠ/MspⅠ)泳道条带数及带型统计分析.
1.3 RNA提取及第一链cDNA合成
用总RNA提取试剂盒(Trizol Reagent, TaKaRa)提取牛耳草叶片总RNA,并用DNaseⅠ(TaKaRa)纯化.按照M-MLV反转录酶(Promega)说明书合成第一链cDNA,作为荧光定量PCR的模板.
1.4 Real-time PCR反应
采用Eppendrof公司Realplex2 PCR仪来实时定量检测基因表达.qPCR反应体系10 μL(2×SYBR® Green Realtime PCR Master Mix 5.0 μL,引物(10 μmol·L-1)各0.4 μL,cDNA模板0.4 μL,RNase-free H2O补足体积到10 μL).反应程序为:95 ℃预变性1 min;95 ℃ 15 s,55 ℃ 15 s,72 ℃ 30 s,进行40个循环.扩增结束后绘制溶解曲线,95 ℃ 15 s,55 ℃ 15 s,从55 ℃逐渐升温至95 ℃ 20 min,95 ℃ 15 s.以18S rRNA为内参基因,qRT-PCR所用引物序列见表2.

表1 MSAP分析中使用的接头、预扩增及选择扩增引物序列Table 1 Adaptors, pre-amplification primers and selective primers in MSAP analysis
1.5 数据处理与分析
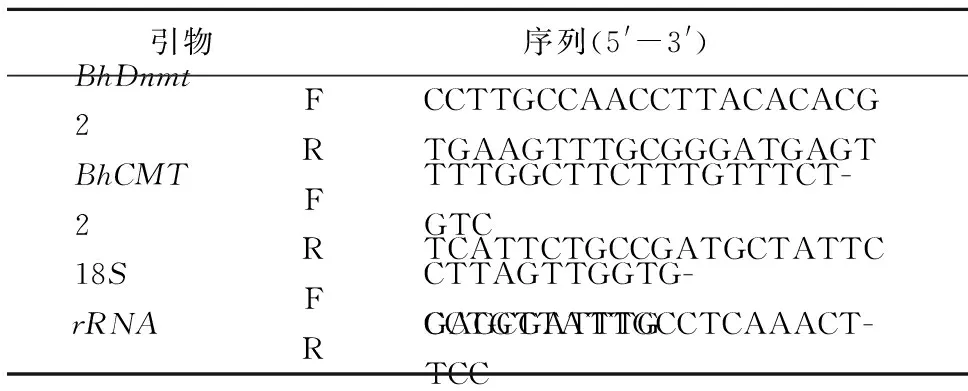
表2 qRT-PCR所用引物Table 2 Primers used in qRT-PCR
在MSAP试验中,只对条带清晰、与引物组合筛选时一致的条带进行统计.在胶板上的1个特定位点,对每个样本来说,有条带的记为“1”,没有条带的记为“0”.本次试验采用3次重复,所有数据处理及分析均使用SPSS 19.0完成,柱状图均由Origin 8.0完成.
2 试验结果
2.1 不同干旱处理条件下牛耳草表型差异
研究表明,干旱处理5 d的牛耳草叶片开始萎蔫;干旱处理14 d的牛耳草失去大部分水分,叶片变得干硬卷缩,呈现一种典型的复苏植物特有的类似脱水休眠的状态;对干旱处理14 d的牛耳草进行复水处理3 d后,牛耳草基本恢复原来的生活状态(图1).

图1 不同处理下牛耳草状态Fig.1 Status of Boea hygrometrica plants during dehydration and rehydration
2.2 不同干旱处理对牛耳草甲基化水平的影响
样品全基因组DNA经EcoRⅠ/HpaⅡ(H)和EcoRⅠ/MspⅠ(M)两组限制性内切酶组合酶切后,其产物经聚丙烯酰胺凝胶电泳检测到3种甲基化类型:Ⅰ.H有带,M无带,代表CCGG位点发生单链外部甲基化(CHG甲基化/半甲基化);Ⅱ.H无带,M有带,代表CCGG位点发生双链内部甲基化(CG甲基化/全甲基化);Ⅲ.H和M组合都有带,代表CCGG位点未甲基化.
利用5条EcoRⅠ引物和5条HpaⅡ/MspⅠ引物组合成25对引物,筛选出5对扩增效果较好且结果稳定的引物组合(表1).对不同干旱复水条件下牛耳草叶片DNA的甲基化水平进行了MSAP分析,分析100~750 bp的条带.结果表明,在所有处理样品中甲基化水平均在50.65%~60.82%范围内.由对照组F结果可知,牛耳草在新鲜状态下CCGG位点的甲基化水平约为50.65%,在干旱处理后,CCGG位点甲基化水平明显升高,并且在干旱5 d时,甲基化水平最高达到60.82%;而干旱14 d时,牛耳草甲基化水平又降低到与新鲜状态相当的水平,为50.94%;复水3 d后甲基化水平略微上升,为53.12%(表3).无论哪种状态,牛耳草叶片甲基化位点均以Ⅰ型CHG(半甲基化)位点为主,占约32.3%~40.2% (图2).

表3 不同干旱条件牛耳草叶片基因组DNA甲基化水平1)Table 3 Levels of major types of MSAP profiles in leaves of B.hygrometrica during dehydration and rehydration
1)F代表新鲜对照,SD5代表干旱5 d,SD14代表干旱14 d,A代表复水3 d.
2.3 不同干旱处理下牛耳草叶片DNA甲基化变化类型

F代表新鲜对照,SD5代表干旱5 d,SD14代表干旱14 d,A代表复水3 d.图2 不同干旱处理下牛耳草叶片DNA甲基化水平Fig.2 Levels of major types of DNA methylation calculated based on the MSAP profiles in leaves of B.hygrometrica during dehydration and rehydration
当同一个CCGG位点的MSAP条带在不同处理条件下发生变化,就说明此位点的甲基化模式发生了变化.为揭示牛耳草在干旱复苏过程中甲基化变化的程度和类型,本文将可检测到的条带变化进一步分为10个亚类(表4).A类是干旱胁迫时牛耳草基因组甲基化水平上升的条带,即发生了甲基化(methylation),包括5个亚类;B类是与对照相比,甲基化水平下降,即发生了去甲基化(demethylation),包括5个亚类.(表3,图3).
以F样品为对照,对SD5、SD14、A的甲基化变化进行统计,从表3中可以看出,SD5、SD14以及A多态性条带数分别为55、64、69条,分别占扩增总条带数的56.70%、60.37%、71.88%.其中SD5时有18条发生甲基化升高,37条发生去甲基化;SD14时有15条发生甲基化升高,49条发生去甲基化;A时有26条发生甲基化升高,43条发生去甲基化.这些甲基化变异主要集中在CG位点,且主要以去甲基化为主(图4).

表4 不同干旱处理下牛耳草CCGG位点甲基化模式的变异1)Table 4 Change of DNA methylation pattern in CCGG sites of B.hygrometrica during dehydration and rehydration
1)+:有带,-:无带;H和M分别表示EcoRⅠ/HpaⅡ和EcoRⅠ/MspⅠ酶切组合.

H代表EcoRⅠ/HpaⅡ限制性内切酶组合酶;M代表EcoRⅠ/MspⅠ限制性内切酶组合酶;F代表新鲜对照,SD5代表干旱5 d,SD14代表干旱14 d,A代表复水3 d.图3 不同干旱处理下牛耳草叶片MSAP示意图Fig.3 Examples of MSAP profiles in leaves of B.hygrometrica during dehydration and rehydration
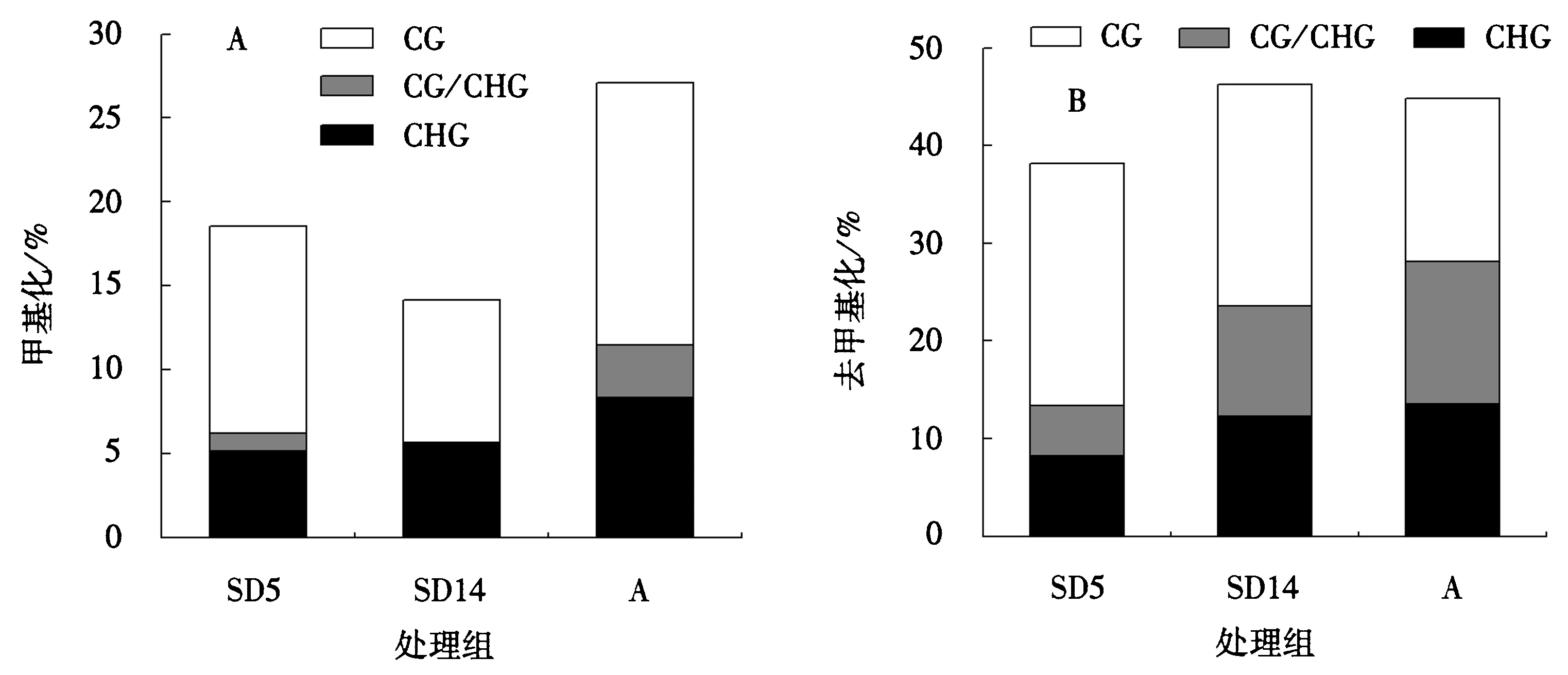
F代表新鲜对照,SD5代表干旱5 d,SD14代表干旱14 d,A代表复水3 d.图4 不同干旱处理下牛耳草叶片CCGG位点甲基化变化类型差异Fig.4 Variation in the major methylation patterns, including CG, CHG, simultaneous CG/CHG types in leaves of B.hygrometrica during dehydration and rehydration
2.4 不同干旱处理下牛耳草DNA甲基化酶相关基因表达情况
利用前文报道的牛耳草芯片数据[8](http://www.ncbi.nlm.nih.gov/geo/,NO.GSE53058)进行分析,发现2个与DNA甲基化相关的酶编码基因在干旱及复苏过程中表达发生变化.根据cDNA序列设计引物,利用实时荧光定量PCR验证上述2个基因在牛耳草干旱及复苏过程中的表达情况,BhDnmt2 (DNA methyltransferase 2)在干旱5 d时表达量急剧上升,而干旱14 d时几乎不表达,复水后表达量又明显增加;而BhCMT2 [DNA (cytosine-5) methyltransferase]在干旱过程中下降,尤其是干旱14 d时,但在复水后表达量有明显升高.这些数据与芯片数据趋势吻合(数据未列出).
3 讨论
牛耳草在遭受干旱胁迫后叶片会迅速失水并向叶柄方向卷曲,以休眠状态生存;当浇水后逆转,恢复正常的生长状态,这种表型变化对于牛耳草适应极端干旱环境具有重要作用.转录组分析发现与信号途径、保护蛋白(包括LEA蛋白和分子伴侣等)、活性氧清除、蛋白质质量控制和自噬等过程相关的大量基因在干旱复水过程中表达发生变化,上调或者下调,从而在转录水平上调控牛耳草抗旱生理反应,最终保障其在脱水状态下存活并在水分条件恢复后复苏[8].

F代表新鲜对照,SD5代表干旱5 d,SD14代表干旱14 d,A代表复水3 d.图5 不同干旱处理下牛耳草DNA甲基化酶相关基因的表达变化Fig.5 The expression changes of DNA methylation related genes in B.hygrometrica during dehydration and rehydration
DNA甲基化水平的变化是影响基因表达的重要表观调控机制.MSAP分析结果显示,牛耳草甲基化水平较高,在50.65%~60.82%之间.研究表明,甲基化水平与物种基因组复杂性密切相关,甲基化的DNA绝大多数由转座子和其他重复序列构成;重复序列多的基因组,甲基化水平程度亦较高[9].而牛耳草基因组较大,约1.691×109bp,包含了75.75%的重复序列(主要是转座子序列)[10],这些重复序列可能发生甲基化而失去活性,因此导致牛耳草的甲基化水平相对较高.
环境刺激,诸如干旱、冷、热、盐害等都会引起植物DNA甲基化状态的改变[11-14].多数胁迫情况下植物总甲基化水平会降低[14-17];但在本研究发现干旱胁迫初期牛耳草DNA甲基化水平却升高,这可能是复苏植物所特有的变化特征,推测有助于一些特定基因的表达抑制;而干旱胁迫后期,DNA甲基化总体水平降低,可能有助于一些脱水特异性基因的表达诱导;复水3 d后,尽管牛耳草表型基本恢复,但CG位点甲基化水平仍明显高于未处理组,这说明牛耳草基因组特定位点仍被DNA甲基化控制.牛耳草基因组DNA甲基化水平在其干旱复苏过程中发生明显改变,表明DNA甲基化很可能参与调控牛耳草干旱相关基因的表达;并且由半甲基化和全甲基化条带比例可推测,半甲基化调控在牛耳草抗旱过程中作用可能更明显.
DNA甲基化和去甲基化,与转录的抑制或激活密切相关;甲基化抑制基因的表达,去甲基化可能激活基因的转录[18-21].而DNA甲基化主要是通过DNA甲基化相关酶来催化完成.目前植物中已鉴定的甲基转移酶亚家族有4类:methylatransferase (MET)、chromomethyltransferse (CMT)、domain-rearranged methyltransferase (DRM)以及DNA methyltransferase homologue 2 (DNMT2),这4类酶共同维持着植物体内DNA甲基化[22].在本研究中,干旱处理过程中甲基化发生变异的位点主要以CG位点为主,且去甲基化程度大于甲基化程度,暗示表达被激活的基因可能多于抑制的基因.通过本研究qRT-PCR检测的BhDnmt2、BhCMT2的表达情况也能看出,牛耳草为了适应干旱胁迫,通过体内多个甲基化相关酶来参与牛耳草基因组DNA甲基化水平调控,进而增强其适应干旱胁迫的能力,但具体各个酶之间是如何协调还应进一步研究.
4 结论与展望
干旱复苏过程中,牛耳草DNA甲基化状态发生变化,总甲基化水平在胁迫初期明显升高,发生甲基化/去甲基化的位点占总扩增位点的50%.这些变化表明DNA甲基化可能参与了牛耳草抗旱复苏过程,是旋蒴苣苔干旱抗性反应系统的一个环节.通过MSAP检测到不同干旱胁迫处理下牛耳草基因组中的胞嘧啶甲基化存在差异,这些差异片段与牛耳草响应干旱胁迫是否有关,具体涉及到哪些基因,尚需进一步研究证实.这就需要对许多5′-CCGG-3′被甲基化修饰的序列进行分离以及基因功能的研究,以便更准确深入地理解干旱胁迫下基因组DNA的甲基化和逆境适应机制的联系.
[1] 刘杰,LIN C T,邓馨.复苏植物耐脱水机制研究进展[J].生物技术通报,2016,32(10):42-51.
[2] CHINNUSAMY V, ZHU J K. Epigenetic regulation of stress responses in plants[J]. Curr Opin Plant Biol, 2009,12(2):133-139.
[3] MIROUZE M, PASZKOWSKI J. Epigenetic contribution to stress adaptation in plants[J]. Curr Opin Plant Biol, 2011,14(3):267-274.
[4] NAYDENOV M, BAEV V, APOSTOLLOVA E, et al. High-temperature effect on genes engaged in DNA methylation and affected by DNA methylation inArbidopsis[J]. Plant Physiol Biochem, 2015,87:102-108.
[5] FOLSOM J J, BEQCY K, HAO X, et al. Rice FIE1 regulates seed under heat stress by controlling early endosperm development[J]. Plant physiol., 2014,pp.113.232413.
[6] MIN L, LI Y, HU Q, et al. Sugar and auxin signaling pathways respond to high-temperature stress during anther development as revealed by transcript profiling analysis in cotton[J]. Plant Physiol, 2014,164(3):1 293-1 308.
[7] 郭大龙,吴正景.一种简单有效的去除植物DNA中多糖等杂质的方法[J].生物技术,2008,18(5):31-33.
[8] ZHU Y, WANG B, PHILLIPS J, et al. Global transcriptone analysis reveals acclimation-primed processes in the acquisition of desiccation tolerance inBoeahygrometrica[J]. Plant Cell Physiol, 2015,56(7):1 429-1 441.
[9] ZHANG X, YAZAKI J, SUNDAREASAN A, et al. Genome-wide high-resolution mapping and functional analysis of DNA methylation inArabidopsis. Cell, 2006,126(6):1 189-1 201.
[10] XIAO L H, YANG G, ZHANG L C, et al. The resurrection genome ofBoeahygrometrica: A blueprint for survival of dehydration[J]. PNAS, 2015,112(18):5 833-5 837.
[11] CORREIA B, VALLEDOR L, MEIJON M, et al. Is the interplay between epigenetic markers related to the acclimation of cork oak plants to high temperatures?[J] PLoS ONE, 2013,8(1):e53543.
[12] SONG Y, JI D, LI S, et al. The dynamic changes of DNA methylation and histone modifications of salt responsive transcription factor genes in soybean[J]. PLoS ONE, 2012,7(7):e41274.
[13] GOHLKE J, SCHOLZ C J, KNEITZ S, et al. DNA methylation mediated control of gene expression is critical for development of crown gall tumors[J]. PLoS Genet, 2013,9(2):e1003267.
[14] CAO D, GAO X, LIU J, et al. Root-specific DNA methylation inChlorisvirgata, a natural alkaline-resistant halophyte, in response to salt and alkaline stresses[J]. Plant Molecular Biology Reporter, 2012,30(5):1 102-1 109.
[15] GAO X, CAO D, LIU J, et al. Tissue-specific and cation/anion-specific DNA methylation variations occurred inC.virgatain response to salinity stress[J]. PLoS ONE, 2013,8(11):e78426.
[16] 黄韫宇,张海军,邢燕霞,等.NaCl胁迫对黄瓜种子萌发的影响及DNA甲基化的MSAP分析[J].中国农业科学,2013,46(8):1 646-1 656.
[17] 唐晓梅,王艳,马东伟,等.干旱胁迫下高羊茅基因组甲基化分析[J].草业学报,2015,24(4):164-173.
[18] GOREN A, SIMCHEN G, FIBACH E, et al. Fine tuning of globin gene expression by DNA methylation[J]. PLoS ONE, 2006,1(1):e46.
[19] TARIQ M, PASZKOWSKI J. DNA and histone methylation in plants[J]. TRENDS in Genetics, 2004,20(6):244-251.
[20] KUMAR S, KUMARI R, SHARMA V, et al. Roles, and establishment, maintenance and erasing of the epigenetic cytosine methylation marks in plants[J]. Journal of genetics, 2013,92(3):629-666.
[21] GRATIVOL C, HEMERLY A S, FERREIRA P C G. Genetic and epigenetic regulation of stress responses in natural plant populations[J]. BBA-Gene Regul Mech, 2012,1819(2):176-185.
[22] PAVLOPOULOU A, KOSSIDA S. Plant cytosine-5 DNA methyltransferases: structure, function, and molecular evolution[J]. Genomics, 2007,90(4):530-541.

