基于EST-SSR及SRAP标记构建苦荬菜品种(系)DNA指纹图谱
班骞,谢彩云,范国华,黄琳凯,张新全*
(1.贵州省农业科学院草业研究所,贵州 贵阳 550006;2.四川农业大学动物科技学院草业科学系,四川 成都611130)
物种种质资源及其遗传多样性是遗传改良和新品种育种的基础[1],而保证品种身份真实可有效遏制我国种业市场上套牌、冒牌、派生品种等恶性现象,同时为保障农民权益提供了有力依据。随着DNA分子标记技术的快速发展,其研究领域已涉及遗传连锁图谱构建[2]、功能基因表达[3]、亲缘关系分析[4]、指纹图谱构建[5]等多个方面,目前应用DNA指纹标记鉴定植物品种[6]已成为一项快捷、准确的有效技术。
简单重复序列(simple sequence repeats, SSR),具有重复性好、共显性、多态丰富等优点[7],近年来ESTs发展迅速,已有不少研究利用EST-SSR构建芦笋[8](Asparagusofficinalis)、白菜[9](Brassicarapa)、紫花苜蓿[10](Medicagosativa)等品种DNA指纹图谱;而SRAP标记主要针对基因阅读框区域(ORFs),即对物种特异扩增内含子和启动子区域进行特异扩增,常用于种质资源评价、重要性状标记、基因定位、指纹图谱构建等研究领域[11],已有不少报道利用此标记技术在菜薹[12](Brassicacampestris)、 国兰[13](Orchidaceaecymbidium)、假俭草[14](Eremochloaophiuroides)等品种上构建DNA指纹图谱,可关于苦荬菜品种DNA指纹图谱的构建尚未见到报道。
苦荬菜(Ixerispolycephala)属一年生菊科青饲牧草,异花授粉植物,是稳定的二倍体物种。因其适口性好,草质鲜嫩常被用于家禽饲喂,在我国具有一定的市场潜力。截至目前,我国审定苦荬菜品种仅为3个,且国内市场以次充优,品种混乱,为解决目前现状,本研究首次利用EST-SSR和SRAP标记对14个苦荬菜品种(系)进行指纹图谱构建,研究结果可为苦荬菜品种快速区分、鉴定和知识产权保护的贡献依据,对重要性状基因挖掘提供参考资料,同时为逐步完善我国苦荬菜种质资源DNA指纹图谱数据库奠定基础。
1 材料与方法
1.1 试验材料
供试14个苦荬菜品种(系)来源如表1所示,CF编号材料由国家草种质资源基因库提供,其余由四川农业大学草学系牧草育种课题组、黑龙江畜牧所提供。2015年7月,种子经4 ℃低温处理,于25 ℃恒温培养箱中发芽,温室盆栽后取幼嫩叶片。
1.2 样品DNA的提取
每份材料选取30个单株混合,选取苦荬菜材料的健康幼嫩新鲜叶片,用蒸馏水冲洗干净后烘干叶片,剪碎后装入2 mL离心管内,把离心管放入液氮中浸泡5~10 min,离心管加入钢珠置于高通量组织研磨器内进行研磨,频率50 Hz,时间60 s。研磨成粉状后,使用植物基因组提取试剂盒(北京天根生化科技有限公司)提取基因组DNA。采用琼脂糖电泳检测DNA的纯度。琼脂糖电泳检测:将DNA样品和已知浓度的标准梯度DNA作为对照,在0.8%琼脂糖凝胶进行电泳,通过凝胶成像系统对比样品和对照的亮度估算样品DNA的浓度。将DNA样品稀释至10 ng·μL-1,4 ℃保存。
1.3 SSR-PCR引物筛选和扩增
所用引物参考近缘种菊苣EST-SSR引物序列信息[15],选取152对引物进行筛选,最终获得24对条带清晰且多态性好的引物。引物由南京金斯瑞生物科技有限公司合成(表2)。
EST-SSR PCR 反应体系为15 μL,包括1 μL(10 ng·μL-1)DNA,上下引物各1 μL (10 pmol·μL-1),0.3 μL (2.5 U·μL-1)Taq DNA 聚合酶,Mix混合液7.5 μL,4.1 μL ddH2O。PCR所用Mix混合液(含有10×PCR buffer、Mg2+、dNTPs),Taq酶和50 bp DNA ladder Marker均购自天根科技生化公司。PCR反应条件为:94 ℃预变性5 min;94 ℃变性1 min,56~64 ℃退火45 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。PCR产物经6%变性聚丙烯酰胺凝胶电泳,采用银染显色后拍照。

表1 供试苦荬菜品种(系)DNA指纹图谱材料信息 Table 1 Information of Ixeris polycephala varieties (lines) for DNA fingerprint
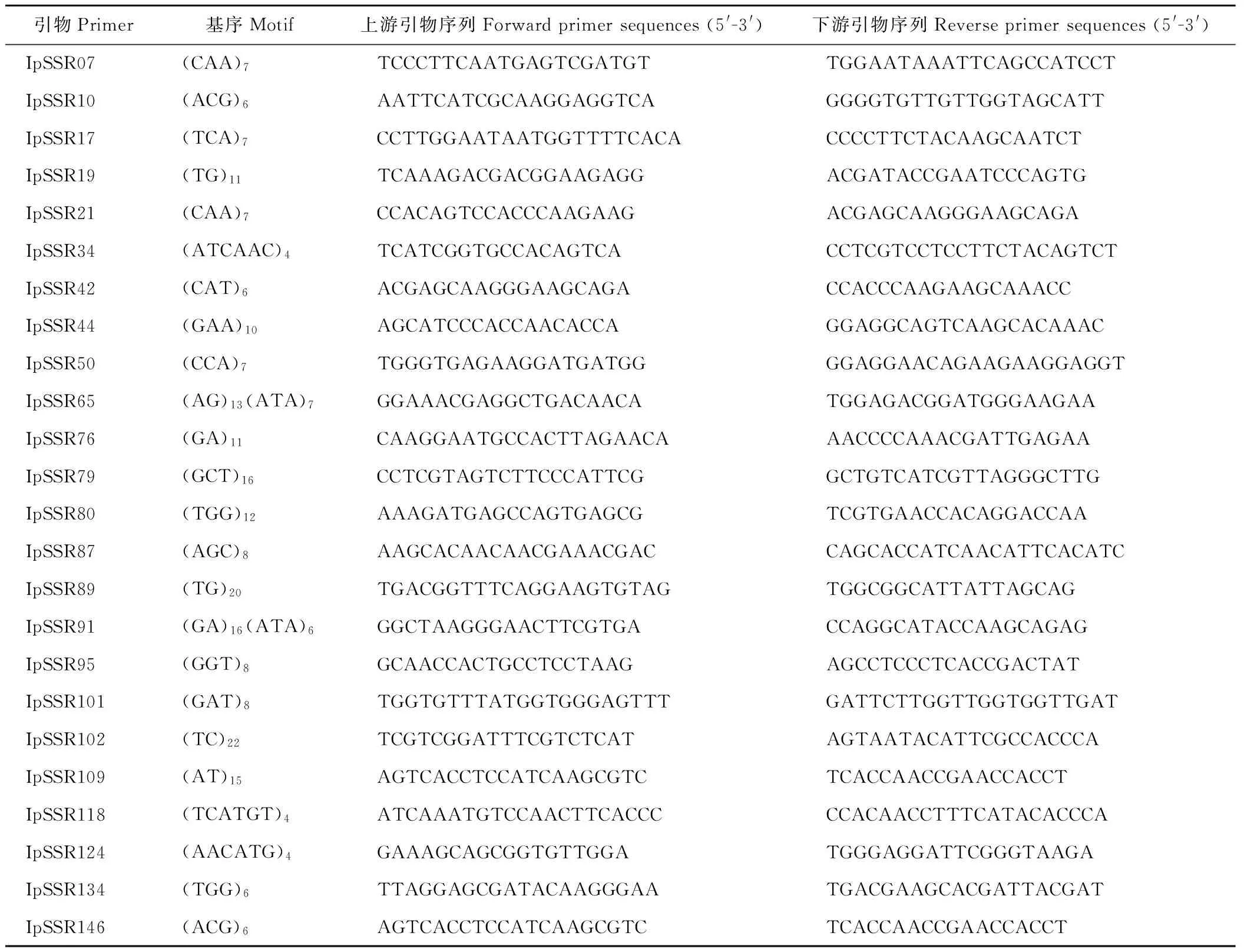
表2 苦荬菜EST-SSR标记引物序列Table 2 EST-SSR markers primer sequences for I. polycephala varieties (lines)
1.4 SRAP-PCR引物筛选和扩增
参考文献[16-17]中SRAP标记的上游引物和下游引物产生的引物组合,筛选供试材料,得到多态性好的引物进行后续扩增,SRAP引物及PCR Mix混合液(含有10×PCR buffer、Mg2+、dNTPs),Taq酶均购于天根科技生化公司。SRAP-PCR扩增反应体系总体积为15 μL,反应最终浓度:包括1 μL(10 ng·μL-1)DNA,引物各0.8 μL (10 pmol·μL-1),0.3 μL (2.5 U·μL-1)Taq DNA 聚合酶,Mix混合液7.5 μL,4.6 μL ddH2O。PCR扩增反应在Biometra PCR仪上进行,扩增程序为:94 ℃预变性5 min;然后94 ℃变性1 min,35 ℃复性1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,50 ℃复性1 min,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;4 ℃保存。PCR产物经6%变性聚丙烯酰胺凝胶电泳,点样6 μL,采用银染显色后拍照保存。缓冲体系为1×TBE,电压200 V,时间约1.5 h。
1.5 数据统计与分析
每对引物重复试验3次,依据扩增后的电泳结果,统计清晰可重复的扩增条带,在相同迁移位点上,有带记为1,无带记为0,建立由0,1组成的原始数据矩阵。据表征矩阵,利用POPGENE 1.31[18]软件计算I值和H值,并观察等位基因数(Na)和有效等位基因数(Ne)等群体遗传参数。PIC参考Nei[19]的方法计算,PIC=1-∑Pi2。其中,Pi为基因座位上第i等位基因的频率。利用Gelpro Analysis软件对凝胶电泳图像进行扩增片段分子量的计算,参考陈昌文等[20]的方法构建苦荬菜主栽品种(系)的DNA指纹编码,利用Excel对0,1矩阵进行转制,构建与指纹编码相对应的苦荬菜品种(系)的DNA指纹图谱标准模式图。
2 结果与分析
2.1 EST-SSR和SRAP标记多态性分析
筛选出24对ESR-SSR引物对部分苦荬菜品种(系)进行后续扩增(图1),由表3得知24对引物一共扩增270个条带,多态性条带223个,平均每对引物扩增出11.3个条带,9.3个多态性条带,PPB变幅为55.56%~100.00%,平均81.61%。PIC变幅为0.765~0.894,基因多样性指数变幅为0.2464~0.4288,均值0.3382,Shannon指数变幅为0.3597~0.6148,均值0.4989,表明供试苦荬菜品种(系)遗传变异较为丰富,EST-SSR引物可以适用于苦荬菜品种(系)相关遗传研究和DNA指纹图谱构建。
筛选出的32对SRAP引物组合对14个苦荬菜品种(系)进行扩增(图1),由表4可知,SRAP标记共扩增出428个条带,其中多态性条带367个,平均每个引物组合扩增出13.4个条带,11.5个多态性条带,PPB变幅在54.54%~100.00%,均值84.42%,PIC均值0.826,基因多样性指数变幅在0.2006~0.3898,均值0.2855,Shannon指数在0.3167~0.5716,均值0.4283,说明苦荬菜品种(系)存在丰富的遗传变异,SRAP标记可以用于苦荬菜遗传多样性研究和DNA指纹图谱构建。
2.2 EST-SSR和SRAP标记核心引物分析
两种标记对苦荬菜扩增出的多态性条带有所不同,且能够区分的苦荬菜品种数量也不同,24 对 EST-SSR引物中可以区分的苦荬菜品种数量为3~14个,平均7.7个(表3),SRAP引物组合能够区分的苦荬菜品种数量在2~12个,平均6.6个(表4)。EST-SSR引物IpSSR19能够扩增出9条多态性条带,引物IpSSR91能够扩增出11条多态性条带,均能一次性区分14个供试苦荬菜品种(系),而引物IpSSR118仅能扩增出5条多态性条带,可鉴定3个苦荬菜品种;SRAP引物组合Me9+Em17能够扩增出14条多态性条带,引物组合Me10+Em5能够扩增出15条多态性条带,最多可以区分12个供试苦荬菜品种(系),而引物组合Me4+Em6能够扩增出6条多态性条带,可以鉴定2个苦荬菜品种;SRAP引物组合虽没有单独引物能够一次性区分全部品种(系),但通过不同的引物有效组合,可以提高对苦荬菜品种的甄别能力,而且运用两种标记的不同引物组合,更能大幅度提高鉴别苦荬菜品种数量,这将对构建准确、高效苦荬菜品种DNA指纹图谱提供重要依据。
采用以上EST-SSR和SRAP引物对14个苦荬菜品种(系)进行分析,利用Gelpro Analysis软件对凝胶电泳图像扩增片段进行定位,并计算分子量大小,9个品种(系)在7个引物扩增条带上具有特征谱带(表5),即只用1个特征引物就能与其他品种区分开。如引物IpSSR91和引物组合Me9+Em17均在品种‘Longmu’上具有特征谱带(图1,图2),其中,品种(系)‘Lvyuan’、‘Treasure’、‘CF6042’、‘川选1号’、‘L6-2’、‘CF023568’、‘CF023569’均有一个特征引物,品种(系)‘Longmu’和‘L5-4’具有两个特征引物;此外Me9+Em17在‘L5-4’、‘川选 1 号’和‘CF023568’上同时具有特征谱带(图2)。表明EST-SSR和SRAP多态性引物能够产生较多的特征谱带,可以有效地应用于构建苦荬菜品种DNA指纹图谱。

表3 EST-SSR引物扩增结果及多态性信息Table 3 Results and the polymorphism information of EST-SSR primers
TNB:Total number of amplified bands;NPB:The number of polymorphic bands;PPB:The percentage of polymorphic bands;PIC:Polymorphic information content;I:Shannon’s information index;H:Nei’s gene diversity;DV:Distinguished varieties;下同The same below.
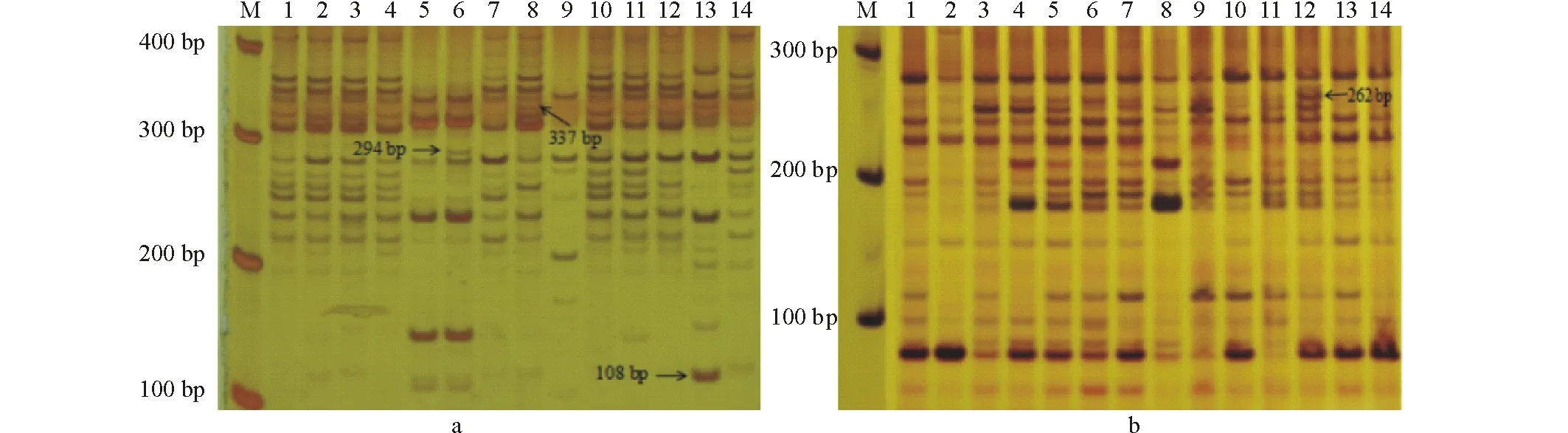
图1 EST-SSR对部分苦荬菜品种(系)扩增Fig.1 EST-SSR amplified of I. polycephala varieties (lines) a:引物IpSSR91扩增 “Longmu”、“川选1号”的特异带Special band of Longmu and Chuanxuan No.1 varieties amplified with primer IpSSR91; b:引物IpSSR102扩增“Treasure”的特异带Special band of Treasure variety amplified with primer IpSSR102.

图2 引物组合Me9+Em17对苦荬菜品种(系)扩增结果Fig.2 Me9+Em17 primer amplified results of I. polycephala varieties (lines)

品种(系)Variety(Line)特征引物Specificprimer品种特征带条带大小Variety-specificbandsize(bp)品种(系)Variety(Line)特征引物Specificprimer品种特征带条带大小Variety-specificbandsize(bp)LvyuanIpSSR80172L5-4Me9+Em17844TreasureIpSSR102262Me11+Em5631CF6042IpSSR50198L6-2IpSSR80164LongmuIpSSR91294CF023568Me9+Em171220Me9+Em17321IpSSR91108川选1号ChuanxuanNo.1IpSSR91337CF023569IpSSR19155
2.3 构建苦荬菜品种(系)DNA指纹图谱
构建指纹图谱尽可能以较少数量的引物组合和高效的甄别方法为准则,最终可获得能够鉴别较多的品种数量。综合考虑24对EST-SSR引物和32对SRAP引物的扩增条带数、条带统计难易程度、PIC值和鉴定品种时效性等多种因素,最终筛选出3个较为高效的EST-SSR引物和2个SRAP引物,分别为IpSSR80、IpSSR91、IpSSR102、Me9+Em17和Me10+Em5。借鉴陈昌文等[20]对中国主要桃品种ID身份指纹图谱编码构建方法,对选择的5个引物的多态性条带进行1~9的整数赋值,根据Gelpro Analysis软件估算出每个引物多态性条带的分子量大小,从小到大依次赋值,最大赋值数为9,不同引物所选择的多态性条带不同,对于多态性条带超过9的,只考虑其中更加高效的9条条带对其编码赋值(表6)。对最终确定的5个引物选择了40个多肽条带供于赋值,其中8个特征谱带可直接用于鉴定当中的某一品种。

表6 特征谱带选择和条带赋值标准 Table 6 Specific bands selection and encoded standard for I. polycephala varieties (lines) (bp)
*引物特征谱带Primer-specific band.
根据表6引物多态性条带的赋值标准对参试苦荬菜品种(系)进行DNA指纹编码(表7),为真实高效地鉴别苦荬菜品种(系),构建的DNA指纹图谱编码数应尽可能少,避免指纹数据过于繁杂,那么每对引物多态性条带的选择重复性好,甄别能力强的多态谱带,引物排列顺序按照扩增条带分子量大小排列,即IpSSR102—IpSSR80—IpSSR91—Me10+Em5—Me9+Em17,其中IpSSR102提供一个多态谱带,IpSSR80、IpSSR91、Me10+Em5、Me9+Em17均提供两个多态性条带,以‘川选1号’为例,表现在引物IpSSR102的248 bp,IpSSR80的145和280 bp,IpSSR91的260和337 bp,Me10+Em5的373和870 bp,Me9+Em17的484和677 bp出现了多态性谱带(表6,表7,图3),能够与其他品种(系)区分开。结果表明,在构建苦荬川选1号: Chuanxuan No.1

表7 苦荬菜品种(系)的指纹数据库编码 Table 7 Fingerprint code of I. polycephala varieties (lines)
菜DNA指纹图谱中,每个品种(系)最终确定指纹编码数为9个,即1~9的整数赋值,每份苦荬菜品种(系)具有特异指纹编码,便于快速直观地区分不同品种。

图3 苦荬菜新品系‘川选1号’指纹图谱标准模式图Fig.3 The DNA fingerprinting standard model pattern of I. polycephala new lines of ‘Chuanxuan No.1’
以DNA指纹数据库(表7)构建苦荬菜品种(系)指纹图谱,图4所示,横坐标表示不同苦荬菜品种(系),纵坐标表示引物扩增谱带位点,黑色条带代表在相应引物扩增位点有谱带出现,且每个苦荬菜品种(系)的指纹图谱均与指纹数据库编码信息相对应,详见表6。每个苦荬菜品种(系)均有不同扩增带,能够快速鉴别其他品种(系)。
3 讨论
EST-SSR标记是基于表达序列标签开发的新型标记,利用已有的ESTs数据库,筛选鉴别SSR,其序列来源于基因转录区[21],其保守性好,在不同物种间通用性强,除去了SSR引物开发过程中的测序、克隆步骤。目前已有不少应用EST-SSR分析不同物种遗传多样性[22],构建品种指纹图谱[23],比较作图等[24]研究。本研究应用24对EST-SSR多态性引物对收集的14份苦荬菜品种(系)一共扩增出270个清晰条带,多态性条带223个, PPB平均81.61%。PIC变幅为0.765~0.894,基因多样性指数变幅为0.2464~0.4288,Shannon指数变幅为0.3597~0.6148,说明供试来源不同的苦荬菜品种(系)在遗传背景中发生了基因水平的丰富变异,同时EST-SSR引物可以适用于苦荬菜品种(系)相关遗传研究和DNA指纹图谱构建。

图4 14份苦荬菜品种(系)指纹图谱Fig.4 Fingerprinting diagram of 14 I. polycephala varieties (lines)
SRAP标记可以通过其独特的引物设计,对植物基因组外显子与内含子之间的序列进行扩增,不同品种(系)之间的序列差异最终导致扩增片段有所差异[25],从而产生多态性。近年来在作物上已经被广泛地应用于遗传图谱构建、多样性研究、种子真实性鉴定等多个方面。本研究从209个引物组合种筛选出32对SRAP引物,对14份苦荬菜品种(系)进行扩增,其中多态性条带367个,PPB变幅为54.54%~100.00%,PIC均值0.826,基因多样性指数变幅为0.2006~0.3898,Shannon指数为0.3167~0.5716,表明了SRAP标记在苦荬菜品种(系)鉴别能力上有着较大的应用潜力,存在丰富的遗传变异,SRAP标记可以适用于DNA指纹图谱构建,获得的基因位点很大可能是某部分相关性状的功能基因,这将为以后的功能基因组学研究及分子辅助育种提供技术支撑。
现在我国草牧业快速发展,家禽养殖业对苦荬菜的需求量日益增多,但目前国内苦荬菜品种资源相对较少,加之市场管理极其混乱,许多产品之间互相掺杂,严重限制了苦荬菜的生产利用,极大程度上地损害了消费者的权益,严重妨碍我国十三五规划的优质牧草推广应用。因此快速地鉴定苦荬菜品种资源显得相当重要,传统的植物学表型鉴定受地理环境影响和人为观测所带来的误差较大,相较于DNA水平上的变异不易受环境和人为的干扰,而开展DNA指纹标记技术日渐成为目前鉴定植物品种的主流方法,目前构建植物品种指纹图谱的主要方法有特征谱带法[26]、单引物法[27]和引物组合法[28-29]。本研究采用3个EST-SSR引物和两个SRAP引物有效组合鉴别14个苦荬菜品种(系),结果显示所有品种(系)能够完全区分,每个品种(系)有一套与指纹图谱上相对应的指纹编码,可以视作其分子水平上的ID身份证,其ID编码差异较小的品种(系),遗传背景也比较相近,反之亦然,如品种‘Prado’和‘Treasure’指纹编码分别是345342646和356243547,两个品种在遗传差异上有一定相似性,可见,构建苦荬菜品种(系)指纹数据库可为苦荬菜选育提供重要依据。
DNA指纹编码相关研究中有着不同的报道,刘红艳等[30]直接采用特征谱带0,1矩阵建立区域试验新品系指纹图谱数据库编码,结果生成的指纹编码过于繁多,不能直观地快速区分品种,王静毅等[31]采用字母代表引物顺序和数字代表扩增的特征谱带顺序号,两者相结合建立品种资源分子身份的DNA指纹图谱,导致指纹编码字符串数值过多,不能更加快捷地区分品种数量。本实验对核心引物谱带进行复制编码,使得第N对引物的扩增谱带与指纹编码数值相对应,选择谱带不超过9个扩增片段进行指纹编码赋值,与前两种统计方法相对比显得更加直观简洁,利于快速准确区分苦荬菜品种(系)。不过本研究因筛选的引物数量有限,用于鉴别品种的特征谱带有一定的材料范围限制[32],即随着品种数量的增加,特征条带很有可能在品种中出现。从理论上,指纹图谱在鉴定物种时有一定的技术优势,但其实在生产使用上还存在局限性,如成本高,鉴定可操作性有限等,今后育种上可以利用新型分子标记技术结合传统鉴定方法辅助利用指纹图谱。我国中央一号文件明确指出要加快发展草牧业,开展优质饲草料种植,国家对草牧业重视程度日渐增高,今后不断有育成的新品种扩编入国家品种资源库当中,这也就预示着苦荬菜品种指纹图谱需根据时事不断补充和更新,也就意味着在今后还需继续扩大引物筛选数量,增加核心引物,不断完善苦荬菜品种(系)指纹数据库,提高指纹图谱的准确性、实用性和兼容性,为苦荬菜品种快速鉴定、管理和知识产权保护等方面提供科学基础资料。
4 结论
本研究筛选24对ESR-SSR引物和32对SRAP引物组合用于14个国内苦荬菜品种(系)的DNA指纹图谱构建,结果表明EST-SSR和SRAP标记可以适用于苦荬菜品种鉴定及遗传分析,来源不同的苦荬菜品种(系)在基因水平上存在着的丰富变异,9个品种(系)在7个引物扩增条带上具有特征谱带,构建的苦荬菜品种(系)指纹图谱能够全部区分所有供试材料,每个苦荬菜品种(系)有一套特异的分子ID身份指纹编码,该试验研究结果可作为鉴定苦荬菜品种的科学依据,为后续苦荬菜品种(系)指纹图谱数据库的扩充奠定基础。
参考文献References:
[1] Qiang H P, Yu G H, Liu H Q,etal. Genetic diversity and population structure of Chinese and American alfalfa (MedicagoSativaL.) germplasm assessed by SSR markers. Scientia Agricultura Sinica, 2014, 47(14): 2853-2862.
强海平, 余国辉, 刘海泉, 等. 基于SSR标记的中美紫花苜蓿品种遗传多样性研究. 中国农业科学, 2014, 47(14): 2853-2862.
[2] Dracatos P M, Cogan N O, Sawbridge T I,etal. Molecular characterisation and genetic mapping of candidate genes for qualitative disease resistance in perennial ryegrass (LoliumperenneL.). BMC Plant Biology, 2008, 9(1): 62.
[3] Singh N, Dang T T M, Vergara G V,etal. Molecular marker survey and expression analyses of the rice submergence-tolerance gene SUB1A. Theoretical and Applied Genetics, 2010, 121(8): 1441-1453.
[4] Shang G F, Re F H, Sai Y,etal. Start codon targeted (SCoT) and target region amplification polymorphism (TRAP) for evaluating the genetic relationship ofDendrobiumspecies. Gene, 2015, 567(2): 182-188.
[5] Archak S. Plant DNA fingerprinting: an overview. AgBiotechNet, 2000, 2: https://www.researchgate.net/publication/228559903.
[6] Nybom H, Weising K, Rotter B. DNA fingerprinting in botany: past, present, future. Investigative Genetics, 2014, 5(1): 1-35.
[7] Chen S Y, Ma X, Zhang X Q,etal. The transferability of SSR and EST-SSR markers of different origins inElymusandRoegneriain theTriticeae(Poaceae). Acta Prataculturae Sinica, 2016, 25(2): 132-140.
陈仕勇, 马啸, 张新全, 等. 不同来源SSR和EST-SSR在披碱草属和鹅观草属物种中的通用性分析. 草业学报, 2016, 25(2): 132-140.
[8] Caruso M, Federici C T, Roose M L. EST-SSR markers forAsparagusgeneticdiversityevaluation and cultivar identification. Molecular Breeding, 2008, 21(2): 195-204.
[9] Li L, He W M, Ma L P,etal. Construction Chinese cabbage (BrassicarapaL.) core collection and its EST-SSR fingerprint database by EST-SSR molecular markers. Genomics and Applied Biology, 2009, 28(1): 76-88.
李丽, 何伟明, 马连平, 等. 用EST—SSR分子标记技术构建大白菜核心种质及其指纹图谱库. 基因组学与应用生物学, 2009, 28(1): 76-88.
[10] Yu L Q, Liu R X, Su D,etal. Genetic diversities of fall SSR and dormancy alfalfa based on EST——SSR. Chinese Journal of Grassland,2009, 31(6): 52-58.
于林清, 刘荣霞, 苏东, 等. 利用SSR和EST-SSR技术研究不同秋眠级苜蓿遗传多样性. 中国草地学报, 2009, 31(6): 52-58.
[11] Xu C, Zhao B H. The development and application of SRAP molecular markers. Life Science Instruments, 2009, 7(4): 24-27.
徐操, 赵宝华. SRAP分子标记的研究进展及其应用. 生命科学仪器, 2009, 7(4): 24-27.
[12] Mao W W, Sun J D, Chen X H. Establishment of finger-print for a hybrid ofBrassicacampestrisL.ssp.Chinensisvar.utilisby ISSR and SRAP markers. Journal of Changjiang Vegetables, 2010, (20): 18-20.
冒维维, 孙敬东, 陈学好. 菜薹杂交种ISSR和SRAP的指纹图谱构建. 长江蔬菜, 2010, (20): 18-20.
[13] Tang Y J, Cao W J, Wu K L. Genetic diversity analysis and molecular identification card construction of ChineseCymbidiumgermplasmsbased on SRAP markers. Scientia Agricultura Sinica, 2015, 48(9): 1795-1806.
唐源江, 曹雯静, 吴坤林. 基于SRAP标记的国兰种质资源遗传多样性分析及分子身份证构建. 中国农业科学, 2015, 48(9): 1795-1806.
[14] Guo H L, Guo A G, Zong J Q,etal. Identification analysis of eight centipedegrass materials by SRAP molecular markers. Acta Agrestia Sinica, 2014, 22(1): 203-207.
郭海林, 郭爱桂, 宗俊勤, 等. SRAP标记对8份假俭草材料的鉴定分析. 草地学报, 2014, 22(1): 203-207.
[15] Liang X Y. Germplasm evaluation, molecular marker development and genetic linkage map construction of chicory. Ya’an: Sichuan Agriculture University, 2013.
梁小玉. 菊苣种质资源评价、分子标记开发及遗传连锁图谱构建. 雅安: 四川农业大学, 2013.
[16] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics, 2001, 103(2/3): 455-461.
[17] Manoj M K, Sandhyarani N, Jayarama. Molecular identification and genetic relationships among coffee species (CoffeaL.) inferred from ISSR and SRAP marker analyses. Archives of Biological Sciences, 2011, 63(3): 667-679.
[18] Yeh F C, Yang R C, Boyle T. Popgene, Version 1.31//Microsoft window-based freeware for population genetic analysis, quick user-guide. University of Alberta: Center for International Forestry Research, 1999.
[19] Nei M. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences, 1973, 70(12): 3321-3323.
[20] Chen C W, Cao K,Wang L R,etal. Molecular ID establishment of main China peach varieties and peach related species. Scientia Agricultura Sinica, 2011, 44(10): 2081-2093.
陈昌文, 曹珂, 王力荣, 等. 中国桃主要品种资源及其野生近缘种的分子身份证构建. 中国农业科学, 2011, 44(10): 2081-2093.
[21] Eujayl I, Sorrells M E, Baum M,etal. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat. Tag. Theoretical & Applied Genetics, 2002, 104(2/3): 399-407.
[22] Bi Q, Mao J F, Guan W. Efficiently developing a large set of polymorphic EST-SSR markers forXanthocerassorbifoliumby mining raw reads from high-throughput sequencing. Conservation Genetics Resources, 2015, 7(2): 423-425.
[23] Wang Q B, Zhang Y Y, Zhuang M,etal. EST-SSR fingerprinting of fifty cabbage representative varieties from China. Scientia Agricultura Sinica, 2014, 47(1): 111-121.
王庆彪, 张扬勇, 庄木. 中国50个甘蓝代表品种EST-SSR指纹图谱的构建. 中国农业科学, 2014, 47(1): 111-121.
[24] Varshney R K, Sigmund R, Börner A,etal. Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice. Plant Science, 2005, 168(1): 195-202.
[25] Zhang L J, Guo Z D, Fang W X,etal. Genetic diversity analysis and cultivar identification of blueberry (Vacciniumspp.) using SRAP marker. Molecular Plant Breeding, 2015, 13(12): 2794-2802.
张鲁杰, 郭照东, 房文秀, 等. 蓝莓品种的SRAP遗传多样性分析及品种鉴别. 分子植物育种, 2015, 13(12): 2794-2802.
[26] Gao Y, Liu F Z, Wang K,etal. Establishment of molecular identity card forMalusMill. originated from China based on the fingerprints of TP-M13-SSR. Journal of Plant Genetic Resources, 2015, 16(6): 1290-1297.
高源, 刘凤之, 王昆, 等. 基于TP-M13-SSR指纹图谱的中国原产苹果属植物分子身份证的建立. 植物遗传资源学报, 2015, 16(6): 1290-1297.
[27] Tang H, Yu H Y, Zhang X M,etal. Analysis on the diversity of DNA fingerprinting of the example varieties used for the test of rice new varieties. Journal of Plant Genetic Resources, 2015, 16(1): 100-106.
唐浩, 余汉勇, 张新明, 等. 水稻新品种测试的标准品种DNA指纹图谱多样性分析. 植物遗传资源学报, 2015, 6(1): 100-106.
[28] Miao H B, Chen F D, Zhao H B,etal. Genetic diversity and construction of fingerprinting ofChrysanthemumcultivars by ISSR markers. Scientia Agricultura Sinica, 2008, 41(11): 3735-3740.
缪恒彬, 陈发棣, 赵宏波, 等. 应用ISSR对25个小菊品种进行遗传多样性分析及指纹图谱构建. 中国农业科学, 2008, 41(11): 3735-3740.
[29] Qi L, Wang W Q, Zhang Z W,etal. DNA fingerprinting analysis of 18 Cassava varieties using sequence-related amplified polymorphism markers. Acta Agronomica Sinica, 2010, 36(10): 1642-1648.
齐兰, 王文泉, 张振文, 等. 利用SRAP标记构建18个木薯品种的DNA指纹图谱. 作物学报, 2010, 36(10): 1642-1648.
[30] Liu H Y, Wu K, Yang M M,etal. DNA fingerprinting of sesame (SesamumindicumL.) varieties (lines) from recent national regional trials in China. Acta Agronomica Sinica, 2012, 38(4): 596-605.
刘红艳, 吴坤, 杨敏敏, 等. 国家芝麻区域试验新品种(系)的DNA指纹分析. 作物学报, 2012, 38(4): 596-605.
[31] Wang J Y, Chen Y Y, Huang B Z,etal. Establishment of fingerprinting for bananas (Musanana) by SSR marker. Journal of Fruit Science, 2009, 26(5): 733-738.
王静毅, 陈业渊, 黄秉智, 等. 部分香蕉品种SSR指纹图谱的构建. 果树学报, 2009, 26(5): 733-738.
[32] Nie X H, You C Y, Li X F,etal. Construction of DNA fingerprinting and analysis of genetic diversity for Xinluzao cotton varieties. Acta Agronomica Sinica, 2014, 40(12): 2104-2117.
聂新辉, 尤春源, 李晓方, 等. 新陆早棉花品种DNA指纹图谱的构建及遗传多样性分析. 作物学报, 2014, 40(12): 2104-2117.

