沙冬青响应非生物胁迫的转录因子基因鉴定与分析
金 曼, 苏彦华
(1. 中国科学院南京土壤研究所 土壤与农业可持续发展国家重点实验室, 江苏 南京 210008; 2. 中国科学院大学, 北京 100049)
沙冬青〔Ammopiptanthusmongolicus(Maxim. ex Kom.) Cheng f.〕隶属于豆科(Fabaceae)沙冬青属(AmmopiptanthusCheng f.),是中国西北阿拉善荒漠地区特有的常绿阔叶灌木,也是第三纪残遗物种,为稀有濒危植物[1]。沙冬青主要分布区生境严酷,气候干旱,年蒸发量为年降水量的20多倍;夏季酷热,高温可达35 ℃以上;冬季严寒,低温可至-30 ℃~-20 ℃[2]。此外,沙冬青在固定流沙以及防止沙漠化方面有重要作用。鉴于沙冬青具有在严苛环境条件下生存的能力及在生态上的重要地位,可将其作为植物抗逆研究的重要材料。
目前已经从沙冬青中分离克隆得到多个参与逆境胁迫的抗性相关基因,这些抗逆相关基因编码的蛋白质可分为2大类:一类为具有保护作用的功能性蛋白质,主要为与逆境胁迫相关的蛋白质,如晚期胚胎发育丰富蛋白AmLEA[3]和脱水素蛋白AmDHN[4]等;与渗透调节物质合成和降解相关的酶类,如肌醇半乳糖苷合成酶AmGolS[5]和脯氨酸转运体AmProT[6]等;与植物毒性物质降解、抗氧化防御能力相关的酶类,如超氧化物歧化酶AmSOD、谷胱甘肽过氧化物酶AmGPX以及抗坏血酸过氧化物酶AmAPX[7]等。另一类为参与逆境条件下信号转导和转录调控相关的蛋白质,主要包括响应及转导胁迫信号的磷酸酶AmCBL[8],以及与胁迫相关的转录因子AmDREB[9]和AmMYB[10]等。
转录因子是一类在转录调控过程中具有十分重要作用的蛋白质,在植物逆境响应调控网络中起关键作用[11]。当植物受到逆境胁迫后,转录因子与下游基因启动子部分特异性结合并对基因的表达进行调控,进而调节细胞内的逆境应答物质含量以响应逆境胁迫。由于转录因子在转录调控过程中具有不可或缺的作用,在对逆境胁迫响应的研究中,当前的研究热点聚焦于转录因子及其与下游靶基因的识别与调控[11]。通过转录组测序技术,可以获得大量的在逆境胁迫下的植物转录本数据,并为筛选响应逆境胁迫的转录因子,以及为后续调控机制的研究提供充足的基因资源。
本研究对非生物胁迫处理前后沙冬青幼苗进行转录组测序及分析,以期为更深入地研究沙冬青响应非生物胁迫的分子机制提供基因资源库,并为进一步鉴定下游的重要非生物响应基因以及研究其调控机制奠定基础。
1 材料和方法
1.1 材料
沙冬青种子由甘肃民勤沙生植物园提供。将沙冬青种子用流动的清水冲洗干净后,挑取大小一致、籽粒饱满的种子用体积分数70%乙醇浸泡30 s,无菌水清洗4~5次后,在黑暗条件下清水浸泡1 d,使种子充分吸胀。将吸胀的沙冬青种子转移到铺满浸润无菌水的吸水纸的灭菌培养皿中,置于25 ℃培养箱中遮光萌发催芽。3 d后,将发芽后长势基本一致的沙冬青幼苗转移至塑料盒中,用改良的1/2Hoagland营养液(pH 5.8)进行培养[6],每隔2 d更换1次营养液。
对在1/2Hoagland营养液中培养3周的沙冬青幼苗进行质量体积分数18%聚乙二醇6000(PEG 6000)模拟的干旱胁迫以及100 mmol·L-1NaCl模拟的盐胁迫。分别于胁迫1、6、12、24和48 h对地上部和地下部的组织取样,并以胁迫0 h的样品为对照,将样品用液氮速冻后置于-80 ℃冰箱中保存。
1.2 转录因子分析及差异表达基因选择
沙冬青转录组数据来源于作者所在实验室前期实验结果。采用RNAiso Plus试剂〔宝生物工程(大连)有限公司〕分别提取正常培养3周以及质量体积分数18%聚乙二醇6000处理6 h的沙冬青幼苗地上部和地下部的总RNA,构建cDNA文库。使用Illumina HiSeqTM2000测序仪(美国Illumina公司)对上述样品进行转录组测序,测序结果经过质量控制过滤去除无效序列,拼接后得到44 959条沙冬青unigenes序列,以fasta格式保存。
利用InterProScan软件(http:∥www.ebi.ac.uk/Tools/pfa/iprscan/)和PlnTFDB 3.0数据库(http:∥plntfdb.bio.uni-potsdam.de/v3.0/)对沙冬青转录组数据中拼接获得的unigenes进行转录因子预测。通过Hmmsearch软件搜寻植物转录因子基因的保守域,并将具有这些植物转录因子基因保守域的unigenes归类到相应的转录因子基因家族中。通过RPKM(reads per kilobase per million reads)法计算沙冬青unigenes的表达丰度(RPKM值),根据在干旱胁迫处理条件与对照条件下基因的表达丰度计算其表达量的差异[12]。将表达量差异倍数大于等于2倍的转录因子视为差异表达转录因子。
1.3 总RNA提取及荧光定量PCR
采用RNAiso Plus试剂〔宝生物工程(大连)有限公司〕分别提取经干旱胁迫和NaCl胁迫的沙冬青幼苗地上部和地下部的总RNA,根据PrimeScriptTM1st Strand cDNA Synthesis Kit试剂盒〔宝生物工程(大连)有限公司〕说明书合成第1链cDNA,用于后续基因定量表达分析。通过Primer Premier 5软件设计6个在转录组中差异表达的AmbHLH基因的荧光定量PCR引物,引物序列见表1,并以编号为comp62392的基因(AmeIF1)[7]作为沙冬青的内参基因,引物由上海英骏生物技术有限公司合成。根据SYBR®PremixExTaqTM试剂盒〔宝生物工程(大连)有限公司〕说明书,在Bio-Rad CFX-96实时定量PCR仪(美国Bio-Rad公司)上进行qRT-PCR反应。反应体系总体积20.0 μL,包含SYBRPremixExTaq10.0 μL、正向引物和反向引物各0.4 μL、ddH2O稀释5倍后的cDNA 5.0 μL及ddH2O 4.2 μL。反应程序为:95 ℃预变性30 s;95 ℃变性10 s、60 ℃退火15 s、72 ℃延伸15 s,40个循环。每个样品3次重复,采用2-ΔΔCT法[13]计算基因的相对表达量。
表1用于沙冬青AmbHLH基因荧光定量PCR扩增的引物序列
Table1PrimersequencesusedforfluorescencequantitativePCRamplificationofAmbHLHgenesfromAmmopiptanthusmongolicus(Maxim.exKom.)Chengf.

基因编号GeneID正向引物序列(5'→3')Sequenceofforwardprimer(5'→3')反向引物序列(5'→3')Sequenceofreverseprimer(5'→3')comp62392(AmeIF1)CTGACATGCGCCGTAGGAACGCCCTGCTTATGCCAGTCTTTTcomp55415TGCAGCATCAAGTGGAGTTCTCATCAACTGGCAACTACT-TCTcomp38040ATTGCTCAGATGGGCGAAGGATGTAGCCGTGCGTAGTGTCcomp58250TGACTTGTCCATCATCTG-GTTTGAGGAGGCTTAGCCATAGACACcomp64918.1GATTTGCTCGCATCACTGGGGGAACAGTGTGACACCAGACAcomp64918.2GCTCATTGACGCCAAATTCTTGGTCTGCGGGTACAAAACAcomp65919AAGAGATGGTTCCCGCTTGGTCTGGAGGGTTTATCCCCGT
2 结果和分析
2.1 干旱胁迫下沙冬青转录因子基因鉴定
对转录组测序拼接的unigenes进行转录因子分析,结果表明:沙冬青中共有1 575个unigenes(即1 575个转录因子基因),归类到49个转录因子基因家族(图1)。在沙冬青各转录因子基因家族中基因数最多的为MYB基因家族,共有222个转录因子基因;之后依次为AP2-EREBP基因家族(144个转录因子基因)、bHLH基因家族(114个转录因子基因)、DBP基因家族(106个转录因子基因)、NAC基因家族(82个转录因子基因)和WRKY基因家族(79个转录因子基因)。
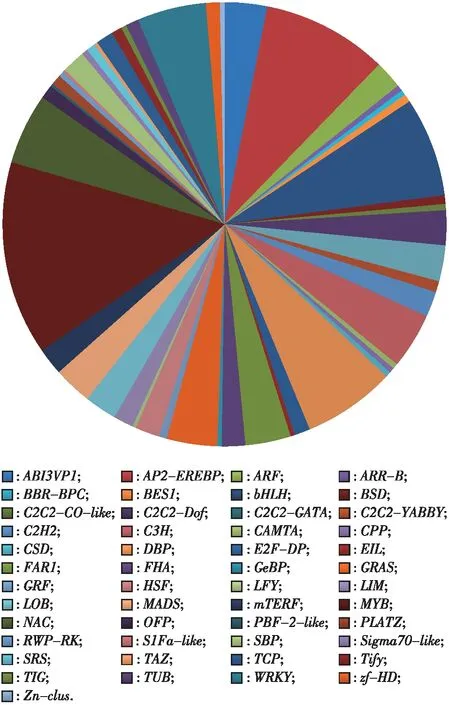
图1 沙冬青转录因子基因家族分类Fig. 1 Classification of transcription factor gene families from Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f.
2.2 干旱胁迫下沙冬青差异表达转录因子基因分析
干旱胁迫下沙冬青地上部共有44个转录因子基因差异表达,其中,33个转录因子基因上调表达,11个转录因子基因下调表达;地下部共有57个转录因子基因差异表达,其中,50个转录因子基因上调表达,7个转录因子基因下调表达(表2)。NAC基因家族中差异表达转录因子基因的数量最多,地上部和地下部分别有8和9个差异表达转录因子基因;之后依次为AP2-EREBP基因家族(地上部和地下部分别有6和9个差异表达转录因子基因)、MYB基因家族(地上部和地下部均有7个差异表达转录因子基因)和WRKY基因家族(地上部和地下部分别有7和2个差异表达转录因子基因)。
干旱胁迫下沙冬青差异表达转录因子基因表达量差异倍数的分布情况见图2。由图2可以看出:干旱胁迫下沙冬青差异表达转录因子基因在地上部和地下部的表达模式不同,地上部表达量的差异倍数为2~5倍的转录因子基因最多,而地下部表达量的差异倍数为5(含5)~10(含10)倍的转录因子基因最多。在地上部,表达量的差异倍数在10(含10)倍以内的转录因子基因有35个,占地上部差异表达转录因子基因总数的79.5%。其中,表达量上(下)调2~5倍的转录因子基因有23个,占地上部差异表达转录因子基因总数的52.3%;表达量上(下)调5(含5)~10(含10)倍的转录因子基因有12个,占地上部差异表达转录因子基因总数的27.3%。表达量的差异倍数在10倍以上的转录因子基因有9个,占地上部差异表达转录因子基因总数的20.5%。
表2干旱胁迫下沙冬青地上部和地下部差异表达转录因子基因分析1)
Table2Analysesondifferentiallyexpressedtranscriptionfactorgenesfromabove-andunder-groundpartsofAmmopiptanthusmongolicus(Maxim.exKom.)Chengf.underdroughtstress1)
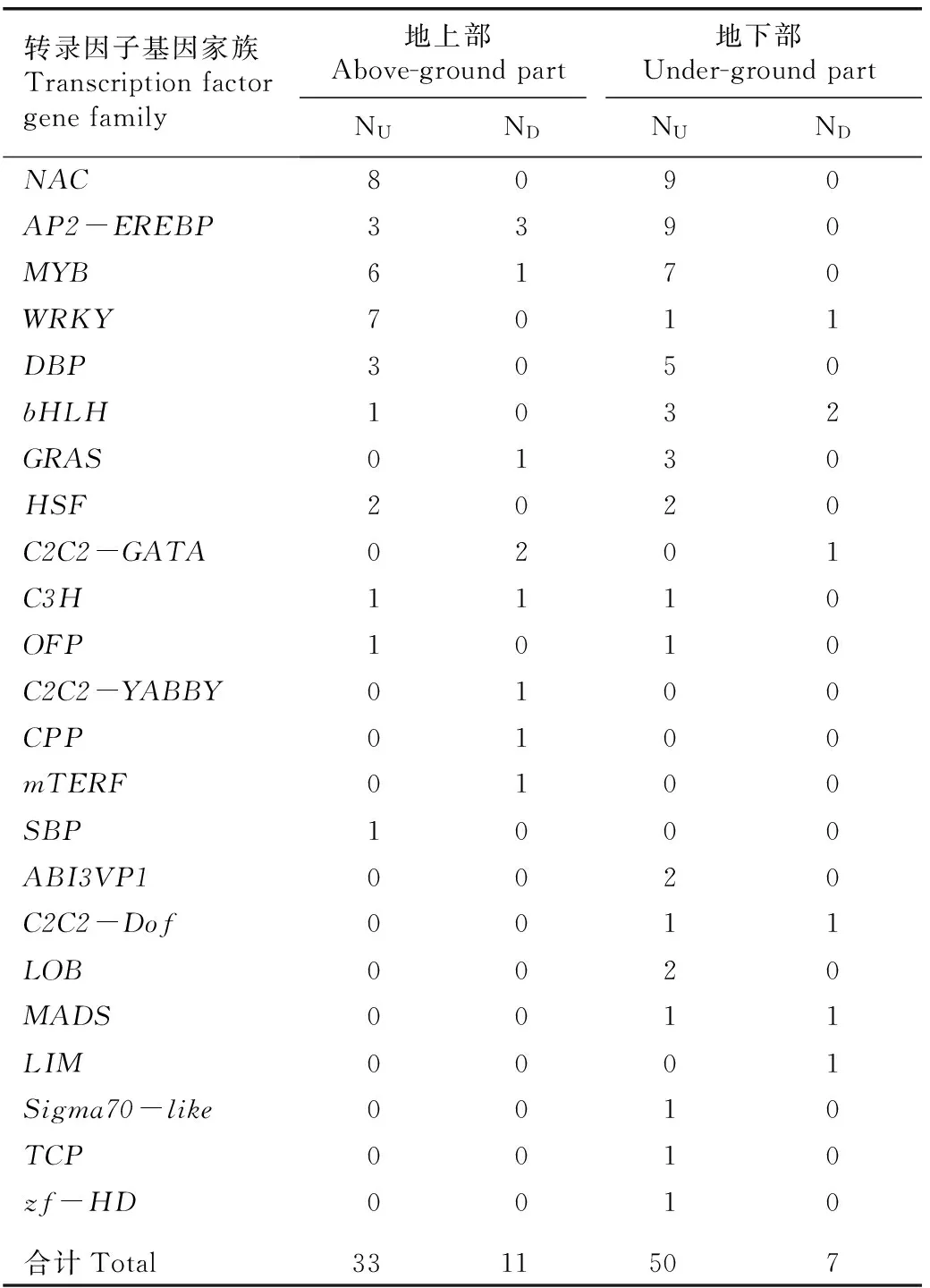
转录因子基因家族Transcriptionfactorgenefamily地上部Above-groundpart地下部Under-groundpartNUNDNUNDNAC8090AP2-EREBP3390MYB6170WRKY7011DBP3050bHLH1032GRAS0130HSF2020C2C2-GATA0201C3H1110OFP1010C2C2-YABBY0100CPP0100mTERF0100SBP1000ABI3VP10020C2C2-Dof0011LOB0020MADS0011LIM0001Sigma70-like0010TCP0010zf-HD0010合计Total3311507
1)NU: 上调差异表达转录因子基因数Number of up-regulated differentially expressed transcription factor genes; ND: 下调差异表达转录因子基因数Number of down-regulated differentially expressed transcription factor genes.
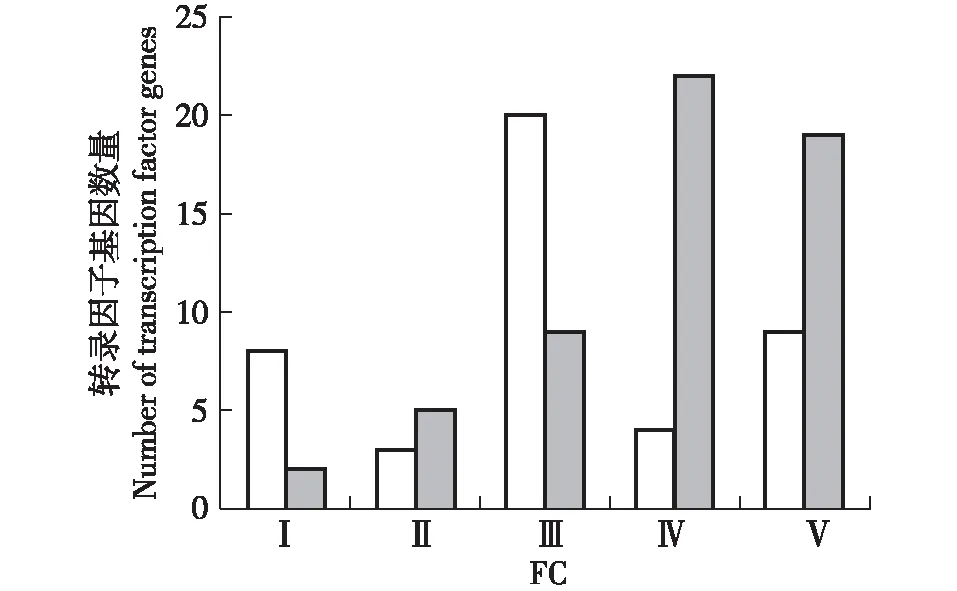
□: 地上部Above-ground part; : 地下部Under-ground part. FC: 差异倍数Fold change. Ⅰ: -10≤FC≤-5; Ⅱ: -5
在地下部,表达量的差异倍数在10(含10)倍以内的转录因子基因有38个,占地下部差异表达转录因子基因总数的66.7%。其中,表达量上(下)调2~5倍的转录因子基因有14个,占地下部差异表达转录因子基因总数的24.6%;表达量上(下)调5(含5)~10(含10)倍的转录因子基因有24个,占地下部差异表达转录因子基因总数的42.1%。表达量的差异倍数在10倍以上的转录因子基因有19个,占地下部差异表达转录因子基因总数的33.3%。
2.3 干旱胁迫下沙冬青差异表达bHLH转录因子的系统进化分析
bHLH类转录因子是植物体内一类重要的转录因子家族,在植物响应逆境胁迫过程中起着重要的调控作用。对沙冬青转录组数据的分析结果显示:沙冬青中存在114个bHLH基因,当受到干旱胁迫后,表达量的差异倍数大于等于2倍的bHLH基因有6个。在转录组数据中对这6个bHLH基因进行搜索,获得各基因序列开放阅读框的全长,这6个bHLH基因在转录组中的编号分别为comp64918.1、comp64918.2、comp55415、comp58250、comp65919和comp38040。利用NCBI的BLASTp软件对这6个bHLH基因编码的氨基酸序列进行同源性比对,分析结果表明:这6个bHLH基因编码的氨基酸序列与拟南芥〔Arabidopsisthaliana(Linn.) Heynh.〕bHLH基因编码的氨基酸序列有不同程度的相似性。
对沙冬青中6个差异表达bHLH基因和拟南芥bHLH基因编码的氨基酸序列进行聚类分析(图3)。结果显示:沙冬青中编号为comp55415的基因编码的氨基酸序列与拟南芥中编号为AT1G73830.1的基因(BEE3,BRenhancedexpression3)编码的氨基酸序列的相似度为46%,编号为comp38040的基因编码的氨基酸序列与编号为AT4G37850.1的基因编码的氨基酸序列的相似度为41%,编号为comp58250的基因编码的氨基酸序列与编号为AT1G10120.1的基因编码的氨基酸序列的相似度为42%,编号为comp64918.1和comp64918.2的基因编码的氨基酸序列与编号为AT3G43790.3的基因(ZIFL2,zincinducedfacilitator-like2)和编号为AT5G13750.1的基因(ZIFL1,zincinducedfacilitator-like1)编码的氨基酸序列的相似度较高,相似度分别为54%和59%,编号为comp65919的基因编码的氨基酸序列与编号为AT4G34530.1的基因(CIB1,cryptochrome-interactingbasic-helix-loop-helix1)编码的氨基酸序列的相似度可达55%。
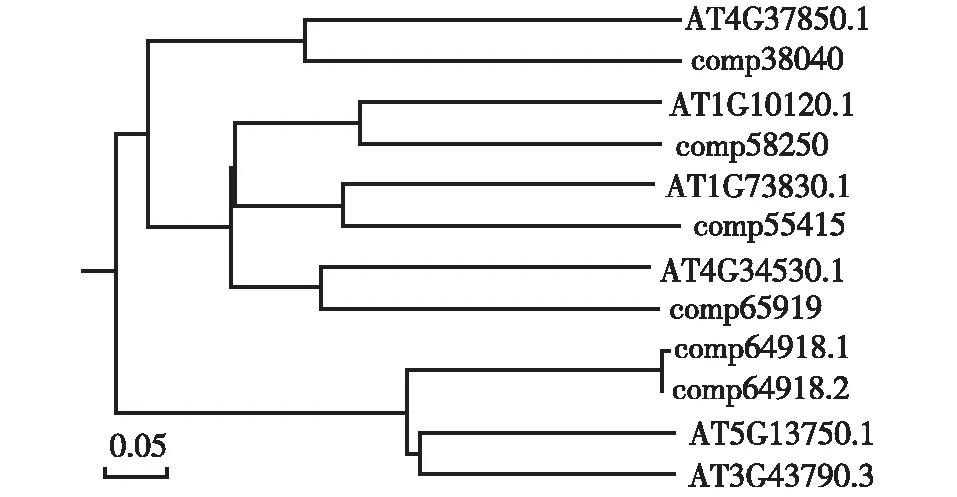
AT4G37850.1,AT1G10120.1,AT1G73830.1,AT4G34530.1,AT5G13750.1,AT3G43790.3: 拟南芥bHLH基因的编号ID of bHLH genes from Arabidopsis thaliana (Linn.) Heynh.; comp64918.1,comp64918.2,comp55415,comp58250,comp65919,comp38040: 沙冬青bHLH基因的编号ID of bHLH genes from Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f.图3 干旱胁迫下沙冬青差异表达bHLH基因和拟南芥bHLH基因编码的氨基酸序列的聚类分析Fig. 3 Cluster analysis on amino acid sequences encoded by differentially expressed bHLH genes from Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f. under drought stress and bHLH genes from Arabidopsis thaliana (Linn.) Heynh.
2.4 干旱胁迫和NaCl胁迫下沙冬青差异表达bHLH基因的表达模式
通过荧光定量PCR对干旱胁迫和NaCl胁迫下沙冬青地上部和地下部6个差异表达bHLH基因的表达模式进行分析,结果分别见图4至图7。
由图4可以看出:随着干旱胁迫时间的延长,沙冬青地上部差异表达bHLH基因的相对表达量略有波动,除编号为comp55415的基因的相对表达量在干旱胁迫1 h明显提高外,其他5个差异表达bHLH基因的相对表达量总体上低于2.0。
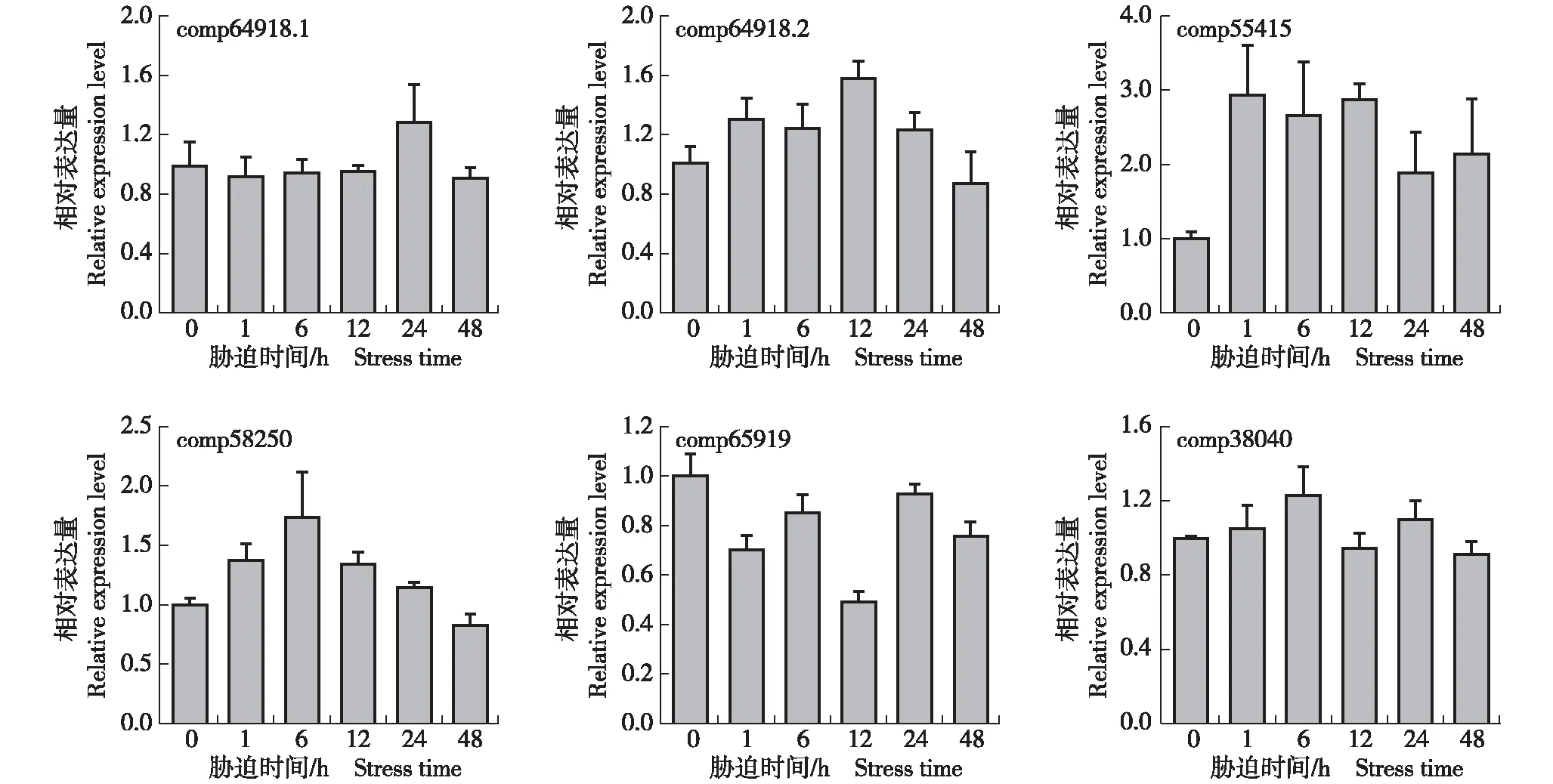
comp64918.1,comp64918.2,comp55415,comp58250,comp65919,comp38040: 基因编号Gene ID.图4 干旱胁迫下沙冬青地上部差异表达bHLH基因的表达模式Fig. 4 Expression pattern of differentially expressed bHLH genes from above-ground part of Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f. under drought stress
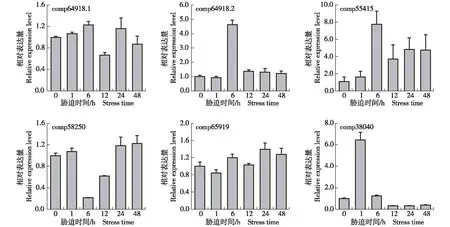
comp64918.1,comp64918.2,comp55415,comp58250,comp65919,comp38040: 基因编号Gene ID.图5 干旱胁迫下沙冬青地下部差异表达bHLH基因的表达模式Fig. 5 Expression pattern of differentially expressed bHLH genes from under-ground part of Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f. under drought stress
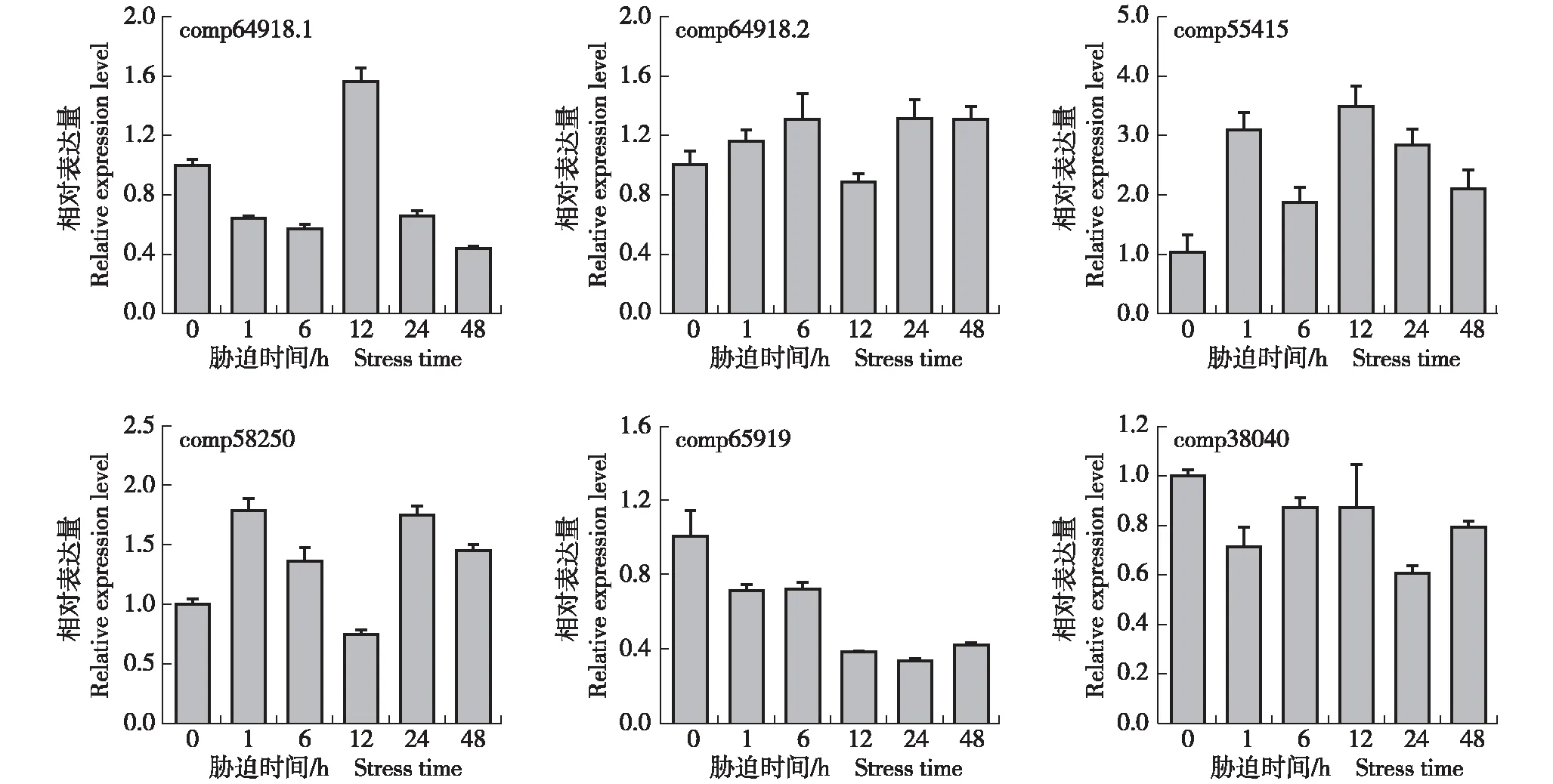
comp64918.1,comp64918.2,comp55415,comp58250,comp65919,comp38040: 基因编号Gene ID.图6 NaCl胁迫下沙冬青地上部差异表达bHLH基因的表达模式Fig. 6 Expression pattern of differentially expressed bHLH genes from above-ground part of Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f. under NaCl stress
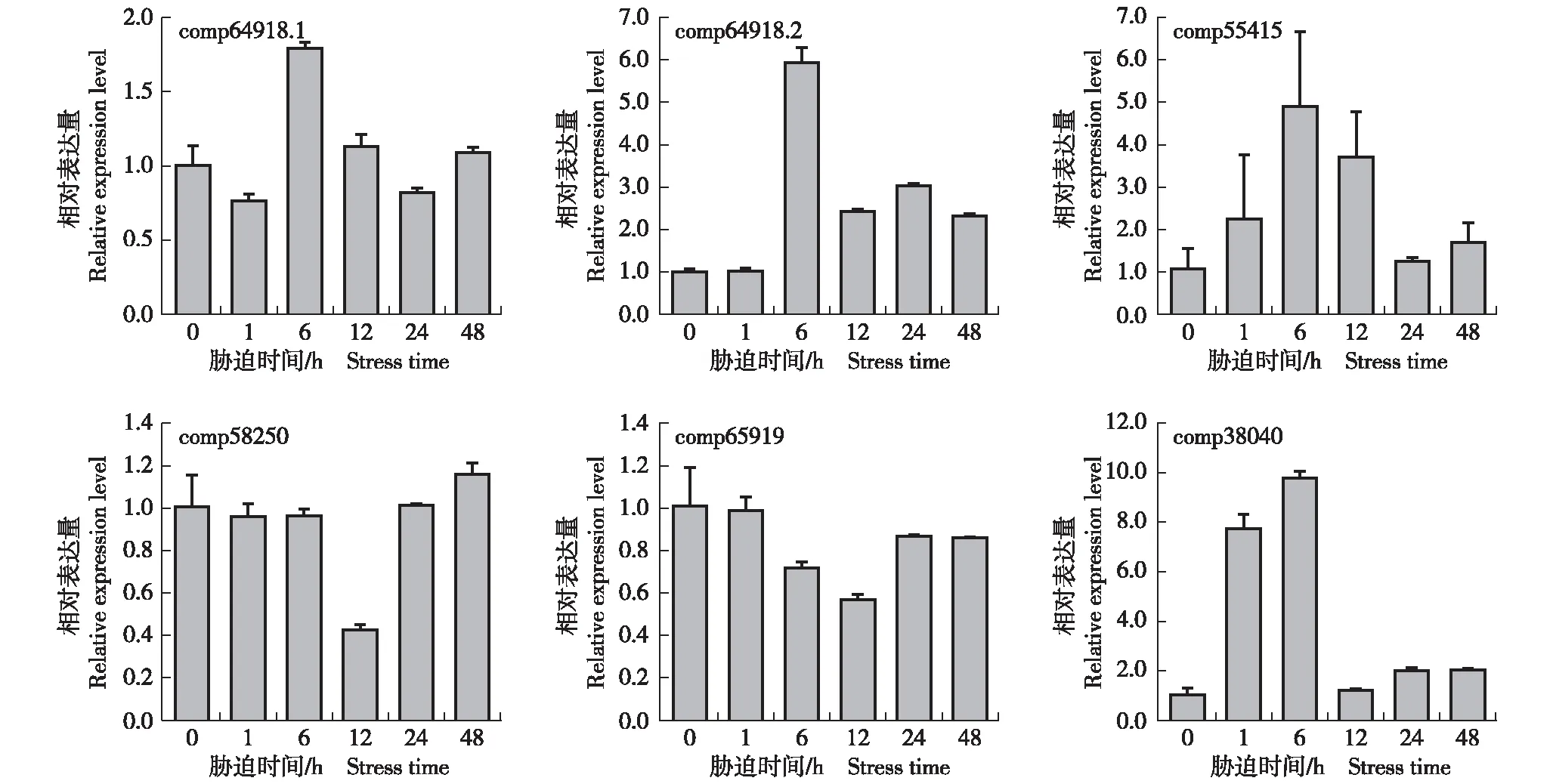
comp64918.1,comp64918.2,comp55415,comp58250,comp65919,comp38040: 基因编号Gene ID.图7 NaCl胁迫下沙冬青地下部差异表达bHLH基因的表达模式Fig. 7 Expression pattern of differentially expressed bHLH genes from under-ground part of Ammopiptanthus mongolicus (Maxim. ex Kom.) Cheng f. under NaCl stress
由图5可以看出:随着干旱胁迫时间的延长,沙冬青地下部编号为comp58250和comp65919的基因的相对表达量呈先降低后升高的趋势,在干旱胁迫处理后期,这2个基因的相对表达量略高于对照;编号为comp64918.1、comp64918.2和comp55415的基因的相对表达量总体上高于对照;而编号为comp38040的基因的相对表达量在干旱胁迫处理前期高于对照,在干旱胁迫处理后期低于对照。此外,编号为comp64918.1、comp64918.2和comp55415的基因的相对表达量在干旱胁迫6 h达到峰值,编号为comp380405的基因的相对表达量在干旱胁迫1 h达到峰值。
由图6可以看出:随着NaCl胁迫时间的延长,沙冬青地上部编号为comp65919和comp38040的基因的相对表达量略有下降;编号为comp64918.2、comp55415和comp58250的基因的相对表达量总体上升高;而编号为comp64918.1的基因的相对表达量在NaCl胁迫12 h高于对照并达到峰值,在其他胁迫时间则低于对照。
由图7可以看出:随着NaCl胁迫时间的延长,沙冬青地下部编号为comp64918.1、comp64918.2、comp55415和comp38040的基因的相对表达量呈先升高后降低的趋势,均在NaCl胁迫6 h达到峰值;编号为comp58250和comp65919的基因的相对表达量无显著变化,均在NaCl胁迫12 h降至最低。
3 讨 论
转录因子在植物响应逆境胁迫的过程中起重要的调控作用,通过调控下游功能基因的表达,从而显著增强植物对逆境的耐受能力。沙冬青是一种古老的沙漠植物,在逆境胁迫条件下具有很强的抗逆能力,是挖掘植物抗逆相关基因的优质物种。本研究的转录组测序结果显示:干旱胁迫下沙冬青NAC、AP2-EREBP、MYB、WRKY和bHLH基因家族的差异表达基因数较多,说明这些基因在响应干旱胁迫的过程中起到重要作用。
在质量体积分数18%聚乙二醇6000模拟的干旱胁迫条件下处理6 h,沙冬青中数个包含NAC和MYB结构域的转录因子基因差异表达。大量研究结果表明:NAC转录因子家族成员参与植物的生长发育、衰老及次生壁合成等生物过程,在逆境应答响应过程中具有重要功能[14-15]。MYB转录因子家族成员对植物的生长发育、次级代谢和激素信号转导以及在响应生物和非生物胁迫过程中均发挥重要的调控作用[16-17]。本研究结果表明:沙冬青在受到干旱胁迫后,其地上部和地下部分别有8和9个NAC基因表达量上调,以及6和7个MYB基因表达量上调。这2类转录因子家族基因在沙冬青受到干旱胁迫后响应强烈,说明其参与了沙冬青干旱胁迫响应的调控过程。
AP2-EREBP是植物特有的一类转录因子家族,可分为AP2和EREBP 2个亚家族[18]。AP2-EREBP转录因子家族成员参与植物逆境响应过程的调控,进而提高植物对逆境的耐受能力[19]。本研究认为,沙冬青地上部有6个AP2-EREBP基因在受到干旱胁迫后差异表达,表达量上调和下调的转录因子基因各有3个;地下部有9个AP2-EREBP基因表达量上调。AP2-EREBP转录因子家族不同基因在干旱胁迫下呈现不同的上调或下调表达模式,表明该转录因子家族中不同成员在干旱胁迫响应过程中的调控方式存在差异。
本研究中,沙冬青地上部有7个WRKY基因表达量上调,地下部有2个WRKY基因差异表达,表达量上调和下调的WRKY基因各1个,说明WRKY转录因子家族在沙冬青干旱胁迫响应过程中起到重要作用。通常,一个WRKY基因能够响应多种胁迫条件,进而对不同的生理过程进行正调控或负调控[20]。研究结果表明:在NicotianabenthamianaDomin中过表达GhWRKY17基因可增强N.benthamiana对干旱和高盐胁迫的耐受能力[21],但是在水稻(OryzasativaLinn.)中过表达OsWRKY45基因则导致水稻对盐胁迫更为敏感[22]。
bHLH转录因子家族具有典型的碱性螺旋-环-螺旋结构域,其不仅参与了植物体内一系列的生长发育调控过程[23-25],而且在植物响应干旱[26-27]、高盐[28-29]、脱落酸[30]、低温[31]、缺铁[32]和低磷[33]等非生物胁迫过程中也发挥着重要作用。本研究结果显示:沙冬青bHLH转录因子家族中有114个基因,其中有6个bHLH基因在干旱胁迫处理下表达量的差异倍数大于等于2倍。这6个bHLH基因不仅在干旱胁迫下差异表达,而且在NaCl胁迫下的表达也受到不同程度的诱导。将沙冬青6个bHLH基因(编号分别为comp64918.1、comp64918.2、comp55415、comp58250、comp65919和comp38040)和拟南芥bHLH基因编码的氨基酸序列进行聚类分析,结果显示:根据氨基酸序列的相似度,编号为comp55415的基因编码的氨基酸序列与拟南芥中编号为AT1G73830.1的基因(BEE3)编码的氨基酸序列最为相似,而编号为AT1G73830.1的基因对应的转录因子调控植物的生长发育,促进下胚轴的伸长[34]。此外,编号为comp58250的基因编码的氨基酸序列与拟南芥中编号为AT1G10120.1的基因编码的氨基酸序列的相似度为42%,编号为comp65919的基因编码的氨基酸序列与拟南芥中编号为AT4G34530.1的基因(CIB1)编码的氨基酸序列的相似度为55%。拟南芥中编号为AT1G10120.1和AT4G34530.1的基因对应的转录因子均参与蓝光信号响应过程,调控花周期开放[35-36]。而与编号为comp38040的基因对应的转录因子最为相似的拟南芥中编号为AT4G37850.1的基因对应的转录因子参与乙烯和生长素的响应过程[37],从而调控逆境胁迫的响应。编号为comp64918.1和comp64918.2的基因编码的氨基酸序列与拟南芥中编号为AT3G43790.3的基因(ZIFL2)和编号为AT5G13750.1的基因(ZIFL1)编码的氨基酸序列的相似度最高,编号为AT3G43790.3和AT5G13750.1的基因与拟南芥体内钾离子的平衡相关,参与调节气孔运动,从而增强拟南芥的抗旱性能[38-39]。
本研究结果可为沙冬青干旱胁迫调控机制研究提供庞大的转录因子基因资源,然而,大多数转录因子基因在干旱胁迫响应中的功能还未明确,其调控的下游基因及其结合位点,以及对下游靶基因的调控机制尚不清楚,仍需进一步深入研究。
4 结 论
对沙冬青转录组的分析结果表明:沙冬青中存在49个转录因子基因家族,共有1 575个转录因子基因。当受到质量体积分数18%聚乙二醇6000模拟的干旱胁迫处理6 h后,沙冬青地上部有44个转录因子基因差异表达,地下部有57个转录因子基因差异表达。bHLH基因家族中6个差异表达的转录因子基因对干旱胁迫以及NaCl胁迫有不同程度的响应,说明其在逆境胁迫调控网络中具有重要作用。
参考文献:
[1]张党权, 宋志丹, 田晔林, 等. 抗逆模式灌木沙冬青的研究进展[J]. 中南林业科技大学学报, 2012, 32(2): 16-22.
[2]LI W R, FENG J C, JIANG T R, et al. Seasonal changes in photosynthetic characteristics ofAmmopiptanthusmongolicus[J]. 植物学报, 1999, 41(2): 190-193.
[3]LIU R, LIU M, LIU J, et al. Heterologous expression of aAmmopiptanthusmongolicuslate embryogenesis abundant protein gene (AmLEA) enhancesEscherichiacoliviability under cold and heat stress[J]. Plant Growth Regulation, 2010, 60: 163-168.
[4]SUN J, NIE L, SUN G, et al. Cloning and characterization of dehydrin gene fromAmmopiptanthusmongolicus[J]. Molecular Biology Reports, 2013, 40: 2281-2291.
[5]SONG J, LIU J, WENG M, et al. Cloning of galactinol synthase gene fromAmmopiptanthusmongolicusand its expression in transgenicPhotiniaserrulataplants[J]. Gene, 2013, 513: 118-127.
[6]岳光振, 金曼, 李俊林, 等. 沙冬青脯氨酸转运体基因的克隆及表达分析[J]. 生物技术通报, 2015, 31(5): 106-112.
[7]SHI J, LIU M, SHI J, et al. Reference gene selection for qPCR inAmmopiptanthusmongolicusunder abiotic stresses and expression analysis of seven ROS-scavenging enzyme genes[J]. Plant Cell Reports, 2012, 31: 1245-1254.
[8]CHEN J H, SUN Y, SUN F, et al. Tobacco plants ectopically expressing theAmmopiptanthusmongolicusAmCBL1 gene display enhanced tolerance to multiple abiotic stresses[J]. Plant Growth Regulation, 2011, 63: 259-269.
[9]李章磊, 高飞, 曹玉震, 等. 蒙古沙冬青AmDREB2.1基因的克隆及表达分析[J]. 生物技术通报, 2015, 31(3): 108-114.
[10]肖自华, 高飞, 孙会改, 等. 蒙古沙冬青AmMYB4-like基因的克隆与表达分析[J]. 生物技术通报, 2017, 33(5): 108-116.
[11]马进, 郑钢, 裴翠明, 等. 南方型紫花苜蓿根系盐胁迫应答转录因子鉴定与分析[J]. 浙江农林大学学报, 2016, 33(2): 201-208.
[12]MORTAZAVI A, WILLIANMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods, 2008, 5: 621-628.
[13]LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod[J]. Methods, 2001, 25: 402-408.
[14]PURANIK S, SAHU P P, SRIVASTAVA P S, et al. NAC proteins: regulation and role in stress tolerance[J]. Trends in Plant Science, 2012, 17: 369-381.
[15]NAKASHIMA K, TAKASAKI H, MIZOI J, et al. NAC transcription factors in plant abiotic stress responses[J]. Biochimica et Biophysica Acta, 2012, 1819: 97-103.
[16]COMINELLI E, TONELLI C. A new role for plant R2R3-MYB transcription factors in cell cycle regulation[J]. Cell Research, 2009, 19: 1231-1232.
[17]ALLAN A C, HELLENS R P, LAING W A. MYB transcription factors that colour our fruit[J]. Trends in Plant Science, 2008, 13: 99-102.
[18]王庆灵, 刘文鑫, 赵嘉平. 山海关杨PdERF-18转录因子的表达特征分析[J]. 浙江农林大学学报, 2014, 31(5): 716-723.
[19]HUSSAIN S S, KAYANI M A, AMJAD M. Transcription factors as tools to engineer enhanced drought stress tolerance in plants[J]. Biotechnology Progress, 2011, 27: 297-306.
[20]CHEN L, SONG Y, LI S, et al. The role of WRKY transcription factors in plant abiotic stresses[J]. Biochimica et Biophysica Acta, 2012, 1819: 120-128.
[21]YAN H, JIA H, CHEN X, et al. The cotton WRKY transcription factor GhWRKY17 functions in drought and salt stress in transgenicNicotianabenthamianathrough ABA signaling and the modulation of reactive oxygen species production[J]. Plant and Cell Physiology, 2014, 55: 2060-2076.
[22]TAO Z, KOU Y, LIU H, et al.OsWRKY45 alleles play different roles in abscisic acid signalling and salt stress tolerance but similar roles in drought and cold tolerance in rice[J]. Journal of Experimental Botany, 2011, 62: 4863-4874.
[23]NESI N, DEBEAUJON I, JOND C, et al. TheTT8 gene encodes a basic helix-loop-helix domain protein required for expression ofDFRandBANgenes in Arabidopsis siliques[J]. The Plant Cell, 2000, 12: 1863-1878.
[24]GROSZMANN M, PAICU T, SMYTH D R. Functional domains of SPATULA, a bHLH transcription factor involved in carpel and fruit development in Arabidopsis[J]. The Plant Journal, 2008, 55: 40-52.
[25]FUJISAWA M, NAKANO T, SHIMA Y, et al. A large-scale identification of direct targets of the tomato MADS box transcription factor RIPENING INHIBITOR reveals the regulation of fruit ripening[J]. The Plant Cell, 2013, 25: 371-386.
[26]KIRIBUCHI K, JIKUMARU Y, KAKU H, et al. Involvement of the basic helix-loop-helix transcription factor RERJ1 in wounding and drought stress responses in rice plants[J]. Bioscience Biotechnology, and Biochemistry, 2005, 69: 1042-1044.
[27]SEO J S, JOO J, KIM M J, et al. OsbHLH148, a basic helix-loop-helix protein, interacts with OsJAZ proteins in a jasmonate signaling pathway leading to drought tolerance in rice[J]. The Plant Journal, 2011, 65: 907-921.
[28]ZHOU J, LI F, WANG J L, et al. Basic helix-loop-helix transcription factor from wild rice (OrbHLH2) improves tolerance to salt- and osmotic stress inArabidopsis[J]. Journal of Plant Physiology, 2009, 166: 1296-1306.
[29]陈李淼, 沙爱华, 张婵娟, 等. 一个大豆脱水胁迫响应的bHLH类转录因子的克隆及功能分析[J]. 中国油料作物学报, 2013, 35(6): 630-636.
[30]ABE H, URAO T, ITO T, et al. Arabidopsis AtMYC2 (bHLH) and AtMYB2 (MYB) function as transcriptional activators in abscisic acid signaling[J]. The Plant Cell, 2003, 15: 63-78.
[31]CHINNUSAMY V, OHTA M, KANRAR S, et al. ICE1: a regulator of cold-induced transcriptome and freezing tolerance inArabidopsis[J]. Gene and Development, 2003, 17: 1043-1054.
[32]ZHANG J, LIU B, LI M, et al. The bHLH transcription factor bHLH104 interacts with IAA-LEUCINE RESISTANT3 and modulates iron homeostasis in Arabidopsis[J]. The Plant Cell, 2015, 27: 787-805.
[33]YI K, WU Z, ZHOU J, et al.OsPTF1, a novel transcription factor involved in tolerance to phosphate starvation in rice[J]. Plant Physiology, 2005, 138: 2087-2096.
[34]CIFUENTES-ESQUIVEL N, BOU-TORRENT J, GALSTYAN A, et al. The bHLH proteins BEE and BIM positively modulate the shade avoidance syndrome in Arabidopsis seedlings[J]. The Plant Journal, 2013, 75: 989-1002.
[35]LIU Y, LI X, LI K, et al. Multiple bHLH proteins form heterodimers to mediate CRY2-dependent regulation of flowering-time inArabidopsis[J]. PLoS Genetics, 2013, 9: e1003861.
[36]LIU H T, WANG Q, LIU Y, et al.ArabidopsisCRY2 and ZTL mediate blue-light regulation of the transcription factor CIB1 by distinct mechanisms[J]. Proceedings of the National Academy ofSciences of the United States of America, 2013, 110: 17582-17587.
[37]STEPANOVA A N, YUN J, LIKHACHEVA A V, et al. Multilevel interactions between ethylene and auxin inArabidopsisroots[J]. The Plant Cell, 2007, 19: 2169-2185.
[38]REMY E, CABRITO T R, BATISTA R A, et al. The major facilitator superfamily transporter ZIFL2 modulates cesium and potassium homeostasis in Arabidopsis[J]. Plant and Cell Physiology, 2015, 56: 148-162.
[39]REMY E, CABRITO T R, BASTER P, et al. A major facilitator superfamily transporter plays a dual role in polar auxin transport and drought dtress tolerance inArabidopsis[J]. The Plant Cell, 2013, 25: 901-926.
——致坚守奋斗的你

