猪繁殖与呼吸综合征病毒类NADC30株CHsx1401感染性克隆的构建
卞 婷,周 磊,宋江伟,盖新娜,郭 鑫,韩 军,杨汉春
(中国农业大学动物医学院农业部动物流行病学重点实验室,北京 100193)
猪繁殖与呼吸综合征病毒(PRRSV)是影响全球养猪业的一种重要病原[1-2]。PRRSV为有囊膜的单股正链RNA病毒,归类于套式病毒目、动脉炎病毒科、动脉炎病毒属[3]。国际病毒分类委员会(ICTV)最新的分类显示,动脉炎病毒科有5个属,PRRSV归类于猪动脉炎病毒属(Porartevirus)(https://talk.ictvonline.org/ictv-reports/ictv_online_report/)。PRRSV基因组全长约15 kb[4-5],至少包含有12个开放阅读框(opening reading frames, ORFs)[6-7]。基于基因组序列及抗原特性的差异,PRRSV被分为两大基因型,即欧洲型(基因1型)和北美洲型(基因2型)[8-10],两大基因型毒株间核苷酸相似性仅为60%左右[11-12]。迄今为止,PRRSV感染及其造成的疫情仍未得到很好的控制,仍然在许多国家流行,尤其是变异或新毒株的出现经常导致疫病的暴发和再流行[13-16]。
自20世纪90年代中期PRRSV在我国被确认以来,已成为严重影响我国养猪业的主要病原之一[17],尤其是2006年出现的高致病性PRRSV(HP-PRRSV)的广泛流行给我国养猪业造成了巨大的经济损失[15, 17]。近年来,基因2型PRRSV的类NADC30毒株在我国出现[18-19],并造成许多地区猪场PRRS疫情的暴发[19-21]。该类毒株的非结构蛋白2(Nsp2)编码区存在与2008年美国分离株NADC30相同的缺失模式[22],并且已有文献报道该类毒株与国内高致病性毒株存在基因重组现象[23]。作为影响中国养猪业的一类新的PRRSV毒株,研究其致病机制为临床疫情的控制提供依据显得尤为重要,而反向遗传操作技术是研究PRRSV变异与演化及分子致病机制的一个重要工具。因此,本研究旨在构建本实验室于2014年分离获得的类NADC30毒株CHsx1401的感染性克隆,以期为深入研究该类毒株的变异与演化以及与其他毒株致病性差异的分子机制奠定基础。
1 材料与方法
1.1 材料
1.1.1病毒和细胞PRRSV 培养所需传代细胞 MARC-145,由中国农业大学农业部动物流行病学重点实验室保存。原代猪肺泡巨噬细胞(pulmonary alveolar macrophages,PAMs)从4周龄SPF仔猪肺泡灌洗液中获得,保存于液氮中备用。类NADC30毒株CHsx1401,分离于我国山西省某暴发 PRRS 的猪场[19],其全序组序列已测定并被GenBank(KP861625)收录。
1.1.2载体和菌株pJET1.2克隆载体试剂盒购于Fisher Scientific公司(Waltham, MA, USA)。改造后的 pWSK-29 低拷贝质粒,用于全长感染性克隆质粒的构建,由中国农业大学农业部动物流行病学重点实验室保存。大肠杆菌(Escherichiacoli,E.coli) Trans10、DMT 感受态购于北京全式金生物技术有限公司。
1.1.3主要试剂KOD 高保真 PCR 扩增酶购于东洋纺上海生物科技有限公司;GIBCOTMOpti-MEM®I Reduced Serum Medium、GIBCOTMRPM/MINI 1640与GIBCOTMDMEM以及 Lipofectamine LTX and plus reagents均购自Fisher Scientific公司;DNA/RNA 提取试剂盒、dNTPs、TIANamp DNA/RNA kit、Fast Site-Directed Mutagenesis Kit 点突变试剂盒及 Trans15K Marker均购于全式金生物技术有限公司;质粒提取试剂盒 Wizard®Plus Midipreps DNA Purification System、M-MLV反转录酶和 T4 DNA 连接酶均购于 Promega公司(Madison, WI, USA);DNA 琼脂糖胶回收试剂盒购自Omega公司;所有限制性内切酶均购于NEB公司(北京,中国)。PRRSV N蛋白单克隆抗体由中国农业大学农业部动物流行病学重点实验室制备并保存。异硫氰酸荧光素(FITC)-羊抗小鼠 IgG购自中杉金桥生物有限公司(北京,中国)。
1.2 方法
1.2.1PRRSV CHsx1401基因组片段cDNA的扩增基于PRRSV CHsx1401的全基因组序列,将其基因组分为四个片段(A、B、C、D),分别设计RT-PCR扩增引物(表1)。取200 μL PRRSV CHsx1401 F8代病毒液,用DNA/RNA 提取试剂盒提取病毒RNA,将提取的病毒基因组RNA分别用设计的扩增各片段的下游引物AR、BR、CR、DR进行反转录,然后进行PCR扩增。
表1扩增PRRSV基因组cDNA片段和回复突变所用引物
Table1PrimersusedforamplificationofcDNAfragmentsofPRRSVgenomeandreversemutations
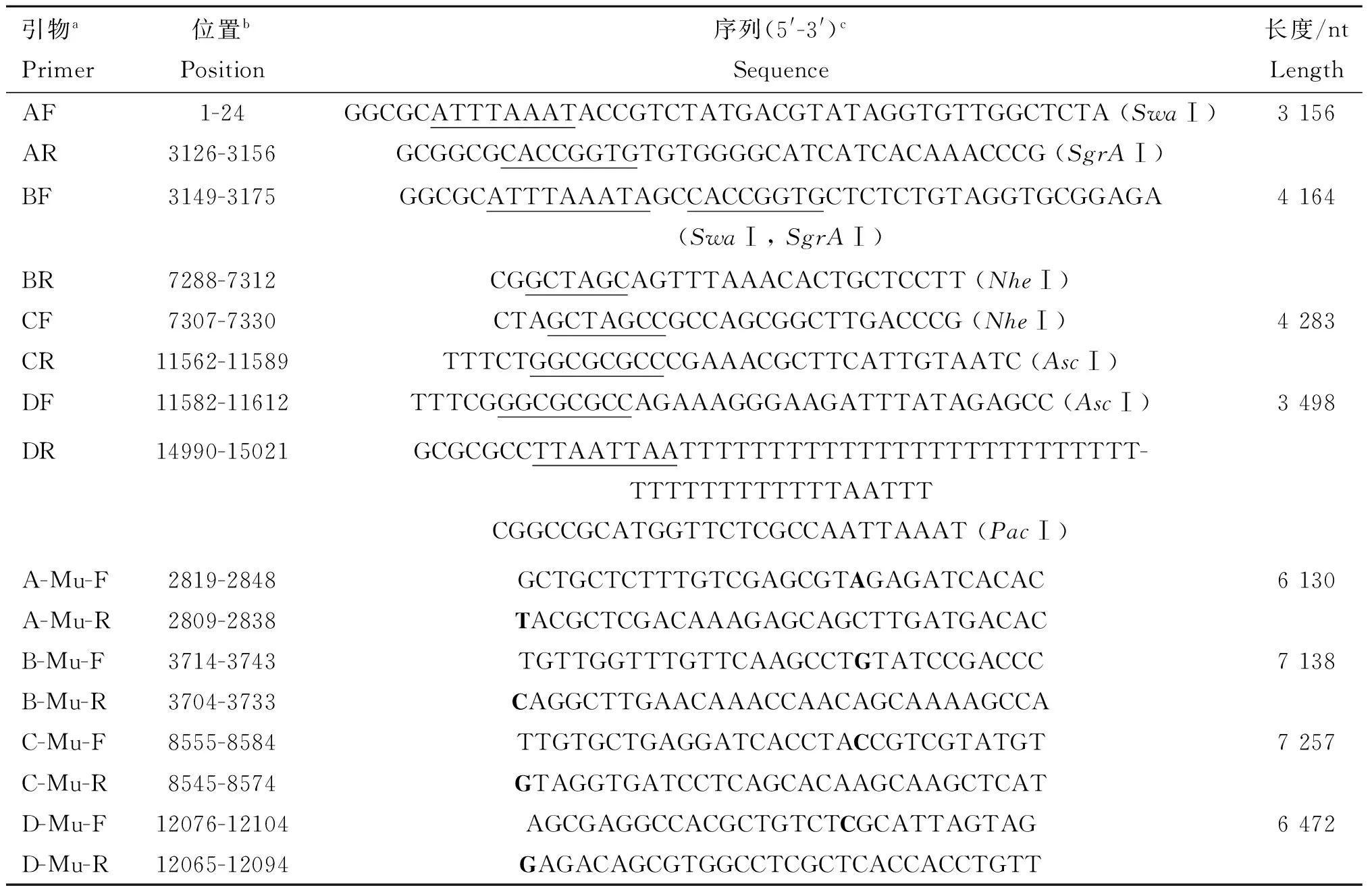
引物aPrimer位置bPosition序列(5'-3')cSequence长度/ntLengthAF1-24GGCGCATTTAAATACCGTCTATGACGTATAGGTGTTGGCTCTA(SwaⅠ)3156AR3126-3156GCGGCGCACCGGTGTGTGGGGCATCATCACAAACCCG(SgrAⅠ)BF3149-3175GGCGCATTTAAATAGCCACCGGTGCTCTCTGTAGGTGCGGAGA(SwaⅠ,SgrAⅠ)4164BR7288-7312CGGCTAGCAGTTTAAACACTGCTCCTT(NheⅠ)CF7307-7330CTAGCTAGCCGCCAGCGGCTTGACCCG(NheⅠ)4283CR11562-11589TTTCTGGCGCGCCCGAAACGCTTCATTGTAATC(AscⅠ)DF11582-11612TTTCGGGCGCGCCAGAAAGGGAAGATTTATAGAGCC(AscⅠ)3498DR14990-15021GCGCGCCTTAATTAATTTTTTTTTTTTTTTTTTTTTTTTTT-TTTTTTTTTTTTAATTTCGGCCGCATGGTTCTCGCCAATTAAAT(PacⅠ)A-Mu-F2819-2848GCTGCTCTTTGTCGAGCGTAGAGATCACAC6130A-Mu-R2809-2838TACGCTCGACAAAGAGCAGCTTGATGACACB-Mu-F3714-3743TGTTGGTTTGTTCAAGCCTGTATCCGACCC7138B-Mu-R3704-3733CAGGCTTGAACAAACCAACAGCAAAAGCCAC-Mu-F8555-8584TTGTGCTGAGGATCACCTACCGTCGTATGT7257C-Mu-R8545-8574GTAGGTGATCCTCAGCACAAGCAAGCTCATD-Mu-F12076-12104AGCGAGGCCACGCTGTCTCGCATTAGTAG6472D-Mu-R12065-12094GAGACAGCGTGGCCTCGCTCACCACCTGTT
a. F表示上游引物,R表示反转录引物或下游引物,Mu表示回复突变所用引物;b. 数字表示在CHsx1401(KP861625)基因组中的位置;c. 括号中加下划线部分表示由PCR引入的限制性内切酶,加粗字母表示回复突变的核苷酸
a. F represents a forward PCR primer, R represents reverse transcription or a reverse PCR primer, Mu represents the primers used for reverse mutations;b. Numbers refer to the nucleotide position within the genome of CHsx1401 (GenBank accession No. KP861625);c.Restriction enzyme sites introduced by PCR are underlined and indicated in parenthesis. The letters in boldface show the reverse mutated nucleotides
1.2.2全长cDNA克隆构建策略参照先前PRRSV感染性克隆构建的策略和方法[24],构建CHsx1401毒株的全长cDNA克隆。在低拷贝质粒pWSK-29中插入CMV启动子和丁型肝炎病毒核酶,将CHsx1401全长分为四个片段,在A片段5′端加入SwaⅠ酶切位点,在A片段的3′端及B片段的5′端加入SwaⅠ和SgrAⅠ两个酶切位点,同时在D片段 3′末端加入Ploy(A)和PacⅠ酶切位点(表1)。利用PCR方法将CHsx1401基因组11582位处T同义突变为G引入一个全长单一酶切位点AscⅠ,该酶切位点既可以用于连接C和D片段,又可以作为拯救病毒的遗传标记(图1)。RT-PCR扩增各片段后分别连接至pJET1.2载体,构建中间质粒,测序无误后通过各片段两端的酶切位点将片段切下后按 DCBA 的顺序依次连接到 pWSK-29,构建全长cDNA质粒。

图1 pWSK-CHsx1401构建策略Fig.1 Construction strategy of pWSK-CHsx1401
1.2.3中间质粒的构建PRRSV CHsx1401基因组中扩增片段产物经琼脂糖电泳鉴定后回收目的条带,将回收产物连接至pJET1.2载体,连接产物转化Trans10感受态,37 ℃过夜培养后挑选菌落进行PCR鉴定,将菌落PCR鉴定为阳性的克隆,送公司测序。如果存在突变点,则使用Fast Site-Directed Mutagenesis Kit 点突变试剂盒进行回复突变。
1.2.4全长cDNA克隆质粒的构建及测序鉴定用两端的PacⅠ和AscⅠ将连接于pJET1.2载体上的D片段切下,再经T4连接酶连于pWSK29载体,连接产物转化Trans10感受态细胞,用D片段引物 DF 和 DR 鉴定选取阳性克隆接种含有200 mL LB/Amp+培养基,28 ℃ 150 r·min-1摇振培养20 h后,提取质粒。C片段的酶切以及连接方法与D片段方法相同。由于B片段插入的两个限制性内切酶的反应条件不同,需分别进行单酶切后再连接到已构建好的pWSK29-SDC载体中。A片段与B片段相似,同样需分别进行单酶切后再连入已构建好的pWSK29-SDCB载体中,最后对构建好的阳性克隆进行全基因组测序以确保序列的准确性。
1.2.5病毒拯救及鉴定将生长旺盛的MARC-145细胞经胰酶消化,以约 106·mL-1的细胞浓度培养于6孔细胞培养板,每孔2 mL。置于含有 5% CO2细胞培养箱,37 ℃条件下培养,当细胞长满至70%~80%时进行转染。用LTX脂质体将全长cDNA克隆转染至MARC-145细胞,96 h后收取全部细胞培养液置于-80 ℃,反复冻融2次后在MARC-145细胞连续传代2次。用间接免疫荧光和RT-PCR及序列测定对P3代拯救病毒进行鉴定。
1.2.6体外增殖动态按常规方法,将亲本病毒与拯救病毒按MOI为0.01分别接种MARC-145细胞和PAMs,分别于感染后12、24、36、48、60、72、84 h收取细胞培养液,测定病毒TCID50。
2 结 果
2.1 PRRSV NADC30-like毒株CHsx1401基因组片段的扩增及中间质粒的构建
用表1中设计的扩增 CHsx1401 全基因组的四个片段相应引物,将病毒基因组RNA反转录为cDNA,用高保真酶进行 PCR 扩增,依次获得病毒基因组的A、B、C 和D 四个扩增片段,与预期大小(分别为3 156、4 164、4 283和3 498 bp)相符(图2)。在A片段5′端加入SwaⅠ酶切位点,在A片段的3′端及B片段的5′端加入SwaⅠ和SgrAⅠ两个酶切位点,同时在D片段3′末端加入Ploy(A)和PacⅠ酶切位点。在C 片段3′端使用 PCR 进行同义突变引入一个单一酶切位点AscⅠ,用于C、D 片段的连接和区分亲本病毒与拯救病毒的遗传标记。A、B、C、D 四个片段的 PCR 产物经琼脂糖电泳切取相应大小的片段,胶回收后连接到平末端克隆载体pJET1.2,分别构建出中间质粒pJET-A、pJET-B、pJET-C和pJET-D。对各中间质粒进行测序鉴定发现各片段均有由PCR扩增非特异性引入的1个突变点,分别为nt2838位的A突变为G,nt3733位的G突变为A,nt8574位C突变为T和nt12094位C突变为T。利用表1中设计的引物以及点突变试剂盒,对突变位点进行回复突变。
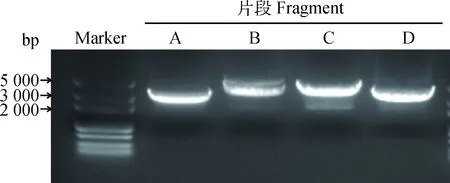
图2 NADC30-like CHsx1401全基因组片段RT-PCR扩增Fig.2 Amplification of four fragments of NADC30-like CHsx1401 genome by RT-PCR
2.2 CHsx1401全长cDNA克隆的构建
将改造后的低拷贝质粒pWSK29和pJET-D分别进行AscⅠ/PacⅠ双酶切,回收相应片段进行连接,转化Trans10感受态细胞。37 ℃ 过夜培养后,用D片段扩增引物 DF/DR对菌落进行 PCR 鉴定,选取阳性菌落进行扩大培养和质粒提取,质粒命名为pWSK-SD。将 pWSK-SD和pJET-C分别进行NheⅠ/AscⅠ双酶切,回收相应片段,转化后用PCR筛选阳性克隆,再经扩大培养和质粒提取,获得质粒pWSK-SDC。用相同方法依次连接B片段和A片段,最终构建出CHsx1401 的全长cDNA克隆质粒 pWSK-SDCBA,全长质粒经序列测定验证正确后,命名为 pWSK-CHsx1401。
2.3 病毒拯救及鉴定
将全长cDNA克隆质粒pWSK-CHsx1401转染MARC-145 细胞,并传代。结果表明,第二代即可观察到典型的 PRRSV 细胞病变(CPE),拯救病毒命名为RvCHsx1401。将拯救病毒 P3 代接种96孔培养板的 MARC-145 细胞,培养48 h 后用预冷的无水乙醇固定,用 PRRSV N蛋白单克隆抗体进行免疫荧光染色。结果表明,拯救病毒感染细胞呈现特异性荧光染色(图3)。将 P3 代拯救病毒培养上清进行 RT-PCR及测序鉴定,确证了遗传标记的存在。
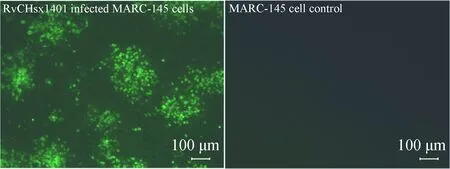
图3 拯救病毒的免疫荧光鉴定Fig.3 Identification of the rescued virus by indirect immunofluorescence assay
2.4 拯救病毒全基因组序列测定
对P3代拯救病毒的全基因组序列进行测定。结果表明,拯救病毒RvCHsx1401的基因组序列无碱基突变,与亲本病毒序列相似性为100%。
2.5 拯救病毒传代稳定性鉴定
将拯救病毒在MARC-145细胞上连续传10代,对3、5、7、9和10代病毒的滴度进行了测定。结果表明,拯救病毒均可在MARC-145细胞上稳定传代,毒价介于105~107.5TCID50·mL-1。同时对各代病毒的遗传标记区域进行测序分析,显示遗传标记稳定存在,未发生突变。
2.6 拯救病毒体外增殖特性
在MARC-145及原代PAMs上的生长曲线显示(图4),拯救病毒RvCHsx1401的增殖动态与亲本病毒CHsx1401相似,仅在MARC-145细胞上各时相的病毒滴度略低于亲本病毒,但无显著性差异。
3 讨 论
反向遗传操作技术即感染性克隆技术,是研究RNA病毒基因组结构与基因功能以及致病机制必不可少的技术平台。PRRSV作为一种影响全球养猪业的重要RNA病原,其感染性克隆技术已广泛应用于其基因功能、复制及分子致病机制等多个领域的研究[24-26]。

图4 拯救病毒在MARC-145细胞(A)和原代PAM(B)上的生长曲线Fig.4 Growth curves of the rescued virus RvCHsx1401 in MARC-145 cells (A) and in primary PAMs (B)
PRRSV严重危害我国养猪生产,其变异与演化快速,毒株多样性不断攀升,新毒株以及重组毒株出现的频率持续增高[27]。近几年,属于基因2型谱系1的类 NADC30 毒株在我国广泛流行,成为我国的优势流行毒株之一。最新的流行病学调查显示,无论猪场免疫何种减毒活疫苗,一旦受到该毒株感染,猪群均会发病[28]。已有研究表明,类NADC30毒株CHsx1401对仔猪具有中等毒力,感染仔猪呈现体温升高、呼吸道症状和肺组织病变,且目前使用的猪繁殖与呼吸综合征活疫苗免疫均不能有效抵抗该毒株的感染[29]。此外,该毒株与我国其他毒株以及疫苗病毒频繁重组[30],加剧了PRRSV的防控难度。显而易见,类NADC30毒株的致病性不同于我国基因2型谱系8的高致病性毒株,且差别很大。因此,研究该类毒株的致病机制以及与我国毒株致病性差异的分子机制十分必要,同时分析其与我国毒株的重组特性与分子机制对于我国养猪生产中PRRSV感染的控制有重要的意义。
本研究成功构建出类NADC30代表性毒株CHsx1401的感染性cDNA克隆,且拯救出与亲本毒体外增殖动态基本一致、传代稳定的克隆病毒。我们将进一步利用类NADC30毒株的感染性cDNA克隆,与我国高致病性毒株进行基因片段的置换,构建嵌合病毒,进而分析相关基因在不同毒株间致病性差异方面的作用和生物学意义。
4 结 论
成功构建出PRRSV类NADC30毒株CHsx1401的感染性克隆,拯救病毒的体外增殖特性与亲本病毒基本一致,为进一步研究该毒株的变异与演化以及致病的分子机制奠定基础。
参考文献(References):
[1]NEUMANN E J, KLIEBENSTEIN J B, JOHNSON C D, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States[J].JAmVetMedAssoc, 2005, 227(3): 385-392.
[2]PEJSAK Z, STADEJEK T, MARKOWSKA-DANIEL I. Clinical signs and economic losses caused by porcine reproductive and respiratory syndrome virus in a large breeding farm[J].VetMicrobiol, 1997, 55(1-4): 317-322.
[3]CAVANAGH D. Nidovirales: a new order comprising Coronaviridae and Arteriviridae[J].ArchVirol, 1997, 142(3): 629-633.
[4]CONZELMANN K K, VISSER N, VAN WOENSEL P, et al. Molecular characterization of porcine reproductive and respiratory syndrome virus, a member of the arterivirus group[J].Virology, 1993, 193(1): 329-339.
[5]MEULENBERG J J M, PETERSEN DEN BESTEN A, DE KLUYVER E, et al. Molecular characterization of Lelystad virus[J].VetMicrobiol, 1997, 55(1-4): 197-202.
[6]JOHNSON C R, GRIGGS T F, GNANANDARAJAH J, et al. Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5 present in all arteriviruses[J].JGenVirol, 2011, 92(5): 1107-1116.
[7]SNIJDER E J, MEULENBERG J J. The molecular biology of arteriviruses[J].JGenVirol, 1998, 79(Pt 5): 961-979.
[8]MARDASSI H, MOUNIR S, DEA S. Identification of major differences in the nucleocapsid protein genes of a Québec strain and European strains of porcine reproductive and respiratory syndrome virus[J].JGenVirol, 1994, 75(Pt 3): 681-685.
[9]MENG X J, PAUL P S, HALBUR P G, et al. Phylogenetic analyses of the putative M (ORF 6) and N (ORF 7) genes of porcine reproductive and respiratory syndrome virus (PRRSV): implication for the existence of two genotypes of PRRSV in the U.S.A. and Europe[J].ArchVirol, 1995, 140(4): 745-755.
[10]NELSON E A, CHRISTOPHER-HENNINGS J, DREW T, et al. Differentiation of U.S. and European isolates of porcine reproductive and respiratory syndrome virus by monoclonal antibodies[J].JClinMicrobiol, 1993, 31(12): 3184-3189.
[11]MURTAUGH M P, ELAM M R, KAKACH L T. Comparison of the structural protein coding sequences of the VR-2332 and Lelystad virus strains of the PRRS virus[J].ArchVirol, 1995, 140(8): 1451-1460.
[12]NELSEN C J, MURTAUGH M P, FAABERG K S. Porcine reproductive and respiratory syndrome virus comparison: divergent evolution on two continents[J].JVirol, 1999, 73(1): 270-280.
[13]HAN J, WANG Y, FAABERG K S. Complete genome analysis of RFLP 184 isolates of porcine reproductive and respiratory syndrome virus[J].VirusRes, 2006, 122(1-2): 175-182.
[14]KARNIYCHUK U U, GELDHOF M, VANHEE M, et al. Pathogenesis and antigenic characterization of a new East European subtype 3 porcine reproductive and respiratory syndrome virus isolate[J].BMCVetRes, 2010, 6: 30.
[15]TIAN K G, YU X L, ZHAO T Z, et al. Emergence of fatal PRRSV variants: unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark[J].PLoSOne, 2007, 2(6): e526.
[16]WANG X, MARTHALER D, ROVIRA A, et al. Emergence of a virulent porcine reproductive and respiratory syndrome virus in vaccinated herds in the United States[J].VirusRes, 2015, 210: 34-41.
[17]ZHOU L, YANG H C. Porcine reproductive and respiratory syndrome in China[J].VirusRes, 2010, 154(1-2): 31-37.
[18]JI G B, LI Y Y, TAN F F, et al. Complete genome sequence of an NADC30-like strain of porcine reproductive and respiratory syndrome virus in China[J].GenomeAnnounc, 2016, 4(2): e00303-16.
[19]ZHOU L, WANG Z C, DING Y P, et al. NADC30-like strain of porcine reproductive and respiratory syndrome virus, China[J].EmergInfectDis, 2015, 21(12): 2256-2257.
[20]LI C, ZHUANG J, WANG J, et al. Outbreak investigation of NADC30-Like PRRSV in South-East China[J].TransboundEmergDis, 2016, 63(5): 474-479.
[21]SUN Z, WANG J, BAI X F, et al. Pathogenicity comparison between highly pathogenic and NADC30-like porcine reproductive and respiratory syndrome virus[J].ArchVirol, 2016, 161(8): 2257-2261.
[22]BROCKMEIER S L, LOVING C L, VORWALD A C, et al. Genomic sequence and virulence comparison of four Type 2 porcine reproductive and respiratory syndrome virus strains[J].VirusRes, 2012, 169(1): 212-221.
[23]ZHAO K, YE C, CHANG X B, et al. Importation and recombination are responsible for the latest emergence of highly pathogenic porcine reproductive and respiratory syndrome virus in China[J].JVirol, 2015, 89(20): 10712-10716.
[24]ZHOU L, ZHANG J L, ZENG J W, et al. The 30-amino-acid deletion in the Nsp2 of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China is not related to its virulence[J].JVirol, 2009, 83(10): 5156-5167.
[25]FANG Y, ROWLAND R R, ROOF M, et al. A full-length cDNA infectious clone of North American type 1 porcine reproductive and respiratory syndrome virus: expression of green fluorescent protein in the Nsp2 region[J].JVirol, 2006, 80(23): 11447-11455.
[26]LI Y, ZHOU L, ZHANG J L, et al. Nsp9 and Nsp10 contribute to the fatal virulence of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China[J].PLoSPathog, 2014, 10(7): e1004216.
[27]杨汉春, 周磊. 猪繁殖与呼吸综合征病毒的遗传变异与演化[J]. 生命科学, 2016, 28(3): 325-336.
YANG H C, ZHOU L. Genetic variation and evolution of porcine reproductive and respiratory syndrome virus[J].ChineseBulletinofLifeSciences, 2016, 28(3): 325-336. (in Chinese)
[28]杨汉春, 周磊. 2016年猪病流行情况与2017年流行趋势及防控对策[J]. 猪业科学, 2017, 34(2): 36-37.
YANG H C, ZHOU L. Swine disease epidemic situation in 2016 and the epidemic trend, prevention and control countermeasures in 2017[J].SwineIndustryScience, 2017, 34(2): 36-37. (in Chinese)
[29]ZHOU L, YANG B N, XU L, et al. Efficacy evaluation of three modified-live virus vaccines against a strain of porcine reproductive and respiratory syndrome virus NADC30-like[J].VetMicrobiol, 2017, 207: 108-116.
[30]BIAN T, SUN Y F, HAO M, et al. A recombinant type 2 porcine reproductive and respiratory syndrome virus between NADC30-like and a MLV-like: genetic characterization and pathogenicity for piglets[J].InfectGenetEvol, 2017, 54: 279-286.

