基于NCBI基因表达综合数据库筛查胃癌关键基因和信号通路的分析
张 鹏, 罗 琴, 汪婷婷, 袁向亮, 沈立松
(上海交通大学医学院附属新华医院检验科,上海 200092)
胃癌是消化道常见的肿瘤之一[1],2015年世界卫生组织发布的《Global Cancer Statistics, 2012》报告指出,胃癌的发病例数高居恶性肿瘤的第4位。根据2015年CHEN等[2]的研究数据显示,2015年我国胃癌新发病例679 100例,死亡病例498 000例。胃癌严重威胁我国人民的健康[3]。
胃癌病因十分复杂,但最终都在不同阶段作用于不同基因,引起相关基因结构及表达水平的改变,这些基因共同作用最终导致胃癌的发生、发展。高通量测序技术及基因芯片技术的快速发展,使基因数据大量累积。美国国立生物技术信息中心(the National Center for Biotechnology Information,NCBI)的基因表达综合数据库(Gene Expression Omnibus,GEO)、美国癌症和肿瘤基因图谱计划(the Cancer Genome Atlas,TCGA)是世界上最大的公共资源基因表达数据库,包含了数以亿计的资源,这种丰富的基因组数据具有很大的潜力,影响未来对疾病的研究模式,癌症基因组学领域的进步正在彻底改变各种癌症的分子表征。2014年《Nature》发文总结了胃腺癌基于基因的亚类分型标准并把其作为TCGA项目的一部分,这些亚型的鉴定为患者分层和靶向治疗提供了指导[4]。这些研究也导致了基于基因的新型胃癌分子分类系统的发展,表明了胃癌发病机制中驱动突变的重要性,并且发现了大量新的驱动基因突变[5]。如何有效地将这一基因组数据为临床和科研提供更好的支持成了一个新的挑战,本研究尝试利用GEO中有关胃癌的数据库,通过生物信息学的方法研究胃癌相关的基因,筛选并预测与胃癌发生、进展、预后相关的基因特征和意义,为肿瘤研究提供新的思路。
1 材料和方法
1.1 研究对象
本研究所有原始数据均从GEO(https://www.ncbi.nlm.nih.gov/)中下载。GEO的纳入标准:具备胃癌组织和正常癌旁对照组织的全基因组测序数据,且数据集覆盖胃癌不同的分期和不同的组织类型。排除对胃癌患者进行药物干预或其他干预的对比测试结果,以及测序数据集包含的样本量过少和没有正常对照的数据集。共采集肿瘤样本1 076份,正常癌旁组织对照样本196份。筛选数据库样本组成为GSE79973、GSE54129、GSE13911,验证数据库样本组成为GSE14210、GSE15459、GSE22377、GSE29272、GSE51105、GSE62254。
1.2 方法
1.2.1 筛选差异基因 从NCBI的GEO下载胃癌相关的芯片数据:GSE79973、GSE54129、GSE13911。使用GEO2R分析平台(https://www.ncbi.nlm.nih.gov/geo/geo2r/),利用R语言程序包limma对表达谱数据进行差异基因提取,导出3个数据集中有差异的基因文件进一步筛选“LogFC>2.0和LogFC<-2.0,且P<0.05的基因”,筛选出的数据集分别命名为GSE79973D1、GSE13911D1、GSE54129D1,以备后续分析所用。
1.2.2 利用韦恩图(VENNY)筛选目标基因 把上述GEO2R分析输出的数据集GSE79973D1、GSE13911D1、GSE54129D1,利用VENNY进一步筛选。本研究选取3个数据两两相交及3个共同表达的基因为研究对象,定义为“至少在2个样本库中表达有差异的基因”,命名为VIG(very important gene),共有基因339个。
1.2.3 基因本体(gene ontology,GO)富集分析 GO是基因功能国际标准分类体系。通过将差异基因做GO富集分析,可以把基因按不同的功能进行归类,达到对基因进行注释和分类的目的。采取的方法是fisher精确检验,数据包采用 clusterProfiler,来自 R/bioconductor。选择标准是落在某个term/GO上差异的基因数目≥4,P<0.05, 按照富集程度的值从大至小降序排列,取前 30 个结果作图。Enrich_factor定义=(某个term中的差异基因数目/总的差异基因数目)/(数据库term中总的基因数目/数据库中总的基因数目)。
1.2.4 京都基因与基因组百科全书(the Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析 KEGG通路分析的信号通路是多个蛋白质之间相互作用,共同调节细胞功能和代谢活动的过程。本研究分析差异基因主要利用公共数据库KEGG来进行分类,对Pathway中的基因进行基于离散分布的显著性分析,得到与实验目的显著相关的Pathway分类。采取的方法是fisher精确检验,数据包采用clusterProfiler,来自 R/bioconductor。选择的标准是落在某个 term/pathway 上差异的基因数目≥4,P<0.05,按照富集程度的值以大小降序排列,取前 30 个结果作图。
1.2.5 生存期分析 利用KM-Plotter数据库(http://kmplot.com/analysis/)验证差异基因的表达和分析关键基因对胃癌患者生存时间的影响。使用数据库中876例胃癌患者信息,根据中位数将目的基因分为低表达组和高表达组,利用缺省设置,在线分析目标基因对胃癌患者总生存的预后价值。
2 结果
2.1 筛选在胃癌组织和癌旁对照组织中表达有差异的基因
本研究首先利用GEO2R分析平台对选取的3个样本集GSE54129、GSE79973、GSE13911的原始数据进行预处理和过滤,筛选出差异基因后进一步筛选出差异的显著性<0.05、差异倍数>2倍的基因。本研究把筛选出的基因合并后得到差异基因1 480个,其中上调基因879个,下调基因601个。取2个以上数据集中有交集的基因共339个(图1),进一步进行生物信息学分析。
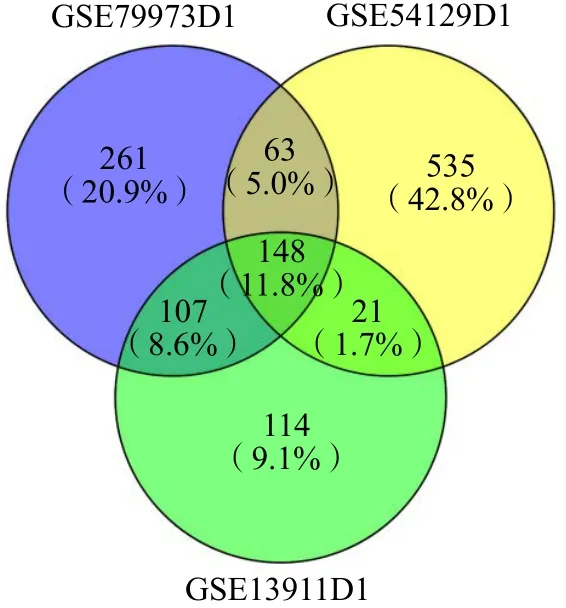
图1 对差异基因进行筛选的VENNY模式图
2.2 胃癌差异基因的生物过程分析
通过GO富集分析显示这些胃癌表达差异基因主要分布在胃、十二指肠、结肠、肌腱、肺、肾等组织。差异基因参与了消化、药物代谢、类黄酮代谢、视黄酸代谢、胶原蛋白分解代谢、酮化合物代谢等1 126个生物过程。其中与消化相关的基因有GKN1、SST、SSTR1等,参与胶原蛋白分解的基因有MMP3、ADAMTS2、COL10A1等,参与多柔比星药物代谢的基因有AKR1B10、AKR1C1、AKR1C2、AKR1C3。富集程度位于前几位的基因主要参与的生物过程是消化、药物代谢、酮类代谢、胶原蛋白代谢。见图2。
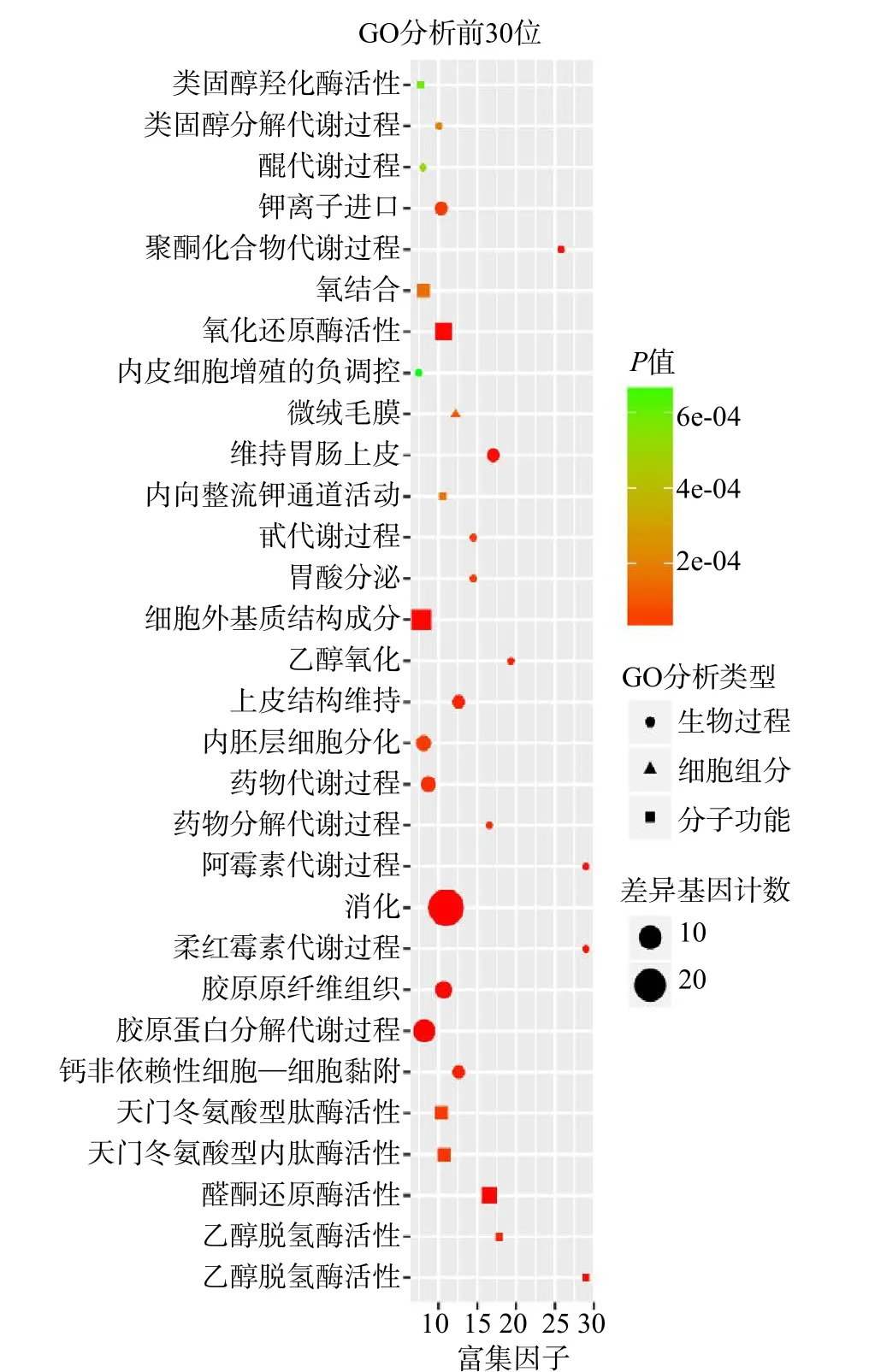
图2 GO生物过程基因富集结果
2.3 胃癌表达差异基因参与的信号通路分析
使用基于KEGG的通路分析发现这些差异基因共涉及信号通路111条,主要的信号通路有:细胞色素P450代谢途径、药物代谢、视黄醇代谢、甾类激素生物合成、酪氨酸代谢、胃酸分泌、血管内皮生长因子受体3信号传导、谷胱甘肽酶、戊糖和葡萄糖醛酸互变等。信号通路的分析与GO功能的分析相吻合,主要集中在消化、药物代谢和类固醇、视黄醇代谢等途径。另外,在通路分析中差异基因在血管内皮生长因子信号通路和细胞外基质受体相互作用中富集也很显著,其中差异基因中富集程度较高的基因主要集中在细胞色素P450家族、葡萄糖醛酸转移酶家族等。见表1。
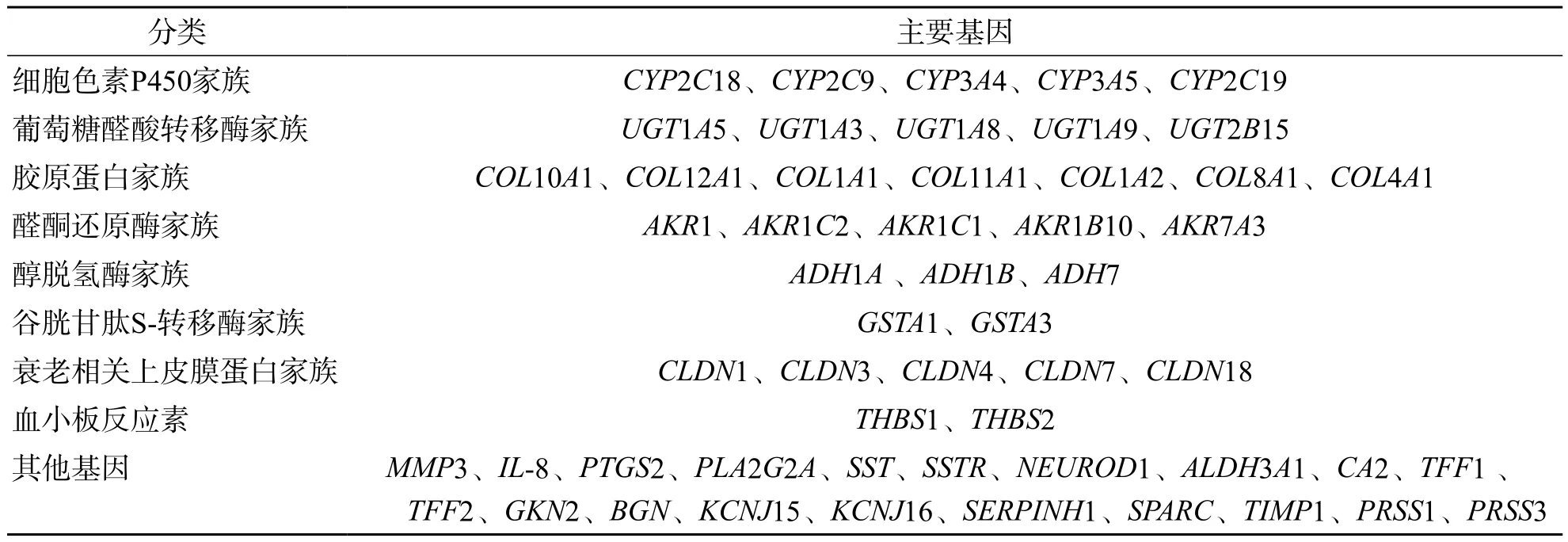
表1 胃癌表达差异基因的信号通路分析
2.4 胃癌关键基因的表达水平及对胃癌患者生存时间的影响
为了验证我们的发现,本研究使用KMPlotter数据库对上述基因在胃癌中的表达水平进行了验证。KM-Plotter数据库包括6个数据集,即GSE29272、GSE51105、GSE14210、GSE15459、GSE22377、GSE62254,共1 051个胃癌样本全基因组测序数据和对应的876例生存时间数据。上述差异基因在KM-Plotter数据库中的表达情况与本研究之前选择的数据集表达一致。本研究对关键基因根据目标基因表达的中位数分为高表达组和低表达组进行生存曲线分析。结果显示细胞色素P450家族2亚科C成员18(cytochrome P450 family 2 subfamily C member 18,CYP2C18)高表达组总生存时间显著高于低表达组(P=0.001 2)。同样,谷胱甘肽S-转移酶家族成员GSTA3、胶原蛋白家族成员COL1A1、醛脱氢酶3家族成员ALDH3A1,高表达组总生存时间均显著降低(图3)。神经元分化因子1(neuronal differentiation 1,NEUROD1)基因在胃癌患者中mRNA的表达水平显著下调,但高表达的胃癌患者生存时间却相对较低(P=0.000 26);前列腺素-内过氧化物合酶2(prostaglandin-endoperoxide synthase 2,PTGS2)基因在胃癌患者中mRNA的表达水平显著上调,但高表达的胃癌患者的生存时间却显著延长(P=0.001 3)。提示这些差异基因在胃癌发生、发展过程中发挥着不同的作用。因此,对筛选出的关键基因进行进一步的功能验证,将会对研究其确切的功能具有重要的意义。
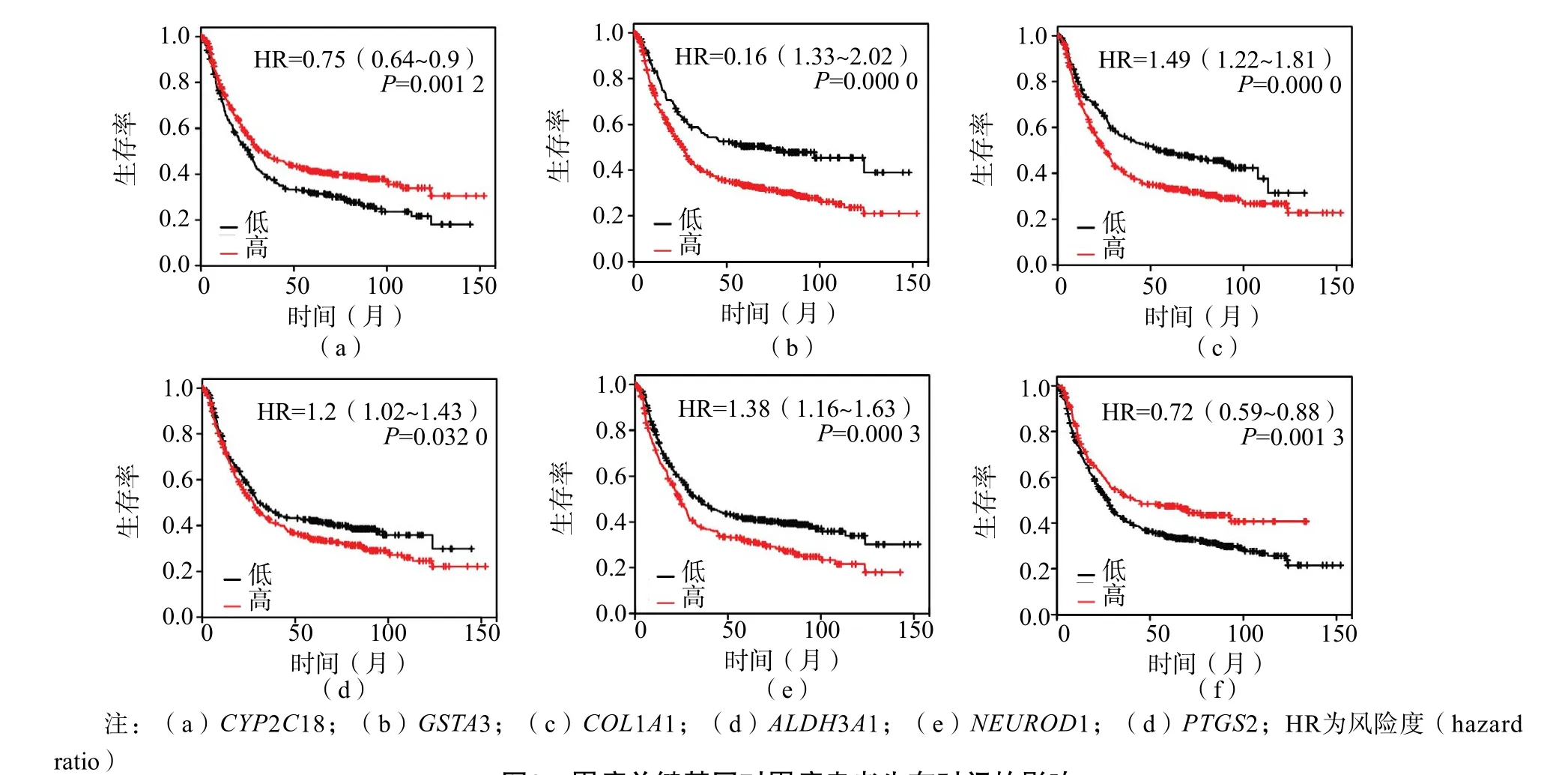
图3 胃癌关键基因对胃癌患者生存时间的影响
3 讨论
本研究利用公共数据资源GEO,使用在线分析平台GEO2R筛选出在胃癌和癌旁组织表达有差异的基因,并对这些差异基因进行了生物过程分析和信号通路分析,发现主要集中于细胞色素P450家族、葡萄糖醛酸转移酶家族等基因簇。对这些关键基因的进一步分析,新发现ALDH3A1、NEUROD1等基因与胃癌的发生、发展密切相关,而且这些关键基因与胃癌生存时间显著相关。
在本研究中,我们鉴定发现的胃癌差异基因包括了细胞色素P450家族和葡萄糖醛酸转移酶家族等基因簇。已有的研究也证实这些基因与胃癌的发生、发展相关。P450家族基因可参与抗肿瘤药物的代谢。环磷酰胺及其异构体异环磷酰胺通过肝脏P450酶催化而活化,提高了此类药物的敏感性[6]。细胞色素P450家族2亚科E成员1 (cytochrome P450 family 2 subfamily E member 1,CYP2E1)基因多态性与胃癌的发生相关[7],其参与胃癌发病的机制可能与其参与亚硝胺及前致癌物N-亚硝基二甲胺和N-亚硝基四吡咯烷的代谢,以及参与黄曲霉属和四氯化碳的活性代谢相关[8]。已有研究表明,COL1A1在胃癌癌变前和恶性组织中的水平显著高于正常组织,并且与肿瘤的进展、大小、淋巴结转移相关[9],而癌变组织中COL1A2的表达水平高于癌前病变和正常组织,因此COL1A1 和COL1A2可以作为胃癌的监测和预后因子。已有研究显示GSTM1和GSTT1基因多态性是胃癌的危险因素[10],GSTM1基因和GSTT1 null基因型的患者癌前病变风险增加,而GSTP1Val等位基因的存在则会减少癌变前损伤的风险[11]。JO等[12]研究认为UGT1A1基因的表达与晚期胃癌患者的治疗相关。WANG等[13]的研究提示UGT1A1多态性可以用来筛选胃癌的风险人群,TYMS、TUBB3和STMN1或可作为预后的潜在生物标志物用于晚期胃癌的化疗指导。这些已有的差异基因研究报道进一步证实本研究的分析筛选模型是有效的,在寻找胃癌相关的基因和蛋白上具有良好的效果。
通过深入检索本研究筛选的这些差异基因,我们发现了一些处于网络核心节点的关键差异基因如ALDH3A1、NEUROD1等,但尚未见其对胃癌的意义的相关报道。ALDH3A1是乙醛脱氢酶3家族成员,PATEL等[14]曾经报道ALDH3A1在肺癌中升高,国内袁青等[15]报道了乙醛脱氢酶2(aldehyde dehydrogenase 2 family,ALDH2)基因多态性及生活习惯与胃癌易感性的相关性分析,认为ALDH2基因多态性与胃癌易感性有关。ALDH3A1在胃癌中意义尚未见报道,本研究分析发现高表达此基因的胃癌患者生存期显著缩短(P=0.032),推测与胃癌进展相关。NEUROD1是转录因子NeuroD家族的成员,已有报道其多与乳腺癌、神经内分泌癌、前列腺癌、糖尿病有关[16-17],尚未发现在胃癌中的研究报道。本研究通过生存时间分析发现,NEUROD1高表达胃癌患者的生存时间显著缩短,说明该基因的异常表达对胃癌患者是有意义的。进一步蛋白交互作用分析证实该基因可能经由胰岛素与AKT1基因有交互作用,推测NEUROD1可能经由胰岛素参与了丝氨酸/苏氨酸激酶介导的血管内皮生长因子信号途径或是通过GPCR信号传导和MAP3K5的磷酸化参与了细胞的凋亡等过程。丝氨酸/苏氨酸激酶1是PI3K/AKT信号通路的重要组成部分。该通路可调节肿瘤细胞的增殖和恶性转化,而且与肿瘤细胞的迁移、黏附、肿瘤血管的生成以及细胞外基质的降解相关[16]。在本研究鉴定发现的差异基因中还有一些基因如PTGS2等在胃癌的发生、发展中可能发挥不同的作用。与PTGS2相关的疾病包括结肠直肠腺瘤和消化性溃疡。在癌细胞中,PTGS2是前列腺素E2生产中的关键步骤的产物。有研究显示,PTGS2等位基因携带者罹患胃癌的风险增加[17]。本研究结果也显示,胃癌患者PTGS2 mRNA表达水平显著上调,这提示该基因与胃癌的发生相关。但生存时间分析发现PTGS2高水平表达的胃癌患者,其生存时间显著延长,这提示该基因在胃癌的进展中可能发挥保护性因子的作用。因此,对PTGS2等基因在胃癌中的表达特征和意义需进一步研究,以明确其在胃癌发生、发展中的功能特征和作用。
本研究建立的筛选分析模型为研究肿瘤提供了一个新的思路:通过公共数据库GEO和TCGA相关肿瘤的全基因组测序数据,对在癌组织和对照组织中表达有差异的基因进行全面分析,找出与肿瘤相关的核心基因,对这些关键基因参与的生物过程和信号通路进行分析,对在主要生物过程和通路中参与的基因进行蛋白交互作用分析,然后对处于网络中心节点的基因进行文献挖掘,寻找研究的突破点,继而用基因突变分析工具和生存时间分析工具来验证该基因对肿瘤患者总生存时间的影响,综合分析某基因在肿瘤中的表达特征和意义,可为进一步开展相关的功能研究提供理论支持和指导。
[1] SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016, 66(1):7-30.
[2] CHEN W, ZHENG R, BAADE P D, et al.Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2):115-132.
[3] PATRU C L, SURLIN V, GEORGESCU I, et al.Current issues in gastric cancer epidemiology[J]. Rev Med Chir Soc Med Nat Iasi, 2013,117(1):199-204.
[4] Cancer Genome Atlas Research Network.Comprehensive molecular characterization of gastric adenocarcinoma[J]. Nature, 2014, 513(7517):202-209.
[5] KATONA B W, RUSTGI A K. Gastric cancer genomics: advances and future directions[J]. Cell Mol Gastroenterol Hepatol, 2017, 3(2):211-217.
[6] WEBER G F, WAXMAN D J. Activation of the anti-cancer drug ifosphamide by rat liver microsomal P450 enzymes[J]. Biochem Pharmacol, 1993, 45(8):1685-1694.
[7] GHOSHAL U, TRIPATHI S, KUMAR S, et al.Genetic polymorphism of cytochrome P450 (CYP)1A1, CYP1A2, and CYP2E1 genes modulate susceptibility to gastric cancer in patients with Helicobacter pylori infection[J]. Gastric Cancer,2014, 17(2):226-234.
[8] ZHANG M X, LIU K, WANG F G, et al.Association between CYP2E1 polymorphisms and risk of gastric cancer: an updated meta-analysis of 32 case-control studies[J]. Mol Clin Oncol, 2016,4(6):1031-1038.
[9] LI J, DING Y, LI A. Identification of COL1A1 and COL1A2 as candidate prognostic factors in gastric cancer[J]. World J Surg Oncol, 2016, 14(1):297.
[10] HIDAKA A, SASAZUKI S, MATSUO K,et al. CYP1A1, GSTM1 and GSTT1 genetic polymorphisms and gastric cancer risk among Japanese: a nested case-control study within a largescale population-based prospective study[J]. Int J Cancer, 2016, 139(4):759-768.
[11] NEGOVAN A, IANCU M, MOLDOVAN V,et al. The interaction between GSTT1, GSTM1,and GSTP1 Ile105Val gene polymorphisms and environmental risk factors in premalignant gastric lesions risk[J]. Biomed Res Int, 2017, 2017:7365080.
[12] JO J C, LEE J L, RYU M H, et al. Phase Ⅱ and UGT1A1 genotype study of irinotecan dose escalation as salvage therapy for advanced gastric cancer[J]. Br J Cancer, 2012, 106(10):1591-1597.
[13] WANG W, HUANG J, TAO Y, et al. PhaseⅡand UGT1A1 polymorphism study of two different irinotecan dosages combined with cisplatin as first-line therapy for advanced gastric cancer[J].Chemotherapy, 2016, 61(4):197-203.
[14] PATEL M, LU L, ZANDER D S, et al.ALDH1A1 and ALDH3A1 expression in lung cancers: correlation with histologic type and potential precursors[J]. Lung Cancer, 2008, 59(3):340-349.
[15] 袁青, 薛亚东, 郑雅萍,等. 乙醛脱氢酶2基因多态性及生活习惯与胃癌易感性的相关性分析[J]. 检验医学, 2016, 31(7):584-587.
[16] SHIMIZU T, TOLCHER A W, PAPADOPOULOS K P,et al. The clinical effect of the dual-targeting strategy involving PI3K/AKT/mTOR and RAS/MEK/ERK pathways in patients with advanced cancer[J].Clin Cancer Res, 2012, 18(8):2316-2325.
[17] LI Y, HE W, LIU T, et al. A new cyclooxygenase-2 gene variant in the Han Chinese population is associated with an increased risk of gastric carcinoma[J]. Mol Diagn Ther, 2010, 14(6):351-355.

