深度学习在数字病理中的应用
闫 雯 汤 烨 张益肇 来茂德 许 燕,*
1(北京航空航天大学生物与医学工程系,北京航空航天大学软件开发环境国家重点实验室,深圳北航新兴产业技术研究院,生物医学工程高精尖创新中心) 2(微软亚洲研究院,北京 100080) 3(浙江大学医学院基础医学院,杭州 310058)
引言
病理切片作为病理诊断的金标准,在临床和科研中都有着十分重要的应用。病理医生通过对病理切片进行镜检,完成病理诊断和预后评估,但是这个过程通常费时费力。病理切片的数字化被认为是病理学发展过程中的重要转折点[1]。数字病理切片的制作首先需要经过组织染色,随后通过显微相机数字化为数字病理切片。但是,由于最初数字切片质量不高,其普及程度受到一定限制。1999年,全片数字化图像(whole slide images, WSI)[1]出现,使病理切片的保存和传输更加方便安全。
随着人工智能技术走进病理分析领域,病理分析不再局限于传统的定性分析,逐渐向定量分析过渡[2-3]。定性分析是对切片性质特点进行概括,并没有形成量化指标,因此定性分析的结果不可复现,且受主观因素影响较大。定量分析是指依据统计数据,建立数学模型,从而计算出与病变相关的各项指标,如有丝分裂数目、肿瘤的实质与间质的比例、黏液湖和癌细胞的比例等定量化指标;并根据定量指标给出病理诊断,其诊断结果更加客观。因此,病理医生也越来越认识到定量分析的重要性。
传统机器学习算法和深度学习算法都可用于定量分析[4]。传统的机器学习算法依赖于人工设计特征表达,即提取图像的形状、大小和纹理等特征并做特征挑选,剔除冗余特征得到最优特征集。但是,这种人工特征的选取依赖于大量专业知识,且难以涵盖图像的全面特征,导致其使用上的局限性。
深度学习预先定义了计算规则,通过层级式网络结构,将数据从输入层传递到输出层,并自动学习图像特征表达,得到图像的低维特征。相比其他机器学习算法,深度学习算法对大数据样本的特征提取能力更强。基于临床上不断积累的WSI数据,深度病理能充分发挥在大数据样本上的优势,推动病理定量分析的发展,辅助医生完成病理诊断。
因此,基于深度学习的病理切片图像的定量分析研究,既是数字病理分析的大势所趋,也是学术界和医学界共同努力的方向。
1 深度学习的基本概念
深度学习是机器学习中一种包含多层隐层的人工神经网络模型,其通过组合低层特征学习到数据的高层特征表达[5]。
在通常情况下,更深的网络深度意味着网络具有更强的特征提取能力。但是,网络深度的加深往往会导致模型难以收敛、计算量巨大等问题。为了克服上述问题,需要一些更强大的技术的辅助,如卷积、pooling、dropout、ReLU函数以及使用GPU训练网络等。
在图像处理与计算机视觉领域,以卷积、pooling和ReLU函数为基础构成的卷积网络(ConvNet)是深度学习最常用的网络结构之一,图1为两层ConvNet构成的CNN。
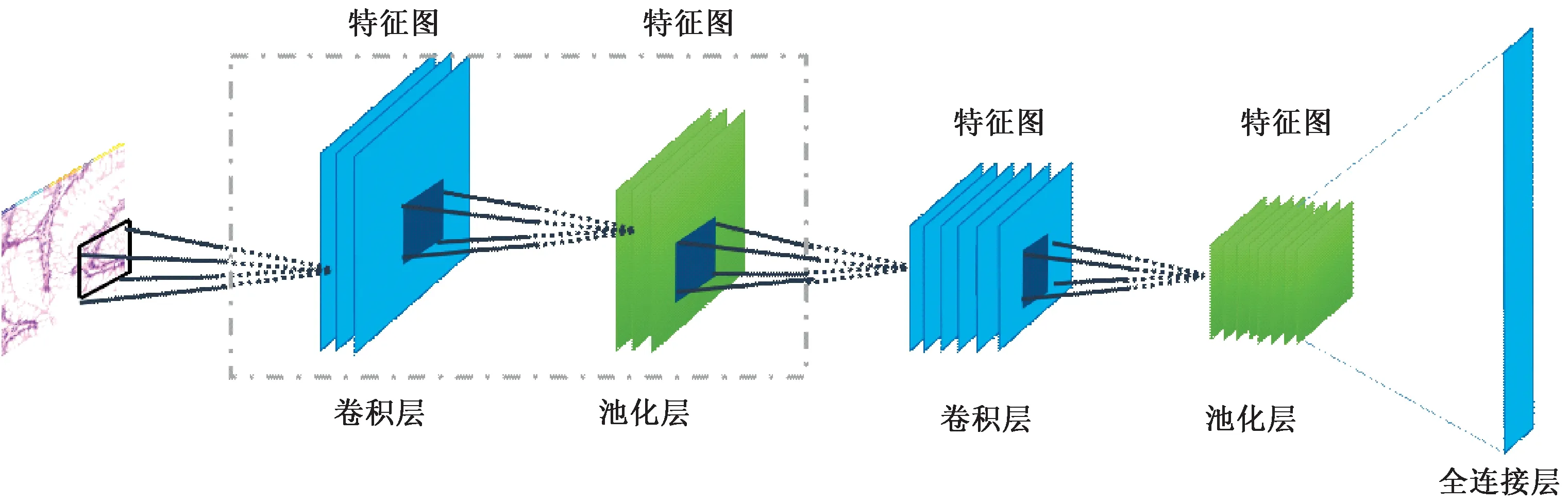
图1 两层卷积神经网络(由卷积层和池化层以及全连接层构成)Fig.1 Two layer convolution neural network, which is constituted by the convolutional layer, the pool layer and the fully connected layer
CNN将图像直接作为网络的输入,避免了传统识别算法中复杂的人工特征设计过程。这种网络结构对平移、比例缩放、倾斜或者其他形式的变形具有高度不变性,在计算机视觉中有广泛的应用。
1.1 卷积神经网络
卷积神经网络(CNN)引入权值共享和局部视野域的概念,大大减少了权值的数量。
1)权值共享。每个隐藏神经元具有同样的偏置和相同的连接到局部感受野的权重。每个隐藏神经元可以学习一种特定的特征映射,最后通过多层堆叠的方式,学习到图片的整体特征表达。
对于l层的第j、k个隐藏神经元,输出为
alj=σ(wljk·a(l-1)k+blj)
(1)
式中,σ是神经元的激活函数,b是偏置的共享值,w是共享权重的数组,a是激活值。
卷积神经网络能够自动学习到合适的权重和偏置,以至于网络的输出y(x)能够拟合所有的训练输入x。为了量化输入x与输出y(x)之间的差距,引入代价函数的概念,有
(2)
式中,w表示网络中所有的权重的矩阵,b是所有的偏置的矩阵,n是训练输入数据的个数,a表示输入为x是输出的激活值,求和则是在所有的输入数据x上进行的。
网络训练的过程其实就是找到代价函数最小的时候对应的权重的值。直接计算代价函数的最小值得计算量十分巨大,1986年,Rumelhart提出反向传播算法[6],大大减少了最小化代价函数时的计算量,为加深网络层数提供了可能性。
2)ReLU函数。该函数是神经网络中最常用的激活函数之一,通常与卷积层同时出现,其公式为
f(x)=max(0,x)
(3)
式中:当输入x<0时,输出为0;当输入x>0时,输出x。
ReLU函数的优点在于梯度不饱和,在反向传播过程中,减轻了梯度弥散的问题,神经网络前几层的参数也可以很快更新;在正向传播过程中,计算速度更快。
3)混合层。除了上面提到的卷积层,卷积神经网络还包含混合层(pooling layer)。混合层通常紧接着卷积层之后使用,其作用是对卷积层的输出信息进行降采样,减少参数的个数,避免过拟合。详细地说,混合层将输出的每个特征映射凝缩成一个新的特征映射,常见的混合层有最大值混合(max-pooling)和均值混合(average-pooling)。以max-pooling层为例,该层提取一定大小窗口内特征图的最大值作为新的特征值输入到下一层,如图2所示。

图2 最大池化层(窗口大小为2×2,依次移动窗口并取该窗口内特征图的最大值,组成下一层的特征图)Fig.2 Gram of the max-pooling layer, the size of sample window is 2x2. The window take the maximum value of feature map within the window, to compose the feature map of next layer
1.2 深度神经网络
2012年,Krizhevsky提出深度卷积网络AlexNet[7],在数据集ImageNet[8]上实现了图像分类任务并将错误率降低到17.0%。在此基础上,Zisserman提出了一种新的深度卷积网络结构——VGG-Net[9]。该网络采用更小的卷积核以及更深的网络以增强网络性能,在ImageNet数据集上将分类错误率降到了7.32%。同年,Szegedy提出了一种带有Inception层CNN结构——GoogleNet,它增大了网络的深度和宽度,在Image Net数据集上测试的错误率为6.67%[10]。
综上所述,卷积神经网络是图像处理领域的研究热点,这种网络结构既具备自动特征的学习能力,也具有强大的分类能力,可同时解决特征提取和分类器设计两方面的问题。在此基础之上,通过设计特定的网络结构,深度学习可以解决多种计算机视觉问题,如图像分类、图像分割、实施目标检测、边缘提取等。
2 深度学习在数字病理切片分析中的应用
计算机辅助算法在病理切片分析中应用十分广泛,可分为传统机器学习算法和深度学习算法[11]。
WSI病理图像包含的信息量很大,包含了大量复杂、冗余的信息。因此,将图片转化为可挖掘的特征数据,是病理分析必要的步骤。特征提取的方法可分为人工特征设计和自动特征学习两种。
人工特征设计是指从图像中挑选并简化出最能有效表达图像内容的低维矢量,这些特征包括灰度直方图、形状特征、纹理结构特征、与周围组织的关系特征等。基于传统机器学习算法的数字病理切片分析需要人工提取特征。但是,人工特征的应用有以下缺点[12]:一是特征选取在很大程度上依靠专业经验,客观性较差,无法表征图片的全面信息;二是缺乏将多种人工特征融合的原则性准则。自动特征学习是指从大量训练数据中学习图像的尺度不变性、形变不敏感的特征表达,能更加全面地表达图片特征,且不受专业因素的限制[13]。深度学习算法既能够学习到数据的特征表达,又可以作为分类器使用,这样的“端对端”结构具有更强的自学习能力和自适应能力,更适合于大数据量的病理分析。
2013年,Ciresan首次证明深度学习算法能够运用在病理切片有丝分裂的检测工作中,并取得巨大成功[14]。愈来愈多的人意识到,深度神经网络在病理切片分析中拥有传统机器学习算法无法企及的巨大潜力。目前,深度学习在病理切片自动分析算法中的应用可以按照解决任务的不同分为3个方面:细胞或组织的分割和检测、组织层面上的分类和癌症分级以及其他应用。
2.1 细胞和组织的检测与分割
病理切片中的腺体形态、有丝分裂数目、细胞核大小等结构都可以作为癌症诊断的指标。针对某些疾病的病理诊断需求,从整张病理切片中分离出上述细胞或组织结构是数字病理分析的基础[15]。下面将分别介绍深度学习在病理切片方面的应用情况,包括细胞和细胞核的检测和分割、腺体的分割、有丝分裂的检测。
2.1.1细胞和细胞核的检测与分割
细胞和细胞核的形态学特征[16-17]、几何学特征[18]、纹理特征[19]、形状特征[20]等是病理分析的重要指标,因此分离出细胞等结构对于病理诊断具有重要意义。病理切片的厚度、杂质、切割均匀度、染色后颜色的深度以及数字图像噪声等因素使切片图像存在异质性[21](见图3),给细胞的分割和检测带来了困难。

图3 细胞核的检测中的不利因素[15]。(a)模糊;(b)重叠;(c)异质性Fig.3 Negative factors in nuclear examination. (a)Blurring;(b)Overlap; (c) Heterogeneity,source
有研究表明,将人工特征与CNN提取的特征相结合,可以有效降低人工特征的敏感性,并减少CNN的计算量[12]。Wang等将人工设计的特征与CNN自动提取的特征相融合,用于乳腺病理切片中细胞核的检测[22],相比当时的其他算法,该算法计算量较小,更符合临床需求。但是,随着计算机性能的增强,在数据集充足的情况下,深度学习也可以直接从大量数据中学习特征表达,并结合一定的预处理和后期处理方法,高效而准确地检测细胞。Xing等和Chen等使用CNN提取特征,并生成切片细胞核位置的概率谱[23-24]。根据所处理切片的特点,Xing等在后期处理中选择迭代区域合并的方法分割独立的细胞核,该算法适用于多种组织的多种染色剂图像。Chen等在预处理和后期处理中分别使用色彩通道的分离和非极大值抑制算法,提取免疫细胞(IHC)的不同色彩通道的特征,用于代表不同细胞结构的特点,计数的结果与人工计数基本持平。
细胞的分割通常比细胞核的分割更加复杂,细胞质的形状不规则以及细胞团的重叠现象都给细胞的分割造成了很大的困难。通过设计网络结构,以上问题可以得到解决。Song等将多尺度卷积网络(MSCN)提取的特征与人工特征相结合,用于宫颈细胞质和细胞核的分割[25]。在此基础上,Song等增加了训练数据量并改进了网络结构,直接使用MSCN学习单个细胞的高级形态特征来分割重叠细胞团[26]。Akram等使用两层级联网络,第一级的卷积网络粗略预测哪些区域包含细胞,第二层池化网络用来准确定位细胞边界[27]。
有监督算法需要大量标记数据集来训练模型,在标记数据集不充足的情况下,可以使用海量的未标记数据集来弥补无监督算法的缺点。Ronneberger等提出了一种网络的训练策略,使用配准方法扩充有限的标记数据的数量,使用多特征通道的上采样层传递上下文信息,帮助提高分割准确率[28]。另外,弱监督也可以弥补标记数据量不足的问题。所谓弱监督,就是对数据做简单的标记,其对专业知识的要求较低,容易获取。Xie等使用弱监督的回归模型检测细胞,适用于尺寸和形状变化较大、有细胞重叠图像的细胞检测[29]。
2.1.2腺体的分割
在临床上,腺体的形态学特征,包括其外部结构和腺体的形成过程,都常作为腺癌诊断的依据,因此,腺体的分割在癌症诊断中有重要意义[30],结肠腺体的病理图像如图4所示。

图4 结肠组织切片的腺体检测[34]Fig.4 Detection of glands in colon tissue[34]
腺体分割任务的挑战性在于:一是腺体形状不规则,染色不均匀、腺体重叠等因素导致其形态特征难以用数学模型描述;二是不同级别的癌症组织中腺体形状特征不同;三是腺体个体分割难以实现。
类似地,CNN在腺体分割中的广泛应用从2015年的腺体分割大赛中可见一斑。在大赛中,主办方提供了结肠腺体的数据集。多个团队采用CNN 网络对腺体候选样本做有无腺体的分类[30],例如:CVML团队使用CNN训练像素级别的分类器;Freiburg团队使用一种端对端的U型深卷积网络;ExB团队使用双路径的CNN网络,分别以不同大小的样本作为输入,用于更好地捕捉多尺度的上下文信息;vision4GlaS使用两个CNN[31],分别用于候选样本和重叠样本的分类。
随后在分类基础上,各团队使用分割算法分割腺体个体:CVML团队以及 vision4GlaS团队使用基轮廓的传统方法分割腺体;ExB额外训练了一个神经网络用于检测腺体轮廓,并结合传统算法分割腺体。不同于以上方法,也有的团队直接使用深度神经网络完成任务。Freiburg团队在CNN网络的基础上,添加由上卷积层和卷积层构成的聚合通路,将每一级的细节特征反向传播到聚合通路,用于捕捉腺体边界;BenTaieb等设计了具有两种损失函数的多任务全卷积神经网络[32],先对候选样本做有无腺体的分类,再对包含腺体的样本分割,分类和分割结果彼此优化;Chen等提出了深轮廓感知网络(DCAN)[33],使用基于多水平特征表达的全卷积神经网络(FCN)分割腺体;Xu等将全卷积神经网络(FCN)与整体边缘探测器(HED)相结合[34],克服了FCN无法识别腺体个体的缺点,实现了腺体个体分割。
2.1.3有丝分裂的检测
在乳腺癌分级中,有丝分裂数是一个重要的参数,有丝分裂的检测可看作是一种特殊的细胞检测。有丝分裂在各个时期形态各异,有着前期、中期、后期和末期4个时期的4种形态,这使得有丝分裂检测更具有挑战性。有丝分裂检测如图5所示。

图5 乳腺组织切片的有丝分裂检测Fig.5 Mitosis detection of Breast histological sections
ICPR数据集[35]是由病理学家手动标记的乳腺病理切片数据集,在有丝分裂检测中被广泛使用。Ciresan使用滑动窗在数据集中截取大量小样本,再用CNN分类模型对样本做有无有丝分裂的二分类,以检测有丝分裂数目[14]。Wang等和Malon等使用人工设计特征,提取细胞核的颜色、形状、纹理等特征,再与CNN自动提取的特征相结合,降低了算法对人工特征的敏感度,在一定程度上降低了有丝分裂多样性给有丝分裂检测带来的问题[22,36]。Chen等使用两级深度卷积级联神经网络,第一级大体定位有丝分裂对象,第二级网络将真正的有丝分裂像从其类似物中识别出来[37]。该算法的第二级网络使用迁移学习的方法,将跨领域学习的特征迁移到病理切片中,解决了训练数据不足的问题。随后,Chen等又提出一种“端对端”的回归网络(DRN)用于有丝分裂检测[38],同样使用了迁移学习的方法。
综上所述,深度学习在细胞、腺体和有丝分裂的病理切片分割和检测等方面的研究日趋成熟。相比人工特征,深度学习的特征表达更全面;相比传统算法,深度学习对于图片噪声的抗干扰能力强,其结果与人工分析的结果大致相同。另外,值得注意的是,有监督训练比无监督训练的性能更好,但是其训练需要大量的标记数据集,而这种数据集的获取花费较大。目前,国际上有许多病理切片算法大赛,并且提供了许多公开的标记数据集,在病例分析算法的研究中起到了重要作用,相关赛事以及数据集如表1所示。
2.2 组织层面上癌症的分类和分级
相比利用病理组织基本单元的特征(如细胞和细胞核、有丝分裂等)来分析病理切片,另一类广泛应用的方法是使用人工智能的算法(如深度学习)学习组织水平上的特征,并对病理切片进行有癌无癌的分类和癌症分级,相关文献见表2。

表1 病理切片大型公开数据集一览表Tab.1 List of pathology pubic dataset

表2 文献及其分析算法Tab.2 List of literature and analysis algorithms
其中,病理学分类(分型)反映了组织细胞的生物行为和形态学特征,病理分级反映了在组织结构和细胞形态上肿瘤组织与正常组织细胞不同程度的形态差异,可用来判断组织是否癌变。下面将介绍深度学习在几种不同组织病理切片的分类和分级任务中的应用情况。
2.2.1乳腺组织切片
Camelyon16数据集是目前最大的乳腺WSI数据集(见表1),包含400张WSI图像,如图6所示。在乳腺前哨淋巴转移瘤的检测中,Wang等和Kovalev等都训练CNN网络得到WSI切片的病变概率图谱,再经过后期处理检测癌变区域[48-49]。
Bayramoglu等和Spanhol等分别使用BreaKHis数据集训练神经网络(该数据集其包含82个病人数据)用于乳腺癌的检测[50-51]。其中,Bayramoglu等训练了两个CNN,用于多尺度地区分肿瘤的良性与恶性[50];Spanhol等选用Alexnet,对乳腺癌切片进行分类[51]。Zejmo等在某医院提供的数据集上,训练GoogLeNet用于乳腺癌的分类[52]。
浸润性导管癌(IDC)是最常见的所有乳腺癌的亚型,如图7所示。2015年,Cruz-Roa等训练了一个3层CNN的二分类模型,对乳腺切片做有癌无癌的检测,结果比使用人工特征的算法高6%[53]。2016年,Shah等为了检测乳腺癌肿瘤扩散情况,使用了多种卷积神经网络(DenseNet、GoogLeNet、LocNet等)进行肿瘤定位、有丝分裂识别以及特征提取,完成肿瘤3个等级的分类[54]。

图7 病理学家标记的浸润性导管癌组织切片(IDC)[53],标记区域为IDC正例,其余为IDC负例Fig.7 WSI with manual annotations from pathologists of invasive ductal carcinoma[53],annotated region correspond to positive example, the other is negative one.
在乳腺癌的研究中,免疫细胞浸润是一个新兴的预后生物学标志。2016年Turkki等以20位乳腺癌病人的病理切片为训练集,训练CNN进行特征提取,而后用SVM检测免疫细胞分布情况,用于乳腺癌诊断[55]。
2.2.2皮肤组织切片
深度学习同样也被应用在了皮肤肿瘤的诊断上。恶性黑色素肿瘤是最常见的皮肤肿瘤之一,但是该类病理图像有标记的数据集较少。2013年,Cruzroa使用深度学习来区分癌症组织和正常组织,并通过数字染色来突出显示待诊断区域,其结果比当时最好的算法提高了7%[56]。Kashif等使用空间约束CNN提取空间特征,该网络可以检测癌变细胞进而分析癌变特征[57]。Masood等提出的使用半监督深度置信网络[58]自监督学习模型,充分利用有限的标记数据集,学习病变特征[59],为解决皮肤病理切片数据问题提供了较好思路。
2.2.3脑组织切片
由于脑组织切片的送检样本通常较小且质地疏松,所以脑组织切片往往难以制成优质的病理切片,导致非专科病理医生诊断困难,该类切片的分析一直是一个比较大的挑战。
根据MICCAI提供的癌症和肿瘤基因图谱(TCGA)标准数据集(见图8),Xu等提出了一种基于迁移学习的CNN,用于脑肿瘤的分割和分类[60]。2015年,Ertosun等将深度学习应用于脑瘤亚型的分级上,第一个8层神经网络将肿瘤区分为第2、3级和第4级两类,第二个19层神经网络进而区分出胶质瘤的2、3级[61]。2016年,Hou等使用多标签的CNN分类模型识别神经胶质瘤细胞的多种形态特征,该方法能识别出常见的胶质瘤细胞核的9个细微的、重要的病理学特征,在癌症诊断中具有良好的准确性[62]。

图8 大脑病理切片[60]。(a)多形性恶性胶质瘤,(b)低级神经胶质瘤Fig.8 Pathology slice of brain tissue[60]. (a) glioblastoma multiforme; (b) Low grade glioma
2.2.4前列腺组织切片
在前列腺癌的分类分级中, Gleason评分系统根据前列腺组织的细胞、腺体等结构的形态特征,将前列腺癌分为5个等级,等级越高恶性程度越高,预后也就越不理想[63]。正确的分级能够帮助医生采取相应的治疗方式,有着重要的意义。
2016年,Litjens等使用基于区块(patch)的CNN计算各区域癌变的概率图谱,再使用基于概率图谱中可能存在病变的区域重新训练神经网络,以便更精确地识别前列前病变[64]。Källén等使用22层神经网络训练模型,得到能够辅助病理医生诊断的Gleason分级模型[65]。该模型能够对Gleason3~5级组织与良性组织进行分类,并根据病理学家的意见调整算法参数,用于辅助病理诊断。
2.3 其他应用
在结肠组织切片中,Sirinukunwattana等使用了空间约束CNN来识别切片中的细胞,再使用周边集成预测的方法对各类结肠细胞进行分类,并判断该细胞是否癌变,从而识别切片中的癌变区域[66]。Xu等使用深度学习自动学习切片的特征,分别使用多实例学习(MIL)和SVM作为分类器,结果表明弱监督(MIL)分类效果更好[67]。
免疫组织化学染色(IHC)是一种广泛使用的技术,用于诊断异常免疫细胞的密度和位置等分布情况,可进一步用于癌症预后预测。由于数据集的限制,目前IHC病理切片的相关研究较少。由于IHC图像的RGB通道对应不同的结构,Chen等将RGB通道分离并选择代表膜标记的通道,利用深度学习检测该通道的免疫细胞分布情况,该结果大体与人工分析结果持平[68]。
由于制作病理切片时相机的差异和染色过程存在许多变量(例如,试样的厚度、染色剂的浓度、制造商、时间和温度的污渍),因此都可能影响最终组织标本的外观。病理医生可以根据自身的经验来加以甄别,而人工智能算法却难以克服这种多样性带来的影响。为了克服图像的多样性带来的影响,Janowczyk等采用稀疏自动编码器(SAE),通过迭代学习生成过滤器来优化重建图像,从而改变目标图像以匹配到模板图像的颜色空间[69]。Sethi等在上皮细胞基质分类中验证了颜色标准化,确实可以为分类带来一定的增益[70]。更具体来说,对于基于超像素分类的算法有较低的增量效益,而对于CNN的分类算法有较高的增益。
3 展望
在本研究中,主要阐释了深度学习理论在病理切片定量分析中的应用情况,如细胞层面上的检测分割、组织层面上的分类与癌症的分级等。CNN针对大数据样本强大的自动特征学习的能力,适用于临床上大量积累的WSI切片的定量分析;同时,CNN在特征提取过程中不需要研究人员具备一定的专业知识,让病理方向的研究对计算机等领域的研究人员开放,有利于病理分析领域的发展。
但是,深度学习在病理切片分析领域的应用仍存在一定的局限性:
1)运算开销大,卷积神经网络需要基于GPU实现,但由于GPU存储容量的限制,WSI切片图像的尺寸过大,无法直接作为输入,神经网络对WSI采样则会损失部分有用信息。
2)目前深度学习广泛应用的卷积神经网络是有监督学习,需要细致地标注图像中癌症组织的位置,而标注数据的获得成本较高。
3)虽然现今的人工智能在病理切片自动分析的研究中取得了一定的进展,但是并没有很好地应用于临床中。结合“云病理平台”等前沿科技,促进科研与临床实践相结合的研究。
4)癌症的分级情况与预后有着十分密切的关系,目前的癌症分期系统对个性化癌症治疗和预后的预测仍不够精准。深度学习有望在预后中取得突破性进展,但是该应用还处于起步阶段,有待加强。
目前,人工智能技术在病理学中应用广泛,定量分析为病理诊断提供了一系列定量化指标,弥补了医生主观经验中的不可预测因素,提高了病情诊断的准确性。在人工智能算法的帮助下,病理切片的计算机辅助诊断的知识体系将不断完善,最终能够为病人提供预后信息和精准的药物治疗指导。
[1] Weinstein RS, Graham AR, Richter LC, et al. Overview of telepathology, virtual microscopy, and whole slide imaging: prospects for the future [J]. Human Pathology, 2009, 40(8):1057-1069.
[2] Kothari S, Phan JH, Stokes TH, et al. Pathology imaging informatics for quantitative analysis of whole-slide images [J]. Journal of the American Medical Informatics Association, 2013, 20(6):1099-1108.
[3] Webster JD, Dunstan RW. Whole-slide imaging and automated image analysis: considerations and opportunities in the practice of pathology[J]. Veterinary Pathology, 2014, 51(1):211-223.
[4] Madabhushi A, Lee G. Image analysis and machine learning in digital pathology: Challenges and opportunities [J]. Medical Image Analysis, 2016, 33:170-175.
[5] Lecun Y, Bengio Y, Hinton G. Deep learning [J]. Nature, 2015, 521(7553):436-644.
[6] Rumelhart DE, Hinton GE, Williams RJ. Learning internal representations by error propagation [M] //Neurocomputing: Foundations of Research. Cambridge:MIT Press, 1988:318-362.
[7] Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks [C]// International Conference on Neural Information Processing Systems. Nevada: Curran Associates Inc, 2012:1097-1105.
[8] Jia Deng, Wei Dong, Socher R, et al. Imagenet: A large-scale hierarchical image database [C] // IEEE Conference on Computer Vision and Pattern Recognition. Miami: IEEE, 2009:248-255.
[9] Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition [J]. Computer Science, 2014.
[10] Szegedy C, Liu Wei, Jia Yangqing, et al. Going deeper with convolutions [C] //IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015:1-9.
[11] Treanor D. Virtual slides: An introduction [J]. Diagnostic Histopathology, 2009, 15(2):99-103.
[12] Malon CD, Eric C. Classification of mitotic figures with convolutional neural networks and seeded blob features [J]. Journal of Pathology Informatics. 2013, 4(1):9-18.
[13] Janowczyk A, Madabhushi A. Deep learning for digital pathology image analysis: A comprehensive tutorial with selected use cases [J]. Journal of Pathology Informatics, 2016, 7:29-38.
[15] Gurcan MN, Boucheron LE, Can A, et al. Histopathological image analysis: A review [J]. IEEE Reviews in Biomedical Engineering, 2009, 2:147-171.
[16] Nedzved A, Ablameyko S, Pitas I. Morphological segmentation of histology cell images [C] // International Conference on Pattern Recognition. Barcelona: IEEE, 2000:500-503.
[17] Chang Hang, Borowsky A, Spellman P, et al. Classification of tumor histology via morphometric context [C] // IEEE Conference on Computer Vision and Pattern Recognition. Portland: IEEE, 2013, 2013:2203-2210.
[18] Huang P, Lee C. Automatic classification for pathological prostate images based on fractal analysis [J]. IEEE Transactions on Medical Imaging, 2009, 28(7):1037-1050.
[19] Kong J, Sertel O, Shimada H, et al. Computer-aided evaluation of neuroblastoma on whole-slide histology images: Classifying grade of neuroblastic differentiation [J]. Pattern Recognition, 2009, 42(6):1080-1092.
[20] Tosun AB, Gunduz-Demir C. Graph run-length matrices for histopathological image segmentation [J]. IEEE Transactions on Medical Imaging, 2011, 30(3):721-732.
[21] Al-Kofahi Y, Lassoued W, Lee W, et al. Improved automatic detection and segmentation of cell nuclei in histopathology images [J]. IEEE Transactions on Biomedical Engineering, 2010, 57(4):841-852.
[22] Wang Haibo, Cruz-Roa A, Basavanhally A, et al. Mitosis detection in breast cancer pathology images by combining handcrafted and convolutional neural network features [J]. Journal of Medical Imaging, 2014, 1(3):034003.
[23] Xing Fuyong, Xie Yuanfu, Yang Lin. An automatic learning-based framework for robust nucleus segmentation [J]. IEEE Transactions on Medical Imaging, 2016, 35(2):550-566.
[24] Chen Ting, Chefd Hotel C. Deep learning based automatic immune cell detection forimmuno-histochemistry images [C] // International Workshop on Machine Learning in Medical Imaging. Boston: Spring, 2014:17-24.
[25] Song Youyi, Zhang Ling, Chen Siping, et al. Accurate segmentation of cervical cytoplasm and nuclei based on multiscale convolutional network and graph partitioning [J]. IEEE Transactions on Biomedical Engineering, 2015, 62(10):2421-2433.
[26] Song Youyi, Tan Elang, Jiang Xudong, et al. Accurate cervical cell segmentation from overlapping clumps in pap smear images [J]. IEEE Transactions on Medical Imaging, 2017, 36(1):288-300.
[27] Akram SU, Kannala J, Eklund L, et al. Cell Segmentation Proposal Network for Microscopy Image Analysis [M] //Phoenix: Springer International Publishing, 2016, 10008:21-29.
[28] Ronneberger O, Fischer P, Brox T. U-Net:Convolutional Networks for Biomedical Image Segmentation [C] // International Conference on Medical Image Computing and Computer Assisted Intervention. Munich: Springer-Cham, 2015:234-241.
[29] Xie Yuanpu, Xing Fuyong, Kong Xiangfei, et al. Beyond classification: structured regression for robust cell detection using convolutional neural network [C] // International Conference on Medical Image Computing and Computer Assisted Intervention. Munich: Springer-Cham, 2015:358-365.
[30] Sirinukunwattana K, Pluim JP, Chen H, et al. Gland segmentation in colon histology images: The glas challenge contest [J]. Medical Image Analysis, 2017, 35:489-502.
[31] Kainz P, Pfeiffer M, Urschler M. Semantic segmentation of colon glands with deep convolutional neural networks and total variation segmentation [J]. Computer Science, 2015.
[32] Bentaieb A, Kawahara J, Hamarneh G. Multi-loss convolutional networks for gland analysis in microscopy [C] // IEEE International Symposium on Biomedical Imaging. Prague: IEEE, 2016:642-645.
[33] Chen Hao, Qi Xiaojuan, Yu Lequan, et al. Dcan: deep contour-aware networks for accurate gland segmentation [C] // IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016:2487-2496.
[34] Xu Yan, Li Yang, Liu Mingyuan, et al. Gland instance segmentation by deep multichannel side supervision [C] // International Conference on Medical Image Computing and Computer-Assisted Intervention. Athens: Springer-Cham, 2016:496-504.
[35] Roux L, Racoceanu D, Lom NN, et al. Mitosis detection in breast cancer histological images: An ICPR 2012 contest[J]. Journal of Pathology Informatics, 2013, 4:8-16.
[36] Malon CD, Cosatto E. Classification of mitotic figures with convolutional neural networks and seeded blob features [J]. Journal of Pathology Informatics, 2013, 4(1):9-17.
[37] Chen Hao, Dou Qi, Wang Xi, et al. Mitosis detection in breast cancer histology images via deep cascaded networks [C] //Proceedings of the Thirtieth AAAI Conference on Artificial Intelligence. Phoenix: AAAI, 2016:1160-1166.
[38] Chen Hao, Wang Xi, Heng Peng'an. Automated mitosis detection with deep regression networks [C] //IEEE International Symposium on Biomedical Imaging. Minsk: IEEE, 2016:1204-1207.
[39] Radboud University Medical Center, and the University Medical Center Utrecht. ISBI challenge on cancer metastasis detection in lymph nodes [EB/OL].https://camelyon16.grandchallenge.org/, 2016-04-14/2017-10-30.
[40] Department of Pathology of the Radboud University Medical Center in Nijmegen. CAMELYON17 [EB/OL]. https://camelyon17.grand -challenge.org/, 2017-05-15/2017-10-30.
[41] Gregory CS. A public domain database for computational anatomy [EB/OL]. http://www.ima-genglab.com/newsite/pddca/, 2016-12-30/2017-10-30.
[42] National Cancer Institute. Genomic data commons data portal [EB/OL]. https://portal.gdc.cancer.gov, 2017-10-24/2017-10-30.
[43] Ludovic Roux. ICPR 2012-Mitosis detection contest [EB/OL]. http://www.ipal.cnrs.fr/event/icpr-2012, 2012-11-15/2017-10-30.
[44] Frédérique C. Mitos-atypia-14 [EB/OL], https://mitosatypia-14.grand challenge.org/dataset/, 2014/2017-10-30.
[45] 21th International Conference on Pattern Recognition. Contest on hep-2 cells classification [EB/OL].http://nerone.diem.unisa.it/hep2contest/dataset.shtml, 2011-11-15/2017-3-24.
[46] Araújo, T. ICPR2018 gland challenge on breast cancer histology images [EB/OL]. https://iciar2018-challenge.grand-challenge.org/dataset/, 2017/11/1-2017/11/21.
[47] Ana PP and Paulode CA. Bioimaging 2015 4th international symposium in applied bioimaging [EB/OL].http://www.bioimaging2015.ineb.up.pt/challenge_overview.html, 2015/11/16/2017/11/21.
[48] Wang Dayong, Khosla A, Gargeya R, et al. Deep learning for identifying metastatic breast cancer [J]. arXiv, 2016,1606:05718.
[49] Kovalev V, Kalinovsky А, Liauchuk V. Deep learning in big image data: Histology image classification for breast cancer diagnosis [C] //International Conference on Big Data and Advanced Analytics. Boston: IEEE, 2016:15-17.
[50] Bayramoglu N, Kannala J, Heikkilä J. Deep learning for magnification independent breast cancer histopathology image classification [C] // International Conference on Pattern Recognition. IEEE. Plymouth: Springer, London, 2017:2440-2445.
[51] Spanhol FA, Oliveira LS, Petitjean C, et al. Breast cancer histopathological image classification using convolutional neural networks [C] // International Joint Conference on Neural Networks. Vancouver: IEEE, 2016:2560-2567.
[53] Cruz-Roa A, Basavanhally A, Gonz Lez F, et al. Automatic detection of invasive ductal carcinoma in whole slide images with convolutional neural networks [C] // SPIE Medical Imaging. San Diego: International Society for Optics and Photonics, 2014:139-144.
[54] Shah M, Rubadue C, Suster D, et al. Deep learning assessment of tumor proliferation in breast cancer histological images [J]. Computer Science, 2016.
[55] Turkki R, Linder N, Kovanen PE, et al. Antibody-supervised deep learning for quantification of tumor-infiltrating immune cells in hematoxylin and eosin stained breast cancer samples [J]. Journal of Pathology Informatics, 2016, 7(1):38.
[56] Cruz-Roa A, Ovalle JEA, Madabhushi A, et al. A deep learning architecture for image representation, visual interpretability and automated basal-cell carcinoma cancer detection [C] // International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer-Berlin-Heidelberg, 2013:403-410.
[57] Kashif MN, Shan EAR, Sirinukunwattana K, et al. Handcrafted features with convolutional neural networks for detection of tumor cells in histology images [C] // IEEE International Symposium on Biomedical Imaging. Prague: IEEE, 2016:1029-1032.
[58] Hinton GE, Osindero S, Teh YW. A fast learning algorithm for deep belief nets [J]. Neural Computation, 2006, 18(7):1527-1554.
[59] Masood A, Al-Jumaily A, Anam K. Self-supervised learning model for skin cancer diagnosis [C] //International IEEE/EMBS Conference on Neural Engineering. Montpellier: IEEE, 2015:1012-1015.
[60] Xu Yan, Jia Zhipeng, Ai Yuqing, et al. Deep convolutional activation features for large scale brain tumor histopathology image classification and segmentation [C] // IEEE International Conference on Acoustics. Brisbane: IEEE, 2015:947-951.
[61] Ertosun M, Rubin DL. Automated grading of gliomas using deep learning in digital pathology images: A modular approach with ensemble of convolutional neural networks [C] // American Medical Informatics Association Annual Symposium Proceedings. San Francisco: AMIA, 2015:1899-1908.
[62] Hou Le, Singh K, Samaras D, et al. Automatic histopathology image analysis with CNNs [C] //New York Scientific Data Summit. New York: IEEE, 2016:1-6.
[63] Källén H, Molin J, Heyden A, et al. Towards grading gleason score using generically trained deep convolutional neural networks [C] //IEEE Inter-national Symposium on Biomedical Imaging. Prague: IEEE, 2016:1163-1167.
[64] Litjens G, Nchez CI, Timofeeva N, et al. Deep learning as a tool for increased accuracy and efficiency of histopathological diagnosis [J]. Scientific Reports, 2016, 6:26286.
[65] Källén H, Molin J, Heyden A, et al. Towards grading gleason score using generically trained deep convolutional neural networks [C] // IEEE International Symposium on Biomedical Imaging. Prague: IEEE, 2016:1163-1167.
[66] Sirinukunwattana K, Raza SEA, Tsang YW, et al. Locality sensitive deep learning for detection and classification of nuclei in routine colon cancer histology images [J]. IEEE Transactions on Medical Imaging, 2016, 35(5):1196-1206.
[67] Xu Yan, Mo Tao, Feng Qiwei, et al. Deep learning of feature representation with multiple instance learning for medical image analysis [C] // IEEE International Conference on Acoustics, Speech and Signal Processing. Florence: IEEE, 2014:1626-1630.
[68] Chen Ting, Chefd'hotel C. Deep learning based automatic immune cell detection forimmuno-histochemistry images [C] // International Workshop on Machine Learning in Medical Imaging. Quebes: Springer-Cham, 2014:17-24.
[69] Janowczyk A, Basavanhally A, Madabhushi A. Stain normalization using sparse autoencoders (StaNoSA): Application to digital pathology [J]. Computerized Medical Imaging & Graphics, 2016, 57:50-61.
[70] Sethi A, Sha L, Vahadane AR, et al. Empirical comparison of color normalization methods for epithelial-stromal classification in H and E images [J]. Journal of Pathology Informatics, 2016, 7(8):468-476.

