慢性肾脏病中Cyr61加权基因共表达网络分析
(青岛大学附属医院肾内科,山东 青岛 266003)
目前,世界范围内慢性肾脏病(Chroic Kidney Disease,CKD)的患病率逐年增高,已经成为全球重要的公共卫生问题之一,美国CKD的患病率约为13%[1-2],而在我国CKD的患病率约为10.8%[3]。CKD会持续进展为终末期肾病,并增加病人心血管事件及死亡风险。对CKD的发病机制进行更深入的研究,将有助于对CKD进行预防和治疗。富含半胱氨酸蛋白61(Cysteine rich angiogenic inducer 61,Cyr61)是一种具有肝素结合活性的分泌蛋白,其作为基质相关信号分子参与细胞增殖、分化、转化、凋亡及应激等生物过程。研究显示,缺血再灌注肾损伤早期,Cyr61蛋白在组织及尿液中均明显升高[4]。我们的前期研究显示,在缺血性急性肾损伤(Acute kidney injury,AKI)早期,缺氧状态下过表达Cyr61能够促进肾小管上皮细胞增殖,抑制细胞凋亡。本文采用生物信息学数据分析方法,利用网络公共数据库基因芯片筛选CKD表达差异基因,构建基因共表达网络,探讨Cyr61基因在CKD肾小球及肾间质中的表达情况,并探讨Cyr61基因是否为CKD枢纽基因。
1 材料与方法
1.1 基因芯片数据的获取
对公共的基因芯片数据库NCBI GEO[5]和Array Express[6]进行CKD相关样本检索,筛选条件:①物种设定人类;②转录组表达谱芯片;③包含正常肾脏对照组;④不重复的数据。得到GEO数据库中GSE47185[7]、GSE37463[8]、GSE35489[9]、GSE3-2592[8]共4个数据集373个样本(平台GPL11670、平台GLP14663),样本的临床信息已被贡献者上传至nephroseq(www.nephroseq.org, 11 2016, University of Michigan, Ann Arbor, MI)。GEO提供原始数据文件(TAR of CEL)和经过预处理的矩阵文件(TXT),由于不同实验对数据的预处理方法不一致,本研究下载并使用原始数据及平台注释进行统一的数据处理。
1.2 方法
1.2.1数据预处理与差异基因筛选 Affy芯片预处理一般分为背景处理、标准化处理及总汇3步。本文应用R软件(v.3.3.1)对所有样本数据进行数据分析,使用Affy包(v.1.50.0)[10]的RMA(Robust Multiarray Average)方法进行三合一预处理与表达基因筛选,根据疾病类型将373个样本分为7个疾病组和正常组(living kidney donors, LD),并按照样本的来源分为肾小球组与肾间质组,使用线性回归模型limma包[11]对各组进行表达差异计算,设定校正后P值(FDR)与对数化表达变化倍数(log2 fold change,logFC)作为筛选差异基因阈值。整理各疾病组基因表达数据,从而得到Cyr61基因的表达情况。
1.2.2加权基因共表达网络分析(Weighted Gene Co-expression Network Analysis,WGCNA) 本实验应用WGCNA对CKD进行基因共表达分析。WGCNA是构建基因共表达网络的典型系统生物学算法,WGCNA首先将在不同的样本中存在高度相关性的基因划分在同一个共表达网络,或称之为属于同一模块,随后将相关模块与外界信息(临床数据、单核苷酸多态性、蛋白组学等)进行关联,获得模块与各外界信息的相关程度,最后在与疾病相关程度高的模块中寻找枢纽基因、预测调控通路等。参考WGCNA使用手册[12],将表达差异基因变化倍数对数化,使用R语言中WGCNA包(v.1.51)进行WGCNA。
1.2.3枢纽基因的筛选及Cyr61相关基因分析基因或蛋白在疾病发生过程中不是单独发挥作用而是共同发挥作用的,有多种方法可以用来描述基因与基因之间的相关性,WCGNA使用拓扑重叠法(Topological overlap measure,TOM)来计算基因之间的相关程度,使用TOM计算的结果更加具有生物学意义[13-14]。本研究使用WGCNA包提供的“networkScreening”函数在与慢性肾脏疾病发生相关性最高的模块中寻找枢纽基因,获得基因与疾病相关性加权P值、矫正后的q值[15]、加权后相关系数cor及FishZ检验结果,其中q数值越小说明该基因与CKD的发生相关性越强。使用“exportNetworkToCytoscape”函数获得Cyr61基因与其他枢纽基因的相关性数据,将数据导入Cytoscape(v.3.4.0)[16],并按照拓扑重叠权重构图,得到与Cyr61基因高度相关的枢纽基因。蛋白互作网络分析使用STRING数据库(http://www.string-db.org)[17]。
1.3 统计学分析
采用Limma软件包以moderated t-statistic[18]进行两组间比较;WGCNA采用Pearson法或者TOM法。以P<0.05为差异有统计学意义。
2 结 果
2.1 Cyr61基因在CKD中的表达
7种CKD及正常对照共373个样本原始数据均来源于NCBI GEO(表1)。为了研究Cyr61基因在肾脏疾病不同部位的表达情况,本研究将样本分为肾小球组与肾间质组,并使用R软件获取未经对数化处理的基因表达数据,结果显示Cyr61基因在CKD肾小球中表达普遍降低(图1),而在肾间质中部分表达降低(图2,表2)。应用线性回归模型limma包对各组进行表达差异计算,获取Cyr61基因在各组中的变化倍数,结果显示Cyr61基因在高血压肾病(hypertensive nephropathy, HTN)、微小病变性肾病(minimal change nephropathy, MCD)、膜性肾病(membranous nephropathy, MGN)、狼疮性肾炎(Lupus nephritis,LN)、局灶阶段性肾小球硬化(lupus nephritis and focal segmental glomerulosclerosis, FSGS)的肾小球及间质中降低,差异具有统计学意义(FDR<0.05,logFC<-0.8),在IgA肾病(IgA nephropathy, IgAN)肾间质中降低,差异具有统计学意义(FDR<0.01,logFC<-1)。
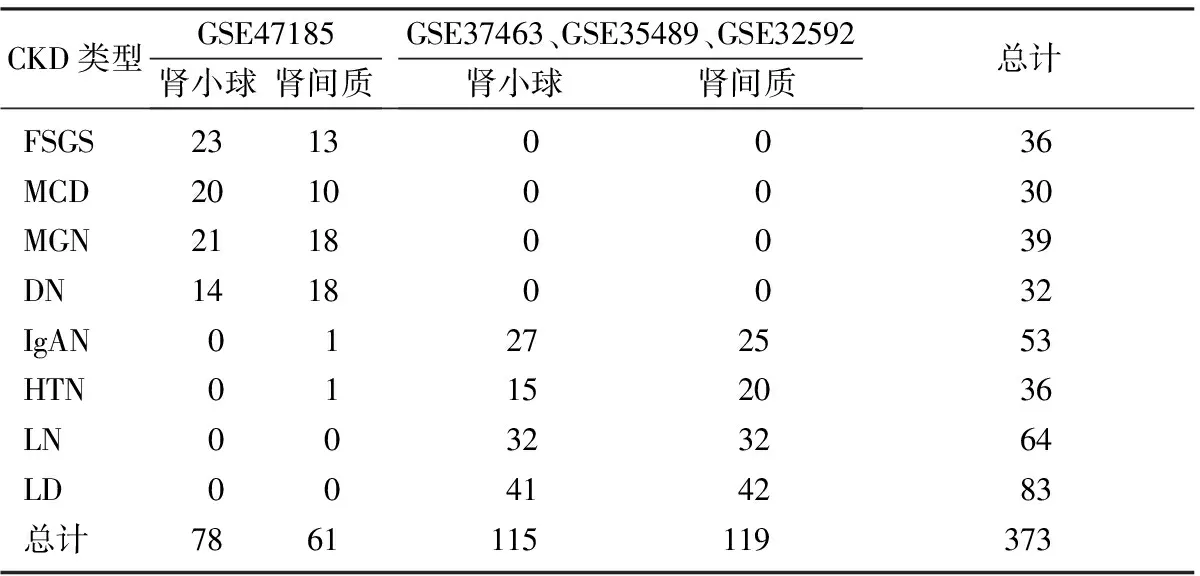
表1 GEO数据库中实验编号及样本的数量
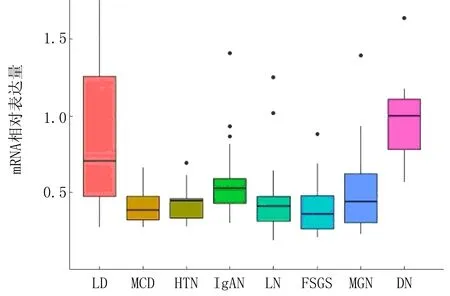
图1 Cyr61基因在CKD肾小球中的相对表达
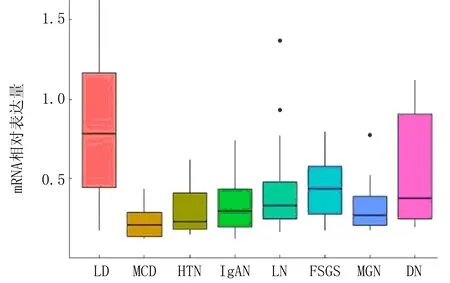
图2 Cyr61基因在CKD肾间质中的相对表达
2.2 Cyr61基因与CKD的关系
由于Cyr61基因在各慢性肾脏疾病间质中普遍表达降低,本研究对GPL14663平台上各肾间质组的2 134个差异基因进行基因共表达分析。按照无尺度网络的标准,本研究选择相关系数等于0.87的邻接矩阵权重参数(软阈值)β=8构建基因模块(图3),并根据动态混合剪切法,共得到9个基因模块(图4,表3),通过计算模块内各基因表达量与样本特征向量的皮尔森相关系数,寻找与CKD发生显著相关的基因模块,其中Brown模块基因的显著性(Gene significance,GS)高于其他模块(图5),因此该模块与CKD相关性最高。随后,应用WGCNA的“networkScreening”函数判断枢纽基因,研究结果显示Cyr61(q<0.01)是CKD发生的枢纽基因(表4)。
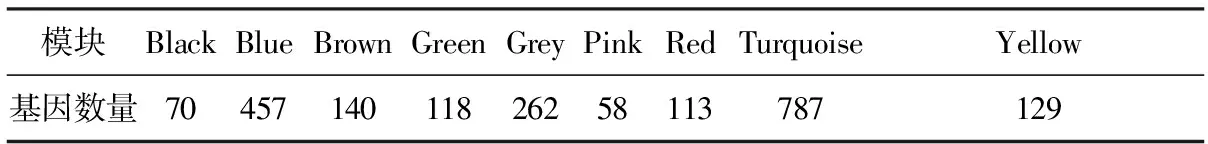
表3 各模块中基因的数量
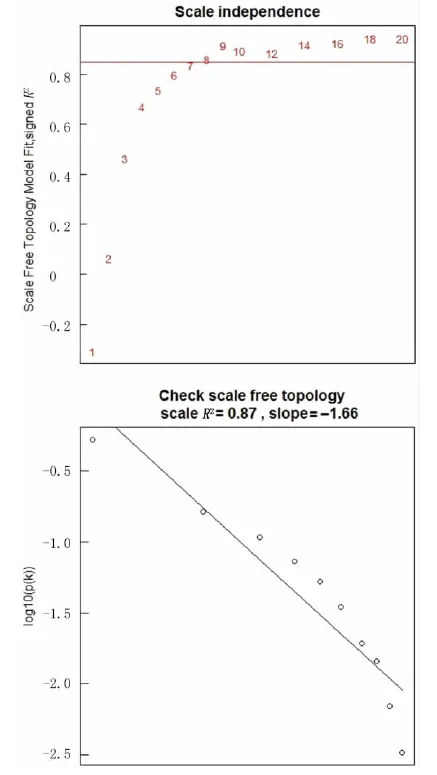
上图红线显示相关系数等于0.85,β=8最接近红线,下图显示β=8时相关系数等于0.87。
图3加权系数β以及无尺度网络特性检验
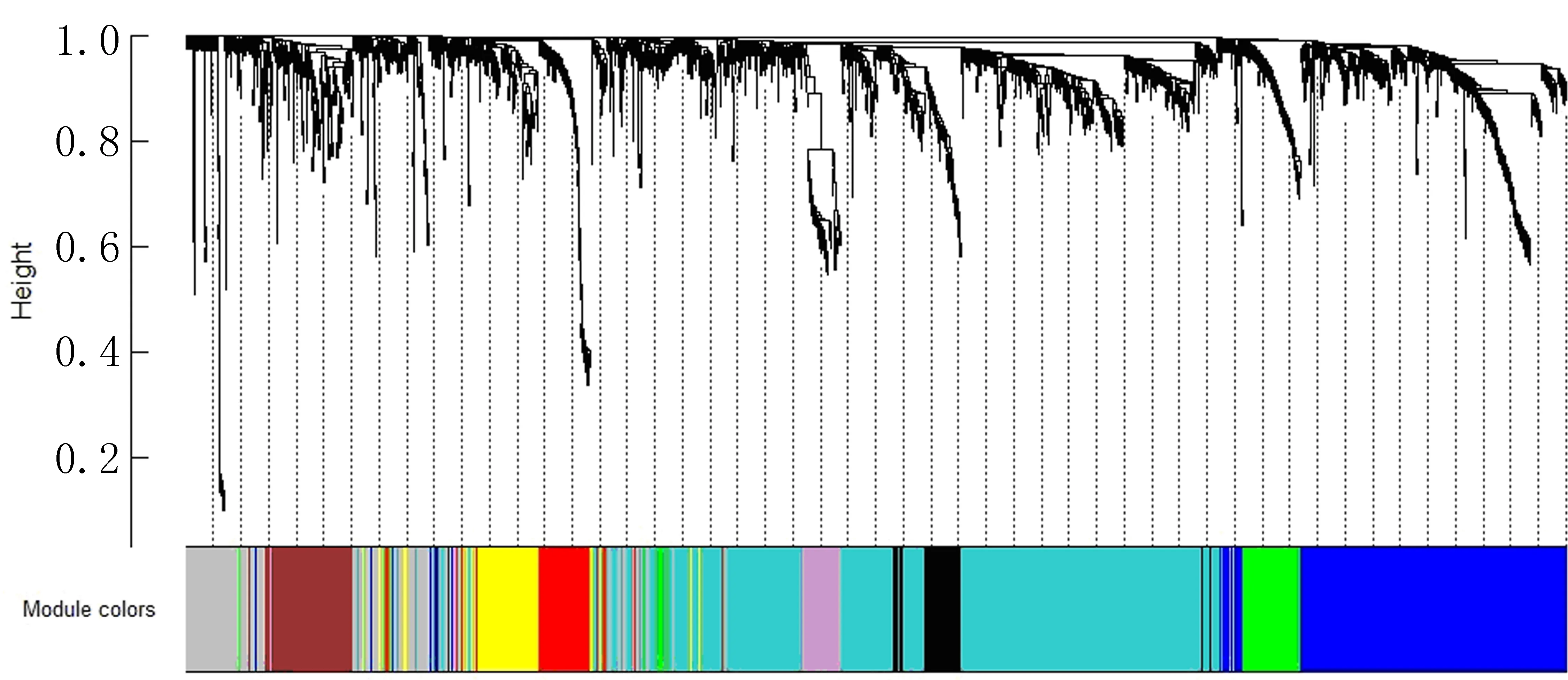
图4 基因层次聚类树状图及对应模块
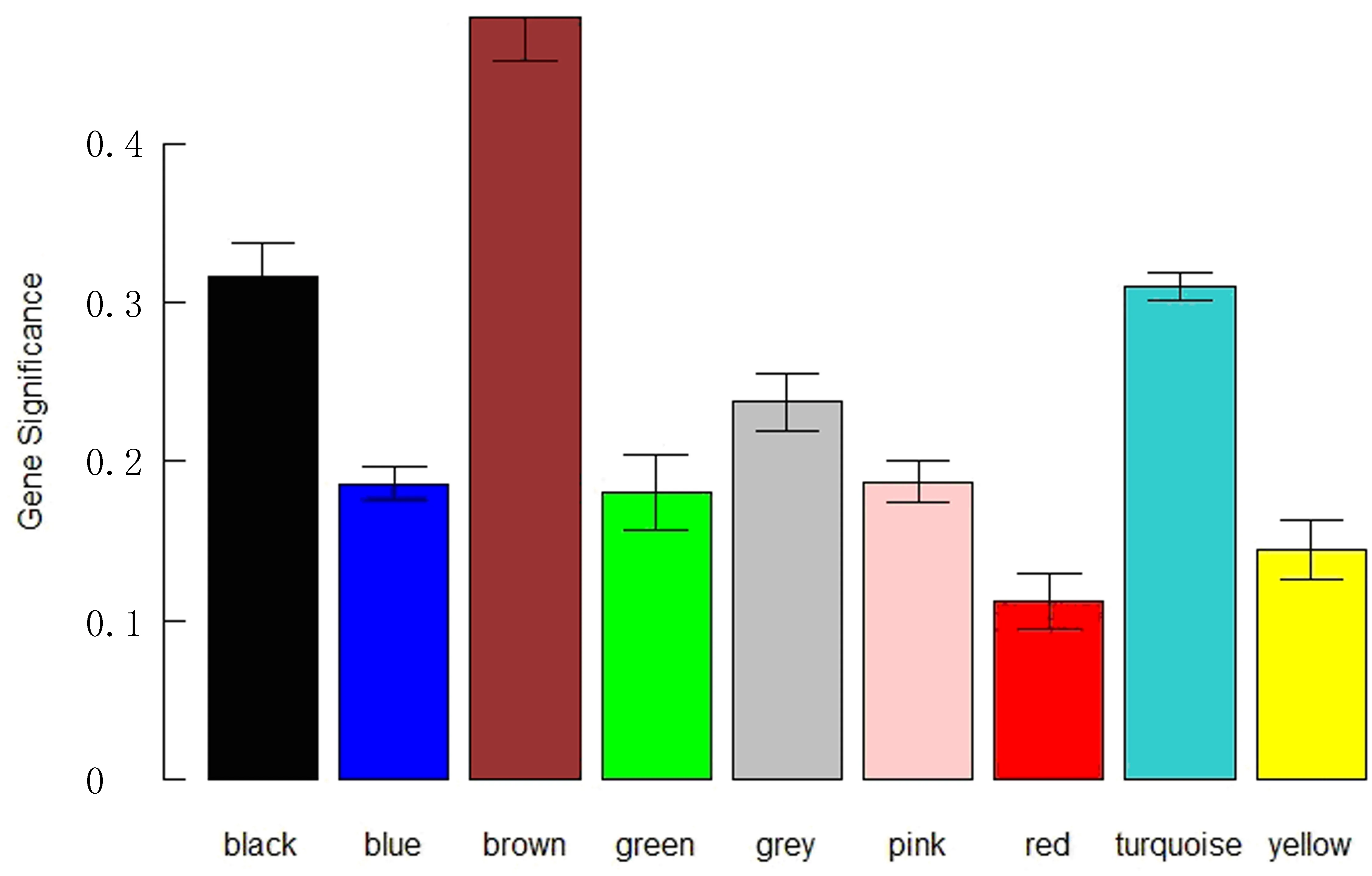
柱状图高度表示整个模块基因显著性的平均值。
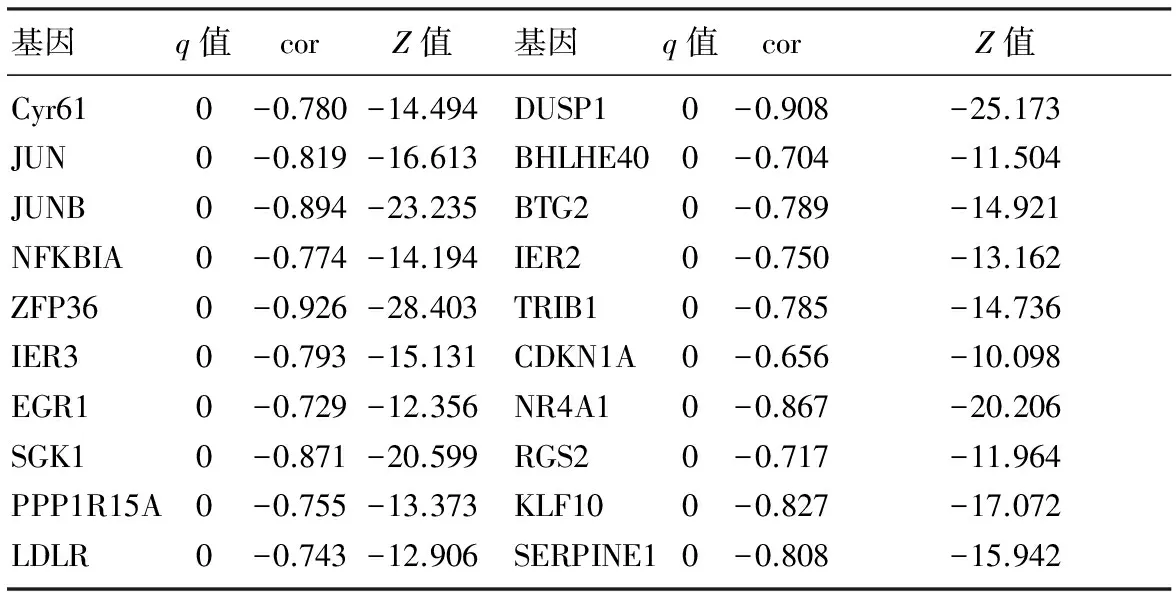
基因q值corZ值基因q值corZ值Cyr610-0.780-14.494DUSP10-0.908-25.173JUN0-0.819-16.613BHLHE400-0.704-11.504JUNB0-0.894-23.235BTG20-0.789-14.921NFKBIA0-0.774-14.194IER20-0.750-13.162ZFP360-0.926-28.403TRIB10-0.785-14.736IER30-0.793-15.131CDKN1A0-0.656-10.098EGR10-0.729-12.356NR4A10-0.867-20.206SGK10-0.871-20.599RGS20-0.717-11.964PPP1R15A0-0.755-13.373KLF100-0.827-17.072LDLR0-0.743-12.906SERPINE10-0.808-15.942
注:仅展示q最小的前20个基因。
2.3 Cyr61与CKD发生过程中相关蛋白的关系
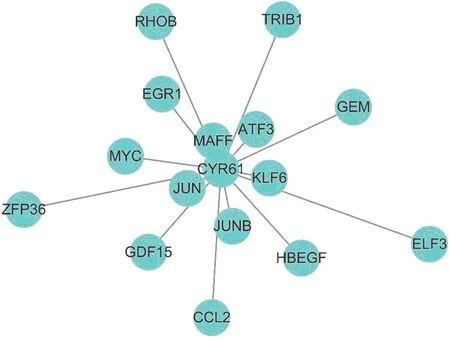
图6a、b展示了q值最小的30个基因之间的相互关系,Cyr61作为枢纽基因在CKD中与众多枢纽基因之间有相互作用。使用Cytoscape按拓扑重叠权重构建Cyr61与其他枢纽基因网状图,图7展示出Cyr61在与CKD发生相关性最高的Brown模块中拓扑重叠最高的15个基因,其中MAFF、JUN、KLF6拓扑重叠系数大于0.10,ATF3、JUNB拓扑重叠系数大于0.09,EGR1、MYC、GDF15、HBEGF、GEM、CCL2、RHOB、TRIB1的拓扑重叠系数大于0.06。本文研究将这15个基因与STRING PPI网络进行对比,其中MAFF、KLF6、GDF15、HBEGF、GEM、CCL2、RHOB以及TRIB1没有与Cyr61基因相关实验。
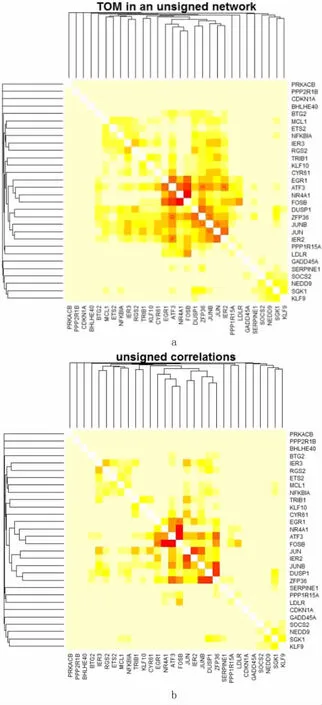
a、b图分别基于拓扑重叠与相关系数,其中每一行及一列分别代表一个基因,颜色越深表示两个基因之间的拓扑重叠或者相关系数越高,两者之间的相关度越高。
图6热图展示CKD中显著性最高的前30个基因
基因之间的距离越近,提示两基因之间的相关度越高。
3 讨 论
已有研究表明,Cyr61在许多疾病及损伤中表达发生变化[19-20],特别是在急性损伤中,Cyr61可以作为早期生物标志物反应疾病的损伤程度和活动性[21-23]。在肾脏疾病的研究中,Cyr61与缺血再灌注肾损伤[4]和肾小球肾炎[24]存在一定的关系。免疫组化结果显示,Cyr61在IgAN和微小病变型肾病肾小球组织中表达降低[24]。本研究结果与其一致,但免疫组化结果显示,Cyr61在各肾脏病肾间质中的表达稳定。而本研究同时显示,在基因芯片上Cyr61基因在各种慢性肾脏疾病间质中的表达是显著降低的,这可能是由于基因的丰富度不一定与翻译产物蛋白质呈线性关系有关。
我们的前期研究显示,Cyr61可抑制转化生长因子-β(transforming growth factor-β,TGF-β)受体表达,对TGF-β诱导的肾小管上皮细胞凋亡有拮抗作用,同时Cyr61还可以参与调解细胞周期蛋白表达,促进细胞增殖[25-27],并且在缺氧条件下,Cyr61表达可以保护肾小管上皮细胞免于凋亡[28]。最新的研究表明,在急性梗阻性肾纤维化模型中,Cyr61参与TGF-β介导的炎症反应,拮抗Cyr61表达可暂时改善间质的炎症反应[29]。以上研究结果证明,Cyr61在肾脏疾病发病过程中发挥一定的作用。在急性肾损伤、CKD中,不同的病理损害以及不同的肾组织中Cyr61表达有差异性,此为预防、诊断和治疗肾脏疾病提供了新的研究方向。
本研究应用生物信息学的方法,整合公共数据库373个样本,从Cyr61的角度分析其在各种CKD中的表达情况,并首次证明Cyr61在HTN、MCD、MGN、LN、FSGS的肾小球及间质中表达降低,在IgAN只有肾间质中表达显著降低。在此基础上,本研究对CKD肾间质的2 134个差异基因进行基因共表达分析,结果显示Cyr61所处的基因模块与CKD的发生相关性最高,在此模块中Cyr61与CKD相关性加权q值<0.01,从而证明Cyr61属于CKD的枢纽基因之一,这说明Cyr61在CKD的发生过程中可能起到重要的作用。而在CKD肾小球中,Cyr61与CKD的发生相关性却较低,这也提示Cyr61在CKD肾间质中参与的功能比在肾小球中多。本实验进一步对Cyr61共表达基因进行研究分析,结果显示MAFF、JUN[30]、KLF6、ATF3[31-32]、JUNB与Cyr61呈高度相关性,目前尚没有相关实验证实MAFF、KLF6、JUNB与Cyr61的相关性,这为进一步研究Cyr61在CKD中的作用提供了理论基础。
目前生物信息学分析广泛应用于医学领域,为疾病的预防、诊断、治疗及预后判断提供了新的方向,使人们对于疾病发生机制有了更深的理解,生物信息学在医学中的应用也是“精准医疗”的要求。随着计算机硬件和软件的不断发展,算法的不断改进,生物信息学将会在医学领域承担更加重要的角色。
[1] COLLINS A J, FOLEY R N, CHAVERS B, et al. United States Renal Data System 2011 Annual Data Report: Atlas of chronic kidney disease & end-stage renal disease in the United States[J]. Am J Kidney Dis, 2012,59(1 Suppl 1):A7,e1-420.
[2] COVIC A, KOTHAWALA P, BERNAL M, et al. Systematic review of the evidence underlying the association between mine-ral metabolism disturbances and risk of all-cause mortality, cardiovascular mortality and cardiovascular events in chronic kidney disease[J]. Nephrol Dial Transplant, 2009,24(5):1506-1523.
[3] ZHANG L, WANG F, WANG L, et al. Prevalence of chronic kidney disease in China: A cross-sectional survey[J]. Lancet, 2012,379(9818):815-822.
[4] MURAMATSU Y, TSUJIE M, KOHDA Y, et al. Early detection of cysteine rich protein 61 (CYR61, CCN1) in urine following renal ischemic reperfusion injury[J]. Kidney Int, 2002,62(5):1601-1610.
[5] BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: Archive for functional genomics data sets-update[J]. Nucleic Acids Res, 2013,41(Database issue):D991-995.
[6] KOLESNIKOV N, HASTINGS E, KEAYS M, et al. ArrayExpress update-simplifying data submissions[J]. Nucleic Acids Res, 2015,43(Database issue):D1113-1116.
[7] JU W, GREENE C S, EICHINGER F, et al. Defining cell-type specificity at the transcriptional level in human disease[J]. Genome Res, 2013,23(11):1862-1873.
[8] BERTHIER C C, BETHUNAICKAN R, GONZALEZ-RIVERA T, et al. Cross-species transcriptional network analysis defines shared inflammatory responses in murine and human lupus nephritis[J]. J Immunol, 2012,189(2):988-1001.
[9] REICH H N, TRITCHLER D, CATTRAN D C, et al. A molecular signature of proteinuria in glomerulonephritis[J]. PLoS One, 2010,5(10):e13451.
[10] IRIZARRY R A, HOBBS B, COLLIN F, et al. Exploration, normalization, and summaries of high density oligonucleotide array probe level data[J]. Biostatistics, 2003,4(2):249-264.
[11] RITCHIE M E, PHIPSON B, WU D, et al. Limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Res, 2015,43(7):e47.
[12] LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008,9:559.
[13] LI A, HORVATH S. Network neighborhood analysis with the multi-node topological overlap measure[J]. Bioinformatics, 2007,23(2):222-231.
[14] YIP A M, HORVATH S. Gene network interconnectedness and the generalized topological overlap measure[J]. BMC Bioinformatics, 2007,8:22.
[15] STOREY J D, TIBSHIRANI R. Statistical significance for genomewide studies[J]. Proc Natl Acad Sci U S A, 2003,100(16):9440-9445.
[16] SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003,13(11):2498-2504.
[17] SZKLARCZYK D, FRANCESCHINI A, WYDER S, et al. STRING v10: Protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Res, 2015,43(Database issue):D447-452.
[18] CUI X, CHURCHILL G A. Statistical tests for differential expression in cDNA microarray experiments[J]. Genome Biol, 2003,4(4):210.
[19] GAO L, FAN Y, HAO Y, et al. Cysteine-rich 61 (Cyr61) upregulated in pulmonary arterial hypertension promotes the proliferation of pulmonary artery smooth muscle cells[J]. Int J Med Sci, 2017,14(9):820-828.
[20] REID S E, KAY E J, NEILSON L J, et al. Tumor matrix stiffness promotes metastatic cancer cell interaction with the endothelium[J]. EMBO J, 2017,36(16):2373-2389.
[21] KLINGENBERG R, AGHLMANDI S, LIEBETRAU C, et al. Cysteine-rich angiogenic inducer 61 (Cyr61): A novel soluble biomarker of acute myocardial injury improves risk stratification after acute coronary syndromes[J]. Eur Heart J, 2017,38(47):3493.
[22] WOO Y J, SEO Y, KIM J J, et al. Serum CYR61 is associa-ted with disease activity in graves’ orbitopathy[J]. Ocul Immunol Inflamm, 2017:1-7.
[23] LIN J, LI N, CHEN H, et al. Serum Cyr61 is associated with clinical disease activity and inflammation in patients with systemic lupus erythematosus[J]. Medicine (Baltimore), 2015,94(19):e834.
[24] SAWAI K, MUKOYAMA M, MORI K, et al. Expression of CCN1 (CYR61) in developing, normal, and diseased human kidney[J]. Am J Physiol Renal Physiol, 2007,293(4):F1363-1372.
[25] 徐岩,国敏,董辉. 含胱氨酸蛋白61对缺氧状态下肾小管上皮细胞的保护作用[J]. 中华肾脏病杂志, 2015,31(6):451-455.
[26] 徐岩,沈学飞,宋年华. 富含半胱氨酸蛋白61对人肾小管上皮细胞增殖与细胞周期的影响[J]. 中华肾脏病杂志, 2013,29(4):273-276.
[27] 徐岩,沈学飞,宋年华. 富半胱氨酸蛋白61对转化生长因子β1诱导肾小管上皮细胞凋亡的影响[J]. 中华肾脏病杂志, 2012,28(10):813-814.
[28] MA R, ZHANG J, LIU X, et al. 7,8-DHF Treatment induces Cyr61 expression to suppress hypoxia induced ER stress in HK-2 cells[J]. Biomed Res Int, 2016, 2016:5029797.
[29] LAI C F, CHEN Y M, CHIANG W C, et al. Cysteine-rich protein 61 plays a proinflammatory role in obstructive kidney fibrosis[J]. PLoS One, 2013,8(2):e56481.
[30] KEENAN S W, HILL C A, KANDOTH C, et al. Transcriptomic responses of the heart and brain to anoxia in the western painted turtle[J]. PLoS One, 2015,10(7):e0131669.
[31] KIM I, LEE S H, JEONG J, et al. Functional profiling of human MeCP2 by automated data comparison analysis and computerized expression pathway modeling[J]. Healthc Inform Res, 2016,22(2):120-128.
[32] JONGBLOETS B C, VAN GASSEN K L, KAN A A, et al. Expression profiling after prolonged experimental febrile seizures in mice suggests structural remodeling in the hippocampus[J]. PLoS One, 2015,10(12):e0145247.

