基于疾病数据库的文本挖掘工具对比研究
, , , , ,
医学研究已进入分子阶段,疾病表型及基因的相似性可能提示分子间的相互作用。由于大多数疾病均为多个基因共同作用的结果,基础医学研究人员通过分子实验确定致病基因的方式费力而耗时,临床研究人员想要针对疾病基因进行治疗也非常困难。新兴的生物信息挖掘技术可以帮助基础医学研究人员在实验前筛选候选基因,也可帮助临床研究人员针对具有相似表型或基因的疾病进行进一步准确诊断治疗及老药新用的尝试[1]。疾病数据库的挖掘对于发现致病基因、阐明分子通路具有重要的意义,这可以通过疾病表型及基因的相似性比较实现。10余年来,科研人员开发了多种疾病数据库文本挖掘工具。本文选取eRAM、PhenUMA 、Gendoo 、G2D 4种性能良好且运行稳定的免费工具进行对比分析,并利用这些工具进行疾病基因发现的实证研究,力求为疾病的遗传学研究提供准确有效的依据,为临床及基础医学研究人员提供有效的参考信息,提高疾病遗传研究的效率。
1 四种文本挖掘工具
精准医学罕见疾病注释百科全书eRAM[2](encyclopedia of Rare Disease Annotation for Precision Medicine)(http://www.unimd.org/eRAM/)是由华东师范大学陈庚等人研发的文本挖掘工具。它整合了10个知名数据库的疾病数据,主要包括罕见病及其用药门户网站(Orphanet)、人类疾病数据库(MalaCards)、NIH-遗传和罕见疾病(Genetic and Rare Diseases,NGRD)、国际罕见病组织数据库(National Organization for Rare Disorders,NORD),为15 942种罕见疾病提供了丰富的临床和分子注释。在其知识库构建过程中将大量的非结构化数据转化为可操作利用的结构化数据,支持基因、表型、疾病间关系的可视化网络构建。分析结果有准确相关基因和全部相关基因两种参考排序方式,两种分析结果中疾病种类及排序不尽相同,为相关疾病预测提供了更多可能。该工具可用于疾病信息检索、基因型检索、表型网络构建、基因网络构建和疾病对网络构建。eRAM提供丰富而准确的知识,不仅有助于研究人员探索罕见疾病的潜在机制,而且有助于临床医生做出准确的诊断和治疗决策。
PhenUMA[3](http://www.PhenUMA.uma.es/)是由西班牙马拉加大学Rocío Rodríguez-López等人基于生物医学和生物分子数据库中的有效信息建成的独立知识库。它以基因功能和疾病表型关系为基础,构建、分析和可视化生物网络,且分析功能多样化,构建网络可视化效果好。该工具可用于研究功能相关基因之间的新的病理学关系,将疾病归类到特定表型的簇中,发现与表型相关的疾病等。PhenUMA有助于临床和基础研究人员重新解释其研究结果,并通过优先考虑表面上非相关的隐含因素来重新设计实验。
Gendoo[4](Gene,Disease Features Ontology-based Overview System)(http://Gendoo.dbcls.jp/)由东京大学Takeru Nakazato 等人研发,通过使用MeSH词汇生成相关药物的特征概况、生物现象和解剖结构描述疾病和基因。该工具可用于说明基因和疾病的特征,分别比较基因和疾病特征之间的差异和相似之处,将加速从生物学和临床角度对组学数据的分析。
G2D[5](Genes to Diseases)(http://g2d2.ogic.ca/)由加拿大渥太华健康研究所Carolina Perez-Iratxeta等人开发。它通过数据挖掘算法评估疾病映射的染色体区域中的基因优先级。如果表型已经与多个位点连锁,则也可检测来自两个基因位点的蛋白质之间的相互作用。G2D指出了查询蛋白质和基因组中序列相似性匹配的位置,并利用了现有的关于假基因预测的信息,对识别疾病相关基因具有极大的帮助。
2 研究方法
2.1 4种工具的对比项目
对比分析eRAM、PhenUMA、Gendoo、G2D在运算原理、数据输入、分析功能以及结果输出4方面的不同。运算原理的对比项目包括知识库来源、运算方法、创建时间、更新周期,数据输入的对比项目包括可录入数据种类、输入格式,分析功能的对比项目包括功能块、分析起始选项、分析项目,结果输出的对比项目包括输出选项、可视化项目、结果下载格式。
2.2 实证研究
以Rett综合征为例,利用上述工具进行疾病基因发现的实证研究。Rett综合征是一种伴X染色体的遗传疾病,多发于女性患者,其发病率为1/10 000~1/15 000。患者常表现为脑部发育迟缓、刻板动作、呼吸障碍、运动障碍以及孤独症样的社交障碍,后期可能伴有癫痫的发生[6]。
当前国际权威的在线人类孟德尔遗传数据库(Online Mendelian Inheritance in Man,OMIM)中,查询到Rett综合征的相关基因仅有1个,为甲基化CpG结合蛋白-2(methyl-CpG binding protein 2,MECP2)。因此将MECP2作为与Rett综合征相关的已知基因。
利用上述4种工具对Rett 综合征的相关基因进行挖掘,选取各工具挖掘结果中得分排位高的前3种基因,筛除已知相关基因MECP2,并去重,进一步验证。
验证方法如下:通过在PubMed、CNKI、万方等数据库中检索相关文献,验证各工具分析结果中所得基因是否与Rett综合征相关;通过在蛋白质数据库UniProt中查询某基因的蛋白参与的生物过程和分子功能;通过查阅该生物过程和分子功能是否与Rett 综合征的病因或症状相关,推断该基因是否可能与Rett 综合征相关。
3 结果与分析
3.1 运算原理的比较
4种工具运算原理比较的结果见表1。

表1 4种文本挖掘工具运算原理比较
如表1所示,eRAM整合了来自13个数据库的知识,相对完整;Gendoo 和G2D知识库来源相对较少。eRAM是最新创建的,G2D创建较早,Gendoo 和G2D更新次数较多,但在近几年内没有更新。
4种工具的运算方法总结如下。
eRAM采用夹角余弦法,通过特征向量对之间的夹角余弦值度量。
PhenUMA采用Resnik法,使用基于Resnik方法的两种不同的语义相似性度量计算基因之间的功能相似性和表型谱之间的表型相似性[7]。这两种测量都基于“信息内容”(Information Concept,IC)的概念,它使用每个术语概率(一个术语的注释数与总注释数的比例)的对数进行计算。如果术语的概率降低,则信息内容增加,因此该术语的特异性和信息含量也增加。Resnik提出,一个给定本体的两个术语之间的语义相似性由最具信息含量的共同祖先(Most Informative Common Ancestor,MICA)的IC决定。通过从术语组中所有可能的术语对中选择出最大MICA来获取术语组之间的相似性分数。
Gendoo采用通过比较OMIM条目的概况和基因表达数据的聚类结果发现基因组之间的相似性,将所开发的特征概况应用于疾病相关基因的分析,但其具体运算方法不详。
G2D的运算方法同PhenUMA。
3.2 数据输入的比较
4种工具数据输入的比较结果见表2。

表2 4种文本挖掘工具数据输入的比较
表2显示,4种工具均允许输入基因ID号,只有eRAM、Gendoo允许输入疾病名称。
eRAM可录入数据种类最多, G2D 相对可录入数据种类较少。
PhenUMA和Gendoo支持录入多个检索词,eRAM和G2D仅可输入单个检索词。
3.3 分析功能的比较
4种工具分析功能的比较结果见表3。

表3 4种文本挖掘工具分析功能的比较
4种工具均支持疾病/表型相关基因检索。eRAM和PhenUMA功能块较多,可分析项目也较多。
eRAM特色功能块为基因/表型/疾病对的网络构建;PhenUMA特色功能块为基因/表型/疾病的网络构建和基因/疾病富集分析,且只有PhenUMA具有富集分析功能;G2D功能块较少,但筛选项较完善。
3.4 结果输出的比较
4种工具结果输出的比较见表4。

表4 4种文本挖掘工具结果输出的比较
4种文本挖掘工具中,eRAM和PhenUMA的分析结果较完全(表4),可视化效果较好,结果中链接稳定(图1、图2)。尤其PhenUMA中可给出4种表现形式的结果图,且具有筛选功能。 G2D、Gendoo可视化效果相对较差,而且结果中的链接有时失效。
Gendoo在结果列表中给出与疾病相似度分数,并把分数划分层级,按颜色区分。
G2D在结果中给出疾病相关的Mesh词、Mesh词出现频率及所在文章和相关基因的本体注释,其结果以列表形式给出。
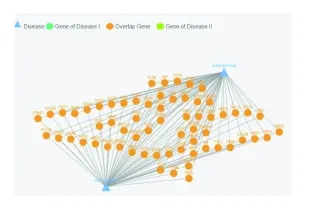
图1 eRAM中Rett综合征与schizophrenia间共享基因网络
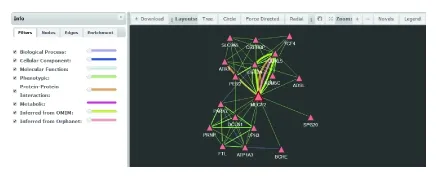
图2 PhenUMA中Rett 综合征的相关基因网络
图2的左侧为网络示意图中不同颜色线段的意义说明,可通过调节滑钮筛选结果。
3.5 实证研究结果与分析
OMIM数据库显示仅有1种基因与Rett综合征相关。由于Rett综合征尚未被认定为单基因疾病,故推测仍有已被认证但未被OMIM收录的相关基因,以及未被认证的相关基因。本文利用上述文本挖掘工具可快速发现Rett综合征的潜在相关基因。
4种文本挖掘工具对Rett综合征的分析结果如下:eRAM预测出6种相关基因,PhenUMA在中度置信水平下预测出21种相关基因,Gendoo预测出56种相关基因,G2D预测出100种相关基因。选取每种工具分析结果中的前3位相关基因(除MECP2,因MECP2是OMIM数据库收录的已知Rett 综合征相关基因):它们分别是eRAM中的EGR2(early growth response 2)、CDKL5(cyclin-dependent kinase-like 5),PhenUMA中的BCHE(butyrylcholin esterase)、CDKL5;Gendoo中的CDKL5、DLX5(distal-less homeobox 5;D2D中的TAZ(tafazzin)、IKBKG(Inhibitor Of Nuclear Factor Kappa B Kinase Subunit Gamma)。
经过去重,得出6种相关基因:EGR2、CDKL5、BCHE、DLX5、TAZ、IKBKG。通过查询PubMed、UniProt中关于这些基因的先验知识,进一步分析它们与Rett综合征相关的可能性。
EGR2为序列特异性DNA结合转录因子,其参与的脑发育、外周神经系统发育、学习与记忆等生物过程与Rett综合征的智力严重低下等症状相关。Swanberg S E等人研究表明,EGR2在出生后的人类皮层中发育增加,并在RTT和自闭症患者皮质中下调[8],故推测EGR2与Rett 综合征相关。
CDKL5介导MECP2的磷酸化,可能调控纤毛生成。Vitorino M等人研究表明CDKL5基因突变导致非典型Rett综合征[9]。
BCHE具有广泛底物特异性的酯酶,有助于神经递质乙酰胆碱的失活,可以降解神经毒性有机磷酸酯,其参与的学习、成神经细胞分化、对糖皮质激素的反应等生物过程与Rett 综合征的智力严重低下、舞蹈样动作、肌张力低等症状相关。
DLX5涉及骨发育的转录因子,其参与的骨形态发生、口腔发育、头部发育等生物过程与生长迟缓、获得性小头、永久性手的失用、进行性行走困难,步态不稳、躯体的失用和共济失调等症状相关。Proudfoot A等人研究表明,DLX5是转录因子,与乳腺癌、肺癌、淋巴瘤、Rett综合征和人类骨质疏松症有关[10]。
TAZ的基因编码是在心脏和骨骼肌中高水平表达的蛋白质。经查阅先验知识,推测TAZ与Rett综合征相关可能性较小。
IKBKG的基因编码kappaB激酶(IKK)是复合物抑制剂的调节亚基,可以激活NF-κB,导致参与炎症、免疫、细胞存活和其它途径的基因的活化。经查阅先验知识,推测IKBKG与Rett综合征相关可能性较小。
OMIM库中仅列1种Rett综合征相关基因MECP2。本文利用上述疾病库文本挖掘工具并结合先验知识推测,除OMIM数据库所列以外的4种相关基因,这是对OMIM的有益补充。
4 结论
eRAM、PhenUMA、Gendoo、G2D 4种工具均可用于快速获取疾病/基因相关信息,并预测疾病与基因的潜在相关关系。eRAM和PhenUMA知识库功能全面,可视化效果好,推荐优先使用。Gendoo和G2D在功能项目上也提供有益的补充,将各工具结合使用可得出更加可信的分析结果。
经实证研究推测基因EGR2、CDKL5、BCHE、DLX与Rett综合征相关,这可作为OMIM数据库对Rett 综合征相关基因阐述的补充。
基于疾病数据库的文本挖掘工具可以有效预测疾病的相关基因,预测疾病、表型、基因间相似和相关关系,有助于疾病病因及治疗等研究。

