热休克蛋白90的分子伴侣调控与癌症研究进展
翟 睿 龚晓云 熊行创 江 游 黄泽建 方 向
(中国计量科学研究院, 北京 100013)
1 引 言
恶性肿瘤已成为严重威胁人类健康的高发病率和高死亡率疾病。据估计,2015年我国新增肿瘤病例429万例,死亡281.42万例,其中肺癌为死亡率最高的癌症,其次为胃癌、食管癌和肝癌[1]。关于癌症的发生及转移的分子机制研究仍是生物医学研究领域需要解决的重要科学问题。热休克蛋白(Heat shock protein,HSP),又名应激蛋白,是机体受到外界不利因素(高温、缺氧、细胞因子释放等)刺激后,迅速合成表达的一类蛋白。热休克蛋白最早于1962年由意大利遗传学家Ferruccio Ritossa在研究果蝇幼虫的唾液腺染色体时发现[2]。当环境温度升高,果蝇唾液腺出现膨突现象,人们将这种现象称为热休克应答(Heat shock response,HSR)。HSP家族成员有6类,根据分子量的不同分为HSP110、HSP90、HSP70、HSP60、HSP40以及小分子热休克蛋白(Small heat shock proteins, sHSPs)。作为一类糖蛋白,HSP在遗传上高度保守,不同种属的同种HSP的核苷酸序列和氨基酸序列具有高度的同源性。在细胞中,HSP最主要的作用之一是作为分子伴侣参与新合成蛋白的正确折叠、帮助受到压力作用后被破坏的蛋白质的重新复性、维持蛋白质的构象和稳定等。随着对肿瘤发生发展分子机制研究的深入,人们发现HSP90由于对肿瘤癌变过程中多条信号通路中的蛋白质起重要调节作用,因而对HSP90的研究已成为抗肿瘤生物学研究的热点。本文对HSP90的分子伴侣调控及参与的肿瘤发生发展及转移调节的研究进展进行了综述。
2 HSP90及其结构特征
HSP90家族是一类ATP依赖的分子伴侣,分子量约90 kDa。HSP90以二聚体形式存在于细胞中,HSP90的二聚化是其行使细胞内功能所必须的[3]。在非应激状态下,HSP90的表达量约占细胞内蛋白总量的1%~2%,是蛋白平均含量的数千倍;在应激条件下,HSP90的含量可升高到细胞蛋白总量的4%~6%[4~6]。在人细胞中,HSP90蛋白分为HSP90α(诱导型)和HSP90β(组成型)。HSP90α在受到热诱导后表达增加,在哺乳动物体内是非必须的,与压力条件下维持细胞稳态相关;而HSP90β可持续表达且在哺乳动物体内必须存在,与哺乳动物的生命活动相关[7,8]。此外,根据蛋白表达位置不同,HSP90的亚型还包括: Gr994(存在于内质网)、TRAP1(位于线粒体)、HSP90C(位于叶绿体)[9]。作为一种重要的分子伴侣,HSP90可以激活不同的底物蛋白,进而参与多种生命活动的调节。
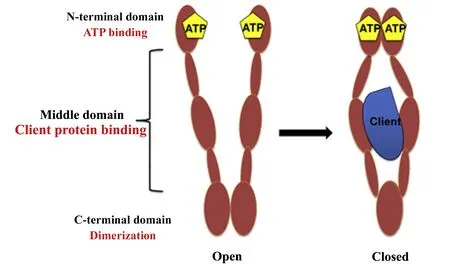
图1 热休克蛋白90的结构示意图[10]Fig.1 Schematic representation of the structure of heat shock protein 90 (HSP90) [10]
HSP90单体包括N端、中间区域和C端3个结构域(图1)[10],每个区域的功能各不相同,无ATP结合时HSP90主要以“V”型开放构象存在[11]。N端结构域为二聚体结构,包含ATP结合位点。HSP90与辅助分子伴侣共同调节ATP的水解过程[12],促使HSP90分子伴侣循环开始,为该过程提供能量。中间区域(M)是底物蛋白和辅助分子伴侣的结合区域[13,14]。连接N端结构域和中间区域的是一段构象易变且带电的连接体,由于其结构不稳定给HSP90全长结构的具体解析带来了很大困难。当HSP90的中间区域催化环变为开放的激活状态时,HSP90才具有ATP酶的活性。中间区域的环状结构含有一个保守的精氨酸残基,其与ATP的γ-磷酸根相互作用,促进HSP90调节的ATP水解供能。C端结构域是另外一个辅助分子伴侣的结合区域,负责HSP90的二聚化[15,16]。C末端具有MEEVD保守序列[17],该序列可与含有TPR(Tetratricopeptide repeat)结构的辅助分子伴侣进行相互作用。当底物蛋白结合到中间区域后,HSP90通过与辅助分子伴侣相互作用和ATP的水解放能共同调节HSP90的N端构象重排,最终HSP90的构象转变为“关闭”状态,此时HSP90才可以行使其分子伴侣的功能[18,19]。
3 HSP90作为分子伴侣
在哺乳动物细胞质中,每毫升细胞液平均约含有300 mg蛋白质[20]。新合成的多肽和不稳定的蛋白质会暴露疏水表面,加剧蛋白质异常折叠和降解的风险,进而引起机体多种疾病。为了使蛋白质行使其正常的生物学功能,分子伴侣可以激活新合成的蛋白质、组装和解聚分子复合物、帮助异常折叠的蛋白质重新折叠、与泛素-蛋白酶体系统协同调节错误折叠蛋白的降解等。HSP90是一类重要的分子伴侣,研究表明,HSP90α和HSP90β可与近2000个来自真核生物的蛋白质进行相互作用[21],因此HSP90广泛地参与了各种生命活动,如类固醇信号转导、蛋白质转运、细胞周期调控、细胞凋亡、免疫应答、癌症发展等过程。
3.1 HSP90的功能调节
3.1.1HSP90的表达调节高温、病毒/细菌感染或缺氧时,机体会启动热休克反应,这一过程在转录水平上受到热休克因子(Heat shock factor 1, HSF1)的调节,同时HSF1也是HSP90的底物蛋白[22]。在正常机体中,HSF1以无活性的单体结合在HSP90上;在压力环境下,HSF1与HSP90形成的复合物解聚,无活性的HSF1被释放,HSF1由单体转变为三聚体,转移至细胞核中与热应激元件(Heat shock element, HSE)结合。随后,HSF1三聚体经过磷酸化转变为有活性的转录因子。至此,HSF1在转录水平上快速提升HSP90的表达水平,同时其它的分子伴侣(如HSP70、HSP40等)和辅助分子伴侣的蛋白表达量也快速升高。当外部压力刺激结束或HSP90表达量达到一定水平后,激活的HSF1再次被抑制[23~25]。
3.1.2辅助分子伴侣对HSP90的功能调节与其它分子伴侣类似,HSP90通常以分子伴侣复合物的形式对底物蛋白进行调节,完成分子伴侣循环。HSP90分子伴侣复合物主要包括其它的分子伴侣(HSP70, HSP40)、辅助分子伴侣(HOP/Sti1, Cdc37, PP5/Ppt1, p23/Sba1和Aha1等)[3,26]及其它作用因子(见表1)。HSP90会根据组织、细胞以及底物蛋白的不同而招募不同的辅助分子伴侣,每种辅助分子伴侣在HSP90帮助蛋白质重新折叠和重新复性过程中发挥独特的作用。在已鉴定到的HSP90辅助分子伴侣中,一些含有TPR结构的蛋白会结合在HSP90的C端保守序列MEEVD上,也有一些蛋白结合在N端结构域和中间区域。HOP/Sti1(HSP70/HSP90 organizing protein, HOP(人); Stress-inducible protein 1, Sti1(酵母))是在HSP90分子伴侣循环中第一个起到HSP90的ATP酶调节作用的辅助分子伴侣。它是一个重要的连接蛋白,其具有3个TPR结构域和2个富含天冬氨酸-脯氨酸(Asp-Pro, DP)的结构域,可以同时与HSP70与HSP90结合,介导底物蛋白从HSP70转移到HSP90形成分子伴侣复合物[27~29]。并且HOP/Sti1还可以抑制HSP90的ATP酶活性,维持HSP90的开放构象。细胞周期分裂蛋白37(Cell division cycle 37, Cdc37)是一种蛋白激酶特异性识别的辅助分子伴侣[30],与HOP/Sti1相似,Cdc37很早就参与到HSP90的分子伴侣循环中。Cdc37的N端可以与蛋白激酶相互作用,中间区域和C端可以与HSP90相互作用,因而Cdc37可以调节细胞内多种激酶的活性,是细胞中必须的蛋白质。Cdc37也可通过抑制HSP90的N端二聚化以及N端与中间区域的对接(ATP水解所需),从而维持HSP90的N端的开放构象。因此,Cdc37可抑制HSP90的ATP酶循环[31~33]。PP5/Ppt1(Protein phosphatase 5, PP5(人);Protein phosphatase T, Ppt1或Ppt(酵母))是具有TPR结构域的HSP90辅助分子伴侣。当PP5/Ppt1与HSP90结合后, PP5/Ppt1的磷酸酶抑制作用解除,进而调节HSP90和Cdc37的去磷酸化作用[34,35]。体内实验表明,由PP5/Ppt1调节的HSP90磷酸化状态的变化直接影响HSP90的功能和底物蛋白的激活[36]。PPIase(Peptidyl-prolyl cis-trans isomerases)也是一类含有TPR结构域的HSP90辅助分子伴侣,包括部分FK506结合蛋白家族成员、亲环蛋白40及Cpr6和Cpr7(酵母)等。除了具备异构酶的活性,PPlase还兼具分子伴侣活性,因此PPlase很可能直接与底物蛋白进行相互作用[37,38],但具体机制仍不清楚。Aha1(Activator of Hsp90 ATPase activity)是近年发现的HSP90辅助分子伴侣,Aha1可以结合在HSP90的中间区域或者N端结构域。Aha1与HSP90的相互作用可极大促进HSP90的ATP酶活性及其构象的转变。然而,形成的Aha1-HSP90复合物对底物蛋白活性的调节机制还不清楚[39,40]。p23/Sba1是一种酸性辅助分子伴侣,存在于哺乳动物大多数组织中,是在研究HSP90与孕激素受体相互作用过程中被发现的。在HSP90分子伴侣循环的后期,p23/Sba1可以代替Aha1结合在HSP90的N端的二聚体上,维持蛋白复合物的稳定以及促进类固醇激素受体的成熟[41,42]。同时,p23/Sba1也可以抑制HSP90的ATP酶活性,促进ATP的结合。当ATP水解和底物蛋白成熟后,p23/Sba1从蛋白复合物上解离[43,44]。在高等真核生物中,HSP70的C末端相互作用蛋白(C terminus of HSP70 interacting-protein, CHIP)与p23/Sba1类似,也在HSP90分子伴侣循环后期起作用。CHIP可以通过自身的TPR结构域与HSP70或HSP90的C端相互作用。由于其具有一个泛素连接酶结构域,当CHIP与HSP90结合后,导致HOP/Sti1和p23/Sba1解离,底物蛋白被降解。敲除CHIP后可使HSP90底物蛋白稳定,而过表达CHIP后可促进蛋白降解[45~47]。因此,CHIP被视为一种释放因子。
表1 HSP90分子伴侣复合物的主要组成[3]
Table 1 Main components of HSP90 chaperone complex[3]
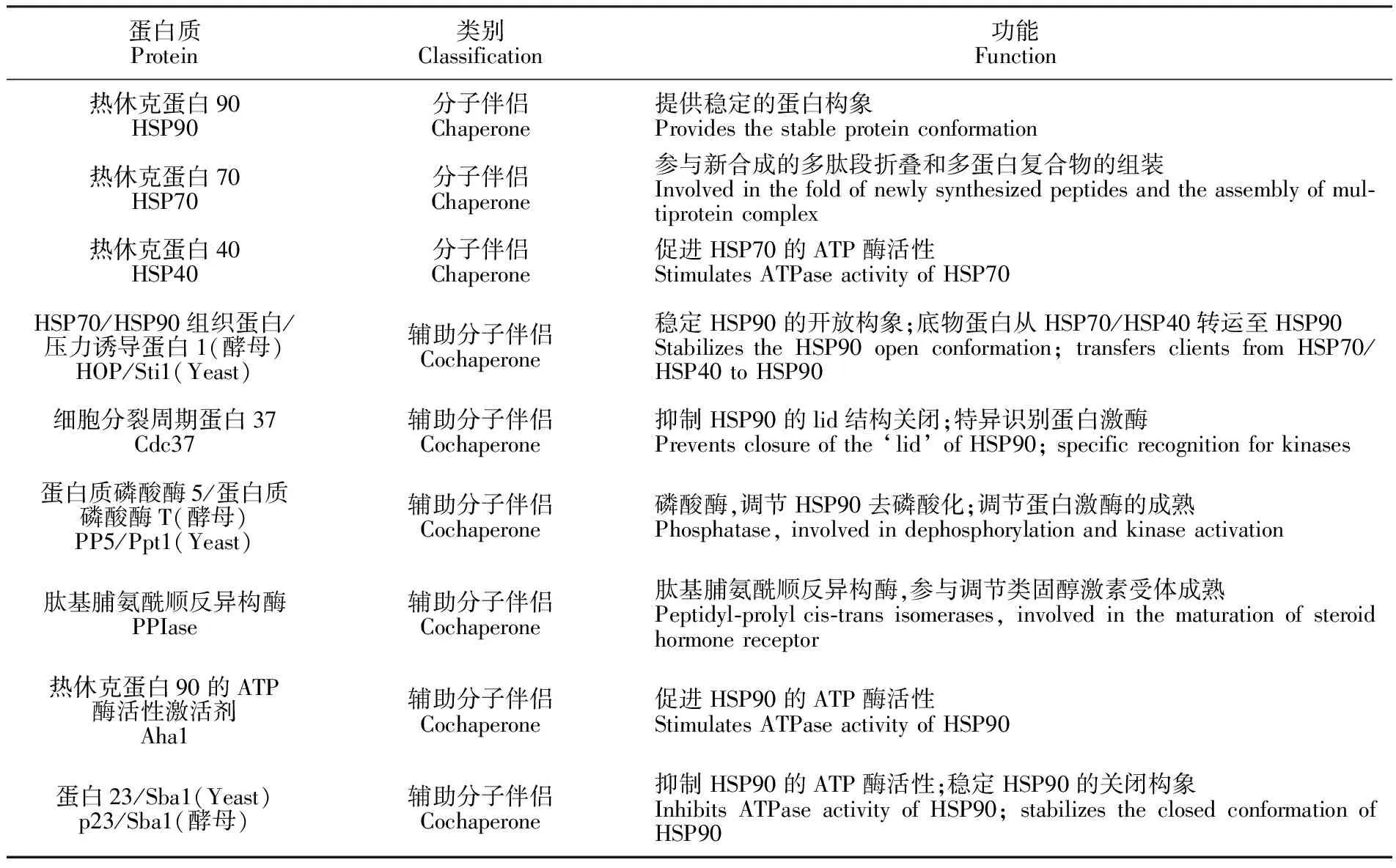
蛋白质Protein类别Classification功能Function热休克蛋白90HSP90分子伴侣Chaperone提供稳定的蛋白构象Providesthestableproteinconformation热休克蛋白70HSP70分子伴侣Chaperone参与新合成的多肽段折叠和多蛋白复合物的组装Involvedinthefoldofnewlysynthesizedpeptidesandtheassemblyofmul⁃tiproteincomplex热休克蛋白40HSP40分子伴侣Chaperone促进HSP70的ATP酶活性StimulatesATPaseactivityofHSP70HSP70/HSP90组织蛋白/压力诱导蛋白1(酵母)HOP/Sti1(Yeast)辅助分子伴侣Cochaperone稳定HSP90的开放构象;底物蛋白从HSP70/HSP40转运至HSP90StabilizestheHSP90openconformation;transfersclientsfromHSP70/HSP40toHSP90细胞分裂周期蛋白37Cdc37辅助分子伴侣Cochaperone抑制HSP90的lid结构关闭;特异识别蛋白激酶Preventsclosureofthe‘lid’ofHSP90;specificrecognitionforkinases蛋白质磷酸酶5/蛋白质磷酸酶T(酵母)PP5/Ppt1(Yeast)辅助分子伴侣Cochaperone磷酸酶,调节HSP90去磷酸化;调节蛋白激酶的成熟Phosphatase,involvedindephosphorylationandkinaseactivation肽基脯氨酰顺反异构酶PPIase辅助分子伴侣Cochaperone肽基脯氨酰顺反异构酶,参与调节类固醇激素受体成熟Peptidyl⁃prolylcis⁃transisomerases,involvedinthematurationofsteroidhormonereceptor热休克蛋白90的ATP酶活性激活剂Aha1辅助分子伴侣Cochaperone促进HSP90的ATP酶活性StimulatesATPaseactivityofHSP90蛋白23/Sba1(Yeast)p23/Sba1(酵母)辅助分子伴侣Cochaperone抑制HSP90的ATP酶活性;稳定HSP90的关闭构象InhibitsATPaseactivityofHSP90;stabilizestheclosedconformationofHSP90
3.1.3蛋白质翻译后修饰对HSP90的功能调节HSP90的功能还受到多种蛋白质翻译后修饰的调节,如磷酸化、乙酰化、类泛素化等。通常,在HSP90的分子伴侣循环中,伴随着一系列不同蛋白的磷酸化作用,直接影响HSP90的ATP酶活性、构象、与辅助分子伴侣及底物蛋白的相互作用。一方面,磷酸化作用对HSP90的分子伴侣循环起到负调节作用。在酵母中,HSP90的Tyr22位点磷酸化可以显著削弱辅助分子伴侣Aha1与HSP90的相互作用[48,49]。Ppt1的缺失可以导致HSP90磷酸化作用的增强,并阻碍一些底物蛋白的成熟[35]。另一方面,磷酸化作用利于HSP90分子伴侣循环的推进,促进底物蛋白成熟,起到正向调节的作用。在以蛋白激酶为底物的分子伴侣循环中(图2)[3],酪蛋白激酶CK2首先磷酸化Cdc37的Ser13位点,进而促进Cdc37与底物蛋白和HSP90的结合[34,50]。随后,在经历磷酸酶PP5/Ppt1调节的Cdc37去磷酸化作用后,HSP90的Tyr197位点以及Cdc37的Tyr298位点的磷酸化可以促进HSP90构象的改变以及Cdc37与HSP90的解离。在这个过程中,PP5/Ppt1既是HSP90的辅助分子伴侣,又可以对HSP90分子伴侣复合物的磷酸化状态进行调节。接下来,HSP90的Tyr313位点的磷酸化与底物蛋白的成熟息息相关,直接导致HSP90的构象变化,转变为易于结合辅助分子伴侣Aha1的结构,促进HSP90的ATP酶活性。最后,HSP90的Tyr627位点的磷酸化导致Aha1、PP5/Ppt1以及成熟的底物蛋白的释放[51]。

图2 磷酸化作用对HSP90的功能调节[3]Fig.2 Regulation of function of HSP90 by sequential phosphorylation[3]
另外,值得注意的是,一些底物蛋白还可以对HSP90进行磷酸化(Akt、PKCγ等)和去磷酸化调节。蛋白激酶C(protein kinase Cγ,PKCγ)是第一个被证明可以直接调节HSP90α活性的底物蛋白激酶[52]。实验结果表明,HSP90α的Thr115/Thr425/Thr603位点可以被PKCγ特异性磷酸化,并且这些位点可以作为HSP90α结合和释放PKCγ的“磷酸化开关”。HSP90α逐渐被PKCγ磷酸化后,导致HSP90α的ATP结合能力、ATP水解能力、与Cdc37结合能力下降。当3个位点完全被磷酸化后,HSP90α失去对PKCγ的结合能力,释放PKCγ。而当HSP90α的这3个位点完全去磷酸化后,恢复对PKCγ强结合能力。因此,在HSP90分子伴侣循环中,蛋白质的磷酸化作用起到多方面的调节作用[52]。
除了磷酸化作用,HSP90的多个位点可以进行乙酰化以及去乙酰化作用,涉及乙酰转移酶p300和去乙酰化酶HDACs(histone deacetylases)家族成员。Yang等鉴定出HSP90α存在7个乙酰化位点:Lys69、Lys100、Lys292、Lys327、Lys478、Lys546和Lys558。除Lys292位点外,其它位点的乙酰化作用导致HSP90结合ATP的能力下降[53]。HSP90的Lys294位点乙酰化可以抑制底物蛋白的成熟和辅助分子伴侣的结合[54]。其它的蛋白质翻译后修饰,如SUMO化(Small ubiquitin-related modifier)、亚硝基化(S-nitrosylation)等也对HSP90的功能有一定调节作用[55,56]。因此,HSP90受到多种翻译后修饰调节,然而人们仍然不清楚这些翻译后修饰之间是否存在相互作用以及其中的分子机制。
3.2 HSP90的底物蛋白及其对底物蛋白成熟的调节
3.2.1HSP90的底物蛋白目前的研究表明,HSP90参与了超过300个蛋白的成熟调控[26](www.picard.ch/downloads),涉及的蛋白种类繁多,因此HSP90在很多重要的生命活动中起到中心调控作用。人们发现的第一个HSP90底物蛋白是病毒激酶v-Src[57],之后HSP90被发现是类固醇激素受体激活所必须的蛋白[58]。早期研究中,蛋白激酶和类固醇激素受体被认为是HSP90的两类主要底物蛋白。然而随着研究的深入,其它底物蛋白也逐渐得到证实,如内皮型一氧化氮和酶、染色质蛋白、转录因子等。此外,最近有关HSP90-底物蛋白相互作用的定量分析结果表明, 60%的人蛋白激酶和7%的转录因子与HSP90结合;人泛素连接酶中与HSP90相互作用的蛋白比例高达30%[59]。根据HSP90对底物蛋白的不同调节作用,可将底物蛋白分为3类:第一类是HSP90促进底物蛋白的构象改变以激活底物蛋白,如蛋白激酶类底物[52];第二类是HSP90帮助多蛋白复合物的组装,如着丝粒的组装等[60];第三类是HSP90促进配体与底物蛋白的结合,如类固醇激素与其受体的结合[61],siRNA与Argonaute 2复合物的结合[62]等。然而,与其它分子伴侣作用机制不同,人们至今仍然不清楚HSP90分子伴侣循环对底物蛋白的激活和成熟的具体分子机制。究其原因,可能有以下4点:(1)通常,HSP90底物蛋白表达水平低,且多具有不稳定的结构;(2)HSP90底物蛋白的多样性。HSP90的底物蛋白涉及种类范围广,且底物蛋白的氨基酸序列和功能均没有相关性;(3)在每一种底物蛋白家族中,一些家族成员可以结合HSP90,而另一些家族成员似乎与HSP90没有任何相互作用;(4)HSP90根据不同情况与不同的辅助分子伴侣相互作用,而辅助分子伴侣可能对底物蛋白具有不同的调节作用,因而增加了HSP90分子伴侣循环的可变性和复杂性,加大了研究的难度。
3.2.2HSP90对底物蛋白成熟的调节HSP90及其辅助分子伴侣帮助底物蛋白折叠和成熟的过程是一个典型的分子伴侣循环[5,63,64]。对于不同的底物蛋白,HSP90会招募不同的辅助分子伴侣形成蛋白复合物。图3为HSP90分子伴侣循环的典型图示[3,26]。首先,HOP/Sti1的一端结合在HSP90的C端MEEVD模体,另一端结合在HSP90中间结构域,以稳定HSP90的“V”型开放构象,进而抑制HSP90的N端二聚化和ATP酶活性;同时,ATP结合在HSP90的N端;底物蛋白与分子伴侣HSP40/HSP70复合物结合。在HOP/Sti1的帮助下,HSP70与底物蛋白复合物结合在HSP90上,至此底物蛋白转移到处于N端开放状态的HSP90上。随后,PPlase结合在HSP90的 C端,Aha1结合在HSP90分子伴侣复合物上以促进HSP90的ATP酶活性以及开放构象的关闭(该状态被称为“Closed 1”),HOP/Sti1和HSP70从分子伴侣复合物上释放,此时HSP90蛋白复合物成熟,底物蛋白保持一种无活性但易被激活的状态。然后,p23/Sba1代替Aha1,导致HSP90构象完全关闭(该状态被称为“Closed 2”)。随着ATP水解供能,成熟的底物蛋白、辅助分子伴侣等从复合物上释放,最后ADP解离,HSP90恢复N端开放状态,一个完整的HSP90分子伴侣循环结束。然而,对于蛋白激酶类底物蛋白,还需要Cdc37对底物蛋白的特异性识别以及PP5/Ppt1对底物蛋白成熟的调节[51]。
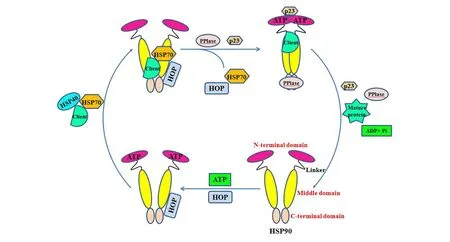
图3 HSP90的分子伴侣循环示意图Fig.3 Diagram of HSP90 chaperone cycle
4 HSP90与肿瘤的关系
在肿瘤细胞中,HSP90的表达量急剧升高,HSP90可以通过对底物蛋白的调节而参与到肿瘤的发生发展以及转移过程中[65]。因此,近年来HSP90成为肿瘤生物学研究的重要靶点。抑癌蛋白p53是HSP90的底物蛋白,在人类50%以上的肿瘤组织中均发现p53的突变。在正常生理条件下,野生型的p53与HSP90分子伴侣复合物仅有短暂的相互作用以维持p53活性并介导其通过泛素化进行降解[66,67]。当p53发生突变,其构象发生变化,其细胞周期检验点活性受到破坏。在肿瘤细胞中,p53与HSP90分子伴侣复合物进行异常相互作用并且持续时间延长,进而抑制p53蛋白的降解,因此在细胞内积累了大量失去抑癌功能的p53蛋白[68~70]。在小鼠为动物模型的研究中,当解除p53与HSP90分子伴侣复合物的相互作用后,可以延长小鼠生存时间并促进肿瘤组织退化[71]。此外,端粒酶、蛋白激酶B(Protein kinase B, PKB)、表皮生长因子受体(Epidermal growth factor receptor, EGFR)等都是HSP90的底物蛋白,这些蛋白的突变或异常调控直接影响细胞增殖、细胞凋亡以及细胞代谢的正常进行。
HSP90除了在细胞内行使分子伴侣的作用,胞内的HSP90还可以被分泌到细胞外,已经证实分泌型HSP90是HSP90α[72]。研究表明,被分泌到细胞外的HSP90存在于多种类型的肿瘤细胞表面,如恶性黑色素瘤细胞、淋巴癌细胞和成纤维肉瘤细胞等[73~75],细胞表面的HSP90可能诱导机体免疫反应,也可以作为分子伴侣通过激活连接肿瘤侵袭性的蛋白,进而促进肿瘤侵袭和进展。Eustace等[75]发现成纤维肉瘤细胞外表达的HSP90是HSP90α,而非HSP90β,并且其可与基质金属蛋白酶(Matrix metalloproteinase, MMP2)相互作用,通过抑制细胞外HSP90α降低MMP2的活性,抑制肿瘤侵袭,说明HSP90α在成纤维肉瘤细胞体外侵袭的重要作用。Wang等[72]发现分泌型HSP90α的C端缺失了EEVD序列,其分泌受到C端EEVD序列与包含TPR域蛋白的相互作用的调节。他们以人乳腺癌细胞为侵袭模型,发现分泌型HSP90α可以依赖MMP2促进肿瘤细胞的侵袭。并且,在多种肿瘤患者的血浆中,HSP90α可被检测到且其水平与肿瘤的恶性程度相关。Yang等[76]发现,HSP90α的质膜表达受到表皮生长因子(Epidermal growth factor, EGF)特异性调节,这一过程是独立于细胞外基质的。他们还发现HSP90α的质膜转运依赖PLCγ1-PKCγ (EGF-mediated activation of phospholipase, PLCγ1)通路。
近年来,研究者逐渐揭示了HSP90与肿瘤发生发展的密切关系,发现抑制HSP90即可调控多种底物蛋白的稳定性及活性进而达到抗癌效果,因此越来越多的团队致力于HSP90抑制剂的研究,目前HSP90抑制剂包括格尔德霉素(Geldanamycin, GA)、根赤壳菌素(Radicicol)、替拉替尼(17-AAG, Tanespimycin)、阿螺旋霉素(17-DMAG, Alvespimycin)、BIIB021等。这些小分子抑制剂大多结合在HSP90的N端的ATP结合区域,维持HSP90的N端的开放状态,因此能够抑制HSP90行使分子伴侣功能所需的ATP酶活性,也阻碍了底物蛋白的折叠。GA是最早发现的HSP90抑制剂,然而由于它的低溶解性以及较大的肝脏毒性妨碍了其临床试验[77]。基于GA的衍生物的合成与研究成为了新药领域的热点,如GA的17位经过化学修饰的衍生物替拉替尼是首个进入临床试验的HSP90抑制剂,然而其溶解性差,对不同肿瘤治疗差异显著。GA的其它衍生物17-DMAG具有良好的溶解性,也已经进入了临床试验[78,79]。然而,作用于HSP90的N端的抑制剂会促进HSF-1解离进而激活热休克反应,削弱抑制剂效果。还有一小部分抑制剂可以结合在HSP90的C端,从而破坏HSP90与某些辅助分子伴侣的相互作用。另外,还有一些抑制剂可以直接结合在HSP90上,如紫杉酚(Taxol)、水飞蓟宾(Silybin)等[65]。最近发展的HSP90抑制剂以复合物的形式破坏HSP90与辅助分子伴侣的相互作用,如雷公藤红素(Celastrol)衍生物可以阻碍Cdc37与HSP90的结合[80]。然而遗憾的是,由于这些抑制剂在应用时会产生一些不良反应,目前还没有抑制剂批准进入临床使用。
5 HSP90的定性定量分析方法
目前,HSP90的定性定量分析方法主要以免疫法为主,包括免疫印迹法和酶联免疫吸附法(Enzyme linked immunosorbent assay, ELISA)。免疫印迹法主要用于HSP90定性分析,通过抗原抗体反应或基于蛋白标签的免疫反应显示HSP90条带。但是,该方法可能存在假阳性及背景干扰等问题,影响HSP90定性分析[52,72]。ELISA法是目前应用最广泛的HSP90定量分析方法,El-Shemi等[81]在大鼠结直肠癌研究中,采用ELISA的方法定量分析结肠组织中HSP90、VEGF等蛋白的表达水平;研究HSP90α在肿瘤侵袭过程中的分泌及功能时,Wang等[72]采用ELISA法定量分析血浆中HSP90α蛋白水平,发现HSP90α表达量与肿瘤的恶性程度相关。然而,由于ELISA法无法获得蛋白质翻译后修饰以及相互作用蛋白的信息, 因此迫切需要发展更快速、高效的HSP90分离方法,以及更精准的定性与定量分析方法。
6 结语与展望
HSP90作为一种重要的分子伴侣,其表面的亲水结构、疏水结构及带电结构等为不同类型蛋白的结合提供了大量相互作用位点。从转录水平、蛋白质翻译后修饰水平到辅助分子伴侣以及底物蛋白,HSP90受到了多种系统不同程度的调节,进而影响HSP90的构象转变、ATP酶活性变化及与其它蛋白的相互作用,最终参与了众多底物蛋白的折叠和成熟,形成了一个巨大而复杂的多功能调控网络。与HSP90相关的肿瘤生物学研究已成为当前热点。
Verba等[82]最近揭示了人HSP90、Cdc37和细胞周期蛋白依赖性激酶(Cyclin dependent kinase 4,Cdk4)复合物的原子分辨率结构,其中Cdc37通过楔入Cdk4的N端半段结构和C端半段结构稳定其开放的激酶构象,使N端半段结构和C端半段结构完全分开,最后HSP90夹住未折叠的β5股并与暴露的N端半段结构和C端半段结构相互作用,维持Cdk4未折叠状态。该研究进一步解释了HSP90与蛋白激酶的相互作用,具有重要的生物学意义。然而,目前仍然有一些问题亟待解决,如HSP90的全长结构解析以及HSP90与底物蛋白的动态构象变化;HSP90识别底物蛋白的具体机制以及其对底物蛋白的调控过程;HSP90与每种辅助分子伴侣相互作用的决定因素、结合位点以及结合能力;在细胞外,是否存在HSP90的新底物蛋白等。此外,HSP90的表达水平与多种肿瘤的恶性程度相关[72],目前HSP90的定性与定量检测方法主要以酶联免疫吸附法为主,然而该方法仍存在一些不足,因此发展快速、稳定的HSP90检测方法具有极其重要的临床价值。这些关于HSP90问题的解决必会带来新的观点和认知,让人们更加全面地了解HSP90参与的生命活动的发展规律,进而针对性地发展HSP90抑制剂,以期在不同的调控水平或生理环境中针对靶点进行精准定位与治疗。
1 Chen W, Zheng R, Baade P D, Zhang S, Zeng H, Bray F, Jemal A, Yu X Q, He J.CA.CancerJ.Clin.,2016, 66(2): 115-132
2 Juliann G K, George C T.Pharmacol.Ther.,1998, 80(2): 183-201
3 Mayer M P, Le Breton L.Mol.Cell,2015, 58(1): 8-20
4 Whitesell L, Lindquist S L.Nat.Rev.Cancer,2005, 5(10): 761-772
5 Taipale M, Jarosz D F, Lindquist S.Nat.Rev.Mol.CellBiol.,2010, 11(7): 515-528
6 Finka A, Goloubinoff P.CellStressChaperones.,2013, 18(5): 591-605
7 Ammirante M, Rosati A, Gentilella A, Festa M, Petrella A, Marzullo L, Pascale M, Belisario M A, Leone A, Turco M C.Oncogene,2008, 27(8): 1175-1178
8 Voss A K, Thomas T, Gruss P.Development,2000, 127(1): 1-11
9 Johnson J L.Biochim.Biophys.Acta,2012, 1823(3): 607-613
10 Sauvage F, Messaoudi S, Fattal E, Barratt G, Vergnaud-Gauduchon J.J.ControlRelease,2017, 248: 133-143
11 Shiau A K, Harris S F, Southworth D R, Agard D A.Cell,2006, 127(2): 329-340
12 Prodromou C, Roe S M, O'Brien R, Ladbury J E, Piper P W, Pearl L H.Cell,1997, 90(1): 65-75
13 Meyer P, Prodromou C, Hu B, Vaughan C, Roe S M, Panaretou B, Piper P W, Pearl L H.Mol.Cell,2003, 11(3): 647-658
14 Hawle P, Siepmann M, Harst A, Siderius M, Reusch H P, Obermann W M.Mol.CellBiol.,2006, 26(22): 8385-8395
15 Minami Y, Kimura Y, Kawasaki H, Suzuki K, Yahara I.Mol.CellBiol.,1994, 14(2): 1459-1464
16 Meyer P, Prodromou C, Liao C, Hu B, Mark Roe S, Vaughan C K, Vlasic I, Panaretou B, Piper P W, Pearl L H.EMBO.J.,2004, 23 (3): 511-519
17 Ramsey A J, Russell L C, Chinkers M.Biochem.J.,2009, 423(3): 411-419
18 Street T O, Lavery L A, Agard D A.Mol.Cell,2011, 42 (1): 96-105
19 Hellenkamp B, Wortmann P, Kandzia F, Zacharias M, Hugel T.Nat.Methods,2017, 14(2): 174-180
20 Ellis R J.Adv.Exp.Med.Biol.,2007, 594: 1-13
21 Echeverría P C, Bernthaler A, Dupuis P, Mayer B, Picard D.PloSone,2011, 6(10): e26044
22 Jiang S, Tu K, Fu Q, Schmitt D C, Zhou L, Lu N, Zhao Y.Tumour.Biol.,2015, 36(7): 4923-4931
23 Bharadwaj S, Ali A, Ovsenek N.Mol.CellBiol.,1999, 19(12): 8033-8041
24 Zou J, Guo Y, Guettouche T, Smith D F, Voellmy R.Cell,1998, 94(4): 471-480
25 Shamovsky I, Nudler E.CellMol.LifeSci.,2008, 65(6): 855-861
26 Röhl A, Rohrberg J, Buchner J.TrendsBiochem.Sci.,2013, 38(5): 253-262
27 Brinker A, Scheufler C, Von Der Mulbe F, Fleckenstein B, Herrmann C, Jung G, Moarefi I, Hartl F U.J.Biol.Chem.,2002, 277(22): 19265-19275
28 Muller P, Ruckova E, Halada P, Coates P J, Hrstka R, Lane D P, Vojtesek B.Oncogene,2013, 32 (25): 3101-3110
29 Röhl A, Wengler D, Madl T, Lagleder S, Tippel F, Herrmann M, Hendrix J, Richter K, Hack G, Schmid A B, Kessler H, Lamb D C, Buchner J.Nat.Commun.,2015, 6: 6655
30 Keramisanou D, Aboalroub A, Zhang Z, Liu W, Marshall D, Diviney A, Larsen R W, Landgraf R, Gelis I.Mol.Cell,2016, 62(2): 260-271
31 Liu W, Landgraf R.Biochemistry,2015, 54(7): 1493-1504
32 Pearl L H.Biopolymers,2016, 105 (8): 594-607
33 Calderwood S K.SubCellBiochem.,2015, 78: 103-112
34 Vaughan C K, Mollapour M, Smith J R, Truman A, Hu B, Good V M, Panaretou B, Neckers L, Clarke P A, Workman P, Piper P W, Prodromou C, Pearl L H.Mol.Cell,2008, 31(6): 886-895
35 Wandinger S K, Suhre M H, Wegele H, Buchner J.EMBOJ.,2006, 25(2): 367-376
36 Soroka J, Wandinger S K, Mäusbacher N, Schreiber T, Richter K, Daub H, Buchner J.Mol.Cell,2012, 45(4): 517-528
37 Li J, Richter K, Buchner J.Nat.Struct.Mol.Biol.,2011, 18(1): 61-66
38 Guy N C, Garcia Y A, Sivils J C, Galigniana M D, Cox M B.SubcellularBiochemistry. Berlin: Springer International Publishing AG,2015, 35-68
39 Schmid S, Hugel T.Mol.Cell,2011, 41(6): 619-620
40 Wolmarans A, Lee B, Spyracopoulos L, La Pointe P.Sci.Rep.,2016, 6: 33179
41 Rehn A B, Buchner J.SubCellBiochem.,2015, 78: 113-131
42 Karagöz G E, Duarte A M, Ippel H, Uetrecht C, Sinnige T, van Rosmalen M, Hausmann J, Heck AJ, Boelens R, Rüdiger S G.Proc.Natl.Acad.Sci.USA,2011, 108(2): 580-585
43 McLaughlin S H, Sobott F, Yao Z P, Zhang W, Nielsen P R, Grossmann J G, Laue E D, Robinson C V, Jackson S E.J.Mol.Biol.,2006, 356(3): 746-758
44 Richter K, Walter S, Buchner J.J.Mol.Biol.,2004, 342(5): 1403-1413
45 Connell P, Ballinger C A, Jiang J, Wu Y, Thompson LJ, Höhfeld J, Patterson C.Nat.CellBiol.,2001, 3(1): 93-96
46 Edkins A L.SubcellularBiochemistry. Berlin: Springer International Publishing AG,2015: 219-242
47 Shang Y, Xu X, Duan X, Guo J, Wang Y, Ren F, He D, Chang Z.Biochem.Biophys.Res.Commun.,2014, 446(1): 387-392
48 Mollapour M, Tsutsumi S, Truman A W, Xu W, Vaughan C K, Beebe K, Konstantinova A, Vourganti S, Panaretou B, Piper P W, Trepel J B, Prodromou C, Pearl L H, Neckers L.Mol.Cell,2011, 41(6): 672-681
49 Mollapour M, Tsutsumi S, Kim Y S, Trepel J, Neckers L.Oncotarget,2011, 2(5): 407-417
50 Ogiso H, Kagi N, Matsumoto E, Nishimoto M, Arai R, Shirouzu M, Mimura J, Fujii-Kuriyama Y, Yokoyama S.Biochemistry,2004, 43(49): 15510-15519
51 Xu W, Mollapour M, Prodromou C, Wang S, Scroggins BT, Palchick Z, Beebe K, Siderius M, Lee M J, Couvillon A, Trepel J B, Miyata Y, Matts R, Neckers L.Mol.Cell,2012, 47(3): 434-443
52 Lu X A, Wang X, Zhuo W, Jia L, Jiang Y, Fu Y, Luo Y.Biochem.J.,2014, 457(1): 171-183
53 Yang Y, Rao R, Shen J, Tang Y, Fiskus W, Nechtman J, Atadja P, Bhalla K.Cancer.Res.,2008, 68(12): 4833-4842
54 Scroggins B T, Robzyk K, Wang D, Marcu M G, Tsutsumi S, Beebe K, Cotter R J, Felts S, Toft D, Karnitz L, Rosen N, Neckers L.Mol.Cell,2007, 25(1): 151-159
55 Mollapour M, Bourboulia D, Beebe K, Woodford M R, Polier S, Hoang A, Chelluri R, Li Y, Guo A, Lee M J, Fotooh-Abadi E, Khan S, Prince T, Miyajima N, Yoshida S, Tsutsumi S, Xu W, Panaretou B, Stetler-Stevenson W G, Bratslavsky G, Trepel J B, Prodromou C, Neckers L.Mol.Cell,2014, 53(2): 317-329
56 Martínez-Ruiz A, Villanueva L, Gonzlez de Ordua C, López-Ferrer D, Higueras M A, Tarín C, Rodríguez-Crespo I, Vzquez J, Lamas S.Proc.Natl.Acad.Sci.USA,2005, 102(24): 8525-8530
57 Brugge J S, Erikson E, Erikson R L.Cell,1981, 25(2): 363-372
58 Joab I, Radanyi C, Renoir M, Buchou T, Catelli M G, Binart N, Mester J, Baulieu E E.Nature,1984, 308(5962): 850-853
59 Taipale M, Krykbaeva I, Koeva M, Kayatekin C, Westover K D, Karras G I, Lindquist S.Cell,2012, 150(5): 987-1001
60 Makhnevych T, Houry W A.Biochim.Biophys.Acta,2012, 1823(3): 674-682
61 Kirschke E, Goswami D, Southworth D, Griffin P R, Agard D A.Cell,2014, 157(7): 1685-1697
62 Iwasaki S, Sasaki H M, Sakaguchi Y, Suzuki T, Tadakuma H, Tomari Y.Nature,2015, 521(7553): 533-536
63 Li J, Richter K, Buchner J.Nat.Struct.Mol.Biol.,2011, 18(1): 61-66
64 Li J, Richter K, Reinstein J, Buchner J.Nat.Struct.Mol.Biol.,2013, 20(3): 326-331
65 Garg G, Khandelwal A, Blagg B S.Adv.CancerRes.,2016, 129: 51-88
66 Müller L, Schaupp A, Walerych D, Wegele H, Buchner J.J.Biol.Chem.,2004, 279(47): 48846-48854
67 Walerych D, Kudla G, Gutkowska M, Wawrzynow B, Muller L, King F W, Helwak A, Boros J, Zylicz A, Zylicz M.J.Biol.Chem.,2004, 279(47): 48836-48845
68 Whitesell L, Sutphin P D, Pulcini EJ, Martinez J D, Cook P H.Mol.CellBiol.,1998, 18(3): 1517-1524
69 Blagosklonny M V, Toretsky J, Bohen S, Neckers L.Proc.Natl.Acad.Sci.USA,1996, 93(16): 8379-8383
70 He K, Zheng X, Zhang L, Yu J.Mol.CancerTher.,2013, 12(11): 2559-2568
71 Alexandrova E M, Yallowitz A R, Li D, Xu S, Schulz R, Proia D A, Lozano G, Dobbelstein M, Moll U M.Nature,2015, 523(7560): 352-356
72 Wang X, Song X, Zhuo W, Fu Y, Shi H, Liang Y, Tong M, Chang G, Luo Y.Proc.Natl.Acad.Sci.USA,2009, 106(50): 21288-21293
73 Becker B, Multhoff G, Farkas B, Wild P J, Landthaler M, Stolz W, Vogt T.Exp.Dermatol.,2004, 13(1): 27-32
74 Sapozhnikov A M, Ponomarev E D, Tarasenko T N, Telford W G.CellProlif.,1999, 32(6): 363-378
75 Eustace B K, Sakurai T, Stewart J K, Yimlamai D, Unger C, Zehetmeier C, Lain B, Torella C, Henning S W, Beste G, Scroggins B T, Neckers L, Ilag L L, Jay D G.Nat.CellBiol.,2004, 6(6): 507-514
76 Yang J, Song X, Chen Y, Lu X A, Fu Y, Luo Y.Traffic,2014, 15(8): 861-878
77 Supko J G, Hickman R L, Grever M R, Malspeis L.CancerChemother.Pharmacol.,1995, 36(4): 305-315
78 Samuni Y, Ishii H, Hyodo F, Samuni U, Krishna M C, Goldstein S, Mitchell J B.Free.Radic.Biol.Med.,2010, 48(11): 1559-1563
79 Rastelli G, Tian Z Q, Wang Z, Myles D, Liu Y.Bioorg.Med.Chem.Lett.,2005, 15(22): 5016-5021
80 Jiang F, Wang H J, Bao Q C, Wang L, Jin Y H, Zhang Q, Jiang D, You Q D, Xu X L.Bioorg.Med.Chem.,2016, 24(21): 5431-5439
81 El-Shemi A G, Refaat B, Kensara O A, Mohamed A M, Idris S, Ahmad J.CancerPrev.Res. (Phila).,2016, 9(6): 491-501
82 Verba K A, Wang R Y, Arakawa A, Liu Y, Shirouzu M, Yokoyama S, Agard D A.Science,2016, 352(6293): 1542-1547

