PEC HPI irp2基因缺失株的构建及蛋白结构功能预测
刘超英,吴程华,高 洪,严玉霖,富国文,赵 汝(云南农业大学动物科学技术学院,昆明 650201)
耶尔森菌强毒力岛(The Yersinia High-pathogeniticity island,HPI)主要含有与摄铁有关的毒力基因簇[1-3]。研究表明,耶尔森杆菌HPI不仅可以通过Ybt的铁载体夺取宿主中的铁元素进而加重机体的感染,而且可在耶尔森菌和E.coli之间水平传播,与致病性E.coli的毒力进化有着密切的关系[4]。irp2基因是HPI中主要的功能基因,在铁缺乏的条件下可表达高分子量蛋白(High Molecular Weight Protein 2,HMWP2)。高分子量铁调节蛋白参与表达耶尔森杆菌素的阳性表型,被认为对铁载体耶尔杆菌素的合成有着非常重要的作用。因此,致病性E.coli的irp2基因缺失株的构建对致病机制的研究至关重要。
大肠杆菌基因敲除的传统方法是利用其自身的RecA同源重组系统编码的RecA和RecBCD蛋白介导DNA的同源重组。但因该系统存在重组效率低等较多缺点,目前主要采用更为简单迅速的Red同源重组系统的方法进行基因敲除[5-6]。利用该方法构建致病性大肠杆菌(PathogenicE.coli,PEC)HPIirp2基因缺失株,不仅为HPI毒力致病机理研究提供技术基础和理论依据,也为大肠杆菌病的防治奠定基础。
1 材料与方法
1.1 试验材料 质粒pKD46、pKD4、pCP20购自优宝生物,5株E.coli(A-E)分离株由实验室分离鉴定保存。试剂及试剂盒主要包括离心柱型质粒小提试剂盒、细菌基因组DNA提取试剂盒、DNA纯化试剂盒、2×power Taq MasterMix、Competent Cell Preparation Kit、Phusion Flash High-Fidelity PCR MasterMix、FastDigest DpnI限制性内切酶等。
1.2 试验方法
1.2.1 DNA的提取和irp2基因的检测 利用细菌基因组DNA提取试剂盒的说明提取CVCC1565、E.coli(A-E)分离株培养液的DNA,产物保存在-20 ℃,作为PCR模版备用。根据irp2基因序列(GenBank登录号:L18881.1)的保守区域设计上下游引物,irp2-F(5'-3'):TTCCTTCAGCATCGCCTGTTA,irp2-R(5'-3'):CAAGCCCGACATACTCAATCT,由华大基因公司合成。以提取的DNA为模版,irp2-F/R为引物,进行PCR扩增。反应条件为:95 ℃预变性5 min;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸30 s,共35个循环;72℃再延伸5 min。扩增结束后,经1.0 %琼脂糖凝胶电泳检测,并在紫外灯下观察结果,拍照。选择E.coli(E)为试验对象,送硕擎公司进行irp2基因测序。
1.2.2 Red同源重组引物的设计 根据pKD 4序列GENEBANK(AY048743.1)设计引物扩增两侧带有FRT位点的卡那霉素抗性基因,在上下游引物5'端加入50 bp和45 bp的irp2基因的同源重组臂,同源重组引物为Q1/Q2(划线部分为同源臂);根据pKD 46序列(GenBank登录号:AY048746.1)设计pKD46质粒鉴定引物bet-F/R;根据pCP 20序列(GenBank登录号:HB393402.1)设计pCP 20质粒鉴定引物FLP-F/R;敲除鉴定引物为A1、A2、B1、B2,引物由华大基因合成。引物序列见表1:

表1 同源重组所用引物Tab 1 Primers of homologous recombination
1.2.3irp2缺失株的构建 以质粒pKD 4为模板,Q1、Q2为引物进行PCR反应,制备打靶DNA片段。制作E.coli(E)细胞感受态,将pKD 46质粒转化后用特异性鉴定引物bet-F/R鉴定pKD 46阳性菌株,转化成功的菌株命名为E.coli(E)/pKD 46。过夜培养后制备E.coli(E)/pKD 46电转化感受态细胞并进行电转化,完成后取100 μL菌液涂布在LB固体培养基(卡那霉素终浓度为50 μg/mL)上,37 ℃培养过夜,然后使用鉴定引物A1、A2和B1、B2挑选卡那霉素抗性重组子(图1)。挑选含有Kan+和Amp+双抗性的阳性转化株通过FLP重组酶删除FRT位点之间的抗性序列。用鉴定引物A1、B2对卡那霉素消失的克隆进行鉴定。鉴定正确的菌种命名为E.coli(E)/Δirp2。

图1 PCR鉴定引物所在位置Fig 1 Scheme of priming sites
1.2.4 HMWP2蛋白结构及功能预测 利用测序得到的irp2基因序列,通过ExPASy开发的在线工具Protparam分析HMWP2的理论分子量和等电点(pI);登陆http://www.predictprotein.org/,对HMWP2二级结构进行预测;使用ExPASy开发的在线工具ProtScale分析HMWP2的亲疏水性;使用ExPASy开发的在线工具TMpred分析HMWP2的可能的跨膜螺旋区,最优拓扑结构显示有9个跨膜螺旋;使用ExPASy开发的在线工具SWISS-MODEL,与已知结构的PDB数据库中的蛋白质序列进行比对,预测HMWP2的三级结构。
2 结果与分析
2.1E.coliirp2基因的PCR扩增E.coli(A-E)及阴性对照的PCR扩增结果如图2(M为2000 bp的DNA Marker;1为阴性对照,无条带;2-6为E.coli(A-E)irp2基因PCR扩增条带,与预期产物大小一致)。
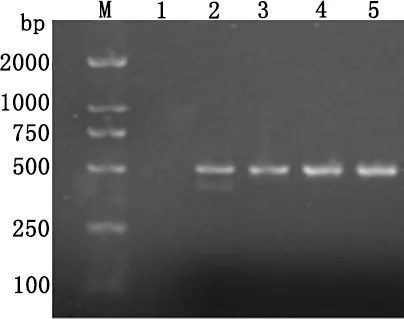
图2 E.coli中HPI irp2基因的PCR检测Fig 2 PCR amplification of HPI irp2 gene of E.coli
2.2 打靶DNA片段的制备和纯化 用于替换irp2基因的打靶片段两端的同源序列分别为45 bp和50 bp,片段中间部分为两侧带有FRT位点的卡那霉素(Kan)抗性基因,有利于重组子的筛选。纯化回收的产物在高保真酶PCR扩增后经2%的琼脂糖凝胶电泳分析,与目的片段的大小一致,见图3(M为2000 bp的DNA Marker,1-3为同源重组DNA片段)。
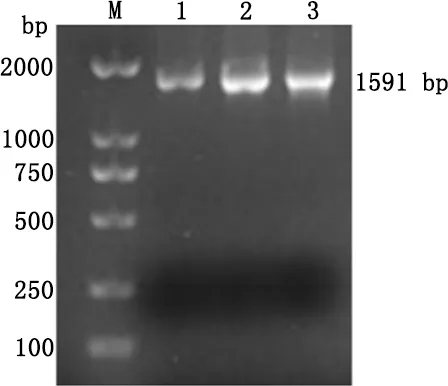
图3 融合PCR构建的同源重组DNA片段Fig 3 DNA homologous recombination fragment builded by fusiohn PCR
2.3 质粒pKD 46转化E.coli(E)后的提取验证 将pKD 46转化导入到E.coli(E)中,挑选单个菌落在Amp+ LB液体培养基中进行扩大培养,提取质粒后进行电泳验证,转化的鉴定结果见图4(M为2000 bp的DNA Marker,1-3为鉴定结果):
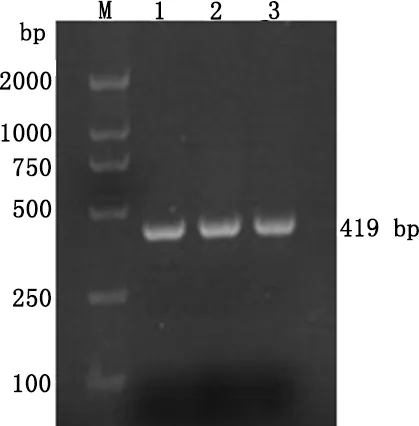
图4 质粒pKD 46转化的提取验证Fig 4 Extraction and Verification of Plasmid pKD 46
2.4 电转化后同源重组的鉴定 电转化后irp2基因被Kan抗性基因片段替换后,如图1所示,引物A1/A2、B1/B2和A1/B2分别用于扩增片段a、b、c。结果显示,有3个片段的长度和预计的一样(图5),说明Kmr基因已经取代irp2基因,缺陷株E.coli(E)/Kmr+/Δirp2构建成功,正确重组率为10%。
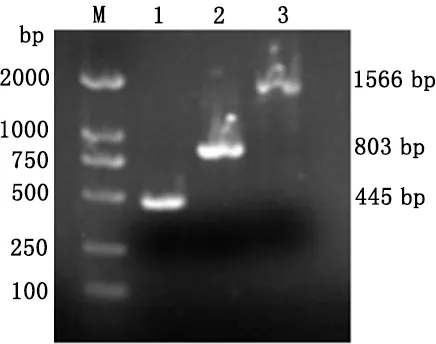
图5 irp2基因敲除的PCR验证Fig 5 Verification of irp2 knockout by PCR
2.5 质粒pCP 20转化E.coli(E)/Kmr+/Δirp2后的提取验证 将质粒pCP 20转化导入缺陷株E.coli(E)/Kmr+/Δirp2,涂布于含Kan+和Amp+双抗性LB平板上,30℃培养筛选阳性转化子,挑选单个菌落在Amp+ Kan+ LB液体培养基中进行扩增,提取质粒后进行电泳验证,转化的鉴定结果见图6(M为2000 bp的DNA Marker,1-3为目的基因)。
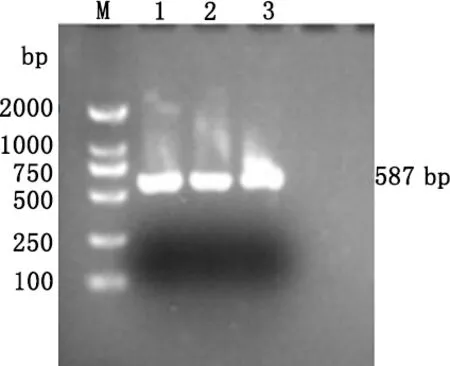
图6 质粒pCP 20转化的提取验证Fig 6 extraction and verification of plasmid pCP 20
2.6 卡那霉素抗性基因的消除鉴定 将成功转化pCP 20的缺陷株E.coli(E)/Kmr+/Δirp2接种于无抗性的LB液体培养基中,42 ℃下传3~5代,再用卡那霉素抗性进行检测,卡那霉素抗性在能够稳定遗传的菌株中已经消除,命名为E.coli(E)/Δirp2。用鉴定引物A1、B2进行验证PCR和测序验证,如图7(M:DNA分子质量标准;1-3:目的基因)。
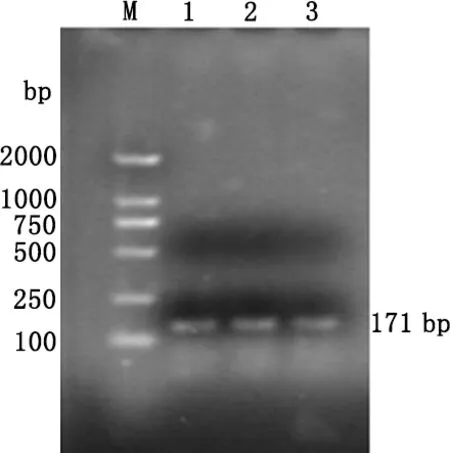
图7 卡那霉素抗性基因消除后PCR鉴定Fig 7 PCR analysis after kanamycin resistance gene eliminated
2.7 HPIirp2基因表达的蛋白的预测分析
2.7.1 HMWP2分子量及等电点的预测分析 使用ExPASy开发的在线工具Protparam分析HMWP2的理论分子量和等电点(pI)分别为:228826.60 Da,5.85。
2.7.2 HMWP2 二级结构的预测分析 登陆http://www.predictprotein.org/,对HMWP2二级结构进行预测,二级结构中α螺旋、β折叠和无规则卷曲所占的百分比分别为:43.5%、10.76%、46.19%,如图8。
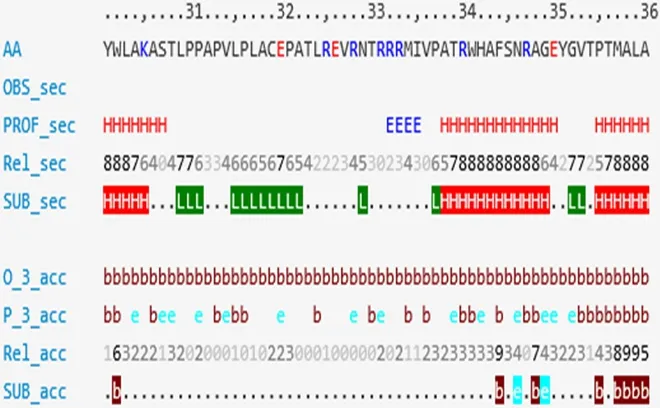
图8 HMWP2二级结构预测Fig 8 Prediction of HMWP2's Secondary Structure
2.7.3 HMWP2亲疏水性分析 使用ExPASy开发的在线工具ProtScale分析HMWP2的亲疏水性。结果显示总平均疏水指数为:-0.221,为疏水性蛋白,如图9。
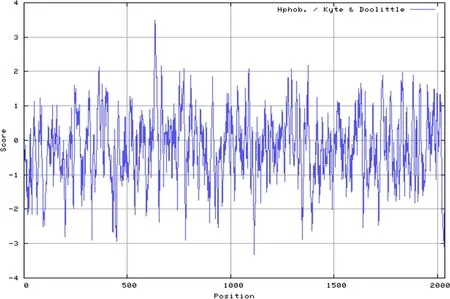
图9 HMWP2疏水性预测Fig 9 Prediction of HMWP2's Hydrophobicity
2.7.4 HMWP2跨膜区分析 使用ExPASy开发的在线工具TMpred分析HMWP2的可能的跨膜螺旋区,最优拓扑结构显示有9个跨膜螺旋,如图10。
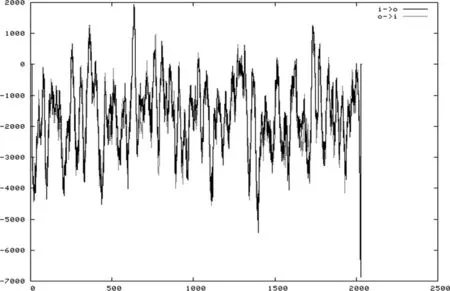
图10 HMWP2跨膜螺旋预测Fig 10 Prediction of HMWP2's Transmembrane Helices
2.7.5 HMWP2三级结构的预测分析 使用ExPASy开发的在线工具SWISS-MODEL,与已知结构的PDB数据库中的蛋白质序列进行比对,来预测HMWP2的三级结构,结果如图11。
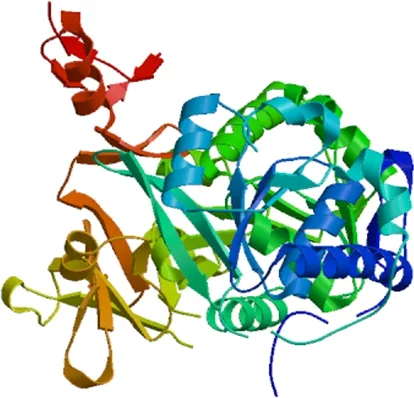
图11 HMWP2三级结构预测Fig 11 Prediction of HMWP2's Tertiary Structure
3 讨论与小结
1998年,Murphy[7]首次报道了将λ噬菌体的exo、bet、gam三个重组功能基因在质粒上进行表达,从而利用λ噬菌体的Red功能在E.coli染色体上进行基因替换。与RecA同源重组体系相比,由于Red重组发生在细胞内,E.coli的复制和修复系统保证了克隆的子代分子完全忠实于亲代分子[8]。Red同源重组系统是由λ噬菌体的分别编码Exo、Beta、Gam三种蛋白质的exo、bet、gam三个基因组成的。Exo蛋白是一种单亚基分子量为24 kD的核酸外切酶,它的活性形式是一种环状的三聚物分子,中间有一个一端能容纳双链DNA(dsDNA),另一端能容纳单链DNA(ssDNA)的中空通道[9]。Exo蛋白可以结合在dsDNA的末端,从双链DNA从5'端向3'端降解,从而产生3'端突出。Beta蛋白是一种单亚基分子量为25.8 kD的退火蛋白,它可以通过自发地形成环状结构而结合到由Exo降解产生的ssDNA的3'突出端,从而促进ssDNA末端与互补链之间的退火,同时Beta蛋白还能防止单链核酸酶降解外源线性DNA[10]。Gam蛋白是Exo和Beta的辅助蛋白,功能是防止外源DNA片段被降解掉[11]。
目前用的最多的Red重组质粒是Datsenko[12]构建的具有高效重组能力的的低拷贝质粒pKD 46。pKD 46在30℃培养时能够正常地复制,当温度高于37 ℃时质粒自动丢失,它含有温敏型的复制起点oriR101和受ParaB启动子调控的exo、bet、gam基因[13],这三个基因携带氨苄青霉素抗性基因作为筛选标记,在L-阿拉伯糖的诱导下表达。pKD 4在Red同源重组系统中作为抗性标记使用,它含有卡那霉素抗性基因,以及在抗性基因的两侧有FRT位点。pCP 20同时含有氨苄青霉素和氯霉素抗性,且含有温敏型的复制起点。pCP 20上还含有一个能产生与FRT位点结合的翻转酶重组酶(Flipase Recombination Enzyme,FLP)的基因,从而使FRT位点发生自身同源重组,消除一个FRT位点和抗性基因。FLP重组酶可以在42 ℃时诱导表达,同时质粒逐渐丢失[12,14]。
HPI是耶尔森菌的一个基因组,含有11个基因的功能核心区,携带的基因与铁载体—耶尔森杆菌素的合成、调节和转运有关。其中,irp2基因为HPI的标志基因,与摄铁功能相关,该基因表达的HMWP2蛋白对Ybt的合成具有重要作用,被认为是Ybt阳性表型表达所必需。预测结果表明,HMWP2蛋白在pH为5.85时溶解度最小,易于析出。其中α螺旋、β折叠和无规则卷曲所占的百分比分别为43.5%、10.76%和46.19%,HMWP2蛋白为疏水性跨膜蛋白。这些性质可能与该蛋白的摄铁转运相关。irp2作为铁调节基因,仅在致病性耶尔森菌中表达,编码的分子量为228 KD的HMWP2蛋白可参与Ybt的合成,诱导鼠疫菌素受体和Ybt表达,与耶尔森菌的铁摄取能力有关。试验利用Red同源重组的方法对irp2基因进行了敲除,并通过测序,对irp2基因表达的HMWP2蛋白进行了结构及功能预测,不仅为研究irp2基因在PEC致病机制中的作用及Red重组系统研究HPI的其他基因奠定了基础,也为制备肠道感染病疫苗和粘膜免疫佐剂等提供了更多的理论依据。
[1] Russell C B,Thaler D S,Dahlquist F W. Chromosomal transformation of Escherichia coli recD strains with linearized plasmids[J]. Journal of Bacteriology,1989,171(5): 2609-2613.
[2] Hamilton C M,Aldea M,Washburn B K,etal. New method for generating deletions and gene replacements in Escherichia coli[J]. Journal of Bacteriology,1989,171(9): 4617-4622.
[3] Link A J,Phillips D,Church G M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: Application to open reading frame characterization[J]. Journal of Bacteriology,1997,179(20): 6228-6237.
[4] Rakin A, Schubert, Guilvout I,etal. Local hopping of IS3 elements into the A+T-rich part of the high-pathogenicity island in Yersinia enterocolitica 1B, O:8[J]. Fems Microbiol Lett,2000,182(2): 225-229.
[5] 吕沈聪,赵颖颖,钟卫鸿. RED同源重组在大肠杆菌基因敲除中的应用[J]. 化学与生物工程,2013,30(6):1-6.
Lv S C, Zhao Y Y, Zhong W H. Application of RED homologous recombination in gene knockout ofE.coli[J]. Chemistry & Bioengineering, 2013, 30(6): 1-6.
[6] 孙 旭,周长林,方宏清. Red同源重组在大肠杆菌基因组修饰中的应用[J]. 生物技术通讯,2011,(6):873-878.
Sun X, Zhou C L, Fang H Q. Application of RED mediated recombination inE.coligenome modification[J]. Letters in Biotechnology, 2011, (6): 873-878.
[7] Murphy K C. Use of bacteriophage lambda recombination functions to promote gene replacement in Escherichia coli[J]. J Bacteriol,1998,180(8): 2063-2071.
[8] Muyrers J P,Zhang Y,Stewart A F. Techniques:Recombinogenic engineering-new options for cloning and manipulating DNA[J]. Trends Biochem Sci,2001,26(5): 325-331.
[9] Kovall R,Matthews B W. Toroidal structure of lambda-exonuclease [J]. Science,1997,277 (5333): 1824~1827.
[10] Smith C E,Bell C E. Domain Structure of the Redβ Single-Strand Annealing Protein: the C-terminal Domain is Required for Fine-Tuning DNA-binding Properties, Interaction with the Exonuclease Partner, and Recombination in vivo[J]. J Mol Biol,2016,428(3): 561-578.
[11] Yu D, Ellis HM, Lee E,etal. An efficitent recombination system for chomosome enjineerinig inEscherichiacoli[J]. Proc Natl Acad Sci USA,2000,97(11): 5978.
[12] Datsenko K A,Wanner B L. One-step inactivation of chromosomal genes inEscherichiacoliK-12 using PCR products[J]. Proc Natl Acad Sci USA,2000,97(12): 6640-6645.
[13] 张穗生,郭 媛,韦廷宗,等. Red重组系统介导下大肠杆菌改造体系的优化[J]. 广西科学,2010,17(2):160-163.
Zhang H S, Guo Y, Wei Y Z,etal. Optimizing the conditions for Redmediated recombinogenic engineering ofE.coli[J]. Guangxi Sciences, 2010, 17(2): 160-163.
[14] Yuan L Z, Rouviere P E, Larossa R A,etal. Chromosomal promoter replacement of the isoprenoid pathway for enhancing carotenoid production inE.coli[J]. Metab Eng,2006,8(1): 79-90.

