基于质谱技术的婴幼儿肠道菌群多样性鉴别方法研究
高丽娟, 高建萍, 马 凯, 李赛娜, 张贵锋
(1. 北京市理化分析测试中心 北京市食品安全分析测试工程技术研究中心, 北京 100089; 2. 中国科学院过程工程研究所, 北京 100190)
微生物检测在应用领域十分广泛,传统技术对于微生物的检测和鉴定技术多基于微生物的形态学、细胞生理生化、脂肪酸组成以及核酸检测等方法,这些方法往往耗时长且灵敏度低,特异性差。近年来发展起来的基于质谱的检测方法具有快速和灵敏度高等优点,采用质谱检测进行鉴定微生物主要基于质谱图中是否存在特异性的生物标记物的分子离子,不同微生物具有不同的质谱图,即微生物指纹图谱[1-3]。微生物的质谱鉴定实验流程包括样本的采集和制备,整个过程在数分钟内完成,并可以实现高通量分析。质谱分析作为一种通用技术,可以检测到所有类型的病原体,包括植物细菌、真菌及其孢子和寄生原生动物等[4-6]。
肠道微生物对人体的健康至关重要,如对人体免疫力、新陈代谢、炎症性疾病甚至心情等都有不同程度的影响。肠道微生物的丰度、多样性以及构成变化对人体健康状况的影响近年来引起广泛关注[7-8]。婴幼儿发育的过程中,肠道微生物菌群也是在逐渐成熟的,受母体、分娩、喂养、医疗、环境等因素影响,研究肠道菌群的动态变化是近年来肠道微生物研究领域的热点。因此,对于成长过程中婴幼儿肠道内动态变化的微生物菌落,则采用单一模式的质谱分析技术进行微生物菌落分析存在一定的局限性[9-12]。
本研究拟采用基质辅助激光解析电离飞行时间质谱(MALDI-TOF)和液相色谱质谱联用技术(HPLC-MS)对不同年龄的婴幼儿肠道微生物组成进行分析。实验首先收集了100个不同年龄的婴幼儿粪便进行检测,按照年龄分成了0~1岁、1~2岁和2~3岁3个年龄段,比较了基于MALDI-TOF MS和HPLC-MS分析结果的差异,为进一步研究婴幼儿发育过程中肠道菌群的动态变化提供参考。
1 材料与方法
1.1 材料与试剂
乙腈购自ThermoFisher公司,α-氰基-4-羟基肉桂酸(CHCA)购自Bruker公司,其他试剂为市售分析纯。实验样本为70个0~1岁、1~2岁和2~3岁3个年龄段婴幼儿的粪便,样本来自于北京儿童医院。
1.2 分析仪器
高效液相色谱仪为美国Agilent 公司的HPLC1100,PDA 检测器为美国热电公司的UV6000 检测器,离子阱质谱为美国热电公司的LCQ DecaXP质谱,MALDI-TOF MS 为德国布鲁克公司的AutoFlex III质谱。
1.3 样本处理方法
1)取婴幼儿粪便1 g,加入5 mL生理盐水,混合均匀后实验80目滤网进行过滤,收集透过液,取1 mL,在12 000 r/min条件下离心10 min,弃去上清液,沉淀物进行质谱分析。
2)MALDI-TOF MS分析样本点靶之前进行涡旋混合,取1 μL样本点靶,自然风干后取2 μL CHCA基质溶液点靶,自然风干,质谱分析前仪器的质量数利用外标法,使用标准蛋白混合物进行校正。
3)HPLC-MS分析中,将粪便样本细胞破碎,静置自然沉降,取100 μL上清液调pH至8.0,与50 μL胰蛋白酶溶液(1 μg/μL,0.05 mol/L NH4HCO3,pH 8.0),混合均匀后在37℃酶解处理24 h,加5 μL 4 mol/L DTT还原二硫键,离心(10 000 r/min,10 min),酶解产物采用HPLC-MS分析。
1.4 分析方法
1)MALDI-TOF质谱分析方法。脉冲激光(337 nm)离子解析电离源;离子加速电压20 kV;平均每次测定的激光脉冲次数为100次/s;质谱信号为多次累加扫描;质谱扫描范围m/z1600~24 000;采用正离子线性模式。选择α-氰基-4-羟基肉桂酸(CHCA)作为基质,用V(0.1% TFA的超纯水)∶V(CH3CN)=1∶1的溶液将CHCA配成浓度为5 g/L的基质溶液,基质溶液现用现配。
2)HPLC-MS分析方法。色谱条件,C18柱 (75 μm ID×15 cm, 5 μm, 120 Å);流动相A为水(含0.1% 甲酸),流动相B为乙腈(含0.1%甲酸);梯度:0~3 min:3%~6%B,3~50 min:6%~25%B,50~58 min:25%~35%B,58~63 min:35%~80%B,63~73 min:80%B;流速300 nL/min;波长280 nm。进样量50 μL。质谱条件:ESI电喷雾离子源,喷雾电压3.5 kV,离子导入电压(Skimmer 电压) 20 V,壳气流速10 arb,辅助气流速2 arb,离子传输毛细管温度300℃。离子监测模式参数中共设立2个扫描段,第一段监测为一级质谱全扫描,扫描范围为m/z=350~1550,采用正离子监测模式,分辨率设为12 000;第二段监测采用数据依赖型二级质谱的扫描(Data dependent MS/MS),用于扫描在第一段中已确定质量数离子的二级质谱,离子带电荷数默认值为2个,二级质谱碰撞诱导裂分中的能量值均设置为35%。质谱数据用Mascot 软件进行检索,数据库为从Swiss-Prot中下载的包括粪便中已发现的全部蛋白质的氨基酸序列。
2 结果与讨论
2.1 MALDI-TOF MS质谱分析
为了完整测定粪便样本的分子量信息,实验将粪便样本涡旋混合之后直接点靶,进行MALDI-TOF分析。取1 μL涡旋混合之后的样本点靶,自然风干后取2 μL CHCA基质溶液点靶,自然风干,进行MALDI-TOF分析。图1~3分别为0~1岁、1~2岁、2~3岁部分样本分子量分布图。
实验对不同年龄段的样本的分子质量分布及检测出峰的个数进行了汇总,结果表明0~1岁婴幼儿粪便中检出峰的个数主要分布在4~7个,其中两个样本出峰最多是11个,平均每个样本出峰5.5个;样本的分子质量分布主要为3000~14 000 u,分子质量最低为2378 u,最高为23 541 u。1~2岁婴幼儿粪便中检出峰的个数主要分布在6~8个,样本出峰最多达13个,平均每个样本出峰6.1个;样本的分子质量分布主要为3000~14 000 u,分子质量最低为1897 u的峰,最高为15 073 u。2~3岁婴幼儿粪便中检出峰的个数主要分布在6~10个,样本出峰最多是15个,平均每个样本出峰6.5个;样本的分子质量分布主要为3000~14 000 u,分子质量最低1182 u的峰,最高为15 022 u。综上所述,2~3岁年龄段的婴幼儿检出峰个数最多,0~1岁年龄段的婴幼儿检测出的分子质量分布范围最广。

图1 0~1岁婴幼儿粪便样本MALDI-TOFMS分析图谱
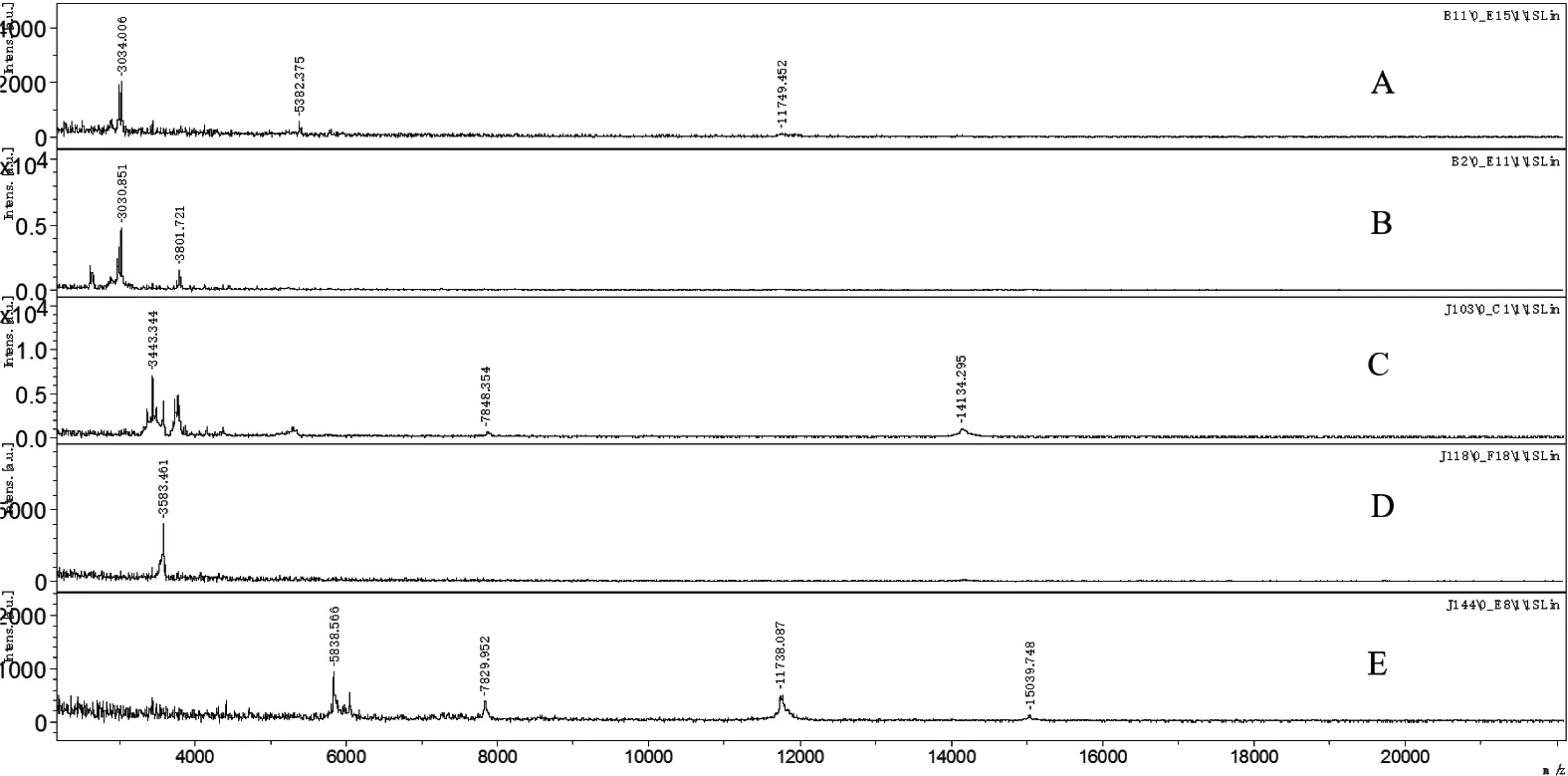
图2 1~2岁婴幼儿粪便样本MALDI-TOFMS分析图谱

图3 2~3岁岁婴幼儿粪便样本MALDI-TOFMS分析图谱
实验对质谱数据进行PCA聚类分析,图4为3组不同年龄段的婴幼儿粪便TOF数据PCA分析图。结果表明,0~1岁婴幼儿粪便样本分布相对集中,1~2岁婴幼儿粪便样本分布比0~1岁的分布范围宽,2~3岁婴幼儿粪便样本的分布最广。
文献报道MALDI-TOF MS技术对菌落进行菌种鉴定过程中,对常见细菌和酵母菌等高丰度微生物的鉴定率较高。由于婴幼儿粪便中不同的微生物丰度存在一定差异,直接进行不同样本分析质谱图中特征离子种类波动范围较大。0~1岁婴幼儿粪便样本中可以确定的微生物主要包括大肠杆菌、酵母菌、白色念珠菌等;1~2岁婴幼儿粪便中主要有红色毛麟菌、大肠杆菌、白色念珠菌、酵母菌等;2~3岁婴幼儿粪便中主要有双歧杆菌、白色念珠菌和大肠杆菌等。

图4 3组不同年龄段的婴幼儿粪便TOF数据PCA分析图

图5 3个不同年龄阶段样本的总离子流图
A: 0~1岁; B:1~2岁; C: 2~3岁

图6 B61样本中m/z 872.41的二级质谱图
2.2 液质联用法识别婴幼儿粪便中微生物
实验首先将婴幼儿粪便样本进行细胞破碎,静置0.5 h,使粪便中的固体颗粒物质自然沉降,取上清液调节pH 值,酶解,HPLC-MS分析。图5为酶解样本的HPLC-MS分析的总离子流图。样本总离子流图中检测出离子m/z872.41的提取离子流图(图6),表明B61样本中存在m/z872.41的离子,其经色谱柱分离后保留时间为11.02 min。
利用Mascot软件搜索离子m/z872.41的pep_expect的值<0.01,表明数据可靠。m/z872.41的二级质谱图与多肽QDAGDAAPK理论二级质谱碎片离子匹配,该多肽序列与土霉菌中的局部序列一致,表明样本中存在土霉菌;实验使用相同的方法对不同样本进行分析,并确定了3个样本中的主要微生物组分,识别结果见表1。
2.3 分析方法比较
鉴于MALDI-TOF MS技术对菌落分析中存在的波动范围大和结果不稳定等问题,试验对样本中的微生物进行了培养,并进一步采用MALDI-TOF MS进行了分析。结果表明菌落种类没有明显的改变,但信号强度会大幅度提升,结果稳定性和重复性也会随着培养时间延长而增加,因此,MALDI-TOF MS对于高丰度的微生物鉴定具有可操作性,但可检测出的微生物数量较为有限。

表1 0~1岁、1~2岁、2~3岁3个年龄段粪便样本中微生物构成
—a,暂无正式中文对照名
HPLC-MS是基于微生物中不同蛋白质的含量变化进行检测的一种方法,其关键因素为不同微生物中以及同一微生物中不同蛋白质的含量。结果(表1)表明,基于HPLC-MS检测出的微生物种类远高于MALDI-TOF MS,其主要因素是基于液质的检测方法具有较高的灵敏度。尽管微生物的酶解产物中不同种类的蛋白质含量存在较大差异,但HPLC-MS是在色谱分离基础上进行的多肽检测,因此可以大幅度减少离子化过程中的质量歧视效应,并增加方法的灵敏度。对于多元混合的微生物检测,HPLC-MS明显可提升可检测的微生物数量,且方法具有较高的稳定性和重复性,因此该方法也适用于未经培养的微生物菌落。
[1]RANDAZZO A, SIMON M, Goffinet P, et al. Optimal turnaround time for direct identification of microorganisms by mass spectrometry in blood culture[J]. Journal of Microbiological Methods, 2016, 130: 1-5.
[3]GONZALEZ M D, WEBER C J, BURNHAMC D. Rapid identification of microorganisms from positive blood cultures by testing early growth on solid media using matrix-assisted laser desorption ionization time of flight mass spectrometry[J]. Diagnostic Microbiology and Infectious Disease, 2016, 85(2): 133-135.
[4]STOPKA S A, MANSOUR T R, SHRESTHA B, et al. Turnover rates in microorganisms by laser ablation electrospray ionization mass spectrometry and pulse-chase analysis[J]. Analytica Chimica Acta, 2016, 902: 1-7.
[6]RIEGEL P, MOUSSAOUI W, SANGLIER S F, et al. Mass spectrometry and microorganism identification[J]. Médecine et Maladies Infectieuses, 2007, 37(S1):S11-S13.
[7]SALADINO F, POSARELLI E, LUZ C, et al. Influence of probiotic microorganisms on aflatoxins B1 and B2 bioaccessibility evaluated with an simulated gastrointestinal digestion[J]. Journal of Food Composition and Analysis, 2017.
[8]TAKI Y, KENZAKA T, OHATA K, et al. Cause and responsible microorganisms of bacteremia after gastrointestinal surgery[J]. Journal of the American College of Surgeons, 2016, 223(4): e92.
[9]HAYASHI A, AOYAGI H, YOSHIMURA T, et al. Development of novel method for screening microorganisms using symbiotic association between insect and intestinal microorganisms[J]. Journal of Bioscience and Bioengineering, 2007, 103(4): 358-367.
[10]KARIM IKHLAS A E, LINDEN G J, ORR F T, et al. Antimicrobial activity of neuropeptides against a range of micro-organisms from skin, oral, respiratory and gastrointestinal tract sites[J]. Journal of Neuroimmunology, 2008, 200(1-2): 11-16.
[11]MORITA H, TOH H, OSHIMA K, et al. Complete genome sequence of Bifidobacterium breve JCM 1192Tisolated from infant feces[J]. Journal of Biotechnology, 2015, 210: 81-82.
[12]KONGNUM K, HONGPATTARAKERE T. Cholesterol-lowering mechanism of lactic acid bacteria andBifidobacteriumsp. isolated from breast milk and infant feces[J]. Journal of Biotechnology, 2014, 185(1):S78.
[13]MORITA H, TOH H, OSHIMA K, et al. Complete genome sequence ofBifidobacteriumpseudocatenulatumJCM 1200(T) isolated from infant feces[J]. Journal of Biotechnology, 2015, 210: 68-69.
[14]MORITA H, TOH H, OSHIMA K, et al. Complete genome sequence ofbifidobacteriumbifidumJCM 1255(T) isolated from feces of a breast-fed infant[J]. Journal of Biotechnology, 2015, 210: 66-67.
[15]DAVOODABADI A, SOLTAN DALLAL M M, FOROUSHANI A R, et al. Antibacterial activity ofLactobacillusspp. isolated from the feces of healthy infants against enteropathogenic bacteria[J]. Anaerobe, 2015, 34: 53-58.
[16]ALP G, ASLIM B. Relationship between the resistance to bile salts and low pH with exopolysaccharide (EPS) production ofBifidobacteriumspp. isolated from infants feces and breast milk[J]. Anaerobe, 2010, 16(2): 101-105.

